用于细胞三维培养的集成微柱阵列的微流控芯片设计与验证
2017-08-14刘军山张洋洋王忠邓嘉翌叶轩薛日
刘军山+张洋洋+王忠+邓嘉翌+叶轩+薛日野+葛丹+徐征


摘 要 设计并验证了一种用于细胞三维培养的集成微柱阵列的微流控芯片。芯片由一片聚二甲基硅氧烷(PDMS)沟道片和一片玻璃盖片组成, 在PDMS沟道片上集成了一个由两排微柱阵列围成的细胞培养室和两条用于输送培养基的侧沟道。微柱间距直接影响了芯片的使用性能, 是整个芯片设计的关键。基于数值模拟和实验验证, 本研究对微柱间距进行了优化设计。优化后的微流控芯片可以很好地实现细胞与细胞外基质模拟材料混合液的稳定注入、培养基中营养物质向培养室内的快速扩散和细胞代谢物的及时排出。在芯片上进行了神经干细胞的三维培养, 证明了芯片上构建的细胞体外微环境的稳定性。
关键词 微流控芯片; 细胞; 三维培养; 微柱; 聚二甲基硅氧烷
1 引 言
微流控芯片具有试剂消耗量少、易于精确控制液体流动和模拟构建细胞体外微环境等优点, 在细胞分析领域得到了广泛应用[1~3]。近年来, 基于微流控芯片的细胞分析研究主要包括:单细胞操控与分析[4]、细胞与细胞[5]及细胞与细胞外基质的相互作用[6]、干细胞诱导与分化[7]、药物筛选[8]和器官芯片[9]等, 而细胞培养是开展上述研究的基础。
微流控芯片上的细胞培养有二维和三维两种方式。二维培养操作简单, 但最近研究表明, 细胞在二维培养环境中无法真实体现其在体内的生物学特性和功能[10]。在微流控芯片上进行细胞三维培养的主要方法是将细胞与细胞外基质模拟材料(例如胶原蛋白)混合, 然后将混合液注入到细胞培养室中, 为细胞搭建一个三维培养空间, 通过微沟道与培养室连接, 为细胞持续提供培养基[5,11]。
芯片上的细胞培养室多数采用一种四周封闭、只有一个入口和一个出口的圆池形结构[12]。这种结构虽然制作简单, 但能够实现的功能有限, 而且在输送培养基时会产生较大剪切力, 对细胞造成伤害[13]。Toh等[14]提出了一种由两排微柱阵列围成的培养室结构, 这种结构既可实现多种复杂功能, 又避免了培养基直接从细胞表面流过, 可大幅减小细胞所受的剪切力, 此种培养室得到了越来越多应用[15]。然而, 有关此类型芯片的优化设计却鲜见报道。微柱间距直接影响芯片使用性能, 间距过大, 在注入细胞与细胞外基质模拟材料混合液时, 混合液会克服流动阻力流出培养室, 使得芯片无法正常工作; 间距过小, 既会增加芯片制作难度, 又会降低培养基中的营养物质和细胞代谢物的扩散速度, 影响细胞正常生长[16,17]。
本研究基于数值模拟和实验验证, 对微流控芯片上的微柱间距进行了优化设计, 并在优化后的芯片上进行了神经干细胞三维培养, 检测了构建的细胞体外微环境稳定性。
2 实验部分
2.1 微流控芯片设计
微流控芯片结构如图1所示, 由一片聚二甲基硅氧烷(Polydimethylsiloxane, PDMS)沟道片(40 mm×20 mm×3 mm)和一片玻璃盖片(50 mm×24 mm×0.15 mm)组成。在沟道片上, 集成了一个由两排微柱阵列围成的细胞培养室、两条用于输送培养基的侧沟道和6个储液池。微柱阵列长20 mm, 两排微柱之间距离为1 mm, 微柱直径为100 μm、高150 μm。侧沟道宽1 mm、深150 μm, 储液池直径为3 mm。
本研究组曾利用鼠尾I型胶原构建了神经干细胞的三维培养微环境[18], 因此本研究所用细胞外基质模拟材料均为鼠尾I型胶原。芯片操作流程如下:首先利用移液器将细胞与胶原混合液从细胞入口注入培养室, 接着将芯片放入细胞培养箱中使胶原溶液聚合形成水凝胶, 然后采用微量注射泵将培养基从两个培养基入口注入侧沟道, 培养基中的营养物质通过扩散方式经过微柱阵列进入培养室, 同时细胞代谢物也通过扩散方式排出培养室。
2.2 微流控芯片数值模拟
基于计算流体动力学, 对不同微柱间距情况下胶原溶液的注入过程和培养基中营养物质的扩散过程进行了数值模拟。
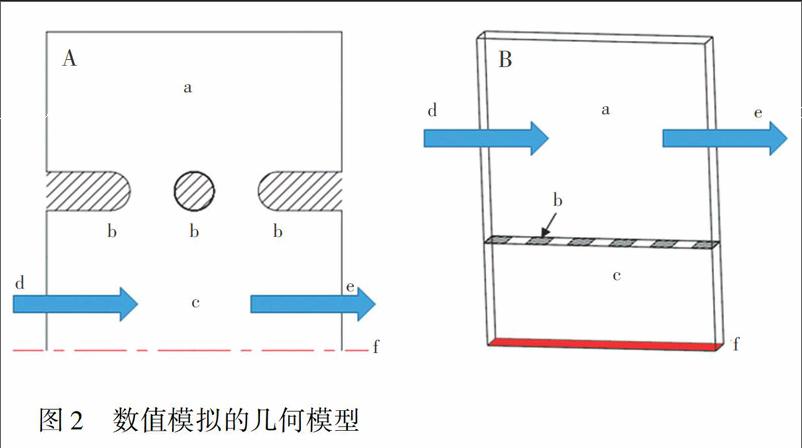
首先, 基于COMSOL Multiphysics的两相流水平集模型, 模拟胶原溶液注入过程。由于侧沟道和培养室沿着芯片水平中心线对称分布, 而且微柱等间距排列, 因此为减少仿真工作量, 建立了一个简化的轴对称二维几何模型(图2A)。
影响细胞与胶原混合液注入过程的主要参数包括:混合液粘度和表面张力系数、注入压力、微沟道表面接触角、微柱间距等。胶原溶液浓度为1 mg/mL, 利用流变仪测得的粘度为0.032 Pa·s, 利用接触角测量仪测得的表面张力系数为44 mN/m。混合液注入压力设定为1 kPa, 由于微沟道出口与外界大气相通, 因此出口压力设定为0。为了提高芯片键合强度和微沟道表面亲水性, 在芯片键合前, 利用等离子体对PDMS沟道片表面进行改性处理。测得胶原溶液在改性后的PDMS表面上接触角为30°, 因此设定微沟道表面接触角为30°。微柱间距共设计了4种, 分别为30、50、80和100 μm。
其次, 基于ANSYS Fluent的組分输运模型, 模拟营养物质扩散过程。同样, 建立一个简化的轴对称三维几何模型(图2B)。影响营养物质扩散过程的主要参数包括:营养物质的浓度和扩散系数、培养基流速、培养室内水凝胶的孔隙率和渗透率、微柱间距等。葡萄糖是培养基中的主要营养物质, 在进行神经干细胞培养时, 葡萄糖浓度为50.83 mmol/L, 远高于其它营养物质的浓度, 因此选用50.83 mmol/L葡萄糖溶液作为模拟培养基。在37℃(细胞培养时的温度)时, 葡萄糖在培养基中扩散系数为9.6×1010 m2/s [19]。微量注射泵的流量设定为1 μL/min, 换成培养基的流速为5.56×105 m/s。 利用排液法, 测得水凝胶孔隙率为0.99, 渗透率设定为2×109 cm2[20]。设计了3种微柱间距, 分别为5、50和100 μm。
2.3 微流控芯片制作
PDMS具有良好的生物兼容性、透光性和透气性, 被广泛用于制作各种细胞分析微流控芯片[7,9]。采用浇注成型的方法制作PDMS沟道片 [16], 然后利用等离子体清洗机对PDMS沟道片和玻璃表面进行氧等离子体處理,然后对准键合在一起。
2.4 仪器与试剂
K1050X等离子体清洗机(英国Quorum公司); STM6工具显微镜(日本Olympus公司); IX71倒置荧光显微镜(日本Olympus公司); PHD 22/2000微量注射泵(美国Harvard公司); Sunrise酶标仪(奥地利Tecan公司); DSA100接触角测量仪(德国Krüss公司); AR2000ex流变仪(美国TA公司)。
鼠尾I型胶原(生友生物技术有限公司); 荧光素钠(天津市大茂化学试剂厂); 葡萄糖和乳酸检测试剂盒(南京建成生物工程研究); 碘化丙啶(Propidium iodide, PI, Sigma公司); 钙黄绿素AM(CalceinAM, Sigma公司); PDMS(Dow Corning公司)。
3 结果与讨论
3.1 数值模拟
在不同微柱间距情况下, 胶原溶液的流动过程如图3所示。胶原溶液的注入过程是压力、毛细力和表面张力共同作用的结果[21]。在培养室内, 压力和毛细力起主要作用, 胶原溶液流动迅速, 仅用1 ms便充满了培养室。然而, 在微柱之间, 胶原溶液受到粘性力和表面张力的阻滞作用, 流速明显衰减。当微柱间距为30和50 μm时, 即使经过1000 ms胶原溶液仍停留在微柱之间, 没能进入侧沟道。随着微柱间距增大, 这种阻滞作用逐渐减弱, 胶原溶液有突破微柱束缚进入侧沟道的趋势。当微柱间距为80 μm时, 经过1000 ms胶原空气界面已明显越过圆柱中心线。当间距继续增大到100 μm时, 胶原溶液克服了阻滞作用, 在200 ms时胶原空气界面进入侧沟道, 在1000 ms时溶液充满侧沟道。
图4显示了微柱间距为50 μm时, 葡萄糖浓度在芯片内随时间的变化情况。葡萄糖溶液在侧沟道中流动迅速, 仅用18 s便到达了侧沟道出口。同时, 葡萄糖会经过微柱阵列向培养室内扩散, 在220 s时扩散过程达到平衡, 浓度在芯片内达到了均匀分布。当微柱间距为5和100 μm时, 葡萄糖浓度达到均匀分布所用的时间分别为237和200 s, 二者相差约40 s。因此, 微柱间距会影响营养物质向培养室内的扩散速度。
结合上述两方面数值模拟结果, 在综合考虑芯片加工难度和使用性能的情况下, 最终选择50 μm作为微柱间距的最优值。之后如无特殊说明, 芯片上微柱间距均为50 μm。
3.2 实验验证
在优化后的微流控芯片上对胶原溶液注入过程进行实验验证。采用0.01 mol/L磷酸盐缓冲液(PBS, pH 7.4)将5 mg/mL胶原溶液稀释到1 mg/mL, 用0.1 mol/L NaOH溶液将pH值调节为中性。然后将20 μL胶原溶液从细胞入口注入培养室。胶原溶液5 s便充满了培养室, 但一直到30 min后仍未突破微柱束缚进入侧沟道, 在微柱之间形成了一个稳定的胶原空气界面(图5)。
对营养物质扩散过程进行实验验证。荧光素钠扩散系数为4.3×1010 m2/s[22], 与数值模拟时用到的葡萄糖扩散系数相近, 为了在显微镜下清楚观测到扩散过程, 采用20 μmol/L荧光素钠溶液代替葡萄糖溶液。利用微量注射泵将荧光素钠溶液从培养基入口注入侧沟道, 流速为0.5 μL/min, 每隔500 ms拍摄一次照片。荧光素钠的流动与扩散过程与葡萄糖溶液的数值模拟结果类似, 仅用5 s便充满了侧沟道, 同时经过微柱阵列向培养室内不断扩散, 在160 s时扩散过程达到平衡(图6)。
上述结果表明, 利用数值模拟优化出的微柱间距不仅可以很好地实现细胞与细胞外基质模拟材料混合液的稳定注入, 有效拦截了混合液向侧沟道中的泄漏, 而且保证了营养物质可以快速扩散到培养室内, 满足了芯片性能要求。
3.3 芯片上的微环境稳定性测试
在细胞培养过程中, 体外微环境的稳定性至关重要, 除了要保持营养物质含量的稳定, 细胞代谢产物也应及时排出。例如乳酸是葡萄糖主要代谢副产物, 乳酸的积累会改变微环境pH值, 对细胞有毒害作用, 因此希望能够维持在较低浓度[17]。
神经干细胞具有自我更新、多向分化等能力, 已成为神经组织工程等领域的研究热点[23]。本研究通过神经干细胞的三维培养, 测试优化设计的微流控芯片所构建的微环境稳定性。
原代神经干细胞取自孕龄为14 天的昆明种小鼠胎脑的海马组织, 待培养至第三代时与胶原溶液混合, 形成细胞密度为1×106 cells/mL的混合液[18]。共制作了3片微流控芯片, 用于培养神经干细胞。同时, 为了对比分析, 在96孔板的3个孔中静态培养神经干细胞, 每个孔中加入50 μL细胞与胶原混合液, 待胶原聚合后加入100 μL培养基, 每24 h更换50 μL培养基。
每隔12 h从芯片废液池和96孔板中取出10 μL培养基, 对其中的葡萄糖和乳酸浓度进行检测(图7)。如图7A所示, 葡萄糖初始浓度为50.83 mmol/L, 由于细胞消耗, 芯片上的葡萄糖浓度会略有下降, 但如3.2节所述, 葡萄糖可以快速扩散到培养室内, 所以浓度仍然可以保持很高, 一直稳定在48~50 mmol/L。相反, 由于96孔板是静态培养, 在每个换液周期内营养物质得不到补充, 因此葡萄糖浓度持续下降, 最低降至41 mmol/L。从图7B可见, 由于芯片上的细胞代谢物可以通过扩散方式快速排出培养室, 因此培养基中的乳酸浓度很低, 在0.6~0.8 mmol/L之间。而在96孔板中, 由于乳酸在每个换液周期内持续积累, 所以浓度一直很高, 即使在换液后也无法有效降低, 始终保持在2.4 mmol/L以上, 最高达到4.3 mmol/L。
从葡萄糖和乳酸浓度的长期检测结果可知, 与96孔板中静态培养相比, 由本研究优化设计的微流控芯片构建的微环境具有更好的稳定性, 可用于细胞长期培养。
培养到第8天, 对神经干细胞进行在线死活荧光染色(图8)。与96孔板相比, 芯片上的干细胞突起更为明显, 突触间相互连接, 向三维空间纵深。根据随机选取的10张不同區域下的照片, 对死活细胞进行了计数, 芯片上和96孔板中的细胞存活率分别约为90%和82%。
4 结 论
利用数值模拟和实验验证相结合的方法, 对集成微柱阵列的微流控芯片进行了优化设计。利用优化后的微流控芯片, 进行了神经干细胞的三维培养。对葡萄糖和乳酸浓度进行了长期连续检测, 结果表明, 由本研究优化设计的微流控芯片所构建的细胞体外微环境具有良好的稳定性, 适合于细胞长期三维培养, 未来可以用于各种细胞分析研究。
References
1 ZHUANG QiChen, NING RuiZhi, MA Yuan, LIN JinMing. Chinese J. Anal. Chem., 2016, 44(4): 522-532
庄琪琛, 宁芮之, 麻 远, 林金明. 分析化学, 2016, 44(4): 522-532
2 Halldorsson S, Lucumi E, GomezSjoberg R, Fleming R M. Biosens. Bioelectron., 2015, 63: 218-231
3 Mazutis L, Gilbert J, Ung W L, Weitz D A, Griffiths A D, Heyman J A. Nat. Protoc., 2013, 8(5): 870-891
4 HUO DanQun, FANG Kejing, HOU ChangJun, YANG Mei, FA HuanBao, HUANG ChengHong, Luo XiaoGang. Chem. J. Chinese Universities,2013, 34(6): 1327-1332
霍丹群, 方可敬, 侯长军, 杨 眉, 法焕宝, 黄承洪, 罗小刚. 高等学校化学学报, 2013 ,34(6):13271332
5 WANG WeiXin, LIU WeiPing, WU Bin, LIANG GuangTie, LIU DaYu. Chinese J. Anal. Chem., 2015, 43(5): 637-642
王伟鑫, 刘未平, 吴 斌, 梁广铁, 刘大渔. 分析化学, 2015, 43(5): 637-642
6 Han S, Yang K, Shin Y, Lee J S, Kamm R D, Chung S, Cho S W. Lab Chip, 2012, 12(13): 2305-2308
7 Yang K, Park H J, Han S, Lee J, Ko E, Kim J, Lee J S, Yu J H, Song K Y, Cheong E, Cho S R, Chung S, Cho S W. Biomaterials, 2015, 63: 177-188
8 Weltin A, Slotwinski K, Kieninger J, Moser I, Jobst G, Wego M, Ehret R, Urban G A. Lab Chip, 2014, 14(1): 138-146
9 Huh D, Matthews B D, Mammoto A, MontoyaZavala M, Hsin H Y, Ingber D E. Science, 2010, 328(5986): 1662-1668
10 Pal R, Mamidi M K, Das A K, Bhonde R. J. Biosci. Bioeng., 2013, 115(2): 200-206
11 ZHANG Qiong, ZHOU XiaoMian, YAN Wei, LIANG GuangTie, ZHANG QiChao, LIU DaYu. Chinese J. Anal. Chem., 2012, 40(7): 996-1001
张 琼, 周小棉, 严 伟, 梁广铁, 张其超, 刘大渔. 分析化学, 2012, 40(7): 996-1001
12 Lei K F, Wu M H, Hsu C W, Chen Y D. Biosens. Bioelectron., 2014, 51: 16-21
13 Leclerc E, David B, Griscom L, Lepioufle B, Fujii T, Layrolle P, Legallaisa C. Biomaterials, 2006, 27(4): 586-595
14 Toh Y C, Zhang C, Zhang J, Khong Y M, Chang S, Samper V D, van Noort D, Hutmacher D W, Yu H. Lab Chip, 2007, 7(3): 302-309
15 Yang K, Park H J, Han S, Lee J, Ko E, Kim J, Cho S R. Biomaterials, 2015, 63: 177-188
16 LIU JunShan, XIAO QingLong, GE Dan, ZHANG YangYang, ZHANG WenZhu, XU Zheng, LIU Chong, WANG LiDing. Chinese J. Anal. Chem., 2015, 43(7): 977-982
刘军山, 肖庆龙, 葛 丹, 张洋洋, 张文珠, 徐 征, 刘 冲, 王立鼎. 分析化学, 2015, 43(7): 977-982
17 Ges I A, Baudenbacher F. Biosens. Bioelectron., 2010, 26(2): 828-833
18 Ge D, Song K D, Guan S, Qi Y L, Guan B, Li W F, Liu J S, Ma X H, Liu T Q, Cui Z F. Appl. Biochem. Biotechnol., 2013, 170(2): 406-419
19 Suhaimi H, Wang S, Thornton T, Das D B. Chem. Eng. Sci., 2015, 126: 244-256
20 Knapp D M, Barocas V H, Moon A G, Yoo K, Petzold L R, Tranquillo R T. J. Rheol., 1997, 41(5): 971-993
21 Huang C P, Lu J, Seon H, Lee A P, Flanagan L A, Kim H Y, Putnam A J, Jeon N L. Lab Chip, 2009, 9(12): 1740-1748
22 Roy A, Banerjee P, Dutta R, Kundu S, Sarkar N. Langmuir, 2016, 32(42): 10946-10956
23 Karimi M, Bahrami S, Mirshekari H, Basri S M M, Nik A B, Aref A R, Akbari M, Hamblin M R. Lab Chip, 2016, 16(14): 2551-2571
