结、直肠肿瘤肠道菌群特异性的临床研究
2017-08-12尹小平黎尉浩朱栋良严海冬王芳元
尹小平 黎尉浩 朱栋良 严海冬 王芳元
·论著·
结、直肠肿瘤肠道菌群特异性的临床研究
尹小平 黎尉浩 朱栋良 严海冬 王芳元
目的 研究肠道菌群变化与结、直肠肿瘤之间的关系。方法 病理检查证实的结、直肠肿瘤患者35例为研究组,同期结肠镜检查排除结、直肠肿瘤的患者35例为对照组。收集两组患者的粪便标本,采用扩增核糖体DNA限制性分析(ARDRA)技术对样本内细菌进行检测,比较两组菌种的变化及规律。结果 两组70例样本共检出389 010条高质量序列,研究组和对照组Chao1指数分别为1.01×103和0.85×103,Shannon指数分别为0.11×102和0.14×102,两组菌群多样性和均匀性比较差异无统计学意义(P>0.05)。而在菌种/属水平,有15种细菌丰度值比较差异有统计学意义(P<0.05)。结论 结直肠肿瘤患者肠道某些菌种与正常人存在差异,研究其变化规律有可能实现对结、直肠肿瘤进行早期的预防、早期诊断,并为微生态制剂作为结、直肠肿瘤的治疗方式提供依据。
肠道菌群; 结直肠肿瘤
流行病学研究显示,结、直肠肿瘤发病有一定的家族聚集现象,20.0%的结、直肠肿瘤发病中遗传因素起重要作用,但>70.0%的发病与环境因素有关[1]。各种肿瘤相关基因,如原癌基因存在但不被活化,在环境等外因作用下,经过表观遗传修饰变化引起该基因活化。人体肠道菌群是结、直肠肿瘤的主要环境因素,宿主肠道菌群变化与结、直肠肿瘤的关系逐渐取代基因成为结直肠肿瘤研究的重点[2]。我们应用扩增核糖体DNA限制性分析(ARDRA)技术,对结、直肠肿瘤患者和正常群体的肠道菌群进行比较,寻求两者之间的差异性和潜在的规律性。
对象与方法
一、对象
实验组35例,为2015年1月以来结肠镜下取材行病理检查证实为结、直肠肿瘤患者,男16例,女19例,平均年龄53.4岁;其中升结肠8例,横结肠6例,降结肠8例,乙状结肠5例,直肠8例。对照组35例,为同期结肠镜检查排除结、直肠肿瘤的患者。两组患者年龄40~60岁,排除结、直肠肿瘤复发或经过放化疗患者;排除合并肠梗阻、肠穿孔、肠炎、糖尿病、严重全身性疾病;所有对象1个月内未使用抗生素,2个月内未使用非甾体抗炎药或益生菌类药物。
二、方法
1.行肠道准备前1天留取新鲜粪便样本1 g,分别悬于9 ml PBS磷酸盐缓冲液中,涡旋至样品充分混匀。4℃ 200 g离心5分钟,收集上清液,此过程重复3次。离心3分钟,收集菌体。最后用PBS磷酸盐缓冲液洗3次,无菌水洗1次,将菌体分装为每管1 ml。
2.样本细菌基因组DNA提取及纯化:每个样品取15 ml洗液收集于离心管中。25℃条件下4000 g离心5分钟收集含菌沉淀物。采用UNIQ-10柱式细菌基因组DNA抽提试剂盒从上述沉淀物中提取细菌基因组DNA,并使用琼脂糖凝胶电泳分析基因组质量。制备的细菌基因组DNA置于-70℃冰箱中保存备用。
3.16S rDNA克隆及纯化:使用通用引物F27(5′-agagtttgatcctggctcag-3′)和R1492(5′-ggttaccttgttacga- ctt-3′)对基因组DNA进行16S rRNA基因扩增。这对引物具有很强的通用性,可扩增出绝大多数细菌的16S rRNA基因片段(长度>1.4 kb)。50 μl PCR反应体系中包含如下成分:基因组DNA(0.1 Ug/μl)2 μl,2 mmol/L dNTPs 5 μl,10 PCR buffer 5 μl,引物(F27和R1492)各10 pmol,TaqPlus DNA聚合酶(Tap+Pfu,Sangon)l U。反应程序如下:94℃ 5分钟后,进入30个循环。最后72℃ 8分钟。琼脂糖凝胶电泳后,使用E.Z.N.ATME胶纯化试剂盒纯化PCR产物。使用TA克隆试剂盒将获得的PCR产物克隆至pGEM-T载体,构建16 rDNA文库,使用蓝一白筛选获得阳性克隆。

图1 部分样本DNA电泳图 图2 部分样本优势菌种PCR扩增16S rRNA基因电泳图
4.ARDRA分析及序列分析:随机选择白色重组子克隆再次进行PCR。本轮PCR的引物对采用载体上的T7(5′-taatacgactcactataggg-3′)和SP6(5′-catacgatttaggtgacactatag-3′)。50 μl PCR反应体系中包含如下成分:质粒DNA,10×PCRbuffer 5 μl,2 mmol/L dNTPs 5 μl,引物(T7和SP6)各10 pmol,TaqPlusDNA聚合酶(Tap+Pfu,Sangon)1U。反应程序如下:94℃ 5分钟,进入30个循环,最后72℃ 8分钟。将所得产物进行扩增性核糖体DNA分析。其中采用Hin6 1和Msp I(MBI)双酶切割,酶切后1.0%琼脂糖凝胶电泳检测,分析OTUs。按照允许2个不匹配的差异进行预聚类,以平均邻近法聚类(97%的相似度)获得运算的分类单位(operationaltaxonomic units,OTUs)数量。随后将优势OTUs进行测序。所得16S rRNA基因序列用BLAST程序在美国生物技术信息中心序列数据库基因序列进行比对。比对中遵循的基本原则是:如果2种细菌的16S rRNA基因序列存在97%以上的相似性,可以视为同一种细菌[3]。
5.数据分析:将每个分类单位内所含的序列标签数进行统计,丰富度指数(Chao1指数)和菌群多样性指数(Shannon指数)代表菌群相对丰度,计算公式如下:Schao1=So+F12/2(F2+1)-F1F2/2(F2+1)2,其中So为观测到的OTU数量,F1为只有一条系列的的OTU数量,F2为有二条系列的的OTU数量;Sshannon=∑i=1fi/nlnfi/n,fi为含有i条系列的的OTU数量,n为系列数。
三、统计学方法
应用SPSS 16.0软件对数据进行分析,采用方差分析和t检验判断细菌种系差异。P<0.05为差异有统计学意义。
结 果
部分样本基因组DNA电泳图见图1。两组70例样本共检出389 010条高质量序列,平均每份样本5557条。基于97%的OTUs相似度,平均每个样本得到98个OUTs,研究组和对照组Chao1指数分别为1.01×103和0.85×103,Shannon指数分别为0.11×102和0.14×102,差异均无统计学意义(P>0.05)。两组样本优势菌属均为拟杆菌属、乳酸菌杆菌属、C.leptum菌属、双歧杆菌属、C.coccoides菌属和肠杆菌属。部分样本优势菌电泳图见图2。
基于菌种/属水平,研究组和对照组粪便菌群有15种菌种差异有统计学意义(P<0.05)。研究组枸橼酸杆菌属、拟杆菌属、消化链球菌属、埃希杆菌属、链球菌属、紫单胞菌属、梭菌属、梭形杆菌属及肠球菌属与对照组比较明显升高,而普氏菌属、罗氏菌属、瘤胃球菌属、酸酐菌属、真杆菌属、变形杆菌属与对照组比较明显降低,见表1。
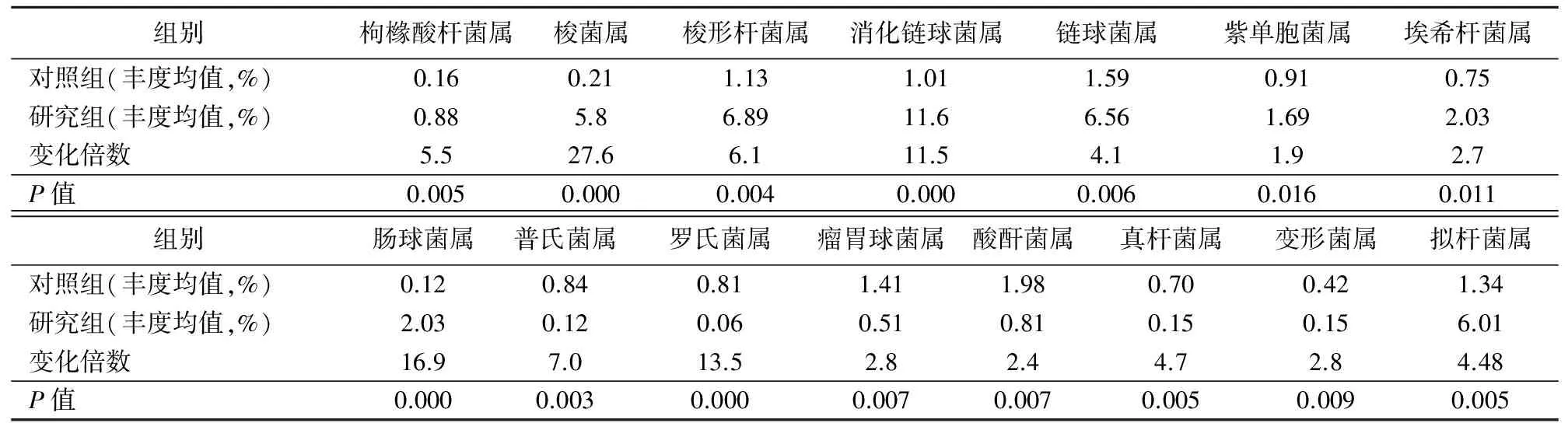
表1 研究组与对照组菌种/属丰度均值对照
讨 论
结、直肠肿瘤是一种常见消化道肿瘤,随着我国人民生活方式及饮食结构的改变其发病率呈现逐年上升的趋势[4-5]。结、直肠肿瘤的主要癌前病变已经确定,异常隐窝-腺瘤-结直肠肿瘤发生途径中的细胞及分子生物学机制已经得到广泛的研究。肠道内菌群与宿主存在着紧密的共生关系,肠道菌群可促进人体免疫系统的发育成熟,参与形成肠道黏膜屏障,防止病原菌定制,还可以解毒肠道致癌因子[6]。一旦正常的肠道菌群结构发生失衡,则会引起代谢改变,进而引发疾病。Chen等[7]研究发现,结、直肠肿瘤患者与健康人比较,总的肠道菌群结构相似,然而多样性降低。Scanlan等[8]的研究结果则显示,结、直肠肿瘤患者与健康人相比肠道菌群稳定性下降、多样性增加。
目前主流学术观点认为,肠道内微生物引起的局部及全身慢性炎症反应是导致结直肠肿瘤发生的主要机制[9]。革兰阴性细菌水平过高可以增加肠道通透性,导致内毒素血症、肥胖及炎性肠病相关的炎症反应[10]。炎症反应过程中产生的细胞因子及趋化因子,包括肿瘤坏死因子-α、白介素-6、白介素17、白介素-23等,可以促进血管形成、抑制抗肿瘤免疫反应,从而促进肿瘤的发生。
本研究发现,与对照组相比,研究组肠道菌群在菌群结构及菌群多样性上无明显差别。可见结、直肠肿瘤患者肠道菌群的改变并不是益生菌的绝对消失或有害菌的绝对增长,而是相对的改变。其中枸橼酸杆菌属、消化链球菌属、埃希杆菌属、链球菌属、拟杆菌属、紫单胞菌属、梭菌属、梭形杆菌属及肠球菌属较对照组明显升高,而普氏菌属、罗氏菌属、瘤胃球菌属、酸酐菌属、真杆菌属、变形杆菌属较对照组明显降低。多数研究认为,结、直肠肿瘤的发生与梭菌及拟杆菌的增加有关,脆弱类杆菌及产气荚膜菌产生的β-葡萄糖醛酸活性明显高于其他细菌,该酶可以将肠道内无毒性物质水解、游离、活化,使之转变为有毒性物质,增加肠道致癌物质的含量[11]。而产丁酸盐菌种对于肠道健康具有重要的意义,可以为肠黏膜细胞呼吸提供能量,保持肠道上皮细胞完整性,调整肠道免疫应答[12]。产丁酸盐菌种的降低而罗氏菌是产丁酸盐的代表菌种。
肠道菌群的结构失衡与结、直肠肿瘤的发生相关,特别是拟杆菌属及产丁酸盐菌种结构的变化可能是结、直肠肿瘤发生的警示。通过对相关肠道菌种的结构优化可能将有助于降低结、直肠肿瘤的发生。
[1] Sanford D,Markowitz,Monica M.Bertaguolli molecular basis of colorectal cancer[J].N Engl J Med,2009,361(25):2449-2460.
[2] Marchesi JR.Shifting from a gene-centric to metabolite·centric strategy to determine the core gut microbiome[J].Bioeng Bugs,2011.2(6):309-314.
[3] Staekebrandt E,Gcebel BM.Taxonomic note:a place for DNA.DNA reassoeiation and 16S rRNA sequence analysis in the present species definition in bacteriology[J].Int J Syst Bacteriol,1994,44:846-849.
[4] 郑树.结直肠肿瘤:基础研究与临床实践.第1版[M].北京:人民卫生出版社,2006,3-14.
[5] Yang L,Parkin DM,Li L,et al.Timetrends in cancer mortality in China:1987~1999[J].Int J Cancer,2003,106(5):771-783.
[6] Gallimore AM,Godkin A.Epithelial barriers,microbiota,and colorectal cancer [J].N Engl J Med,2013,368(3):282-284.
[7] Chen W,Liu F,Ling Z,et al.Human intestinal lumen and mucosa—associated microbiota in patients with colorectal cancer[J].PLoS ONE,2012,7(6):739-743.
[8] Scanlan PD,Shanahan F,Clune Y,et al.Culture—independent analysis of the gut microbiota in colorectal cancer and polyposis[J].Environmental Microbiology,2008,10(3):789-798.
[9] Konstantinov SR,Kuipers EJ,Peppelenbosch MP.Functional genomic analyses of the gut microbiota for CRC screening[J].Nat Rev Gastroenterol Hepatol,2013,10(12):741-745.
[10]Vajro P,Paolella G,Fasano A.Microbiota and gut liver axis:their influences on obesity and obesity-related liver disease[J].Pediatr Gastroenterol Nutr,2013,56(5):461-468.
[11]Nalini N,Manj u V,Menon VP.Effect of coconut cake on the bacterial enzyme Activity in 1,2-dimethyl hydrazine induced colon cancer[J].Clin Chim Acta,2004,342(1-2):203-210.
[12]罗明,李建明,陈海生,等.大肠癌虚证实证与CD44v6、PCNA表达的相关性研究[J].中国中西医结合外科杂志,2012,18(3):234-237.
(本文编辑:杨泽平)
Clinical study on the specificity of intestinal flora in colorectal cancer
YINXiaoping,LIWeihao,ZHUDongliang,etal.
(DepartmentofGastrointestinalSurgeryⅡ,XianningCenterHospital,Xianning437100,China)
Objective To study the relationship of microbial community structure in the intestine with colorectal cancer.Methods 70 patients were choosen randomly and divided into two groups on average,the control group and research group.The research group was who were diagnosed as colorectal cancer by colonoscopy and pathology,the control group was who were excluded as colorectal cancer.We collected their stool samples,and detect the bacteria in it by Amplified ribosomal DNA restriction analysis(ARDRA).Then comparedand analyze their difference.Results 389 010 high quality sequences were detected in 70 samples.The Chao1 index and Shannon index of the control group were 0.85×103and 0.14×102,the research group were 1.01×103and 0.11×102.There was no significant difference between the two groups on these indexs(P>0.05).But 15 bacterial genera were underrepresented or overrepresented in the research group′s stool samples(P<0.05).Conclusion There was a significant difference between some strains of colorectal cancer patients and the normal.It is possible to study the change law and realize the early prevention,early diagnosis and early diagnosis of colorectal cancer,and provide the basis for microbial preparation as the treatment of colorectal cancer.
intestinal flora; colorectal cancer
10.3969/j.issn.1005-6483.2017.07.014
湖北省自然科学基金资助项目(2014CFC1085)
437100 湖北省咸宁市中心医院/同济咸宁医院 湖北科技学院附属第一医院胃肠外科二病区
2016-09-29)
