盐胁迫下盐穗木DNA甲基化程度与去甲基化酶基因(Ros1)表达的相关性研究
2017-07-19张丽丽张富春
杜 驰,张 冀,张丽丽,张富春
(新疆生物资源基因工程重点实验室/新疆大学生命科学与技术学院,乌鲁木齐830046)
盐胁迫下盐穗木DNA甲基化程度与去甲基化酶基因(Ros1)表达的相关性研究
杜 驰,张 冀,张丽丽,张富春
(新疆生物资源基因工程重点实验室/新疆大学生命科学与技术学院,乌鲁木齐830046)
【目的】甲基化和去甲基化协同调控的DNA甲基化直接影响逆境胁迫相关基因的表达,植物的DNA去甲基化主要由去甲基化酶基因Ros1(Repressor of Silencing 1)介导的碱基切除修复实现。开展盐穗木(Halostachys caspica)DNA甲基化程度与HcRos1表达动态变化的分析,有助于阐明DNA甲基化应答盐胁迫的分子机制。【方法】利用qRT-PCR测定盐胁迫下盐穗木幼苗同化枝和根基因组DNA的甲基化程度,探讨DNA的甲基化程度与去甲基化酶基因HcRos1表达的相关性。【结果】在相同NaCl浓度胁迫不同时间下盐穗木同化枝和根中DNA甲基化程度呈现先升高后降低的趋势,盐穗木同化枝中的DNA甲基化程度大多高于根中的基因组甲基化程度,且均在24 h达到最高DNA甲基化程度。而在不同浓度NaCl胁迫处理24 h时,盐穗木同化枝和根中DNA甲基化程度也是先升高后降低的趋势,盐穗木同化枝中的基因组甲基化程度高于根中的基因组甲基化程度,且多在100 mmol/L达到最高DNA甲基化程度。HcRos1的基因表达量在低浓度NaCl胁迫下变化不大,但在700 mmol/L NaCl胁迫72 h时则显著升高。【结论】HcRos1表达量与DNA甲基化水平呈明显的负相关,盐胁迫能够提高HcRos1的表达,降低基因组DNA的甲基化程度,增强植物的耐盐性。
盐生植物;基因组;NaCI处理;甲基化和去甲基化;相关性
0 引言
【研究意义】植物生长发育过程中直接会受到干旱、低温、盐碱及辐射等外界环境因素的影响,这些逆境胁迫不利于植物的生长[1,2]。由于植物的固着生活方式,不能像动物选择逃避,不能像微生物选择休眠,为了生存,植物进化出抵抗逆境胁迫的适应机制。植物抗逆的生理和分子机制的研究已经表明,植物能够通过多种信号通路来调控基因的表达和修饰合成蛋白,调整逆境胁迫引起的代谢紊乱,从而增强植物的耐受性[3]。而DNA甲基化修饰是植物在非生物胁迫在转录和转录后水平调节基因表达的重要机制之一,植物能够在DNA甲基化水平呈现出的遗传表型变化往往相伴着表观遗传多态性,表明盐胁迫影响苜蓿甲基化的水平[4]。【前人研究进展】DNA甲基化(DNA methylation)在高等植物中普遍存在,主要发生部位是二核苷酸序列5'-CpG-3',生成5-甲基胞嘧啶(5 mC)[5]。DNA去甲基化(DNA demethylation)是指5 mC被胞嘧啶(C)所代替[6]。外界环境胁迫的刺激会引发植物基因组DNA甲基化程度和模式发生改变[7,8]。甲基化与去甲基化的转化由主动去甲基化基因Ros1(Repressor of Silencing 1)和DNA甲基化酶(DRM)被动抑制两种方式调控[9-11]。在干旱、低温、盐、重金属、辐射等非生物胁迫下,DNA甲基化对基因表达进行动态调控[12-14]。不同类型的胁迫会产生不同的DNA甲基化程度的动态变化,植物DNA甲基化变异是应对胁迫的重要保护措施,通过DNA甲基化的变异,刺激体内抗逆基因表达,进而增强植物的抗逆性。例如盐胁迫诱导的DNA甲基化主要发生在不同基因型水稻根中[15]。【本研究切入点】盐穗木(Halostachys caspica)是新疆典型的盐生植物,耐盐能力强,分布广泛[16-17]。研究尝试从盐胁迫下盐穗木DNA甲基化水平的检测[18]及DNA甲基化调控基因HcRos1的动态表达,分析盐穗木HcRos1的表达变化与DNA去甲基化的相关性。【拟解决的关键问题】明确盐穗木盐胁迫响应中DNA甲基化和HcRos1的表达的变化规律[19]。了解盐穗木DNA甲基化应答盐胁迫的变化特征,阐明盐穗木在盐胁迫下DNA甲基化的调控作用机制。
1 材料与方法
1.1 材料
1.1.1 盐穗木
盐穗木(Halostachys caspica)种子采集于新疆五家渠103团盐碱地(E87.31°N44.29°),保存于实验室4℃冰箱。将种子播种在装有基质(花土∶蛭石∶珍珠岩=3∶1∶1,植物营养液拌湿)花盆中,保持温度(24±2)℃,保持光照3 000 lx(350 μmol/(m2·s),16 h/8 h:昼/夜),相对湿度控制在40%~60%条件下,采用1/4 MS培养液培养4个月左右。
1.1.2 试剂
MethylFlash Methylated DNA Quantification Kit (D2485,USA),QuantiFast SYBR Green RTPCR Kit 400(Qiagen,Germany),百泰克总RNA提取试剂盒(RP3402,北京),RNA-Free DNase I,Reverse Transcriptase M-MLV(RNase H-)均购自Takara,NaCl为分析纯试剂。
1.1.3 仪器
Tanon-2500全自动数码凝胶图像分析系统(上海天能)、NanoDrop 2 000分光光度计(Thermo,USA)、150℃烘箱、ABI Prism®7500实时荧光定量PCR仪(Applied Biosystems,USA)。
1.2 方法
1.2.1 盐穗木的盐胁迫处理
选取生长了4个月左右的盐穗木幼苗进行盐胁迫处理,处理前72 h在托盘中施以自来水,以去除干旱胁迫因素。待托盘中自来水吸净无多余水分,分别将0、100、300、500和700 mmol/L NaCl溶液充分淋浇在基质上,直到溶液充满基质,自上而下淋出至于托盘。将托盘保持于处理前的培养环境,分别采集各处理浓度下不同时间点0、24和72 h,7和14 d的盐穗木样品。
1.2.2 Total DNA提取
将0、100、300、500和700 mmol/L/NaCl胁迫处理0 h、24 h、72 h、7 d和14 d后的盐穗木根和同化枝各称取200 mg,分别用Omega植物基因组DNA提取试剂盒(D2485,USA)提取基因组,提取方法参照试剂盒说明书。用ND-2000和15%变性PAGE胶检测总DNA的质量和浓度,每份组织重复3次。
1.2.3 DNA甲基化水平检测
以不同NaCl胁迫浓度处理0 h、24 h、72 h、7 d和14 d后的盐穗木根和同化枝基因组DNA为模板,稀释至10 ng/μL/,分别取10 μL基因组DNA,用基因组DNA甲基化检测试剂盒(MethylFlash Methylated DNA Quantification Kit,USA)检测盐胁迫下盐穗木根和同化枝基因组DNA甲基化水平。基因组DNA甲基化检测试剂盒的原理是DNA被固定在一种有DNA吸附能力的孔上,甲基化的片段被捕获剂以及检测抗体识别,甲基化的DNA的量与荧光强度值成线性关系,用荧光分光光度计读取微孔板的荧光强度来定量,操作方法参照试剂盒说明书。
1.3 数据处理
应用GraphPad Prism 5.0软件进行处理效应的方差(ANOVA)分析,如果处理有效,运用Turkey test进行多重比较。不同大写字母表示同一浓度胁迫不同时间下差异极显著(P<0.01),不同小写字母表示不同胁迫浓度胁迫同一时间下差异显著(P<0.05)。
2 结果与分析
2.1 相同盐浓度胁迫不同时间下盐穗木同化枝和根中全基因组DNA甲基化比较
相同盐浓度胁迫不同时间下检测盐穗木同化枝和根中DNA甲基化程度,分析时间变化对DNA甲基化程度的影响。结果发现在相同的盐胁迫浓度时,不论高盐浓度和低盐浓度,随着处理时间的延长,盐穗木同化枝和根中全基因组DNA甲基化程度呈现先升高后降低的趋势。在100 mmol/L/NaCl胁迫下时盐穗木同化枝的DNA甲基化程度最高,在24 h达到最高值,24 h、72 h、7 d和14 d的处理时间分别是对照组的2.71、2.42、1.96、1.98倍,并且随着胁迫时间延长同化枝的DNA甲基化程度降低。而在100 mmol/L/NaCl胁迫下,盐穗木根中DNA甲基化水平程度与同化枝相似,也是在24 h达到最高值,24 h、72 h、7 d和14 d的处理时间分别是对照组的3.07、2.37、2.54、2.11倍,随着盐胁迫浓度升高根的DNA甲基化程度逐渐降低。在同一NaCl浓度胁迫不同时间下盐穗木同化枝中的DNA甲基化程度大多高于根中的基因组甲基化程度,且均在24 h达到最高DNA甲基化程度。图1
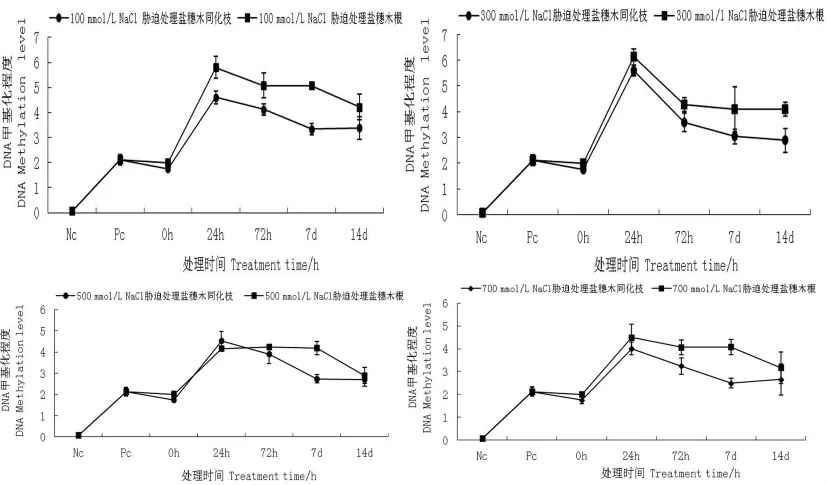
图1 相同盐浓度胁迫不同时间下盐穗木同化枝和根DNA甲基化程度变化Fig.1 Analysis of the DNA methylation between assimilating shoots and roots of Halostachys caspica under the same salt stress treatedwith different time
2.2 相同时间不同盐浓度胁迫下盐穗木同化枝和根中全基因组DNA甲基化比较
在相同时间进行100、300、500和700 mmol/ L不同浓度NaCl胁迫处理检测盐穗木同化枝DNA甲基化程度,分析盐胁迫浓度变化对DNA甲基化程度的影响。结果发现在相同的盐胁迫浓度时,不论处理时间的变化,随着盐浓度的提高,盐穗木同化枝和根中全基因组DNA甲基化程度也是呈现先升高后降低的趋势。除在处理24 h时300 mmol/L NaCl胁迫时DNA甲基化程度达到最高值外,而在72 h、7 d和14 d的处理时间,则均是100 mmol/L NaCl胁迫时DNA甲基化程度达到最高值。在100、300、500和700 mmol/L不同浓度盐浓度胁迫处理24 h时,盐穗木同化枝的DNA甲基化程度分别是对照组的2.71、3.29、2.66、2.36倍,随着盐浓度升高,甲基化程度降低。而盐穗木根中的DNA甲基化程度分别是对照组的2.83、3.07、2.08和2.25倍,同样随着盐浓度升高,甲基化程度降低。结果表明,在不同浓度NaCl胁迫处理24 h时盐穗木同化枝中的基因组甲基化程度高于根中的基因组甲基化程度,且多在100 mmol/L达到最高DNA甲基化程度。图2
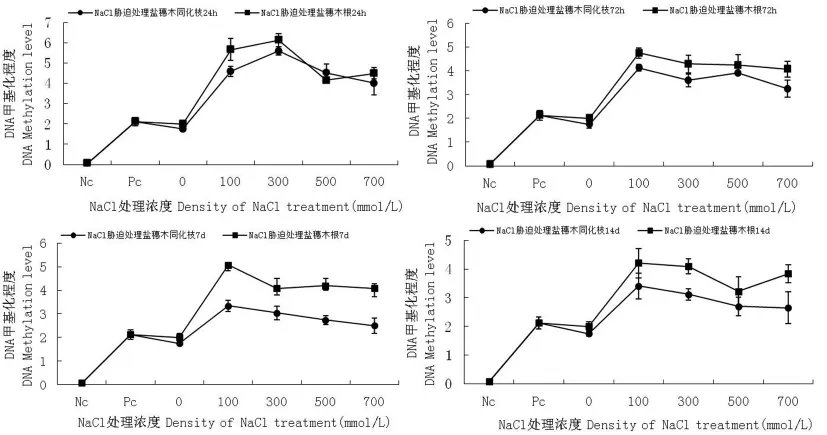
图2 不同浓度盐胁迫下盐穗木同化枝和根DNA甲基化程度的变化Fig.2 The DNA methylation level changes under different salt concentrations treated with same time between assimilating shoots and roots of Halostachys caspica
2.3 盐胁迫下HcRos1基因的表达
植物的DNA去甲基化可以由甲基化酶基因Ros1介导的碱基切除修复实现。因此盐胁迫下Ros1基因表达量会影响植物基因组DNA的甲基化程度。由于NaCI处理导致盐穗木同化枝中全基因组DNA甲基化程度呈现先升高后降低的趋势,是否与Ros1基因表达量变化具有相关性值得分析。采用qRT-PCR方法,选取β-actin基因作为内参基因,检测不同盐浓度胁迫处理不同时间后盐穗木同化枝中HcRos1 mRNA转录水平的变化。研究结果显示700 mmol/L NaCl胁迫处理后72 h时,盐穗木同化枝中HcRos1的表达量的表达量显著上调(P<0.01),在700 mmol/L NaCl胁迫24 h后则急剧增加,在胁迫72 h时表达量达到最高值,约为对照组的8.13倍,在NaCl盐胁7、14 d后,表达量略有下降,但仍然维持在较高水平。在100、300、500 mmol/L NaCl胁迫下,HcRos1的表达量则无显著性变化。结果表明,HcRos1基因的表达量在盐胁迫下较为稳定,只有在高盐浓度时才会响应盐胁迫诱导呈现高表达量。图3
2.4 盐胁迫下DNA甲基化程度与HcRos1基因表达的相关性
对HcRos1基因相对表达量和盐穗木DNA甲基化程度在不同盐浓度胁迫处理不同时间下进行相关性分析,发现盐穗木HcRos1基因表达量与DNA甲基化程度在不同NaCl胁迫处理时间下,与盐浓度表现出较高的负相关性。在不同浓度NaCl胁迫处理下,随盐胁迫浓度升高,不同胁迫时间下HcRos1表达量与DNA甲基化水平相关系数逐渐增大,分别为:-0.732 5、-0.179 5、-0.065 2和-0.028 7,负相关性降低。在盐胁迫处理24 h、72 h、7 d和14 d下,随NaCl胁迫浓度升高,相关系数变化不大,分别为:-0.648 1、-0.530 5、-0.548 2和-0.518 0,保持较高的负相关性。表1
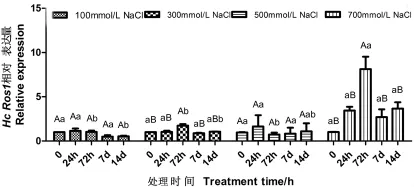
图3 盐穗木同化枝中HcRos1响应盐胁迫的相对表达量Fig.3 The relative expression of HcRos1 in assimilating shoots of Halostachys caspica response to salt stress
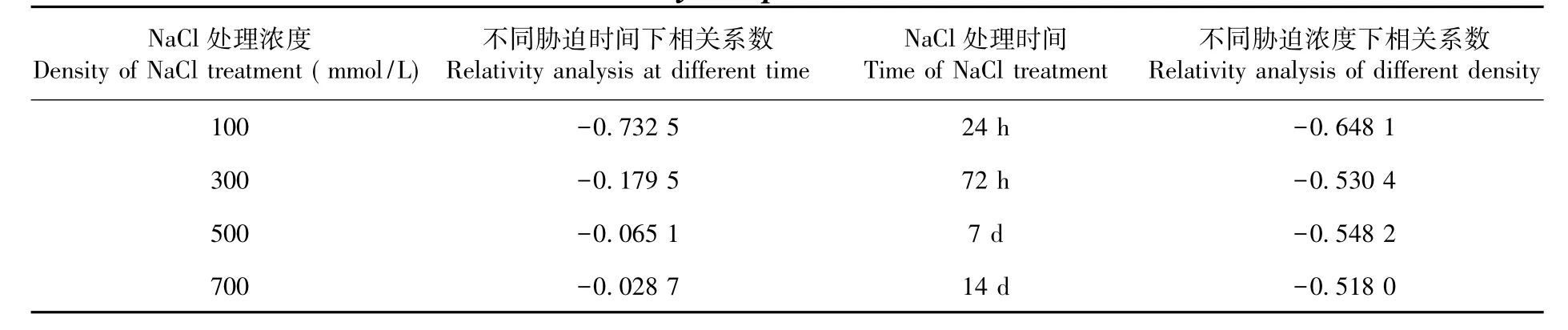
表1 盐胁迫下HcRos1基因表达和盐穗木DNA甲基化水平的相关系数Table1 The correlation coefficient of HcRos1 gene expression and DNA methylation level of Halostachys caspica under salt stress
3 讨论
DNA胞嘧啶转变为甲基化胞嘧啶是表观遗传修饰的方式之一,不仅参与调控植物的生长发育,还参与调控基因表达、基因沉默、基因组防御等表观遗传[20]。已有研究表明在不同的非生物胁迫刺激下,植物表现出DNA甲基化水平动态变化的现象,与植物抗逆基因的表达、逆境胁迫耐受性有着密切的关系[21],DNA甲基化和去甲基化酶基因的高表达影响草莓果实成熟过程颜色的变化,且在热、冷,干旱和盐逆境条件下也会增加DNA去甲基化酶基因的表达[22]。
研究发现,冷胁迫诱导植物发生DNA去甲基化,HPLC检测表明玉米在低温胁迫下基因组的DNA甲基化程度下降超过10%,Choi等也在烟草中发现了同样显著的去甲基化现象[7]。樊洪泓等[13]利用MSAP技术,研究干旱胁迫对棉花甲基化的影响,结果显示经PEG6000处理的棉花植株DNA甲基化水平降低,且与PEG6000浓度呈现负相关性。转海蓬子DREB2A基因烟草的甲基化程度明显提高,表明DREB基因可能在逆境胁迫下通过调控植物基因组甲基化发挥作用[23]。重金属胁迫引起植物的DNA甲基化变异比较复杂,并随不同的植物物种出现不同的动态变化。Labra等[12]用Cr6+等胁迫引起油菜(Brassica campestris),出现同样的DNA甲基化升高的现象,而Aina等[24]研究发现,大麻(Cannabis sativa)和三叶草(Trifolium repens)受到Cd2+、Ni2+和Cr6+等重金属胁迫后,基因组DNA甲基化程度反而下降,并且随不同处理剂量而甲基化动态变化不同(Aina et al,2004)。因此,推测不同植物应对重金属胁迫可能存在不同的甲基化机制。盐胁迫诱导主要使植物发生去甲基化现象,即通过基因组DNA的去甲基化抵御盐胁迫。如Aina发现苜蓿盐浓度与DNA甲基化程度呈负相关,李雪林[14]利用NaCl刺激棉花,研究表明在棉花根部组织中,基因组DNA甲基化比率随盐浓度上升而下降。以上研究结果与实验盐穗木盐胁迫下DNA甲基化的变化趋势类似。对比盐胁迫不同植物检测DNA甲基化水平的结果发现,植物抵御盐胁迫的过程中采取了类似的方式。
DNA去甲基化主要有两种方式:一种是被动去甲基化,是由于DNA甲基化酶活性被抑制或是植物中缺乏该酶而发生DNA去甲基化。另一种是由Ros1或Dme(Demeter)移除DNA甲基化的胞嘧啶,基因得以表达,称为主动去甲基化[25,26],Ros1具有DNA糖苷酶活性和AP裂解酶活性。DNA糖苷酶可切断DNA分子骨架和5 mC之间的N-糖苷键,AP裂解酶将脱碱基位点裂解,而后通过碱基切除修复机制(Base excision repair,BER)修复完成DNA去甲基化进程,当然后期还需要DNA聚合酶和DNA连接酶的参与。在拟南芥胚乳的发育过程中检测到这一重要生物过程,同时拟南芥ros1突变体显示高度甲基化胞嘧啶,并且多个转座子是AtRos1的目标物[27]。因此,认为Ros1对引起去甲基化反应有重要的作用。根据该课题组前期对盐胁迫下盐穗木的转录组的测序结果,调取了盐穗木HcRos1基因的EST序列进行实时定量检测,发现HcRos1基因的表达量与DNA甲基化程度呈动态负相关性。因此研究认为HcRos1基因对引发盐穗木基因组DNA发生主动去甲基化有重要作用。
4 结论
盐生植物盐穗木低甲基化状态能够间接地调控基因表达来增强盐迫的响应,在同一浓度盐胁迫下的不同时间,盐穗木同化枝和根DNA的甲基化均出现先升高后降低的变化,在24 h达到最高水平。随着盐浓度的不断增加同化枝和根DNA的甲基化程度逐渐降低,这表明DNA的甲基化程度下降强化了盐穗木的耐盐能力。而在同一时间不同浓度的盐胁迫下,盐穗木同化枝和根DNA的甲基化均也出现先升高后降低的变化,除24 h时盐穗木同化枝和根DNA的甲基化程度在300 mmol/L达到最高外,其余时间均在低盐浓度100 mmol/L达到最高值,低盐浓度的胁迫能够提高了DNA的甲基化程度,随着盐浓度增加DNA的甲基化逐渐下降。同时DNA的去甲基化又与HcRos1基因的表达密切相关,在700 mmol/L胁迫处理72 h达到最高,说明HcRos1基因的表达只有在高盐浓度时才能够响应发挥作用。因此,需要进一步研究HcRos1基因调控DNA去甲基化的具体机制,并进一步确定盐穗木DNA甲基化水平与具体耐盐基因的特异性位点之间的影响机制,才能更确切的证实DNA甲基化与植物耐盐相关基因调控的相关性。
References)
[1]Moffat,A.S.(2002).Plant genetics.finding new ways to protect drought-stricken plants.Science,296(5571):1,226-1,229.
[2]Munns,R.(2002).Comparative physiology of salt and water stress.Plant Cell&Environment,25(2):239-250.
[3]Jiang,Y.,&Huang,B.(2001).Drought and heat stress injury to two cool-season turfgrasses in relation to antioxidant metabolism and lipid peroxidation.Crop Science,41(2):436-442.
[4]Allawati,A.,Albahry,S.,Victor,R.,Allawati,A.H.,&Yaish,M.W.(2015).Salt stress alters dna methylation levels in alfalfa(medicago spp).Genetics&Molecular Research Gmr,15 (1):1-16.
[5]Saze,H.,Scheid,O.M.,&Paszkowski,J.(2003).Maintenance of cpg methylation is essential for epigenetic inheritance during plant gametogenesis.Nature Genetics,34(1):65-69.
[6]Bauer,M.J.,&Fischer,R.L.(2011).Genome demethylation and imprinting in the endosperm.Current Opinion in Plant Biology,14(2):162-167.
[7]Choi,C.S.,&Sano,H.(2007).Abiotic-stress induces demethylation and transcriptional activation of a gene encoding a glycerophosphodiesterase-like protein in tobacco plants.Molecular Genetics and Genomics,277(5):589-600.
[8]王瑞娴,徐建红.基因组DNA甲基化及组蛋白甲基化[J].遗传,2014,36(3):191-199.
WANG Rui-xian,XU Jian-hong.(2014).Genomic DNA methylation and histone methylation[J].Hereditas,36(3):191-199.(in Chinese)
[9]Gong,Z.,Moralesruiz,T.,Ariza,R.R.,Roldánarjona,T.,David,L.,&Zhu,J.K.(2002).Ros1,a repressor of transcriptional gene silencing in arabidopsis,encodes a dna glycosylase/ lyase.Cell,111(6):803-814.
[10]Mingguang,L.,Huiming,Z.,Russell,J.,Kai,T.,Shaojun,X.,&Jian-Kang,Z.(2015).Regulatory link between dna methylation and active demethylation in arabidopsis.Proceedings of the National Academy of Sciences of the United States of America,112(11):3,553-3,567.
[11]Zhu,J.,Kapoor,A.,Sridhar,V.,&Agius,F.J.(2007).The dna glycosylase/lyase ros1 functions in pruning dna methylation patterns in arabidopsis.Current Biology,17(1):54-59.
[12]Labra,M.,Grassi,F.,Imazio,S.,Fabio,T.D.,Citterio,S.,&Sgorbati,S.,et al.(2004).Genetic and dna-methylation changes induced by potassium dichromate in brassica napus l.Chemosphere,54(8):1,049-1,058.
[13]樊洪泓,李廷春,李正鹏,等.PEG模拟干旱胁迫对石斛DNA表观遗传变化的MSAP分析[J].核农学报,2011,25 (2):363-368.
FAN Hong-hong,LI Ting-chun,LI Zheng-peng.(2011).MSAPanalysisofepigeneticchangesinDendrobiumhuoshanenseunder PEG simulated drought stress[J].Acta Agriculturae Nucleatae Sinica,25(2):363-368.(in Chinese)
[14]李雪林,林忠旭,聂以春,等,2009.盐胁迫下棉花基因组DNA表观遗传变化的MSAP分析[J].作物学报,2009,35 (4):588-596.
LI Xue-lin,LIN Zhong-xu,NIE Yi-chun,et al.(2009).MSAP Analysis of Epigenetic Changes in Cotton(Gossypium hirsutumL.)under Salt Stress[J].Acta Agronomica Sinica,35 (4):588-596.(In Chinese)
[15]Wang,W.,Huang,F.,Qin,Q.,Zhao,X.,Li,Z.,&Fu,B.(2015).Comparative analysis of dna methylation changes in two rice genotypes under salt stress and subsequent recovery.Biochemical&Biophysical Research Communications,465(4):790-796.
[16]常丹,张霞,张富春.盐穗木HcPEAMT基因的克隆及表达分析[J].西北植物学报,2014,34(8):1,522-1,528.CHANG Dan,ZHANG Xia,ZHANG Fu-chun.(2014).Cloning and expression analysis of HcPEAMT gene from Halostachys caspica[J].Acta Botanica Boreali-Occidentalia Sinica,34(8):1,522-1,528.(in Chinese)
[17]张立新,李生秀.甜菜碱与植物抗旱/盐性研究进展[J].西北植物学报,2004,24(9):1,765-1,771.
ZHANG Li-xin,LI Sheng-xiu.(2004).Research progress on relationships betaine and drought/salt resistance of plants[J].Acta Botanica Boreali-Occidentalia Sinica,24(9):1,765-1,771.(In Chinese)
[18]RAUSCH,S.,LAUBINGER,S.(2016).Rapid Assessment of DNA Methylation Changes in Response to Salicylic Acid by Chop-qPCR[J].Methods in Molecular Biology,1,398:345.
[19]Keshet,I.,Yisraeli,J.,&Cedar,H.(1985).Effect of regional dna methylation on gene expression.Proceedings of the National Academy of Sciences of the United States of America,82(9):2,560-2,564.
[20]Chinnusamy,V.,&Zhu,J.K.(2009).Epigenetic regulation of stress responses in plants.Current Opinion in Plant Biology,12 (2):133-139.
[21]Mirouze,M.,&Paszkowski,J.(2011).Epigenetic contribution to stress adaptation in plants.Current Opinion in Plant Biology,14(14):267-274.
[22]Gu,T.,Shuai,R.,Wang,Y.,Han,Y.,&Yi,L.(2016).Characterization of dna methyltransferase and demethylase genes in fragaria vesca.Molecular Genetics and Genomics,291(3):1,333-1,345.
[23]More,P.,Agarwal,P.,Mastan,S.G.,&Agarwal,P.K.(2016).Msap marker based dna methylation study in salicornia brachiata,dreb2a transgenic tobacco.Plant Gene,(6):77-81.
[24]Aina,R.,Sgorbati,S.,Santagostino,A.,Labra,M.,Ghiani,A.,&Citterio,S.(2004).Specific hypomethylation of dna is induced by heavy metals in white clover and industrial hemp.Physiologia Plantarum,121(3):472-480.
[25]Ponferradamarín,M.I.,Roldánarjona,T.,&Ariza,R.R.(2009).Ros1 5-methylcytosine dna glycosylase is a slow-turnover catalyst that initiates dna demethylation in a distributive fashion.Nucleic Acids Research,37(13):4,264-4,274.
[26]Schoft,V.K.,&Tamaru,H.(2011).Function of the demeter dna glycosylase in the arabidopsis thaliana male gametophyte.Proceedings of the National Academy of Sciences of the United States of America,108(19):8,042-8,047.
[27]Lister,R.,O'Malley,R.C.,Tontifilippini,J.,Gregory,B.D.,Berry,C.C.,&Millar,A.H.,et al.(2008).Highly integrated single-base resolution maps of the epigenome in arabidopsis.Cell,133(3):523-536.
Correlation Analysis of DNA Methylation and Expression of Demethylation Enzyme Gene(Ros1)in Halostachys caspica under Salt Stress
DU Chi,ZHANG Ji,ZHANG Li-li,ZHANG Fu-Chun
(Technology,Xinjiang University and Xinjiang Key Laboratory of Biological Resources and Genetic Engineering,College of Life Science,Urumqi 830046,China)
【Objective】The DNA methylation,which is regulated by methylation and demethylation coordinately,directly affects the expression of stress-related genes.DNA demethylation in plants is mainly mediated by demethylase gene Ros1(Repressor of silencing 1)to finish the base excision repair.Analysis of the dynamic changes of the DNA methylation of Halostachys caspica and gene expression changes of Ros1 will be helpful to elucidate the molecular mechanism of DNA methylation in response to salt stress.【Method】qRT-PCR was used to measure the degree of genomic DNA methylation in assimilation shoots and roots of H.caspica,the correlation between DNA methylation and HcRos1 gene expression was also analyzed.【Result】The results showed that the degree of genomic DNA methylation in assimilation shoots and roots of H.caspica increased firstly and then decreased under the same concentration of NaCl stress treated with different times,and DNA methylation in assimilating shoots was higher than in roots and reached the highest at 24 h.However,the degree of genomic DNA methylation in assimilation shoots and roots of H.caspica also increased firstly and then decreased under the different concentrations of NaCl stress treated with the same time,DNA methylation in assimilating shoots was higher than in roots and reached the highest under concentration of 100 mmol/L NaCl.The gene expression of HcRos1 did not changed under low concentration of NaCl stress,but increased significantly under concentration of 700 mmol/L at 72 h.【Conclusion】Correlation analysis demonstrated that the expression of HcRos1 was negatively correlated with the degree of DNA methylation.Salt stress can increase the expression of HcRos1,reduce the methylation degree of genomic DNA and enhance the salt tolerance of plants.
halophyte;genome;NaCI treatment;methylation and demethylation;correlation analysis
ZHANG Fuchun(1962-),Male,Professor,Doctor,Engaged in molecular biology research,(E-mail:)zfcxju@xju.edu.cn
Q756;S188
A
1001-4330(2017)05-0878-08
10.6048/j.issn.1001-4330.2017.05.011
2017-02-27
新疆重点实验室专项资金项目“盐穗木盐胁迫响应的转录组研究及重要基因的功能鉴定”(2014KL001);国家“973”计划前期研究专项“新疆荒漠盐生植物的耐盐生理及分子机理”(2012CB722204)
杜驰(1991-),女,硕士研究生,研究方向为植物分子生物学,(E-mail)924301992@qq.com
张富春(1962-),男,教授,博士,研究方向为分子生物学,(E-mail)zfcxju@xju.edu.cn
Supported by:Special funds for Xinjiang key laboratory"The salt-stress responsive transcriptome of the halophyte halostachyscaspica and the functional identification of the major salt related genes"(2014KL001)and the national 973 pre-research project"Physiological and molecular mechanisms of salt tolerance of desert halophytes in Xinjiang"(2012CB722204)
