CRISPR/Cas9介导的基因组编辑技术及其在RNA编辑中的潜在应用
2017-02-20任文琳徐桂霞牟亚妮吴松青
任文琳, 张 俊, 徐桂霞, 牟亚妮, 楼 杨, 吴松青*
1.福建农林大学生命科学学院, 福州 350002; 2.福建农林大学林学院, 福州 350002; 3.福建农林大学植物保护学院, 福州 350002
CRISPR/Cas9介导的基因组编辑技术及其在RNA编辑中的潜在应用
任文琳1, 张 俊2, 徐桂霞2, 牟亚妮1, 楼 杨3, 吴松青2*
1.福建农林大学生命科学学院, 福州 350002; 2.福建农林大学林学院, 福州 350002; 3.福建农林大学植物保护学院, 福州 350002
化脓链球菌里的 CRISPR-Cas9系统作为基因编辑工具已经得到广泛的应用。现有研究表明这个系统具有作为通用核酸识别技术的潜在价值,即Cas9系统也能靶向识别活细胞中的RNA。将RCas9与不同的蛋白因子融合,可能会实现基因表达调控、控制RNA的定位、改变RNA的组成和使RNA可视化等。在介绍CRISPR-Cas9系统介导的免疫机制的基础上,重点比较了RCas9和先前的RNA研究方法,从转录动力学、再生医学以及合成生物学等方面,综述了其潜在的应用价值,并对该系统在未来生命科学研究的应用进行了展望,以期为CRISPR-Cas9系统在RNA编辑方面的研究提供参考。
CRISPR-Cas9;RCas9;RNA生物学;RNA靶向技术
从十多年前人类基因组计划完成后,分子生物学的研究方向就转向了阐述功能基因的重要性[1]。虽然一个正常生物体的细胞核DNA是相同的,但是基因转录活性的高低造成了细胞的分化,从而使细胞执行不同的功能。于是,以细胞为单位对RNA的检测不仅可以区分不同的细胞,也可以用来判断健康或疾病状态。例如,自闭症患者神经细胞的变异程度与转录过程中一个基因联合表达模板的表达程度密切相关[2]。过去认为非编码RNA不能编码蛋白质,只起到调控基因表达的作用,但德克萨斯大学西南医学中心的最新研究表明骨骼肌中一种特异性生物长链非编码RNA(lncRNA)编码了一个包含46个氨基酸的微肽,这种小型蛋白在心脏肌肉收缩中扮演关键角色[3]。这些都表明对RNA的研究将会是疾病模型构建、临床诊断和治疗的关键。CRISPR-Cas(clustered regularly interspaced short palindromic repeats and CRISPR associated)系统是细菌和古细菌长期进化过程中形成的一种适应性免疫系统[4]。本文介绍了CRISPR-Cas9系统介导的免疫机制,综述了从转录动力学到再生医学再到合成生物学等方面其潜在的应用价值,并对该系统在未来生命科学研究领域的应用进行了展望,以期为CRISPR-Cas9系统在RNA编辑方面的研究提供参考。
1 RCas9的研究现状
1.1 CRISPR/Cas9的作用机制
目前研究得最深入的CRISPR-Cas系统是Ⅱ型CRISPR-Cas系统,通常由Cas9核酸酶和一对RNA,即反式CRISPR RNA(tracrRNA)和CRIPSR RNA(crRNA)构成,细菌在抵御噬菌体等外源DNA入侵时,tracrRNA和crRNA配对形成RNA二聚体,进而和Cas9形成核糖核蛋白复合物介导外源DNA的切割[5]。通过人工可以将tracrRNA和crRNA组合成单导向RNA(sgRNA),化脓性链球菌的Ⅱ型CRISPR-Cas系统就能通过sgRNA定位哺乳动物的DNA[6]。Cas9靶向定位DNA需要两个元素:除了与靶向DNA序列互补的sgRNA之外,还需要一个NGG序列(其中n为任何核苷酸),也被称为前间区序列邻近基序(PAM)[7]。PAM是CRISPR-Cas系统识别外源DNA的必要条件。当噬菌体首次入侵细菌时,对PAM序列进行扫描后,其邻近的序列才能插入宿主菌CRISPR基因座形成间隔序列。并且在外源靶向DNA位点附近存在PAM,而宿主细胞的这些靶向位点缺乏PAM,这样就可以避免宿主菌产生自我免疫[8]。由于利用sgRNA和PAM能够很容易地定位靶DNA序列,使Cas9迅速成为一种通用的编辑基因和调控转录的工具。
1.2 RCas9在体内应用面临的主要挑战
近期对RCas9系统的研究表明,在体外条件下Cas9对RNA有很强的特异性结合能力,且随后Cas9可以使单链RNA裂解[9]。Cas9靶向定位RNA同样需要sgRNA和PAM这两个元素,不同的是RCas9系统中的PAM是由杂交的反义寡核苷酸(the PAMmer)提供,与靶RNA杂交后与sgRNA的互补序列相邻。O′Connell等[9]的实验表明,要使sgRNA能够识别特定RNA,需要将PAM序列的5′端延伸。如果在5′-NGG-3′上游添加2~8个核苷酸使5′端延长,将会使sgRNA和RNA的结合获得较高的特异性,并且可以避免定位到对应的模板DNA上。缺少这种5′端延伸的短PAMmers,无法激活Cas9与sgRNA的结合。这种机制可能会抵消PAMmer 与RNA解螺旋带来的高能量损耗。
Cas9和同源sgRNA的运输已通过各种方法实现,包括利用可以编码Cas9和sgRNA的病毒,利用药物诱导使Cas9在转基因动物中表达[10],以及通过阳离子脂质体实现Cas9蛋白和sgRNA的运输[11]。同时要保证一个有效手段将RCas9的 PAMmer运输到正确的位置。虽然这些方法已用于在体内运输的RCas9系统中的1个或2个部件,但目前仍然需要探索载体以哪种方式与3个组件(Cas9、sgRNA和PAMmer)结合更有利于其在体内的有效重建。
O′Connell等[9]在研究过程中发现,DNA-RNA杂交链会被细胞内的RNase H降解。早期的研究表明,被修饰的DNA寡核苷酸可以消除RNase H的活性[12],如对PAMmer进行2′-羟基甲基化修饰可以有效地防止靶RNA被降解。对其长度进行细微调整或修饰也可以避免启动RNAi机制。
上述研究只在体外进行,Cas9在活体细胞内或生物体内定位RNA的强度和特异性尚不清楚。例如,虽然RCas9在体外不剪切DNA,但在体内是否会出现定位DNA的情况还是未知的。RCas9能否成功地在体内应用从根本上取决于它的特异性和是否能作用于靶RNA,为了评估其作为一种细胞内RNA编辑工具的潜能,还需要对RCas9的特异性进行广泛验证。
2 RCas9系统的潜在应用
2.1 调控转录后的基因表达
RCas9最普遍的应用是利用Cas9内切酶活性使特定的转录产物裂解,从而下调基因表达。虽然RNA干扰(RNAi)也能进行RNA识别和切割,但当RNAi机制不存在或者不活跃时[13],基于RCas9的基因敲除在细胞中将发挥重要作用,并且RNAi在作用过程中存在着诸多局限性。2003年,Jackson等[14]使用基因芯片技术研究人体细胞内siRNA介导的基因沉默时,发现siRNA对其不完全匹配的mRNA也具有抑制作用,同时mRNA局部二级结构[15]和长dsRNA[16]也会影响siRNA的作用效果。相比之下,RCas9对RNA有高亲和力且能被sgRNA和PAMmer双重识别,比siRNA能降解更多的靶向RNA。具体比较见表1。
另外,使基因表达上调的途径是难以获得的。CRISPR-Cas系统的另外一种形式称为CRISPR干扰(CRISPRi),CRISPRi依赖转录调节剂与无核酸酶活性的Cas9(dCas9)融合[17]调控基因的表达。dCas9虽然失去了剪切DNA的能力,但依然能和sgRNA共表达,形成一种DNA识别复合物。由此,研究人员试图将dCas9转化为能激活基因表达的转录因子,即在RCas9系统中,将dCas9与翻译增强因子融合,在不改变RNA丰度的同时可能会达到增强特定基因表达的目的。
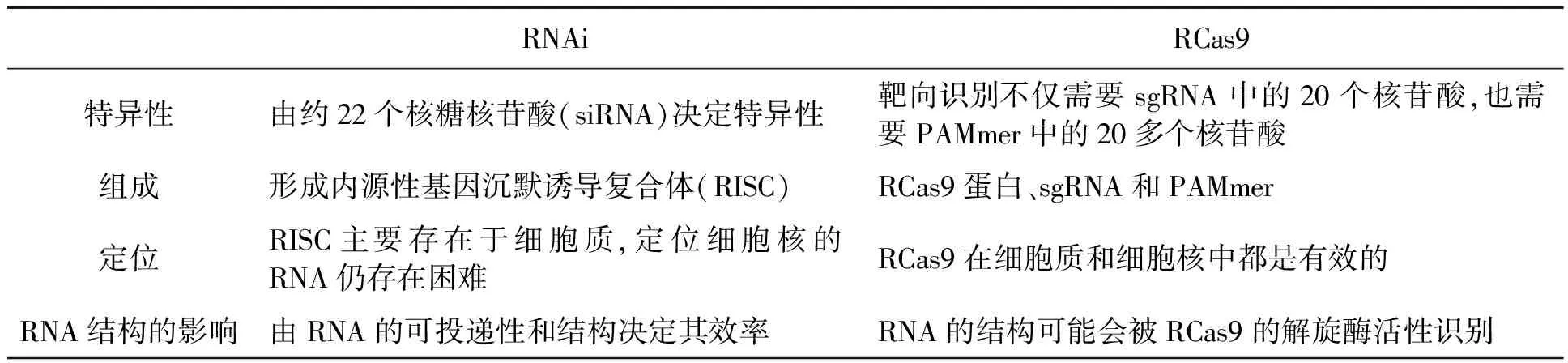
表1 RNAi与RCas9在基因表达调控中的比较
2.2 应用于再生医学
控制RNA的定位可能是再生治疗的一个重要方法。例如在神经元中,mRNA将参与突触的建成活动,但突触间隙会给积累足够浓度的突触蛋白带来挑战。一些证据表明编码细胞骨架成分的RNA的定位是轴突再生的关键[18],通过与转运因子融合,结合了这些特定RNA的RCas9系统会被运输到某个选定区域,如突触前端或末端,使突触蛋白积累到正常的生理浓度。即在RCa9系统的参与下有可能实现损伤后神经元的再生。
RCas9也可以用来改变RNA的组成。前体mRNA的可变剪接是mRNA合成的关键步骤,剪接因子在前体mRNA上的不同作用位点可以产生多种mRNA。剪接因子与RCas9融合后,其作用位点的选择可能会受到RCas9与靶向mRNA结合位点的影响,即产生特定的剪接因子。这种有针对性的剪接可以用来逆转异常剪接引起的各种疾病[19]。例如,脊髓性肌萎缩是由运动神经元生存基因SMN1缺失导致的神经元死亡引起的,但有证据表明,在SMN1基因缺失的细胞中改变SMN2的剪接可以产生1个同类型的SMN2,从而重新获得SMN1基因的活性[20]。这不仅促进了疾病模型的建立,也为再生医学药物的应用奠定了基础。
2.3 应用于RNA成像
2.3.1 不同的RNA成像方法 近些年发展起来的几种核酸识别工具已经能够使活细胞中的特异性核酸成像,但也存在一些缺陷[21]。类似于与荧光蛋白融合使蛋白质可视化的方式,一些RNA识别系统通过序列标签与靶向RNA的结合使RNA可视化,这些RNA标签可以被某些蛋白质特异性识别。目前应用最为广泛的技术是通过MS2噬菌体的外壳蛋白(MCP)与荧光蛋白[22]融合,识别RNA序列中的发卡结构。这是一种使高丰度RNA在活细胞中成像的高效方法[23],但游离的MS2荧光蛋白会引起较高的背景值[22],也可能影响mRNA的稳定性[24]。另一种方法是将分子信标与外源性小分子荧光结合[25],当核酸适配体和靶向RNA发生杂交后,通过荧光基团与荧光淬灭剂分离或利用荧光能量共振转移技术(FRET)产生荧光信号[26]。第3种方法是基于将荧光蛋白拆分为两个结构域,分别与RNA结合结构蛋白融合[27]。当该复合体与靶向RNA结合时,通过两个荧光发射团的重组可实现RNA的可视化成像。这种方法可以有效抑制背景荧光[28]。
虽然这3种方法被广泛用于研究RNA动力学,但面临着高背景噪声、运输繁琐、序列标签影响RNA活性等问题。RCas9可以在保证高特异性和低噪声的同时,通过直接识别未标记的RNA来避免这些问题。
RCas9系统具有同时使一个或多个内源性RNA丰度和定位可视化的潜能。通过将dCas9与荧光蛋白融合,可以显示特定的RNA的定位。另外,将1对Cas9蛋白分别与一半荧光蛋白融合,如黄色荧光蛋白Venus[29],可以定位到RNA的邻近位点。这可能会比一个完整的荧光蛋白产生更低的背景值,从而实现RNA位置的可视化或单个细胞中RNA含量的测量。这种利用部分荧光蛋白的方法还可以用于在不同的选择性剪接过程中定位相邻的外显子,对于表达特定RNA剪切异构体的细胞进行鉴定和分离。这种利用Cas蛋白和sgRNA的正交鉴定技术可以同时定位多个转录,从而进行RNA位置和丰度的测量,也可以用于个体活细胞RNA动力学的综合性研究。
2.3.2 应用于干细胞研究 活细胞内源性RNA定位和丰度测量的应用非常广泛。例如目前阐明干细胞的特性仍然存在困难,因为只存在少数几个表面标记用于细胞识别和纯化,基因表达图谱是目前鉴定干细胞类型的最有效方法[30]。例如CD44是一种肿瘤干细胞(CSCs)标记,位于细胞表面,参与细胞间的相互作用、细胞的黏附和迁移[31]。为了研究CD44基因在肝癌干细胞中的整体功能,来自北京大学医学部和南方医科大学的研究人员[32]使用CRISPR/Cas9系统,敲除了人肝细胞癌细胞系C3A中的CD44,证实了CD44通过保持低分化的肿瘤细胞群,在肝癌干细胞中发挥重要功能。RCas9同样可以满足这种无损检测,所以少量干细胞可以实现保存、增殖和分离。
2.3.3 用于应激颗粒的研究 RNA定位在细胞对损伤、应激和某些行为的应答中也具有重要意义。应激颗粒是一种RNA和蛋白质的聚合体,通常在应对氧化应激、高温、病毒感染或缺氧时形成[33]。许多疾病都与RNA颗粒的异常形成有关:如登革热病毒在侵染宿主后降低了宿主细胞整体的mRNA含量,抑制了应激颗粒的形成[34];而脊髓灰质炎病毒在感染早期可诱导应激颗粒的产生,后期病毒剪切应激颗粒使之降解[35],从而有利于其自身mRNA的复制。但是,对于应激颗粒中RNA的研究才刚刚起步,利用RCas9系统使应激颗粒中的内源性RNA成像将有助于弄清应激颗粒在健康和疾病中的重要性。如利用RCas9在病变神经元中追踪RNA运输,检测应激颗粒形成,以鉴别出这些疾病的分子特征[36]。这表明RCas9系统介导的RNA定位将在受损组织再生中发挥作用。
2.4 应用于合成生物学
合成生物学发展的重点在于建立基于基因组和蛋白质组的模块化的资源库和技术平台,从而应用于设计或改造生物大分子、生物部件、代谢途径或发育过程。RCas9系统的高度模块化和可编程性使其在合成生物学中可以作为潜在的技术平台。例如,Cas9蛋白可以与裂解酶融合,在识别靶向RNA后使裂解酶恢复活性。例如检测出和癌症相关的RNA后,Cas9可以使分裂的死亡诱导蛋白恢复活性。甚至,通过Cas9蛋白和RNA的相互作用,可能会重新设计涉及连续蛋白质之间的相互作用途径。在基因表达的过程中,RCas9在蛋白质之间的相互作用中可能起支架作用以控制信号传输。例如杜克大学的研究人员利用dCas9将酶携带至基因组中的特异位点,将一种表观遗传标记——乙酰基添加到了组蛋白上,结果发现靶基因的表达量上调[37]。原则上,Cas9蛋白和靶向RNA之间强有力的结合效果可以优于连续蛋白质的相互作用,从而达到一个新的调控水平。这表明RCas9系统也具有阐明表观遗传学修饰的功能和遗传性,协助开发控制细胞表型和干涉基因组调控新策略的潜能[38]。
3 展望
从RNA结合蛋白的结构域作为RNA序列特异性的决定因素到RCas9通过简单的核酸杂交进行靶向识别,RNA靶向技术的研究取得了和DNA靶向技术相当的发展。ZFN(锌指技术)和TALEN(转录激活样效应因子核酸酶技术)为Cas9介导的DNA识别技术奠定了基础。在定位DNA的应用中,Cas9及其sgRNA在哺乳动物细胞内表现得很稳定且无毒性,但编码RNA的识别技术则处于起步阶段,RCas9系统的3个组件(Cas9、sgRNA和PAMmer)是否能够有效传递并成功结合RNA还有待观察。RCas9系统通过简单的碱基配对识别无标记的内源RNA的能力,是RNA识别技术中取得的主要成就,在诊断和治疗中的应用尤为重要。如果在可以避免定位基因组DNA和转录脱靶的同时,将RCas9系统提供给细胞进行RNA识别,可以预见该系统在基础应用生物学和医学领域将会有很多新的应用。
[1] Consortium T E P. An integrated encyclopedia of DNA elements in the human genome [J]. Nature, 2012, 489(7414):57-74.
[2] Voineagu I, Wang X, Johnston P,etal.. Transcriptomic analysis of autistic brain reveals convergent molecular pathology [J]. Nature, 2011, 474(7351):380-384.
[3] Anderson D, Anderson K, Chang C L,etal.. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance [J]. Cell, 2015, 160(4):595-606.
[4] Mohan B, Natalia B, Robert F,etal.. A dual function of the CRISPR-Cas system in bacterial antivirus immunity and DNA repair [J]. Mol. Microbiol., 2011, 79(2):484-502.
[5] Sander J D, Joung J K. CRISPR-Cas systems for editing, regulating and targeting genomes [J]. Nat. Biotechnol., 2014, 32(4):347-355.
[6] Mali P, Yang L, Esvelt K M,etal.. RNA-guided human genome engineering via Cas9 [J]. Science, 2013, 339(6121):823-826.
[7] Mojica F J, Díez-Villaseor C, García-Martínez J,etal.. Short motif sequences determine the targets of the prokaryotic CRISPR defence system [J]. Microbiology, 2009, 155(3):733-740.
[8] Marraffini L A, Sontheimer E J. Self versus non-self discrimination during CRISPR RNA-directed immunity [J]. Nature, 2010, 463(463):568-571.
[9] O′Connell M R, Oakes B L, Sternberg S H,etal.. Programmable RNA recognition and cleavage by CRISPR/Cas9 [J]. Nature, 2014, 516(7530):263-266.
[10] Kasap C, Elemento O, Kapoor T M. Drug target SeqR: a genomics- and CRISPR-Cas9-based method to analyze drug targets [J]. Nat. Chem. Biol., 2014, 10(8):626-628.
[11] Steyer B, Carlson-Stevermer J, Angenent-Mari N,etal.. High content analysis platform for optimization of lipid mediated CRISPR-Cas9 delivery strategies in human cells[J]. Acta Biomater., 2015, 34:143-158.
[12] Wu H, Lima W F, Crooke S T. Properties of cloned and expressed human RNase H1[J]. J. Biol. Chem., 1999, 274(40):28270-28278.
[13] Xie K. RNAi in budding yeast [J]. Science, 2009, 326(5952):544-550.
[14] Jackson A L, Bartz S R, Schelter J,etal.. Expression profiling reveals off-target gene regulation by RNAi[J]. Nat. Biotechnol., 2003, 21(6):635-637.
[15] Birmingham A, Anderson E M, Reynolds A,etal.. 3′ UTR seed matches, but not overall identity, are associated with RNAi off-targets [J]. Nat. Methods, 2006, 3(6):199-204.
[16] Kulkarni M M, Booker M, Silver S J,etal.. Evidence of off-target effects associated with long dsRNAs in Drosophila melanogaster cell-based assays [J]. Nat. Methods, 2006, 3(10):833-838.
[17] Qi L, Larson M, Gilbert L,etal.. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5):1173-1183.
[18] Donnelly C J, Willis D E, Xu M ,etal.. Limited availability of ZBP1 restricts axonal mRNA localization and nerve regeneration capacity[J]. EMBO J., 2011, 30(22):4665-4677.
[19] David C J, Manley J L. Alternative pre-mRNA splicing regulation in cancer: pathways and programs unhinged [J]. Genes Dev., 2010, 24(21):2343-2364.
[20] Hua Y, Vickers T A, Okunola H L,etal.. Antisense masking of an hnRNP A1/A2 intronic splicing silencer corrects SMN2, splicing in transgenic mice [J]. Am. J. Human Genet., 2008, 82(4):834-848.
[21] Rath A K, Rentmeister A. Genetically encoded tools for RNA imaging in living cells [J]. Curr. Opin. Biotechnol., 2015, 31:42-49.
[22] Bertrand E, Chartrand P, Schaefer M,etal.. Localization of ASH1, mRNA particles in living yeast[J]. Mol. Cell, 1998, 2(4):437-445.
[23] Park H Y, Lim H, Yoon Y J,etal.. Visualization of dynamics of single endogenous mRNA labeled in live mouse [J]. Science, 2014, 343(6169):422-424.
[24] Lange S, Katayama Y, Schmid M,etal.. Simultaneous transport of different localized mRNA species revealed by live-cell imaging[J]. Traffic, 2008, 9(8):1256-1267.
[25] Strack R L, Disney M D, Jaffrey S R. A superfolding Spinach2 reveals the dynamic nature of trinucleotide repeat-containing RNA [J]. Nat. Methods, 2013, 10(12):1219-1224.
[26] Shin I, Ray J, Gupta V,etal.. Live-cell imaging of Pol Ⅱ promoter activity to monitor gene expression with RNA IMAGE tag reporters [J]. Nucl. Acids Res. Adv. Access, 2014, 42(11):2247-2258.
[27] Magliery T J, Wilson C G M, Pan W,etal.. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: scope and mechanism [J]. J. Am. Chem. Soc., 2005, 127(1):146-157.
[28] Rackham O, Brown C M. Visualization of RNA-protein interactions in living cells: FMRP and IMP1 interact on mRNAs [J]. EMBO J., 2004, 23(16):3346-3355.
[29] Ozawa T, Natori Y, Sato M,etal.. Imaging dynamics of endogenous mitochondrial RNA in single living cells [J]. Nat. Methods, 2007, 4(5):413-419.
[30] Zhang J, Nuebel E, Daley G,etal.. Metabolic regulation in pluripotent stem cells during reprogramming and self-renewal[J]. Cell Stem Cell, 2012, 11(5):589-595.
[31] Leung E L, Fiscus R R, Tung J W,etal.. Non-small cell lung cancer cells expressing CD44 are enriched for stem cell-like properties [J]. PLoS ONE, 2010, 5(11):e14062.
[32] Han S, Guo J H, Liu Y N,etal.. Knock out CD44 in reprogrammed liver cancer cell C3A increases CSCs stemness and promotes differentiation [J]. Oncotarget, 2015, 6(42):44452-44465.
[33] Sonenberg N, Hinnebusch A G. Regulation of translation initiation in eukaryotes: Mechanisms and biological targets [J]. Cell, 2009, 136(4):731-745.
[34] Xia J, Chen X, Xu F,etal.. Dengue virus infection induces formation of G3BP1 granules in human lung epithelial cells[J]. Arch. Virol., 2015, 160(12):2991-2999.
[35] Fitzgerald K D, Semler B L. Poliovirus infection induces the co-localization of cellular protein SRp20 with TIA-1, a cytoplasmic stress granule protein [J]. Virus Res., 2013, 176(1-2):223-231.
[36] Nelles D A, Fang M Y, O′Connell M R,etal.. Programmable RNA tracking in live cells with CRISPR/Cas9 [J]. Cell, 2016, 165(2):488-496.
[37] Hilton I B, D′Ippolito A M, Vockley C M,etal.. Epigenome editing by a CRISPR/Cas9-based acetyltransferase activates genes from promoters and enhancers [J]. Nat. Biotechnol., 2015, 33(5):510-517.
[38] Doudna J A, Gersbach C A. Genome editing: the end of the beginning [J]. Genome Biol., 2014, 16(1):1-3.
CRISPR/Cas9-mediated Gene Editing Technology and Potential Applications in RNA Editing
REN Wenlin1, ZHANG Jun2, XU Guixia2, MOU Yani1, LOU Yang3, WU Songqing2*
1.CollegeofLifeSciences,FujianAgricultureandForestryUniversity,Fuzhou350002,China; 2.CollegeofForestry,FujianAgricultureandForestryUniversity,Fuzhou350002,China; 3.CollegeofPlantProtection,FujianAgricultureandForestryUniversity,Fuzhou350002,China
The CRISPR-Cas9 system, fromStreptococcuspyogenes, has gained widespread application as a genome editing tool. Previous studies showed that CRISPR-Cas9 system has the potential utility as a universal nucleic acid-recognition technology, that is Cas9 system can also allow RNA identification and editing in live cells. By fusing RCas9 to different protein factors, it may be possible to modulate gene expression, control the localization of RNA, alter the composition of RNAs and enable imaging of specific RNA. In this paper, the mechanism of CRISPR-Cas9 system-mediated immunity was introduced, in particular, we compared RCas9 to previous methods for RNA targeting and discussed potential uses ranging from live imaging of transcriptional dynamics to regenerative therapies and applications in synthetic biology, and finally prospected the application of this new system in life science, which was expected to provide reference for CRISPR-Cas9 system application in RNA editing.
CRISPR-Cas9; RCas9; RNA biology; RNA targeting
2016-06-22; 接受日期:2016-07-18
国家林业公益性行业科研专项(201304401);中央财政林业科技推广示范项目[Min(2015)TG017];福建省科技厅自然科学基金项目(2016J01097); 福建省教育厅科技项目(JA15161);福建农林大学重点项目建设专项(6112C035003);福建农林大学科研基金项目(2014xjj12)资助。
任文琳,本科生,主要从事微生物农药研究。E-mail:863507710@qq.com。*通信作者:吴松青,讲师,博士,主要从事微生物农药研究。E-mail: dabinyang@126.com
10.3969/j.issn.2095-2341.2017.01.01
