条斑紫菜Mu型谷胱甘肽S-转移酶基因克隆与分析
2016-12-19易乐飞李信书阎斌伦
易乐飞,郝 伟,李信书,阎斌伦
( 淮海工学院 海洋学院,江苏 连云港 222005 )
条斑紫菜Mu型谷胱甘肽S-转移酶基因克隆与分析
易乐飞,郝 伟,李信书,阎斌伦
( 淮海工学院 海洋学院,江苏 连云港 222005 )
谷胱甘肽S-转移酶是动植物重要的解毒酶,分布广泛,类型众多。本研究利用生物信息学方法和RT-PCR技术获得了条斑紫菜一条新谷胱甘肽S-转移酶(PyGST2)基因的cDNA序列,该基因含有完整的开放阅读框,并且受到铅胁迫的诱导表达。PyGST2蛋白具有谷胱甘肽S-转移酶家族的保守结构域和保守氨基酸残基,与Mu型谷胱甘肽S-转移酶的序列一致性最高,与Alpha、Sigma和Pi型谷胱甘肽S-转移酶的次之,在进化树上远离高等植物的各型谷胱甘肽S-转移酶,而与动物的Mu型谷胱甘肽S-转移酶聚为一支。该Mu型谷胱甘肽S-转移酶的克隆为后续研究条斑紫菜抗逆机制奠定了基础。
条斑紫菜;谷胱甘肽S-转移酶;克隆;表达
谷胱甘肽S-转移酶(glutathione S-transferase,GST,E.C2.5.1.18)是一类分布广泛且功能多样的蛋白超家族[1]。谷胱甘肽S-转移酶主要负责催化还原型谷胱甘肽与非极性化合物结合,因此谷胱甘肽S-转移酶的主要功能是通过硫醚氨酸途径参与外源性有毒物质的解毒过程[2]。谷胱甘肽S-转移酶还参与了内源性过氧化物的清除、芳香族氨基酸的降解、类固醇激素的合成、花生四烯酸的合成与失活、信号转导等过程[2-3]。
谷胱甘肽S-转移酶蛋白家族成员间序列相似性低,型别众多。根据氨基酸序列一致性、基因结构和免疫活性等将谷胱甘肽S-转移酶家族成员进行了分类,哺乳动物可溶性谷胱甘肽S-转移酶分为Sigma、Theta、Zeta、Omega、Alpha、Mu和Pi型,植物可溶性谷胱甘肽S-转移酶分为Theta、Zeta、Lambda、Phi、Tau和DHAR(dehydroascorbate reductases)型,昆虫可溶性谷胱甘肽S-转移酶分为Sigma、Theta、Zeta、Omega和Delta型,细菌可溶性谷胱甘肽S-转移酶分为Theta和Beta型[4]。有些谷胱甘肽S-转移酶型别是各类生物共有的,有些是特异的,比如Alpha、Mu和Pi型谷胱甘肽S-转移酶为动物特有。与高等动植物相比,藻类(特别是海藻)谷胱甘肽S-转移酶研究相对薄弱。2008年Hervé等[5]首次对藻类谷胱甘肽S-转移酶进行了系统研究,结果显示红藻和褐藻的谷胱甘肽S-转移酶与高等植物所有谷胱甘肽S-转移酶的亲缘关系都较远,而与动物的Sigma型亲缘关系较近,并指出这些藻类谷胱甘肽S-转移酶构成了谷胱甘肽S-转移酶家族的一个新类型。
红藻在进化上分化早,较为原始[6],这暗示着红藻可能还具有不同于高等植物的新的谷胱甘肽S-转移酶类型。为了验证这一推测,笔者以红藻中较原始的条斑紫菜(Pyropiayezoensis)为材料,综合运用生物信息学和RT-PCR技术手段,克隆和分析了条斑紫菜谷胱甘肽S-转移酶家族的一个新成员,并命名为PyGST2。与预期相符,PyGST2与高等植物所有谷胱甘肽S-转移酶的亲缘关系均较远,而与动物的Mu型谷胱甘肽S-转移酶亲缘关系较近。暗示着条斑紫菜可能具有不同于高等植物的解毒机制,该Mu型谷胱甘肽S-转移酶的克隆为后续研究条斑紫菜抗逆机制奠定了基础。
1 材料与方法
1.1 试验材料
从连云港高公岛紫菜养殖筏上采集条斑紫菜叶状体。在室内充气培养,控制培养温度为10 ℃、光照为100 μmol/m2·s、光周期为12L∶12D。暂养5 d后进行铅胁迫处理,Pb2+质量浓度为10 mg/L,处理2、6 h和12 h后取样,以不胁迫的为对照。
1.2 总RNA抽提与反转录
用TRIzol试剂(Invitrogen)抽提条斑紫菜叶状体总RNA,接着用DNase Ⅰ(MBI Fermentas)处理总RNA,以降解残留在总RNA中的gDNA。纯化后的总RNA用普通琼脂糖凝胶电泳和核酸蛋白测定仪(Bio-Rad)检测完整性和纯度。以RandomPrimer为反转录引物,用RevertAidTMH Minus First Strand cDNA Synthesis Kit(MBI Fermentas)对总RNA进行反转录,得到第一链cDNA。
1.3 基因克隆与序列分析
[7]的方法对PyGST2进行电子克隆,从GenBank的EST数据库中筛选出了27条条斑紫菜EST序列,这些EST拼接后得到了1条长1045 bp的contig序列。根据电子克隆结果设计引物,上游引物:5′TGGTTCGACACGCTTTCC 3′,下游引物:5′CCCTCGCCTGCTCACAT 3′,预计扩增产物长838 bp,包含PyGST2的完整编码区。PCR反应总体系为20 μL,包含2 μL 10×PCR buffer、2 mmol/L MgCl2、200 μmol/L dNTP、500 nmol/L上下游引物、1 U Dream Taq(MBI Fermentas)和1 μL第一链cDNA。PCR反应条件为:先95 ℃预变性5 min,然后循环扩增,每个循环包括95 ℃变性30 s、53 ℃退火30 s、72 ℃延伸50 s,共30个循环。反应结束后电泳检测并送上海生工测序,测得的序列已提交至GenBank,索引号为HM182105。
采用DNAStar软件分析PyGST2的基本理化特性,使用Blast[8]和CDD[9]对PyGST2保守结构域进行了分析。参考Hervé等[5]的方法,选取了人(Homosapiens)、小鼠(Musmusculus)、斑马鱼(Daniorerio)、果蝇(Drosophilamelanogaster)、拟南芥(Arabidopsisthaliana)、水稻(Oryzasativa)、大肠杆菌(Escherichiacoli)以及多种藻类的各型谷胱甘肽S-转移酶共计93条谷胱甘肽S-转移酶与PyGST2一起进行了多对序列比对,多序列比对采用用MAFFT[10]进行。鉴于谷胱甘肽S-转移酶家族庞大、成员间相似性低,所以采用PhyML[11]构建进化树。
1.4 表达分析
采用荧光定量PCR技术检测PyGST2基因在重金属胁迫下的表达。定量PCR反应总体系为20 μL,包含10 μL 2×SYBR®Premix ExTaqTMⅡ(Tli RNaseH Plus)(TaKaRa)、300 nmol/L正向引物(5′ GGGTCATCCTCCGCTTCATCG 3′)、300 nmol/L反向引物(5′ CGCCGCCTCCATATCCTCTG 3′)和1 μL第一链cDNA。在IQ5 PCR仪(Bio-Rad)上进行扩增,先95 ℃预变性30 s,然后循环扩增,每个循环包括95 ℃变性10 s,63 ℃延伸30 s,循环结束后,制作熔解曲线。内参基因及其定量检测方法参考文献[12]的方法进行设置。反应设阴性对照和无模板对照,每个反应设3个复孔。应用LinRegPCR[13]计算扩增效率,用Pfaffl[14]建立的数学模型对定量PCR数据进行处理。
2 结 果
2.1 PyGST2克隆及基本理化性质
PyGST2的cDNA序列扩增产物电泳后呈现一条长约800 bp的特异性条带,与预期大小接近。在此cDNA序列上起始密码子(AUG)旁侧序列符合典型的kozak规则,cDNA序列上包含一个长654 bp的完整编码区,编码蛋白长217 AA。PyGST2蛋白分子量为24.6 ku,其碱性、酸性、疏水性和极性氨基酸含量分别为25、37、80和36个,等电点为4.86。PyGST2第1~78位氨基酸序列构成谷胱甘肽S-转移酶的N端结构域,其属于类硫氧还蛋白超家族,含有谷胱甘肽特异结合位点;第89~198位氨基酸序列构成C端结构域,含有结合疏水底物的位点(图1)。
2.2 PyGST2型别
首先用Blast程序初步分析了PyGST2的型别,即用PyGST2的氨基酸序列对GenBank中的蛋白质数据库进行Blastp搜索,结果显示数据库中与PyGST2比对上的序列全部都是谷胱甘肽S-转移酶,而且在谷胱甘肽S-转移酶的各种型别中,Mu型谷胱甘肽S-转移酶与PyGST2比对得到的E值相对较小,序列一致性相对较大。
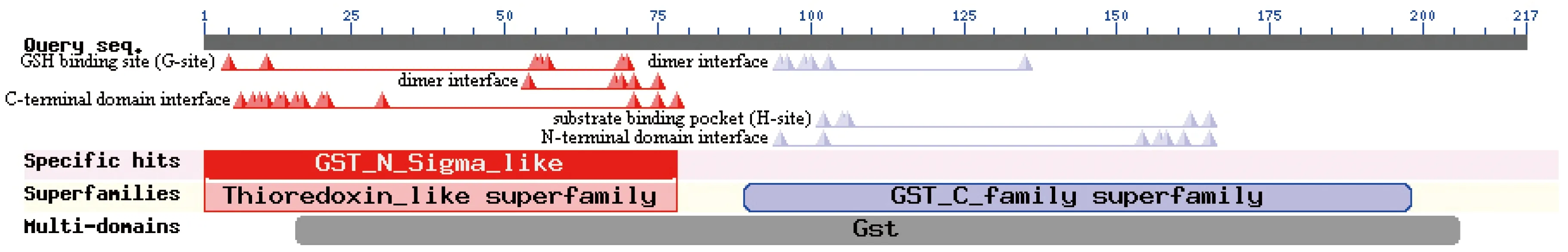
图1 PyGST2的保守结构域
在参与比对的各型谷胱甘肽S-转移酶中,PyGST2与条斑紫菜另一条谷胱甘肽S-转移酶序列(GQ415054)的序列一致性并不高,仅为21.3%,与植物的各型谷胱甘肽S-转移酶的序列一致性也不高。相反,与动物Mu型谷胱甘肽S-转移酶的序列一致性最高,为23.2%~29.4%;与动物Alpha、Sigma和Pi型谷胱甘肽S-转移酶的序列一致性次之,为18.8%~26.2%;与藻类的类Sigma型谷胱甘肽S-转移酶的序列一致性为21.3%~27.1%;与其他型别的谷胱甘肽S-转移酶序列一致性为11.5%~20%。
在多序列比对的基础上构建了系统进化树(图2),在进化树上所有谷胱甘肽S-转移酶首先按型别聚类,然后才是按物种类别聚类,即不同物种来源的同型别谷胱甘肽S-转移酶都单独聚类在一起。第一个被克隆的谷胱甘肽S-转移酶(GQ415054)与其他藻类谷胱甘肽S-转移酶以及Sigma型谷胱甘肽S-转移酶聚为一支,共同构成了Sigma型谷胱甘肽S-转移酶。本克隆的PyGST2与红藻(Cyanidioschyzonmerolae)的谷胱甘肽S-转移酶(XP_005539154)以及Mu型谷胱甘肽S-转移酶聚为一支。这些数据清楚地表明PyGST2属于Mu型谷胱甘肽S-转移酶家族成员之一。
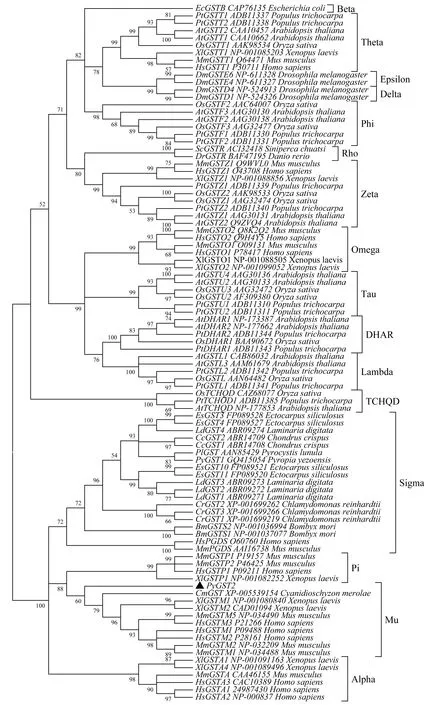
图2 不同型别谷胱甘肽S-转移酶的进化树用PhyML构建了谷胱甘肽S-转移酶家族成员的进化树,节点前的数值表示分支的统计支持率,GenBank索引号和物种名一并显示在图上.
2.3 PyGST2的定量表达
定量PCR仪记录到的扩增曲线呈规则的“S”形,溶解曲线呈现单一特异峰,PyGST2基因和内参基因的扩增效率分别是1.894和1.895,无模板对照的扩增曲线呈水平直线,这些表明定量检测体系性能良好。条斑紫菜叶状体在Pb2+胁迫2、6 h和12 h后,PyGST2基因的表达量上升,分别是对照的1.75、1.25和1.51倍(图3),表明PyGST2基因表达受到重金属胁迫的诱导。

图3 条斑紫菜PyGST2基因在重金属胁迫下的相对表达量
3 讨 论
谷胱甘肽S-转移酶是一个庞大的蛋白超家族,其成员间的序列组成变异较大,例如水稻的Phi型谷胱甘肽S-转移酶成员间的序列一致性23%~82%,通常同一型别谷胱甘肽S-转移酶成员间的平均序列一致性略高于40%,不同型别谷胱甘肽S-转移酶成员间的序列一致性在动物中通常小于25%,在植物中小于20%[4]。虽然不同谷胱甘肽S-转移酶的序列差异很大,但其彼此间仍具有一些共同的特征。首先,谷胱甘肽S-转移酶具有非常相似的二级和高级结构,谷胱甘肽S-转移酶一般以二聚体形式出现,每个亚基都包含2个结构域。结构域Ⅰ位于N端,比较保守,由β折叠和α螺旋构成类硫氧还蛋白折叠(βαβαββα),含有谷胱甘肽特异结合位点以及保守的丝氨酸或酪氨酸残基;结构域Ⅱ位于C端,变异较大,由4~7个α螺旋构成,含有结合疏水底物的位点[1,3-4]。其次,相似的二级和高级结构暗示着这些序列差异较大的谷胱甘肽S-转移酶都执行着相似的功能,即在功能上谷胱甘肽S-转移酶主要参与解毒过程。谷胱甘肽S-转移酶能催化有害物质的亲电子基团与还原型谷胱甘肽的巯基结合,形成更易溶解且无毒性的衍生物,便于其排出体外或分解[1,4]。多序列比对结果清晰地显示PyGST2与其他谷胱甘肽S-转移酶具有较低的序列一致性,因此为了充分确认PyGST2就是谷胱甘肽S-转移酶家族的成员之一,本文也从二级结构和生理功能这两个层面上开展了研究。在二级结构方面,PyGST2具有谷胱甘肽S-转移酶的2个结构域和保守的氨基酸残基,在生理功能方面,PyGST2基因受到重金属胁迫的诱导表达,这些数据清楚地表明PyGST2是谷胱甘肽S-转移酶家族的成员之一。
1961年Booth等[15]首次在动物中发现了谷胱甘肽S-转移酶,随后1970年Frear等[16]在植物中也发现了谷胱甘肽S-转移酶。之后的40年里从动植物和微生物中分离鉴定了大量的谷胱甘肽S-转移酶,于是谷胱甘肽S-转移酶的分类也就随之展开。随着新的谷胱甘肽S-转移酶的不断发现,对谷胱甘肽S-转移酶的分类也在不断发展和变化着。以植物谷胱甘肽S-转移酶为例,最初植物谷胱甘肽S-转移酶均归类于异质性较高的Theta型,随着测序计划的开展更多植物谷胱甘肽S-转移酶被克隆,于是1997年植物谷胱甘肽S-转移酶被分成了3类(即Ⅰ、Ⅱ、Ⅲ型,即后来的Phi、Zeta和Tau型),次年新增了第4类(即Ⅳ型,Theta型),2002年又新增了Lambda型和DHAR[17]。虽然早期的研究认为只有动物才具有Sigma型谷胱甘肽S-转移酶,只有哺乳动物具有Alpha、Mu和Pi型谷胱甘肽S-转移酶[4],但是上述谷胱甘肽S-转移酶分类研究主要集中在动植物,缺乏对低等藻类的研究。2008年Hervé等[5]开展了红藻和褐藻谷胱甘肽S-转移酶的分类研究,并指出这些藻类谷胱甘肽S-转移酶构成了谷胱甘肽S-转移酶家族的一个新类型。随后周向红等[12]的研究也支持了这一观点,认为这些藻类谷胱甘肽S-转移酶属于Sigma型谷胱甘肽S-转移酶。本研究得到的进化树与Hervé等[5,15]的结果基本一致,不仅支持了藻类也含有Sigma型谷胱甘肽S-转移酶的观点,而且进一步发现了条斑紫菜等红藻还含有Mu型谷胱甘肽S-转移酶。条斑紫菜Mu型谷胱甘肽S-转移酶的克隆不仅完善了谷胱甘肽S-转移酶的分类研究,还暗示着条斑紫菜可能具有与高等动物类似的解毒机制,为后续的条斑紫菜抗逆研究奠定了基础。
参考文献:
[1]雷安平,陈欢,黎双飞,等. 谷胱甘肽S-转移酶的功能、应用及克隆表达[J].环境科学与技术,2009,32(12):85-91.
[2]Hayes J D,Flanagan J U,Jowsey I R.Glutathione transferases [J].Annual Review of Pharmacology and Toxicology,2005,45(1):51-88.
[3]胡廷章,周大祥,罗凯.植物谷胱甘肽转移酶的结构与功能及其基因表达[J].植物生理学通讯,2007,43(1):195-200.
[4]Frova C.Glutathione transferases in the genomics era:new insights and perspectives [J].Biomolecular Engineering,2006,23(4):149-169.
[5]Hervé C,de Franco P-O,Groisillier A,et al.New members of the glutathione transferase family discovered in red and brown algae [J].Biochemical Journal,2008,412(3):535-544.
[6]Stiller J W,Hall B D.Sequences of the largest subunit of RNA polymerase Ⅱ from two red algae and their implications for rhodophyte evolution [J].Journal of Phycology,1998,34(5):857-864.
[7]易乐飞,王萍,周向红,等. 条斑紫菜半胱氨酸合成酶基因的电子克隆与分析[J].中国水产科学,2009,16(4):518-524.
[8]Altschul S F,Madden T L,Schaffer A A,et al.Gapped BLAST and PSI-BLAST:a new generation of protein database search programs [J].Nucleic Acids Research,1997,25(17):3389-3402.
[9]Marchler-Bauer A,Lu S,Anderson J B,et al.CDD:a Conserved Domain Database for the functional annotation of proteins [J].Nucleic Acids Research,2011,39(D):225-229.
[10]Katoh K,Standley D M.MAFFT Multiple Sequence Alignment Software Version 7:improvements in performance and usability [J].Molecular Biology and Evolution,2013,30(4):772-780.
[11]Guindon S,Gascuel O.A simple,fast,and accurate algorithm to estimate large phylogenies by maximum likelihood [J].Systematic Biology,2003,52(5):696-704.
[12]周向红, 李信书, 王萍, 等.高温胁迫下条斑紫菜叶状体HSP70基因的表达谱分析[J].水产科学,2011,30(4):233-237.
[13]Ruijter J M,Ramakers C,Hoogaars W M H,et al.Amplification efficiency:linking baseline and bias in the analysis of quantitative PCR data [J].Nucleic Acids Research,2009,37(6):45.
[14]Pfaffl M W.A new mathematical model for relative quantification in real-time RT-PCR [J].Nucleic Acids Research,2001,29(9):2002-2007.
[15]Booth J,Boyland E,Sims P.An enzyme from rat liver catalysing conjugations with glutathione[J].Biochemical Journal,1961,79(3):516-524.
[16]Frear D S,Swanson H R.Biosynthesis of S-(4-ethylamino-6-isopropylamino-2-s-triazino) glutathione:partial purification and properties of a glutathione S-transferase from corn [J].Phytochemistry,1970,9(10):2123-2132.
[17]Frova C.The plant glutathione transferase gene family:genomic structure,functions,expression and evolution [J].Physiologia Plantarum,2003,119(4):469-479.
[18]de Franco P-O,Rousvoal S,Tonon T,et al. Whole genome survey of the glutathione transferase family in the brown algal modelEctocarpussiliculosus[J].Marine Genomics,2009,1(3/4):135-148.
CloningandAnalysisofaMuClassGSTinLaverPorphyrayezoensis
YI Lefei,HAO Wei,LI Xinshu,YAN Binlun
( School of Marine Science & Technology,HuaiHai Institute of Technology,Lianyungang 222005,China )
Glutathione S-transferases (GSTs) are cellular multifunctional detoxification enzymes,and ubiquitously found in all types of organisms with a highly diverse family of proteins.In the present study,a novel member (PyGST2) of GST family in laverPorphyrayezoensisUeda was cloned and analyzed using RT-PCR and bioinformatical tool.It was found that PyGST2 gene contained a continuous complete open reading frame encoding a polypeptide of 217 amino acids.The Pb2+stress induced the expression of PyGST2 gene which contained conserved domains and amino acid residues of GST family.PyGST2 shared the highest identities with Mu class GSTs,and higher identities with Alpha,Sigma and Pi class GSTs among all kinds of GSTs.Phylogenetic analysis showed that PyGST2 was distinct from GST class of plant,but was most closely related to the Mu class GSTs.The findings of PyGST2 laid the foundation for further understanding of the molecular mechanisms of stress tolerance in the laver.
Porphyrayezoensis;GST;cloning;expression
Q785
A
1003-1111(2016)01-0067-05
10.16378/j.cnki.1003-1111.2016.01.012
2015-04-22;
2015-08-31.
江苏省海洋生物技术重点实验室开放课题项目(2012HS013).
易乐飞(1975-),男,硕士,副教授;研究方向:水生生物分子遗传.E-mail:yilf@hhit.edu.cn.通讯作者:阎斌伦(1962-),男,教授;研究方向:海洋生物应用技术.E-mail:yanbinlun1962@163.com.
