生存资料的二次研究系列之一:应用R软件实现生存曲线中数据提取与生存曲线的合并
2016-08-19孟详喻靳英辉程小柯张永刚曹越曾宪涛
孟详喻,靳英辉,程小柯,张永刚,曹越,曾宪涛
• 循证理论与实践 •
生存资料的二次研究系列之一:应用R软件实现生存曲线中数据提取与生存曲线的合并
孟详喻1,靳英辉2,程小柯3,张永刚4,曹越1,曾宪涛1
涉及生存资料的Meta分析最常用的方法是对两组比较的风险比(hazard ratio,HR)进行合并,其数据基础是原始研究的HR及其95%可信区间(95%CI)。然而,原始研究中HR及其95%CI未报道或不完整报道的情况并不少见,这限制了基于HR的Meta分析的开展。绝大多数涉及生存结局的原始研究均提供了生存曲线,通过对生存曲线进行合并能对人群的生存结局进行全面评估。本文介绍利用R软件实现生存曲线数据提取并对其进行合并以生成汇总生存曲线的方法。
生存资料;生存曲线;R软件
涉及生存资料的Meta分析最常用的方法是以倒方差法对(hazard ratio,HR)进行合并[1,2]。该类型的Meta分析主要着眼于两组比较的评价,能区分优劣但很难反映全局问题。此外,原始研究中
HR及其95%可信区间(95%CI)未报道或不完整报道的情况并不少见,这限制了基于HR的Meta分析的开展[3,4]。另一方面,对特定人群的生存曲线进行合并能在扩大样本量的基础上全面反映出研究人群的特定生存结局的总体状况。绝大多数涉及生存结局的原始研究均提供了生存曲线,从而为此类研究的开展提供了便利[5]。国外已有方法学文献报道了通过乘积限估计法计算合并生存率的方法,并开发了相关的R软件包以支持其应用[6]。本文拟通过实例介绍利用R软件digitize程序包对生存曲线进行标准化提取的方法,并介绍利用MetaSurv程序包对自曲线提取出的生存数据进行合并以生成汇总后的生存曲线估计。所使用R软件的版本为3.1.3。
1 程序包简介
因程序包研发者及维护者未能及时回复CRAN(The Comprehensive R Archive Network)工作组的联系,digitize程序包现已自CRAN存储空间移除。我们自GitHub获取该程序包最近一次更新的源代码并使用RStudio重新打包后经检测可在最新版本的R软件成功安装并加载运行。MetaSurv程序包目前也无法通过R官网途径获取。读者可自武汉大学中南医院循证与转化医学中心的官网(http://www.whuzncebtm.com/)中的“科学研究→资料下载”栏中下载这两个程序包的zip格式的安装文件,安装方法详见曾宪涛主编的著作[7]。
由于digitize程序包的运行需调用jpeg程序包,因此在安装digitize程序包之前,首先安装及加载jpeg程序包,命令如下:
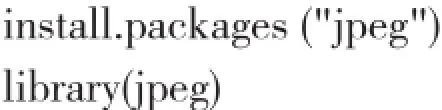
该程序包安装完成后即可使用下述命令加载digitize程序包和MetaSurv程序包:

2 利用digitize程序包自K-M曲线提取数据
2.1提取前准备 提取前的主要准备工作包括自原始文献电子文档截取生存曲线,并利用Photoshop软件添加等分辅助线,建议每条辅助线在时间轴的位置对应至单月,具体方法在此不做赘述。本处所选用的原始文献为2013年发表于《Lancet Oncology》杂志上的一项比较泊马度胺联合低剂量地塞米松与高剂量地塞米松单药治疗复发及难治性多发性骨髓瘤患者的Ⅲ期随机对照试验,所选取的生存曲线为比较两种药物无进展生存期(PFS)的K-M曲线[8]。
3 生存曲线的合并

图3 对生存曲线进行取点

表1 提取出的生存数据列表
我们通过一实例操作介绍使用MetaSurv程序包合并自不同单个研究的提取出的生存数据并生成汇总估计的生存曲线。具体方法学模型原理有兴趣的读者请参阅相关方法学文献[6]。
本实例所纳入的研究为5项以复发与难治性多发性骨髓瘤患者为对象的对照组为地塞米松或地塞米松联合安慰剂的随机对照试验[8-12]。按照前述方法提取各对照组各月的总生存(OS)K-M曲线生存数据。整理好的原始数据表见表2。数据表表头“Study”为研究编号,研究1至5分别对应2005 Richardson、2007 Dimopoulos、2007 Weber、2011 Kropff与2013 San-Miguel(发表年份+第一作者英文名);“Time”为时间点(月份);“Survival”为各时间点所对应的生存率;“NbRisk”为各时间段事件风险人数,该数据自文献中提取或根据Thiery等的方法学假定截尾率在随访期内恒定而计算得出[13]。

表2 示例5项研究的数据
接下来使用下述代码进行合并分析:

命令运行后,结果如表3所示。
结果中包含了固定效应模型与随机效应模型下汇总后的平均生存时间与中位生存时间及其95%可信区间,还显示反应异质性的统计量Cochran Q、H2与I2值。
从表3还可以看出,Cochran Q=186.248778,H2= 1.565116,I2= 36.106963%。根据Higgins等的方法学还可对异质性的统计显著性进行检测,命令代码如下:
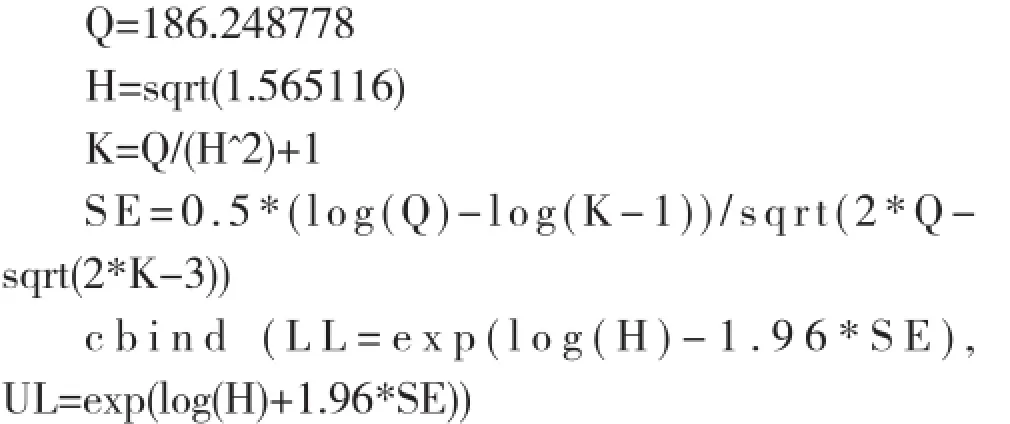
命令运行后,结果如下:

由结果可知,H值可信区间下限大于1 (LL=1.222318),检测结果有统计学显著性,故后续合并后生存曲线的绘制采用随机效应模型的数据。
输入以下命令代码进行后续操作:


表3 示例的计算结果

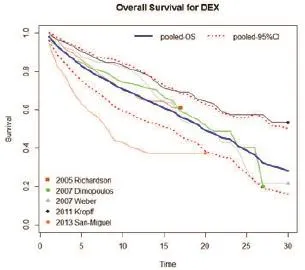
图4 根据各研究所提取数据生成的相应曲线及合并后的OS曲线与95%可信区间
命令运行后,最终生成的图形如图4所示。图正中蓝色粗实线为合并后的OS曲线,该曲线两侧的红色点线为其95%可信区间上下限。其余线条为根据单个研究所提取的生存数据得出的近似曲线,各曲线末端标注了其随访终点。
4 小结
HR值等关键数据的报道缺失是生存资料Meta分析过程中常常遇见的问题,自K-M曲线提取生存数据后根据相关方法学文献进行估算是解决这类问题较为可靠的方法之一[14]。本文详细介绍了利用R软件digitize程序包进行生存曲线数据提取的操作方法。该方法通过添加等分辅助线以单位时间间隔提取数据保证了数据质量及后续计算结果的稳定可靠。但该方法需逐一手动取点,相对费时。
在生存资料汇总分析过程中,单纯通过HR合并虽然能区分优劣但很难反映全局问题。对某些治疗组的生存曲线进行合并能提供很多其他有意义的信息。本文介绍了使用R软件MetaSurv程序包实现生存曲线数据合并的方法,并通过一实例进行了具体介绍。我们希望这一方法能为今后更多的研究所采用,既是对HR分析很好的补充,更有助于对生存资料进行更为全面的反映。
[1] Spruance SL,Reid JE,Grace M,et al. Hazard ratio in clinical trials[J]. Antimicrob Agents Chemother,2004,48(8): 2787-92.
[2] 曾宪涛. 系统评价/Meta分析理论与实践[M]. 北京: 军事医学科学出版社,2012:117-9.
[3] Michels S,Piedois P,Burdett S,et al. Meta-analysis when only the median survival times are known: a comparison with individual patient data results[J]. Int J Technol Assess Health Care,2005,21(1): 119-25.
[4] Parmar MKB,Torri V,Stewart L. Extracting summary statistics to perform meta-analyses of the published literature for survival endpoints[J]. Stat Med,1998,17(24): 2815-34.
[5] Arends L,Hunink M,Stijnen T. Meta-analysis of summary survival curve[J]. Stat Med,2008,27(22):4381-96.
[6] Combescure C,Foucher Y,and Jackson D. Meta-analysis of singlearm survival studies: a distribution-free approach for estimating summary survival curves with random effects[J]. Stat Med,2014,33(15):2521-37.
[7] 曾宪涛,张超. R与Meta分析[M]. 北京: 军事医学科学出版社,2015:26.
[8] San Miguel J,Weisel K,Moreau P,et al. Pomalidomide plus lowdose dexamethasone versus high-dose dexamethasone alone for patients with relapsed and refractory multiple myeloma (MM-003): a randomised, open-label, phase 3 trial[J]. Lancet Oncol, 2013, 14(11): 1055-66.
[9] Kropff M,Baylon HG,Hillengass J,et al. Thalidomide versus dexamethasone for the treatment of relapsed and/or refractory multiple myeloma: results from OPTIMUM,a randomized trial[J]. Haematologica,2012,97(5): 784-91.
[10] Weber DM,Chen C,Niesvizky R,et al. Lenalidomide plus dexamethasone for relapsed multiple myeloma in North America[J]. N Engl J Med,2007,357(21): 2133-42.
[11] Dimopoulos M,Spencer A,Attal M,et al. Lenalidomide plus dexamethasone for relapsed or refractory multiple myeloma[J]. N Engl J Med,2007,357(21):2123-32.
[12] Richardson PG,Sonneveld P,Schuster MW,et al. Bortezomib or highdose dexamethasone for relapsed multiple myeloma[J]. N Engl J Med,2005,352(24):2487-98.
[13] Tierney JF,Stewart LA,Ghersi D,et al. Practical methods for incorporating summary time-to-event data into meta-analysis[J]. Trials, 2007, 8: 16.
[14] Earle C,Wells G. An Assessment of Methods to Combine Published Survival Curves[J]. Medical Decision Making,2000,20:104-11.
本文编辑:田国祥
Extracting data from survival curve and pooled survival curves by using R software
MENG Xiang-yu*,JIN Ying-hui, CHENG Xiao-ke, ZHANG Yong-gang, CAO Yue, ZENG Xian-tao. *Center for Evidence-Based and Translational Medicine, Zhongnan Hospital, Wuhan University, Wuhan 430071, China.
ZENG Xian-tao, E-mail: zengxiantao1128@163.com
The most common method in Meta-analysis involving survival data is pooled hazard ratios (HR), and the data base is HR in original studies and 95% confidence interval (95%CI). However, non-reports or incomplete reports of HR in original studies and 95%CI are not uncommon, which limits the conducting of Meta-analysis based on HR. The most of original studies related to survival outcomes can provide survival curves, and the survival outcomes can be completely reviewed through pooling survival curve in the study population. This paper introduces the approach of extracting and pooling survival curve data for generating a pooled survival curve by using R software.
survival data; survival curve; R software
至此生存数据提取过程结束,可将所提取的数据用于后续分析处理。
R4
A
1674-4055(2016)01-0002-05
1430071 武汉,武汉大学中南医院循证与转化医学中心;2300193 天津,天津中医药大学护理学院循证护理中心;3475000开封,河南大学淮河医院循证医学中心;4610041 成都,四川大学华西医院期刊社
10.3969/j.issn.1674-4055.2016.01.01
曾宪涛,E-mail:zengxiantao1128@163.com.
准备好的图像文件如图1所示。将其命名为“PFS_2013Miguel.jpg”,并存储于桌面“Rwork”文件夹中(注:可通过代码“>setwd ( )”设定工作文件夹,括号中为其路径)。
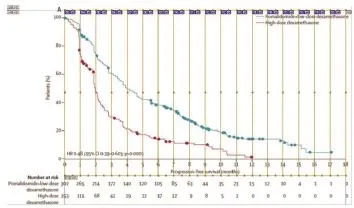
图1提取前预处理好的图像文件
2.2数据提取 运行R软件,输入以下命令代码进行数据处理:
library(digitize)
cal<-ReadAndCal('PFS_2013Miguel.jpg')
第二行命令为读取目标图像文件“PFS_2013Miguel.jpg”并在图上选定横轴X与纵轴Y坐标标定点,下图中横轴两标记点与纵轴两标记点分别对应Xmin、Xmax、Ymin及Ymax,标定点的信息存入对象“cal”中。程序运行界面如图2。

图2读取目标图像文件并在横轴与纵轴选取标定点

该命令是对目标曲线(本实例中对红色的地塞米松组曲线提取数据)取点提取各月所对应的生存率数据,“col='blue'”意指以蓝色空心圆点标记各数据点,结果数据记入对象curv中,如图3所示。取点完毕后单击右键,出现弹窗选择“stop”结束取点。

该行命令是对数据进行标定,curv代表上一步取点所得的数据集,cal即标定点集合,最后四个数字分别代表标定点Xmin、Xmax、Ymin、Ymax的坐标值,将数据标定后的结果记为“data”。

该行命令为将数据集data中的x栏数值取整,记为“Time”即时间(月)。
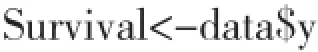
该行命令为将d a t a中的y栏数值记为“Survival”即生存率。

该行命令为将“Time”与“Survival”组合为新的数据集,命名为“PFS_2013Miguel”。
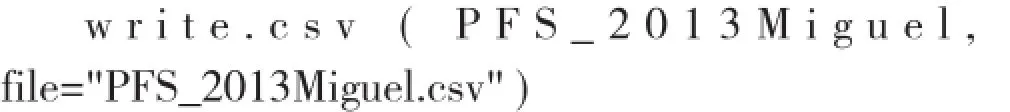
该行命令为将“PFS_2013Miguel”导出为csv格式的文件,命名为“PFS_2013Miguel.csv”,默认存储于Rwork文件夹。该文件可用Excel打开,如表1所示。
