厌氧和好氧处理过程中四环素抗药基因的丰度
2016-06-07刘苗苗北京交通大学土木建筑工程学院市政环境工程系水中典型污染物控制与水质保障北京市重点实验室北京00044中国科学院生态环境研究中心环境水质学国家重点实验室北京00085
任 佳,姚 宏,刘苗苗*,张 昱,杨 敏(.北京交通大学土木建筑工程学院市政环境工程系,水中典型污染物控制与水质保障北京市重点实验室,北京 00044;.中国科学院生态环境研究中心,环境水质学国家重点实验室,北京 00085)
厌氧和好氧处理过程中四环素抗药基因的丰度
任 佳1,姚 宏1,刘苗苗1*,张 昱2,杨 敏2(1.北京交通大学土木建筑工程学院市政环境工程系,水中典型污染物控制与水质保障北京市重点实验室,北京 100044;2.中国科学院生态环境研究中心,环境水质学国家重点实验室,北京 100085)
摘要:为了了解抗生素生产废水不同处理过程(厌氧生物处理和好氧生物处理)中抗药基因的行为,本文以两种四环素(土霉素、金霉素)生产废水处理系统为调查对象,采用PCR和实时定量PCR(qPCR)方法考察厌氧和好氧处理过程中常见的6种四环素抗药基因(tet(A), tet(C), tet(G), tet(Q), tet(W), tet(X))及2种转移因子(I型整合子intI1, 异常插入序列ISCR3)的丰度特征.结果表明,tet(C)在所有样品中均未检出,其它基因在所有样品中检出.四环素生产废水处理系统中,厌氧污泥中tet(A)、tet(G)、tet(X)的相对丰度(与16S rRNA基因的比值)范围为(1.25±0.16)×10-4~(4.52±0.002)×10-2,显著低于好氧污泥[(9.88±0.67)×10-5~(2.70±0.29)×10-1],而tet(Q)、tet(W)在厌氧污泥中的相对丰度为(1.66±0.03)×10-2~(7.48±1.22)×10-2,比好氧污泥中[(1.94±0.12)×10-3~(2.85±0.16)×10-2]高1个数量级;转移因子intI1和ISCR3在厌氧污泥中相对丰度范围为(1.48±0.01)×10-3~(2.61±0.31)×10-2,显著低于好氧污泥[(1.18±0.15)×10-1~(8.99±0.75)×10-1],表明厌氧处理过程中由这两种转移因子介导的水平转移潜力较小.研究表明,好氧处理促进了tet(A)、tet(G)、tet(X)的传播,但对tet(Q)和tet(W)有控制效果,而厌氧处理过程与之相反.抗药基因的分布与水平转移因子、抗药机制、群落结构有关.
关键词:四环素生产废水;四环素抗药基因;转移因子;厌氧处理;好氧处理
* 责任作者, 讲师, lmmhit@163.com
随着抗生素的大量生产和广泛使用,其在环境中的残留被频繁检出.抗生素残留会诱导抗药菌和抗药基因的产生和增殖,威胁公共健康[1].人们已在污水处理厂[2]、养殖水域[3]、河流[4-5]、沉积物[5]和土壤[6-7]等环境中检测出抗药基因和抗药菌.近年来,在中国、印度等地开展的研究发现,抗生素生产废水中有很高的抗生素残留[8-9],远高于其它环境,如城市污水厂、河流等.
抗生素生产废水是一种高浓度有机废水,目前主要采用生物法进行处理.由于该系统中抗生素浓度高,选择压力大,同时微生物数量多,已成为抗药基因的重要污染源[8,10-11].从某土霉素生产废水处理系统筛选的细菌中,94.2%携带四环素类抗药基因(tet基因)[9].定量研究发现,该系统中tet抗药基因的相对丰度比城市污水厂高1~4个数量级[12].从某青霉素生产废水处理系统筛选的细菌中,17.3%(31/179)携带有β-内酰胺类抗药基因[13].某螺旋霉素生产废水处理系统中检测出8种大环内酯-林可霉素-链阳菌素(MLS)抗药基因,其丰度比城市污水厂高1.4~2.3个数量级[14].
迄今为止,关于不同处理工艺中抗药基因的差别已有一些报道,但尚未发现一致规律.某螺旋霉素生产废水厌氧污泥中MLS抗药基因的相对丰度低于好氧污泥[14].Tao等[15]发现养猪废水处理过程中,tet(A)和tet(W)在厌氧污泥中的相对丰度高于好氧污泥.此外,在某制革废水处理系统中发现,磺胺类抗药基因(sul1和sul2)、四环素抗药基因(tet(G),tet(X))在厌氧污泥中丰度低于好氧污泥;四环素抗药基因tet(M),tet(C)在厌氧污泥中丰度高于好氧污泥[16].Christgen等[17]比较了城市污水厌氧、好氧、厌氧-好氧处理过程中抗药基因在水相中的去除情况,发现厌氧-好氧联合处理具有最高的去除效果,但经过好氧处理后,抗药基因的比例升高.在城市污水厂剩余污泥消化过程中发现,厌氧消化对抗药基因的控制效果优于好氧消化[18].不同处理工艺中抗药基因的行为和机制尚不明确,需要进一步研究.
本文以内蒙古某金霉素和某土霉素制药厂生产废水处理系统(厌氧-好氧组合工艺)为对象,使用PCR、实时定量PCR等方法调查了进水、出水、厌氧污泥、好氧污泥中tet基因的种类及丰度,比较了厌氧和好氧处理过程中tet基因的差异,分析了进水和出水中tet基因的变化,预期从抗药基因控制角度为选择制药废水处理工艺提供参考.
1 材料与方法
1.1 样品采集

表1 样品信息及采样编号Table 1 Sample information and number
于2013年7~12月对内蒙古某金霉素和某土霉素制药废水处理系统进行了1~2次采样.土霉素制药厂(K厂)的土霉素产量为5000t/y,该废水处理系统采用UASB + 生物接触氧化池工艺;金霉素制药厂(J厂)的金霉素产量为41500t/y,该废水处理系统采用UASB + AO + CASS工艺.采集的样品包括:处理系统的进水、出水;厌氧和好氧生物处理单元的活性污泥.此外以大豆蛋白发酵废水处理系统为对照体系,该系统采用UASB +SBR处理工艺.每次采样持续3d,每个采样点每天取3个平行样并均匀混合.对于污水和污泥样品,分别取100mL和1mL样品,4℃下10000r/min离心10min,将沉积物保存到-20℃,用于DNA提取.具体样品编号及采样信息见表1.
1.2 水样分析
按照《水和废水监测分析方法》[19]国家标准方法分析常规化学指标,测定了水样的CODCr,氨氮,TP和TOC,测定均有3个平行,最后计算平均值.通过超高效液相色谱串联质谱(UPLC-MS/ MS,Waters,USA)对系统中的土霉素、金霉素进行定量分析[9].
1.3 DNA提取及PCR
对于离心收集到的污水和污泥样品,使用Fast DNA Spin Kit for soil (MpBio,美国)提取DNA.选取了6种tet基因(外排泵类tet(A)、tet(C)、tet(G)、核糖体保护类tet(Q)、tet(W)、酶修饰tet(X))[20]和2种转移因子(intI1和ISCR3)进行了PCR扩增.扩增反应采用50µL体系,包括5µL 10×PCR buffer、4µL dNTPs、20pmol/L上下游引物各1µL、1.25U Taq酶0.25µL、1µL 20mg/L牛血清蛋白溶液、37µL无菌水以及1µL DNA模板.样品DNA浓度范围为79.3~120.8ng/µL,稀释10倍作为PCR模板,阴性对照以无菌水作为DNA模板.反应条件为95℃解链7min;95℃解链35s,退火35s,72℃延伸45s(循环35次);72℃延伸10min,不同tet基因的引物及退火温度见文献[3,12].PCR产物通过1%(质量/体积)的琼脂糖凝胶电泳进行检验.
1.4 qPCR
对于普通PCR检出的5种tet基因[tet(A)、tet(G)、tet(Q)、tet(W)、tet(X)]、2种转移因子及细菌的总16S rRNA基因进行拷贝数定量分析,实验中标准曲线的相关系数R2>0.99,扩增效率均在90%~110%的范围内.详细操作方法如下:使用GeneJET Gel Extraction Kit(天根,北京)对PCR产物进行切胶纯化.将纯化的PCR产物与载体质粒PMD 18-T Vector(Takara,大连)在16℃连接过夜,转化到E.coil TOP10感受态细胞(Takara,大连)中扩大培养后涂平板,每个样品随机挑选30个白色菌落,用载体质粒引物M13进行PCR检测,每种基因选择1~2株阳性菌液进行测序.将克隆测序得到的DNA序列与NCBI数据库比较,确定是目标基因片段后,用TIANprep Mini Plasmid Kit(天根,北京)提取质粒,作为定量PCR的标准模板.使用ABI 7300 SYBR-Green实时定量PCR仪(ABI,美国)对tet基因、转移因子和细菌16S rRNA基因进行定量.反应采用25µL体系,包括1×SYBR Green I, 1×Dye(Takara,大连),200nM上下游引物各1µL, 0.5mg/mL牛血清蛋白,2 µL DNA模板.对于16S rRNA和ISCR3,温度程序为:95℃,30s;95℃,10s,退火温度,35s,循环40次;并于退火温度下收集荧光信号;自动熔解曲线过程.对于其他基因,温度程序为:95℃,30s;95℃,10s;退火温度,10~15s;72℃, 10s;78~83℃,26s(收集荧光信号);循环40次;自动熔解曲线过程.定量PCR的退火温度均比普通PCR高3~5℃.
将克隆测序得到的标准质粒稀释10~108倍,作为模板构建标准曲线.样品作为定量PCR模板时,进行适当稀释(10~100倍),并以无菌水作为阴性对照.所有样品均3个平行,最终计算平均值.不同样品之间由于生物量及DNA提取效率不同,论文中将得到的抗药基因和转移因子的丰度对16S rRNA基因进行归一化,采用tet基因和转移因子的相对丰度(tet基因和转移因子的拷贝数/16S rRNA基因的拷贝数)进行分析讨论.
2 结果与讨论
2.1 废水水质
土霉素及金霉素制药厂废水处理系统常规物理化学指标见表2.
K厂进水中土霉素浓度为25.4mg/L,UASB出水中土霉素浓度为23.1mg/L,生物接触氧化池出水中土霉素浓度为1.1mg/L,该系统中土霉素浓度高于城市污水厂(0.06~1.10µg/L)[20];J厂进水中金霉素浓度为100mg/L,可能由于金霉素水解周期短[21],在各级出水中均未检出金霉素.总的来看,K厂和J厂废水处理系统中的抗生素浓度高于城市污水厂[22].

表2 两种四环素生产废水的化学指标Table 2 Chemical characteristics of the two tested tetracycline production wastewater
2.2 tet基因及转移因子PCR检测
采集的各样品提取DNA后,用PCR扩增检测6种tet基因及2种转移因子的出现,检测结果见表3.据表3可以看出,比较选择的6种tet基因以及2种转移因子,在抗生素生产废水处理系统中,除tet(C)没有检出外,其他均有检出,且tet(A)、tet(Q)、ISCR3和intI1在所有样品中均被检出,表明该系统中抗药基因普遍存在.而在大豆蛋白废水处理系统中,厌氧污泥中只检出核糖体保护类tet(Q)和tet(W),好氧污泥中则只有外排泵类tet(A)、tet(C)未被检出.
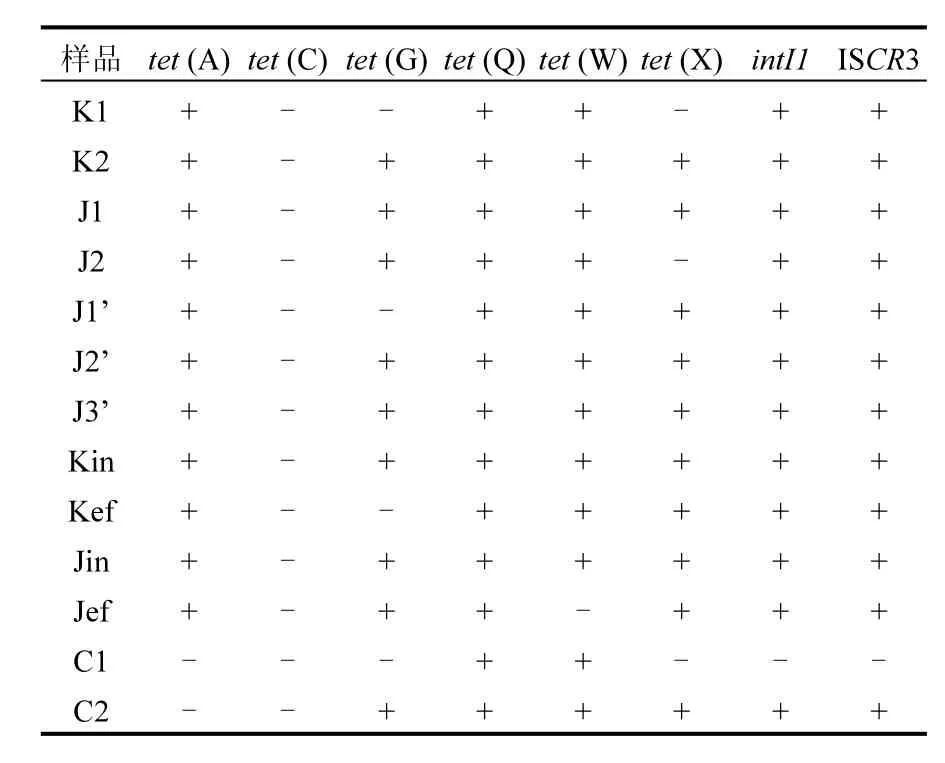
表3 PCR检测结果Table 3 Results of PCR detection
2.3 tet基因及转移因子的丰度
土霉素废水处理系统中,厌氧污泥(K1)中tet基因的丰度范围为(1.73±0.04)×10-4~(4.52±0.02)× 10-2;好氧污泥(K2)中tet基因的丰度范围为: (2.91±0.05)×10-3~(2.70±0.29)×10-1;金霉素废水处理系统中,厌氧污泥(J1, J1’)中tet基因的丰度范围为:(1.25±0.16)×10-4~(5.70±0.001)×10-2;好氧污泥(J2, J2’, J3’)中tet基因的丰度范围为: (9.88± 0.67)×10-5~(1.38±0.41)×10-1.但不同抗药基因在厌氧和好氧污泥中的分布不同,因此需要更全面的调查研究.大豆蛋白生产废水处理系统中,厌氧污泥(C1)中tet基因的丰度范围为:(6.84±0.87)×10-6~(1.82±0.13)×10-2;好氧污泥(C2)中tet基因的丰度范围(4.64±0.19)×10-5~(1.66±0.09)×10-2.对于两种转移因子,四环素生产废水处理系统活性污泥中,intI1的丰度范围为(3.99±0.14)× 10-3~(8.99±0.75)×10-1; ISCR3的丰度范围为(1.48±0.08)×10-3~(1.81± 0.13)×10-1,均高于大豆蛋白生产废水处理系统,丰度范围分别为(1.84± 0.27)×10-4~(1.46±0.18)×10-2和(1.96±0.11)×10-4~(1.30±0.11)×10-3.抗生素生产废水处理系统中抗药基因和转移因子丰度比大豆蛋白废水处理系统高1~3个数量级,表明高浓度的抗生素残留水平促进抗药基因的产生和增殖.
为了进一步阐明厌氧生物处理和好氧生物处理过程中抗药基因的差异,着重比较了厌氧污泥和好氧污泥中tet基因以及转移因子的丰度(图1).3种tet基因tet(A)、tet(G)、tet(X)在厌氧污泥中的丰度[(1.25±0.16)×10-4~(4.52±0.02)× 10-2]比好氧污泥中的丰度[(9.88±0.67)×10-5~(2.70±0.29)×10-1]低1~2个数量级,而另外两种tet基因tet(Q)、tet(W)则是好氧污泥中丰度[(5.76± 0.58)×10-3~(2.85±0.16)×10-2]比厌氧污泥[(1.66± 0.03)×10-2~(7.48±1.22)×10-2]低1个数量级.已有研究发现在城市污水厂活性污泥的厌氧消化过程中tet(G)、tet(X)的丰度降低,tet(A)、tet(W)的丰度升高[23],由于抗药基因的分布与细菌群落有一定的关系[23],而处理工艺的不同运行条件均可能造成微生物群落差异,从而产生不同的抗药基因分布.此外,江苏某养猪废水经过厌氧发酵后,外排泵类tet基因[tet(A)、tet(C)、tet(G)]丰度下降,核糖体保护类tet基因[tet(M)、tet(O)、tet(Q)、tet(W)]丰度升高[24].在本文中,好氧污泥中核糖体保护类tet基因的丰度较低,而厌氧污泥中外排泵类tet基因的丰度较低.

图1 四环素生产废水系统厌氧污泥和好氧污泥中抗药基因的相对丰度Fig.1 The relative abundance of tet genes (normalized to 16S rRNA genes) in the anaerobic and aerobic sludge of tetracycline production wastewater treatment systemsA图为K厂第1次采样;B图为J厂第1次采样; C图为J厂第2次采样
造成不同种tet基因分布差异的原因主要有以下方面.首先,tet基因的分布与其宿主分布可能存在直接联系.已有研究表明,革兰氏阳性(G+)细菌主要携带核糖体保护类tet基因以及少数几种外排泵类tet基因[tet(K)、tet(L)、tet(Z)和tet(T)],而革兰氏阴性(G-)细菌则主要携带外排泵类tet基因[tet(A)、tet(G)][25].好氧活性污泥中G-细菌占据主导,因此导致tet(A)、tet(G)经过好氧处理后丰度甚至升高.而在厌氧活性污泥中,G+细菌占据主导,因此导致tet(Q)、tet(W)丰度较高,在好氧处理后丰度降低.

图2 四环素抗药基因和intI1及ISCR3的相关性分析;intI1与ISCR3的相关性分析Fig.2 Correlation analysis between tet genes and intI1, tet genes and ISCR3, intI1and ISCR3
其次,转移因子(如整合子、转座子、质粒等)介导的抗药基因水平转移也会影响抗药基因的分布[26].本研究发现好氧污泥中intI1和ISCR3的丰度比厌氧污泥中的丰度高1~2个数量级,intI1 和ISCR3在好氧污泥中的丰度范围分别为(1.32±0.13)×10-1~(8.99±0.75)×10-1和(1.18±0.29)×10-1~(1.81±0.29)×10-1,高于厌氧污泥中的丰度范围[(3.9±90.14)×10-3~(2.40±0.07)×10-2和(1.48±0.08) × 10-3~(2.61±0.31)×10-2] (图1),表明好氧污泥中抗药基因水平转移的可能性更大.为了进一步说明转移因子对tet基因分布的影响,对tet基因和转移因子进行相关性分析,发现tet(A)、tet(G)、tet(X)均与intI1呈现显著的正相关关系(R2=0.93,0.71, 0.51,P< 0.05) (图2A),表明intI1可能在这3种抗药基因的产生、传播和增殖中起到重要作用.此外,关于抗药基因与intI1相关性的报道已有不少,已有研究发现北京某公园土壤中tet(G)与intI1存在显著相关性(R2=0.62;P<0.01),表明intI1可能对tet(G)的传播起到促进作用[27].
本文首次调查了制药废水中异常插入序列ISCR3的丰度,并通过相关性分析发现tet(A)、tet(G)、tet(X)与ISCR3均存在显著的正相关关系(R2=0.35,0.70,0.38,P<0.05) (图2B),且ISCR3与intI1之间也存在显著的正相关关系(R2= 0.59,P< 0.05) (图2C).尽管至今未在intI1的任何基因盒区域检测到tet基因,但目前已发现整合子可与异常插入元件(insertion sequence common region, ISCR)结合成为更复杂的整合子,实现抗药基因的共同转移[28].在临床筛选的抗药性沙门氏菌中发现,ISCR3与intI1相连形成的复杂整合子携带tet(G)[29].综上所述,异常插入序列ISCR3和整合子intI1很可能在这3种tet基因的传播过程中具有协同作用,但作用机理还需要通过筛菌等方式进行进一步研究.
与进水相比,出水中外排泵类tet基因tet(A)、tet(G)、酶修饰类tet基因tet(X)相对丰度升高1~2个数量级,而核糖体保护类tet基因的tet(Q)、tet(W)相对丰度降低1~2个数量级(图3),抗药基因总相对丰度总体降低1个数量级,由进水中的(4.34±0.24)×10-1降低为出水中的(7.19±0.03)× 10-2.Christgen等[17]使用宏基因组方法调查了不同处理工艺对城市污水中抗药基因去除效果的影响,并比较了不同抗药机制的抗药基因在处理过程中的变化,结果发现,各种处理均导致出水中外排泵抗药基因的比例升高;同时,靶位点修饰类抗药基因(如核糖体保护类抗药基因)则在处理之后下降;而酶修饰类抗药基因在厌氧处理之后比例升高,好氧处理之后比例有所下降.本论文的结果与之相似,出水与进水中tet基因的分布差异也可能与抗药机制有关.外排泵类基因[tet(A)、tet(G)]通常与其它抗药基因(如抗重金属基因、抗消毒剂基因等)共同存在于转移因子上[30],其存在有助于细菌群落获得多抗性,而污水处理系统中残留的抗生素和重金属促进外排泵类抗药基因的选择.

图3 四环素生产废水处理系统进水和出水中抗药基因的相对丰度Fig.3 The relative abundance of ARGs (normalized to 16S rRNA genes) in the influent and effluent of tetracycline production wastewater treatment systems
就总体排放风险而言,土霉素和金霉素废水处理系统中5种tet基因在进水中的总绝对丰度为(1.49±0.26)×109~(2.52±0.17)×109copies/mL,出水中5种tet基因总绝对丰度降低,总绝对丰度为(2.06±0.63)×107~(1.42±0.27)×108copies/mL,去除率为94.4%~98.6%.据报导,某城市污水处理厂进水中tet抗药基因的去除率甚至超过99.9%[23].Christgen等[17]研究发现经过厌氧-好氧组合工艺,水相中抗药基因的去除率达到85.3%,但大部分抗药基因只是从水相转移到污泥相中,并没有得到实质性去除.为了进一步深入了解污水处理系统对抗药基因的削减程度,在后续研究中有必要对整个处理系统(包括污水和污泥)进行抗药基因通量研究.
3 结论
调查了两种四环素类抗生素(金霉素和土霉素)生产废水生物处理系统(厌氧-好氧组合工艺)中抗药基因的产生和排放,比较了厌氧污泥和好氧污泥中抗药基因及转移因子的分布,并结合宿主分布和水平转移因子分析了抗药基因的分布机理.主要得到以下结论:金霉素和土霉素生产废水处理系统的活性污泥和出水中均含有较高丰度的抗药基因,是重要的抗药基因污染源和储存库.厌氧污泥中,外排泵类和酶修饰类tet基因的丰度较低,而好氧污泥中,核糖体保护类tet基因的丰度较低,该分布特征与细菌群落组成、转移因子及抗药机制有关.从抗药基因传播角度来看,整合子intI1和异常插入序列ISCR3可能对某些抗药基因的传播起到促进作用;好氧处理过程中抗药基因通过这两种转移因子传播的可能性更大.
参考文献:
[1] 徐冰洁,罗 义,周启星,等.抗生素抗性基因在环境中的来源,传播扩散及生态风险 [J].环境化学, 2010,29(2):169-178.
[2] 李侃竹,高 品,王 凯,等.污水中抗生素与重金属对红霉素抗药性基因的选择性效应 [J].中国环境科学, 2015,35(3):889-896.
[3] Zhu Y G, Johnson T A, Su J Q, et al.Diverse and abundant antibiotic resistance genes in Chinese swine farms [J].Proceedings of the National Academy of Sciences, 2013,110(9): 3435-3440.
[4] 欧丹云,陈 彬,陈灿祥,等.九龙江下游河口水域抗生素及抗性细菌的分布 [J].中国环境科学, 2013,33(12):2243-2250.
[5] Pruden A, Pei R, Storteboom H, et al.2006.Antibiotic resistance genes as emerging contaminants: Studies in Northern Colorado [J].Environmental Science and Technology, 40:7445-7450.
[6] Wu N, Qiao M, Zhang B, et al.Abundance and diversity of tetracycline resistance genes in soils adjacent to representative swine feedlots in China [J].Environmental Science & Technology, 2010,44(18):6933-6939.
[7] 彭 双,王一明,林先贵.连续施用发酵猪粪对土壤中四环素抗性基因数量的影响 [J].中国环境科学, 2015,35(4):1173-1180.
[8] Larsson D G J.Pollution from drug manufacturing: review and perspectives [J].Philosophical Transactions of the Royal Society B: Biological Sciences, 2014,369(1656):20130571.
[9] Li D, Yu T, Zhang Y, et al.Antibiotic resistance characteristics of environmental bacteria from an oxytetracycline production wastewater treatment plant and the receiving river [J].Applied and Environmental Microbiology, 2010,76(11):3444-3451.
[10] Zhang Y, Yang M, Liu M, et al.Antibiotic pollution from Chinese drug manufacturing-antibiotic resistance [J].Toxicology Letters, 2012,211:S16.
[11] 张 昱,杨 敏,王春艳,等.生产过程中抗生素与抗药基因的排放特征,环境行为及控制 [J].环境化学, 2015,1:001.
[12] Liu M, Zhang Y, Yang M, et al.Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system [J].Environmental Science & Technology, 2012,46(14):7551-7557.
[13] Li D, Yang M, Hu J, et al.Antibiotic-resistance profile in environmental bacteria isolated from penicillin production wastewater treatment plant and the receiving river [J].Environmental Microbiology, 2009,11(6):1506-1517.
[14] Liu M, Ding R, Zhang Y, et al.Abundance and distribution of Macrolide-Lincosamide-Streptogramin resistance genes in an anaerobic-aerobic system treating spiramycin production wastewater [J].Water Research, 2014,63:33-41.
[15] Tao C W, Hsu B M, Ji W T, et al.Evaluation of five antibiotic resistance genes in wastewater treatment systems of swine farms by real-time PCR [J].Science of the Total Environment, 2014, 496:116-121.
[16] Wang Z, Zhang X X, Huang K, et al.Metagenomic profiling of antibiotic resistance genes and mobile genetic elements in a tannery wastewater treatment plant [J].Plosone, 2013,8(10): e76079.
[17] Christgen B, Yang Y, Ahamed Z, et al.Metagenomics shows low energy anaerobic-aerobic treatment reactors reduce antibiotic resistance gene dissemination from domestic wastewater [J].Environmental Science & Technology, 2015.
[18] Diehl D L, LaPara T M.Effect of temperature on the fate of genes encoding tetracycline resistance and the integrase of class 1integrons within anaerobic and aerobic digesters treating municipal wastewater solids [J].Environmental Science &Technology, 2010,44(23):9128-9133.
[19] 水和废水监测分析方法 [M].北京:中国环境科学出版社, 2002.
[20] Batt A L, Kim S, Aga D S.Comparison of the occurrence of antibiotics in four full-scale wastewater treatment plants with varying designs and operations [J].Chemosphere, 2007,68(3): 428-435.
[21] 李伟明,鲍艳宇,周启星.四环素类抗生素降解途径及其主要降解产物研究进展 [J].应用生态学报, 2012,23(8):2300-2308.
[22] Karthikeyan K G, Meyer M T.Occurrence of antibiotics in wastewater treatment facilities in Wisconsin, USA [J].Science of the Total Environment, 2006,361(1):196-207.
[23] Yang Y, Li B, Zou S, et al.Fate of antibiotic resistance genes in sewage treatment plant revealed by metagenomic approach [J].Water Research, 2014,62:97-106.
[24] Chen B, Liang X, Nie X, et al.The role of class I integrons in the dissemination of sulfonamide resistance genes in the Pearl River and Pearl River Estuary, South China [J].Journal of Hazardous materials, 2015,282:61-67.
[25] Roberts M C.Tetracycline resistance determinants: mechanisms of action, regulation of expression, genetic mobility, and distribution [J].FEMS Microbiology Reviews, 1996,19(1):1-24.
[26] Ochman H, Lawrence J G, Groisman E A.Lateral gene transfer and the nature of bacterial innovation [J].Nature, 2000,405(6784): 299-304.
[27] Wang F H, Qiao M, Lv Z E, et al.Impact of reclaimed water irrigation on antibiotic resistance in public parks, Beijing, China [J].Environmental Pollution, 2014,184:247-253.
[28] Toleman M A, Bennett P M, Walsh T R.ISCR elements: novel gene-capturing systems of the 21st century?[J].Microbiology and Molecular Biology Reviews, 2006,70(2):296-316.
[29] Schnabel E L, Jones A L.Distribution of tetracycline resistance genes and transposons among phylloplane bacteria in Michigan apple orchards [J].Applied and Environmental Microbiology, 1999,65(11):4898-4907.
[30] Shi P, Jia S, Zhang X X, et al.Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water [J].Water Research, 2012,47(1):111-120.
致谢:本实验的采样工作是在课题组左陆珅和王钰锴师兄的帮助下完成,在此表示感谢.
Characteristics of tetracycline resistance genes in the anaerobic and aerobic treatment of tetracycline production wastewater.
REN Jia1, YAO Hong1, LIU Miao-miao1*, ZHANG Yu2, YANG Min2(1.Department of Civil Engineering, Beijing Jiaotong University, Beijing 100044, China;2.State Key Laboratory of Environmental Aquatic Chemistry, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China).China Environmental Science, 2016,36(1):268~275
Abstract:In order to understand the characteristics of antibiotic resistance genes (ARGs) in the anaerobic and aerobic treatment processes of two tetracycline production wastewater, the abundances and distribution of six frequently reported tetracycline resistance genes (tet(A), tet(C), tet(G), tet(Q), tet(W), and tet(X)) and two types of mobile elements (intI1and ISCR3) were determined by PCR and quantitative PCR (qPCR).tet(C) was detected in none of the samples, while the others were discovered in all of the samples.In the systems of the tetracycline production wastewater, the relative abundances (normalized to 16S rRNA genes) of tet(A), tet(G), tet(X) in the anaerobic sludge((1.25±0.16)×10-4~(4.52± 0.002)×10-2) were lower than those in the aerobic sludge ((9.88±0.67)×10-5~(2.70±0.29)×10-1), while the relative abundances of tet(Q) and tet(W) in the anaerobic sludge ((1.66±0.03)×10-2~(7.48±1.22)×10-2) were significantly higher than those in the aerobic sludge (1.94±0.12)×10-3~(2.85±0.16)×10-2).Besides, the relative abundances of intI1 and ISCR3 in the anaerobic sludge ((1.48±0.01)×10-3~(2.61±0.31)×10-2) were significantly lower than those in the aerobic sludge ((1.18±0.15)×10-1~(8.99±0.75)×10-1), showing that the potential of horizontal gene transfer mediated by the mobile elements is lower in the anaerobic treatment process.It is indicated that aerobic treatment may facilitate the spread of tet(A), tet(G) and tet(X) but control the spread of tet(Q) and tet(W) in tetracycline production wastewater, with the opposite situation in the anaerobic treatment.The different distribution of tet genes was related to mobile genetic elements, resistancebook=269,ebook=272mechanism, and community structure.
Key words:tetracycline production wastewater;tetracycline resistance genes;mobile elements;anaerobic treatment;aerobic treatment
中图分类号:X835
文献标识码:A
文章编号:1000-6923(2016)01-0268-08
收稿日期:2015-08-31
基金项目:中央高校基本科研业务费专项资金资助(KCJB14046536);环境模拟与污染控制国家重点联合实验室(中国科学院生态环境研究中心)专项经费(14K01ESPCR);中国博士后科学基金项目(KCI14005531)
作者简介:任 佳(1993-),女,山西朔州人,硕士研究生,研究方向为环境中的抗药基因污染.
