椎间盘退变相关的基因芯片表达谱数据研究
2016-03-06万中元宋芳任大江单建林赵广民李放
万中元,宋芳,任大江,单建林,赵广民,李放
(1.北京军区总医院骨科,北京 100700;2.第二炮兵总医院口腔科,北京 100088)
椎间盘退变相关的基因芯片表达谱数据研究
万中元1,宋芳2,任大江1,单建林1,赵广民1,李放1
(1.北京军区总医院骨科,北京 100700;2.第二炮兵总医院口腔科,北京 100088)
目的 通过对已公开基因芯片表达谱数据进行研究,探究正常及退变椎间盘组织间的基因表达差异,并对差异表达基因所涉及的生物学过程进行预测分析。方法经GEO数据库选取与椎间盘退变相关的基因芯片表达谱数据GSE23130,对GSE23130包含的样本数据进行质量分析,选取GSM569833等10例样本数据纳入实验。采用GeneSpring 13.0软件对正常及退变样本间差异表达基因进行筛选,针对差异表达基因构建聚类分析树及热图,并采用GO分析、KEGG PATHWAY分析及DAVID功能注释簇集分析等方法对差异表达基因进行生物信息学分析。结果筛选出差异表达基因3 182个,其中1 735个基因在退变椎间盘内表达上调,1 447个表达下调。针对差异表达基因进行的生物信息学分析发现了VEGF信号通路、激活T细胞的核因子等数个存在研究价值的生物学过程。结论本研究结果显示椎间盘退变过程中存在大量基因的异常表达,这些差异表达基因相关的生物学过程也出现明显异常,某些生物学过程的异常改变可能在椎间盘退变过程中扮演了重要角色。
椎间盘退变;纤维环;基因芯片;生物信息学
颈椎病、腰椎间盘突出症等脊柱退行性疾病是影响中老年人生活质量的重要疾病,这些疾病不仅会带来严重的躯体痛苦,还给患者及其家庭带来了沉重的经济负担。椎间盘退变是引起这些疾病的主要原因,尽管针对椎间盘退变的研究已进行了多年,但到目前为止人们对椎间盘退变机制的认识尚不全面。椎间盘退变的过程伴随着细胞凋亡、胞外基质降解、基质生成异常等一系列改变[1],在这些改变的背后是相关蛋白编码基因的表达异常[2]。
基因芯片是近十余年来兴起的一项基因检测技术,利用基因芯片可以快速获得样本内所有基因同一时间点的表达信息,这些尤其适用于差异表达基因的筛选[3]。随着基因芯片的广泛应用,产生了海量的芯片数据,这些数据被储存于某些公共数据库中,对这些数据进行分析可以帮助我们揭示疾病背后隐藏的基因秘密。本研究以椎间盘退变相关基因芯片数据作为研究对象,通过统计学方法及生物信息学工具对椎间盘退变相关的差异表达基因进行了研究。
1 材料与方法
1.1 材料 本研究中所用椎间盘退变相关基因芯片表达谱数据来源于美国国立生物技术信息中心(The National Center for Biotechnology Information)基因表达数据库(Gene Expression Omnibus,GEO),基因系列号为GSE23130,所研究标本来源于正常及退变椎间盘纤维环组织。采用GeneSpring 13.0(Agilent Technologies,Santa Clara,CA,US)对GSE23130样本数据进行质量分析,最终10例样本数据(GSM569833、GSM569839、GSM569840、GSM569841、GSM569842为正常纤维环 组织,GSM569848、GSM569849、GSM569850、GSM569851、GSM569852为退变纤维环组织)被纳入实验。
1.2 方法 将样本数据导入GeneSpring 13.0软件,随后对正常及退变纤维环组织间差异表达基因进行筛选,差异表达筛选条件设置为成组Student′s t检验P<0.05(双尾),且基因表达水平相差超过两倍[4]。采用GeneSpring13.0以筛选获得的差异表达基因标准化数据为对象,进行聚类分析并构建热图。对差异表达上调及下调基因分别进行GO分析,从分子功能、生物过程和细胞组分三个角度对其进行富集分析及属性归属。经GO数据库默认的统计学算法计算得到与GO术语富集程度有关的P值,P值越小表示此生物学通路越重要,以P<0.05认定为差异具有统计学意义。将差异表达上调及下调基因分别导入KEGG PATHWAY数据库进行生物学通路分析,经数据库默认的统计学算法计算P值,P值小于0.05的生物学通路认定为具有统计学意义。同时采用DAVID(The Database for Annotation,Visualization and Integrated Discovery,DAVID,version 6.7)数据库对筛选出的差异表达上、下调基因分别进行功能注释簇集分析,分析时选择DAVID数据库默认的注释类别和“高”簇集分类标准选项,簇集评分大于1.3的认定为具有显著性[5]。
2 结 果
2.1 针对差异表达基因进行的GO分析结果 表达上调基因涉及173个GO生物过程条目、87个GO细胞组分条目、55个分子功能条目;表达下调基因涉及46个GO生物过程条目、14个GO细胞组分条目、17个分子功能条目,见表1。

表1 差异表达基因相关的GO分析条目(前5位条目)
2.2 差异表达基因相关的KEGG PATHWAY生物学通路差异和DAVID功能注释富集分析簇集 GSE23130筛选出差异表达基因3 182个,表达上调的基因有1 735个,表达下调的有1 447个,其中116个基因表达上调超过10倍,28个表达下调超过10倍。图1为根据差异表达基因数据构建的聚类分析树及热图,红色条块表示相关基因表达下调,绿色条块表示相关基因表达上调。针对上述两个基因表达谱数据系列进行的KEGG PATHWAY生物学通路分析发现了12个与上调表达基因相关的生物学通路,此外还发现了3个与下调表达基因相关的生物学通路(见表2)。针对GSE23130进行的DAVID功能注释富集分析发现与表达上调基因相关的簇集22个,此外还发现了12个与表达下调基因有关的簇集(见表3)。

表2 差异表达基因相关的KEGG PATHWAY生物学通路
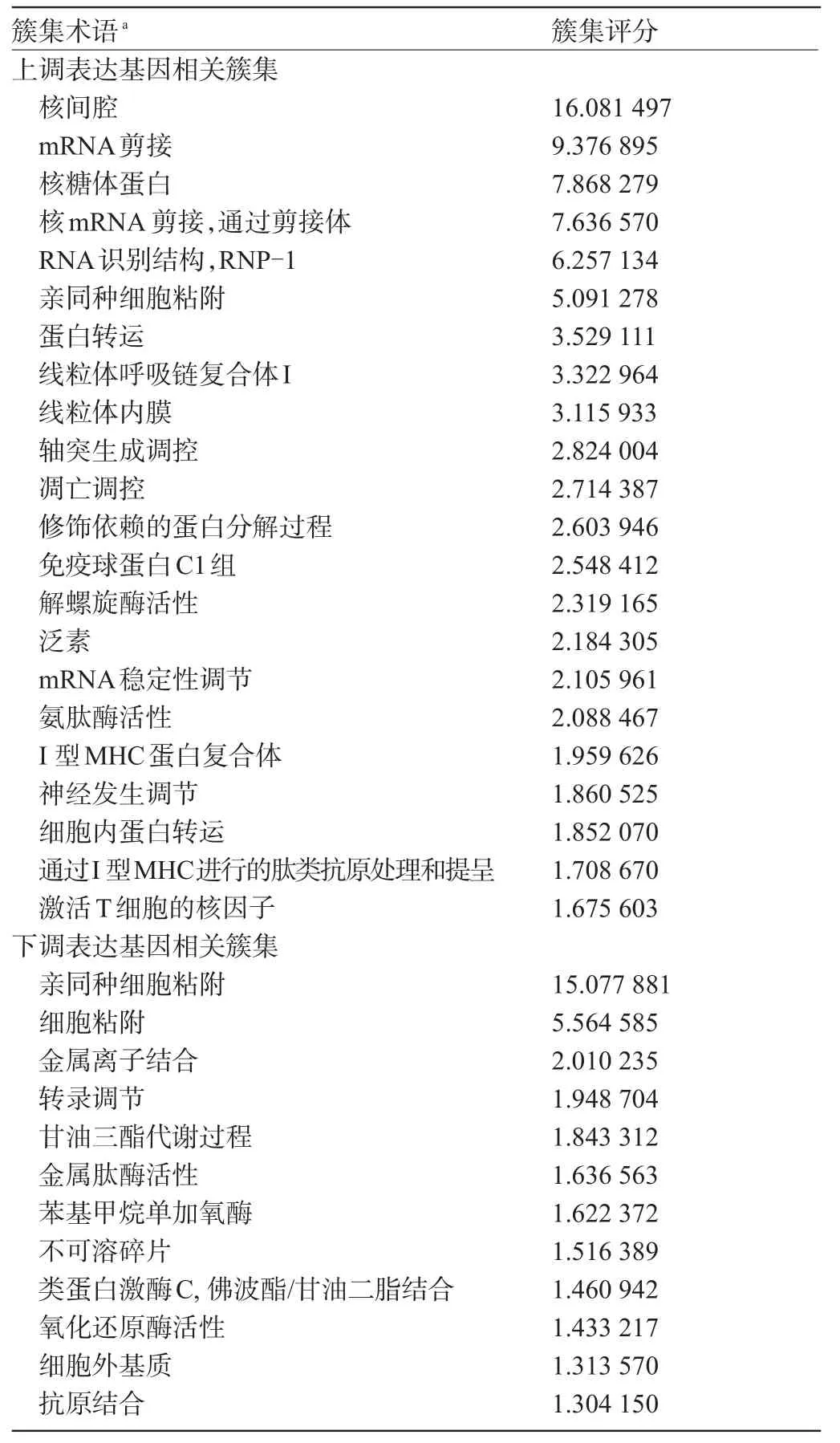
表3 差异表达基因相关的DAVID功能注释富集分析簇集
3 讨 论
本研究通过对GSE23130基因表达谱数据进行深入分析,发现了一批在退变和正常椎间盘组织间差异表达的蛋白编码基因。与此类似,过去针对椎间盘退变进行的基因表达谱芯片研究也发现椎间盘退变过程中存在大量基因的异常表达[6]。针对这些差异表达基因进行功能注释,能够帮助我们了解这些基因所参与的生物学过程。基于这一考虑,本研究采用GO、KEGG PATHWAY、DAVID功能注释簇集等生物信息学工具对差异表达的基因进行了分析。
结果显示表达上调的基因参与了TGF-β信号通路,过去的研究指出TGF-β信号通路的激活在椎间盘细胞生长、增殖、基质产生等方面发挥了重要作用[7],这些基因还参与了Wnt信号通路,椎间盘内Wnt通路的激活被认为与细胞外基质破坏有关[8],而TGF-β具有对抗Wnt信号通路激活的作用[9]。由于椎间盘中TGF-β信号通路的异常改变,TGF-β对Wnt的拮抗作用消失,导致椎间盘损伤后难以进行自我修复,加速了退变的发生。
椎间盘退变时椎间盘内细胞停止有丝分裂、提前进入衰老状态是造成椎间盘退变的因素之一。机械应力、炎性因子均可引起椎间盘细胞的提前衰老,此外,氧自由基引起的损伤也是诱发细胞衰老的原因之一[10-11]。退变时经由外周渗透入椎间盘内的营养成分和氧气减少,同时代谢废物排出障碍,引起氧自由基堆积、细胞氧化应激损伤,继而导致细胞周期调节障碍,细胞提前衰老。这一过程在本研究中即有所体现,结果显示上调表达基因参与了氧化磷酸化、细胞周期通路,也与线粒体呼吸链复合体Ⅰ等簇集有关,而下调表达基因在甘油三酯代谢过程、氧化还原酶活性等过程中发挥了作用。此外上调表达的基因还与凋亡调节簇集密切相关,以前的研究指出营养缺乏、氧化应激损伤不仅会引起细胞衰老,最终也会导致细胞的凋亡[12]。
自体免疫损伤是导致椎间盘退变的一个重要因素,本研究结果显示表达上调的基因与通过Ⅰ型MHC进行的肽类抗原处理和提呈、激活T细胞的核因子、Ⅰ型MHC蛋白复合体簇集有关,提示T细胞在椎间盘免疫损伤的过程中发挥了关键作用。正常情况下椎间盘处于免疫赦免状态,当椎间盘受损时其免疫赦免状态被打破,椎间盘内成分被机体免疫系统认定为外源抗原,经抗原提呈细胞处理后提呈给T细胞,从而激活T细胞针对椎间盘的免疫损伤。此外结果还显示上调基因与免疫球蛋白C1组、B细胞受体信号通路有关,表达下调的基因也参与了自然杀伤细胞介导的细胞毒作用,提示在椎间盘的自身免疫损伤过程中,不仅有T细胞的参与,B细胞及NK细胞均扮演了重要角色[13]。
除上述结果外,差异表达的基因还与剪接体、核糖体通路有关,提示退变时基因转录及蛋白翻译过程均出现很大改变。而黏合连接、黏着斑的异常提示退变时细胞同细胞间及细胞同基质间的关系发生了异常,这些改变会影响细胞的迁移、增殖等过程。结果中还涉及到数个与椎间盘退变时神经、血管侵入有关的生物学通路或簇集条目,例如VEGF信号通路、神经发生、神经生长调控。此外泛素介导的蛋白降解、修饰依赖的蛋白分解过程等生物学过程的异常变化提示背后的异常表达基因可能参与了退变椎间盘胞外基质降解这一过程。
本研究通过对椎间盘退变相关的基因芯片表达谱数据进行研究,发现了一系列和椎间盘退变相关的异常表达基因,这些基因相关的生物学过程也存在明显异常,针对这些基因进行深入研究将有助于我们加深对椎间盘退变机制的理解。
[1]Rahul J,Mike YC.Regenerative biology of the spine and spinal cord [M].Landes Bioscience and Springer Science+Business Media, 2012,CHAPTER 8:114-133.
[2]Chen K,Wu D,Zhu X,et al.Gene expression profile analysis of human intervertebral disc degeneration[J].Genet Mol Biol,2013,36 (3):448-454.
[3]Yan B,Wang ZH,Guo JT.The research strategies for probing the function of long noncoding RNAs[J].Genomics,2012,99(2):76-80.
[4]Ziats MN,Rennert OM.Aberrant expression of long noncoding RNAs in autistic brain[J].J Mol Neurosci.2013,49(3):589-593.
[5]Gillett A,Maratou K,Fewings C,et al.Alternative splicing and transcriptome profiling of experimental autoimmune encephalomyelitis using genome-wide exon arrays[J].PLoS One,2009,4(11):e7773.
[6]Gruber HE,Hoelscher GL,Ingram JA,et al.Genome-wide analysis of pain-,nerve-and neurotrophin-related gene expression in the degenerating human annulus[J].Mol Pain,2012,8:63.
[7]Jin H,Shen J,Wang B,et al.TGF-β signaling plays an essential role in the growth and maintenance of intervertebral disc tissue[J].FEBS Lett,2011,585(8):1209-1215.
[8]Hiyama A,Sakai D,Risbud MV,et al.Enhancement of intervertebral disc cell senescence by WNT/β-catenin signaling-induced matrix metalloproteinase expression[J].Arthritis Rheum,2010,62 (10):3036-3047.
[9]Hiyama A,Sakai D,Tanaka M,et al.The relationship between the Wnt/β-catenin and TGF-β/BMP signals in the intervertebral disc cell [J].J Cell Physiol,2011,226(5):1139-1148.
[10]Dai SM,Shan ZZ,Nakamura H,et al.Catabolic stress induces features of chondrocyte senescence through overexpression of caveolin 1:possible involvement of caveolin 1-induced down-regulation of articular chondrocytes in the pathogenesis of osteoarthritis[J].Arthritis Rheum,2006,54(3):818-831.
[11]Homma Y,Tsunoda M,Kasai H.Evidence for the accumulation of oxidative stress during cellular ageing of human diploid fibroblasts[J]. Biochem Biophys Res Commun,1994,203(2):1063-1068.
[12]Ahsan R,Tajima N,Chosa E,et al.Biochemical and morphological changes in herniated human intervertebral disc[J].J Orthop Sci, 2001,6(6):510-518.
[13]Sun Z,Zhang M,Zhao XH,et al.Immune cascades in human intervertebral disc:the pros and cons[J].Int J Clin Exp Pathol,2013,6 (6):1009-1014.
Study of gene expression profile of an intervertebral disc degeneration-related microarray.
WAN Zhong-yuan1, SONG Fang2,REN Da-jiang1,SHAN Jian-lin1,ZHAO Guang-min1,LI Fang1.1.Department of Orthopedics,General Hospital of Beijing Military Command,Beijing 100700,CHINA;2.Department of Stomatology,General Hospitalof Second Artillery Corps,Beijing 100088,CHINA
ObjectiveTo investigate the difference between gene expression profiles of normal and degenerated intervertebral disc by employing released microarray gene expression profile,and to predict and analyze the biological process related to the differentially expressed genes.MethodsAn intervertebral disc degeneration(IDD)related microarray dataset GSE23130 was selected from the Gene Expression Omnibus(GEO).Quality filtering were applied for the samples of GSE23130,and 10 samples such as GSM569833 were included.GeneSpring 13.0 software was applied to screen the differentially expressed genes between normal and degenerated samples.A hierarchical clustering tree and heat map was mapped for the differentially expressed genes.GO analysis,KEGG PATHWAY analysis and DAVID functional annotation clustering were performed for the genes.ResultsA total of 3 182 genes were found aberrantly expressing in the degenerated discs,of which 1 735 were up-regulated and 1 447 were down-regulated.Several interesting items,such as VEGF signaling pathway,nuclear factor of activated T cells(NFAT)were noted in the results of bioinformatics analysis.ConclusionThe current study shows a great number of genes are aberrantly expressed in IDD.Subsequent bioinformatics analysis indicates that biological process of these genes are abnormal.Some of these biological process might be the key links in IDD deserving further investigation.
Intervertebral disc degeneration;Annulus fibrous;Microarray;Bioinformatics
R681.5+3
A
1003—6350(2016)06—0870—04
10.3969/j.issn.1003-6350.2016.06.004
2015-10-15)
李放。E-mail:FANGL6722@sina.com
