水稻MADS-box基因研究进展
2016-02-14苏亚丽刘梦佳李海峰西北农林科技大学农学院旱区作物逆境生物学国家重点实验室陕西杨凌7200新疆农业职业技术学院新疆昌吉8300
苏亚丽,刘梦佳,李海峰,2*(.西北农林科技大学 农学院/旱区作物逆境生物学国家重点实验室,陕西 杨凌 7200;2.新疆农业职业技术学院,新疆 昌吉 8300)
水稻MADS-box基因研究进展
苏亚丽1,刘梦佳1,李海峰1,2*
(1.西北农林科技大学 农学院/旱区作物逆境生物学国家重点实验室,陕西 杨凌 712100;2.新疆农业职业技术学院,新疆 昌吉 831100)
MADS-box是水稻生长发育过程中重要的转录调控因子,其参与调控植物生长发育及胁迫响应。阐述了水稻MADS-box转录因子的结构、分类和染色体分布,从花器官发育、开花时间、根系发育、种子发育等方面综述了水稻MADS-box基因功能,并分析了水稻MADS-box基因功能研究中存在的一些问题及其发展前景。
水稻; MADS-box; 转录因子; 花发育; 开花时间
在植物中,MADS-box转录因子家族成员参与调控植物生长发育的许多关键方面,包括调控花器官的生长发育、根的生长发育甚至根瘤的形成、雌雄配子的发育、胚胎发育、种子发育以及果实的发育、成熟和开裂等,也参与调控光合作用、营养代谢、顶端分生组织分化以及激素信号转导等。水稻是我国乃至世界上的主要粮食作物之一,MADS-box基因是水稻生长发育过程中重要的转录调控因子。迄今,许多学者对水稻MADS-box基因进行了研究并取得了很大进展,特别是在水稻的花发育方面。综述了水稻MADS-box转录因子的结构、分类和染色体分布及其参与调控植物生长发育方面的生物学功能,并提出其功能研究中存在的一些问题,以期为我国水稻转基因育种提供基因资源。
1 水稻MADS-box基因家族成员结构、分类和染色体分布
植物MADS-box基因结构比较保守,都含有一个高度保守的MADS-box结构域,编码约58个氨基酸,分布在蛋白质的N端区域,可以对特异的DNA序列进行识别。MADS-box基因名字的缘由即为含有保守的MADS-box结构域[1]。
MADS-box基因家族根据其家族成员的蛋白质结构域可以分为type Ⅰ和type Ⅱ 两大类型[2]。其中,type Ⅰ型基因只含有MADS-box结构域[3],没有K结构域和内含子,并且在转录水平上其丰度很低。在植物中,大部分已知功能的MADS-box基因都属于type Ⅱ 型,它所编码的蛋白质包括MADS、I、K、C 4个结构域,所以又可称为MIKC型MADS-box基因[4]。根据Ⅰ结构域蛋白质序列的差异,MIKC型又可以分为MIKCC和 MIKC*2个类型[3,5],大多数植物中分离得到的是MIKCC型MADS-box基因,而水稻兼有2种类型[6]。植物中大部分的MIKC型MADS-box基因是由7个外显子和6个内含子组成,也有少数基因是由7个内含子和8个外显子组成,所具有的外显子和内含子结构相似。MADS结构域序列高度保守,由基因的外显子1编码,大多数MADS-box基因的上游有一段使MADS蛋白与DNA保守序列结合的核心序列(CC-A-rich-GG),称为CArG盒;Ⅰ结构域是DNA结合二聚体分子的重要结构,由基因的外显子2编码;外显子3—5编码半保守K结构域,K包括K1、K2和K3 三个α螺旋[7],通过调控蛋白质间的相互作用来控制基因表达;C结构域由外显子6 和 7 编码,其保守性弱,可能是 MADS-box 基因的转录激活区域,与MADS-box 蛋白复合物的形成有关[8]。在蛋白因子二聚体与DNA结合以及二聚化的过程中,MADS、I、K和C结构域起的作用不同[9]。
MADS-box基因广泛存在于动物、植物和真菌中,它们形成了一个庞大的基因家族,目前的基因组数据显示,水稻中已经有75个MADS-box基因被分离出来,并且分别被定位在不同的染色体上[10]。其中,1号染色体上分布最多,达16个;其次是4号染色体,有11个;2、3、6号染色体上分布也较多,分别为7、8、9个;8和12号染色体上均有6个MADS-box基因;5、7、9号染色体上分布较少,分别为4、3、3个;10号和11号染色体上最少,各自分布1个MADS-box基因。
2 水稻MADS-box基因的生物学功能
MADS-box是水稻生长发育过程中重要的转录调控因子,其参与调控花器官的生长发育、根的生长发育、种子发育、开花时间、顶端分生组织分化、对光周期的反应、激素信号转导等过程,除此之外,一些MADS-box基因在胁迫条件下也会出现一定的响应。主要从花发育、开花时间、根系发育、种子发育等方面阐述水稻MADS-box基因的生物学功能。
2.1 调控水稻花器官发育
20世纪90年代初,通过研究双子叶模式植物拟南芥(Arabidopsisthaliana)和金鱼草(Antirrhinummajus)花器官突变体的表型,深入阐释了双子叶植物花器官发育的遗传控制机制,在此基础上建立了花器官发育的ABC模型[11]。后来,Angenent等[12]从矮牵牛(Petunia)中分离出控制胚珠发育的D类基因FBP7(F-box protein 7)和FBP11,ABC模型被完善为ABCD模型。另外发现,拟南芥SEP1(SEPALLATA1)、SEP2和SEP3也是一类花器官同一性基因,是拟南芥花瓣、雄蕊和心皮正常发育必不可少的,水稻OsMADS1是雄蕊和浆片正常发育所需的,它们属于同一类基因,是一种新的花同源异型基因,称其为E类基因[13],即产生了ABCDE模型或AE模型。从此,花器官发育的充要条件也得到较全面的阐释[14-15]。近年来,随着基因组测序的完成,人们从水稻中已克隆出不少与花器官发育相关的 MADS-box基因,它们分属于ABCDE模型中的A~E类基因。其中,A类基因4个,B类基因3个,C类基因2个,D类基因2个,E类基因5个。
水稻花器官发育A类基因包括OsMADS14、OsMADS15、OsMADS18和OsMADS20,具有决定花分生组织特性的功能。水稻小穗原基形成之后,OsMADS14基因只在其内外稃和颖片中表达,在小穗成熟后,其只在心皮和雄蕊中表达[16]。OsMADS15的表达模式与OsMADS14不同,OsMADS15在小穗发育顶端分生组织及小穗器官分化之后的外稃、内稃和浆片中表达,表明OsMADS15在水稻花分生组织的识别中具有决定作用[17]。OsMADS18在根、叶、花序和所有花器官原基中都表达,利用RNAi沉默该基因后,植株没有明显的表型改变,这说明它很可能与其他的A类基因(如OsMADS15)存在功能冗余[18]。OsMADS20在种子发芽5 d后的茎中和生长的种子中表达,在生长的穗中不表达[19]。这与A类基因在花发育进化中具备的保守功能是相违背的。
水稻花器官发育B类基因包括OsMADS2、OsMADS4和OsMADS16。OsMADS16又被称为SPW1(SUPERWOMAN1),该基因在水稻的浆片和雄蕊中表达,SPW1隐性突变会导致雄蕊转变为心皮、浆片转变为内稃类结构[20]。OsMADS16基因被沉默后,会得到类似于spw1的突变表型;异位过量表达OsMADS16后,会导致心皮转变为雄蕊类器官,但是对外轮器官没有明显的影响[21]。当敲除OsMADS2基因后,浆片变长且具有外颖和稃片,表明OsMADS2对浆片的形成起重大作用[22]。反义OsMADS4转基因植株中,雄蕊变为心皮类的结构,浆片变为稃片类结构,说明OsMADS4主要决定雄蕊的形成[23]。
到目前为止,水稻中共发现了2个C类基因,即OsMADS3和OsMADS58,主要参与心皮和雄蕊的发育。OsMADS3的功能主要是决定雄蕊的形成,并且对浆片的发育具有抑制作用[24],而OsMADS58则主要控制颖花的分化和心皮的正常发育[25]。最新研究发现,水稻DFO1(DEFORMED FLORAL ORGAN 1)通过表观遗传抑制OsMADS58基因的表达来调节花器官身份。DFO1的功能表现在与水稻PcG(polycomb group)蛋白协同作用时,以组蛋白H3第27位赖氨酸三甲基(H3K27me3)作为一个表观抑制标记,调节OsMASD58基因上H3K27me3介导的表观抑制过程,以此保持水稻花器官形态[26]。这一发现意义重大,因为在水稻花器官发育过程中,表观遗传因素的作用还没有被详细地挖掘。
水稻花器官发育D类基因包括OsMADS13和OsMADS21。OsMADS13主要是在胚珠中表达,调控胚珠的发育;同时OsMAS13可以与OsMADS24、OsMADS45、OsMADS6 蛋白相互作用[27]。OsMADS21与OsMADS13具有较高的同源性,但它们的表达模式不一致,前者在发育的种子中表达,并且在心皮壁和胚珠中也有微弱的表达[28]。进一步研究发现,osmads21突变体没有表现出任何明显的表型变化,而且osmads13osmads21双突变体也不存在胚珠缺陷的加性效应;敲除osmads21后植株表型正常,这说明在进化过程中,OsMADS21可能丧失了功能[29]。总之,对水稻D类基因的研究还需进一步加强。
水稻花器官发育的E类基因主要包括:参与控制水稻内外稃分化的OsMADS1及控制心皮发育的OsMADS7、OsMADS8和OsMADS34等基因。OsMADS1在颖花的分生组织中表达,但不在颖片中表达,在内、外稃中强烈表达[17]。Wang等[30]对OsMADS1基因的MADS区域的第24、27位氨基酸进行突变,所得突变体有叶状的内外稃,雄蕊数目减少,偶尔还会见到有附加的雌蕊或小花生成。Hu等[31]研究发现,在特定的水稻花器官和分生组织身份识别中,OsMADS1调节功能多种多样,这可能是通过其遗传和物理因素与不同的花同源异型调节因子相互作用的结果。水稻OsMADS7 和OsMADS8基因在小穗分生组织和内外稃原基中表达[32],它们在水稻中过量表达会引起早花以及植株矮化[16,33]。OsMAD34基因在植株的根、叶、花序原基以及穗部器官组成型表达,其作用主要是控制花和穗的形成[34]。另外,对osmads34osmads1双突变体进行分析表明,该基因有专化内外稃、浆片、雄蕊和心皮等花器官特性的作用[35]。
另外,目前水稻中研究比较透彻的一个花发育基因是AGL6(AGAMOUOS LIKE 6)基因OsMADS6。该基因主要在花分生组织、内稃、浆片和胚珠中表达。OsMADS6突变体osmads6-1和mfo1(MOSAIC FLORAL ORGANS 1)小花多轮花器官表现出发育异常:内稃发育成了外稃状器官;浆片同源转化成了稃状结构;雄蕊数目减少的同时,部分形成了浆片-花药嵌合体;部分小花心皮数目增加,胚珠发育异常[36-38]。在花器官发育异常的同时,部分小花花分生组织失去了花分生组织特征,部分获得了花序或者小穗分生组织属性,在小花内形成新的小花[36-38]。中国科学院遗传与发育研究所薛永彪研究团队报道的MADS6强突变体osmads6-5表现出更强的表型:所有花器官都形成了外稃状结构,同时花分生组织失去了确定性,小花内不断形成一轮又一轮的外稃状结构[39]。对MADS6和水稻MADS1、MADS16、MADS3、MADS13、DL等基因的一系列双突变体进行研究表明,水稻花发育的一个重要调控因子是OsMADS6,通过转录水平上的调控等作用方式和其他花发育基因一起调控花分生组织和花器官的发育[37,39]。
2.2 调控水稻开花时间
开花是生殖生长阶段的起始,在水稻由营养生长向生殖生长过渡的这一过程中,有许多MADS-box基因起重要的调控作用。其中OsMADS14、OsMADS15、OsMADS45、OsMADS50、OsMADS51都是促进水稻开花的MADS-box基因[40]。
水稻OsMADS14、OsMADS15是编码成花素基因Hd3a(heading date 3a)和RFT1(RICE FLOWERING LOCUS T 1)的下游调控基因[41]。OsMADS14被认为是主要负责提前开花的调节因子,其表达水平提高是水稻由营养生长向生殖生长过渡的标志;在转基因水稻中,过量表达OsMADS14会严重缩短水稻抽穗期,形成极早花表型,说明OsMADS14可能在花分生组织确定中起作用[16]。过表达另外一个在下游调控水稻开花的OsMADS15基因,会使转基因水稻早期节间伸长,株高降低,提早开花[42]。
对于水稻OsMADS45基因,异位表达该基因不会改变转基因水稻基因的振荡节律,但会提前(大约20 d)上调2个成花素基因Hd3a和RFT1的表达。即转基因水稻在早期发育阶段,异位表达OsMADS45会激活上游基因Hd3a和RFT1,导致水稻提早开花。但OsMADS45过量表达并不影响Hd1和Ehd1的上游开花调节基因的表达,比如OsGI(OsGIGANTEA)、Ehd2(early heading date 2)/OsId1 (indeterminate 1)/RID1(rice indeterminate 1)和OsMADS50[43]。
在长日照条件下,OsMADS50会促进水稻开花,而OsMADS56会抑制水稻开花,即OsMADS56对开花时间的决定作用与OsMADS50相反。OsMADS50和OsMADS56在水稻调控开花时间网络的上游,通过一定的相互作用形成复合物,协同调控下游基因OsLFL1(LEC2 and FUSCA3 like protein 1)-Ehd1来控制水稻开花[44]。
对于水稻OsMADS51基因,其无义突变体在短日照条件下开花较正常植株晚2周,但是在长日照条件下该基因的缺失对开花只有很小的影响;而异位表达OsMADS51基因,在短日照条件下,导致水稻开花提前7 d[45]。研究表明,OsMADS51主要是介导调控下游OsMADS14、Hd3a和Ehd1基因的表达,并在短日照条件下促使水稻提早开花[46]。
2.3 调控水稻根系发育
在拟南芥硝酸盐信号途径中,ANR1(ARABIDOPSIS NITRATE REGULATED 1)是调控侧根发育的重要调节基因,隶属于MIKC型MADS-box家族。ANR1启动子-GUS融合试验表明,ANR1主要在侧根原基、主根和侧根的顶端分生组织中表达[47]。水稻中有OsMADS23、OsMADS25、OsMADS27、OsMADS57和OsMADS61五个ANR1的同源基因,都属于type Ⅱ MTKCCMADS-box家族,是AGL17家族基因中的进化分支[48]。对这5个受氮(N)、磷(P)和硫(S)诱导的基因进行克隆,利用分子生物学技术对它们调控根系生长的分子机制进行了研究,发现它们的响应机制各不相同。
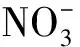
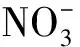
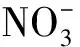
另外,水稻中的小RNA miR444,从感染病毒到OsRDR1(RNA-dependent RNA polymerase 1)表达的过程中,是传递抗病毒信号的一个关键因子[51]。miR444不仅通过硝酸盐信号途径调控根系发育,参与硝态氮的积累过程,而且对P饥饿有响应[52]。研究表明,水稻条纹叶枯病毒(RSV)侵染会增加miR444的表达,过表达miR444会提高水稻抵御RSV侵染的抗性,同时伴随OsRDR1表达的上调;进一步研究发现,3个miR444的靶标OsMADS23、OsMADS27a和OsMADS57之间能够形成同源二聚体或者异源二聚体,并通过直接与OsRDR1启动子的CArG基序结合,进而抑制OsRDR1的表达。因此,miR444水平增加会降低OsMADS23、OsMADS27a和OsMADS57对OsRDR1转录的抑制作用,因而激活OsRDR1介导的抗病毒RNA沉默通路[51]。
2.4 调控水稻种子发育
水稻中的MADS-box转录因子除了调控花发育、开花时间、根系发育,也可调控种子和胚乳的发育[53],且可能与雌雄配子的发育有一定的关联[54]。除此之外,OsMADS13作为D类基因STK(SEEDSTICK)、FBP7、FBP11的同源基因,与矮牵牛的胚珠特异基因FBP7、FBP11序列具有极高相似性[55]。对OsMADS13基因的突变体进行分析发现,突变体雌花败育,胚珠发育为心皮状结构[29]。
研究发现,OsMADS29主要在雌性生殖器官中表达,包括胚珠、胚珠维管束和除了果皮外层细胞的整个种子[56]。如果敲除OsMADS29基因,其相应株系表现出种子败育,同时在胚乳中也会表现出淀粉累积不足。组织切片观察OsMADS29敲除植株异常种子时发现,共质体(提供营养的珠心和胚珠维管束迹)运输连续性受到影响。另外,与野生型相比,所有OsMADS29敲除植株种子中的母体组织(包括果皮、维管束、珠被、珠心)的退化都被阻止。以上研究结果表明,调控水稻种子发育的OsMADS29基因是通过控制母体组织中的细胞退化来调控种子发育的。
最新的研究发现,另一个MADS-box基因OsMADS87与水稻种子发育密不可分。调节OsMADS87表达水平,可以调节水稻种子大小。通过对过表达、RNAi干扰OsMADS87和野生型植株的表型观察发现,抑制OsMADS87表达,会加速胚乳细胞化;过表达OsMADS87植株的种子热应力敏感性较低,且种子变小,OsMADS87被确定为提高水稻生殖发育期间种子热弹性潜在的目标基因[57]。
2.5 胁迫响应
水稻中的一些MADS-box转录因子对胁迫是有响应的。Arora等[10]研究发现,在水稻幼苗期进行冷、干旱和盐3种胁迫处理,4 h后,一些MADS-box基因的表达量发生变化,其中OsMADS18、OsMADS22、OsMADS26、OsMADS27表达量上升,OsMADS2、OsMADS30、OsMADS55表达量下降。后来,Khong等[58]进一步研究发现,水稻OsMADS26基因负调控水稻稻瘟病、白叶枯病抗性和抗旱性,这些表型均在控制环境和野生2种条件下被发现。
3 问题与展望
随着植物分子生物学的不断发展,水稻MADS-box转录因子家族的功能研究在各方面都有了很大的进展,但仍存在一定的不足。如大部分MADS-box基因都是通过转化拟南芥进行功能鉴定的,并且大多功能鉴定试验都是在实验室内进行的,这在一定程度上会限制一些基因的功能研究,因为同一家族基因在不同植物上的功能可能有所不同,且在不同试验条件下(大田、室内)鉴定的功能强弱可能也不同。
经过多年的研究,水稻一些基因功能得到验证,作用机制也得到解析。这些结果为进一步深入研究MADS-box基因家族奠定了良好的基础。随着更多MADS-box基因功能的解析和基因间相互作用的分析,使得建立一个MADS-box基因相互作用的调控网络,明确每个基因在调控网络中的位置和作用成为可能。另外,随着水稻部分MADS-box基因功能得到解析,这些基因将成为作物遗传改良育种的重要基因资源。
[1] De Bodt S,Raes J,Florquin K,etal.Genome wide structural annotation and evolutionary analysis of the type Ⅰ MADS-box genes in plants[J].Journal of Molecular Evolution,2003,56(5):573-586.
[2] Elena R A,Soraya P,Sarah J L,etal.An ancestral MADS-box gene duplication occurred before the divergence of plants and animals[J].PNAS,2000,97(10):5328-5333.
[3] Nam J,Kim J,Lee S,etal.Type Ⅰ MADS-box genes have experienced faster birth-and-death evolution than type Ⅱ MADS-box genes in angiosperms[J].PNAS,2004,101(7):1910-1915.
[4] Ma H,Yanofsky M F,Meyerowitz E M.AGL1-AGL6,anArabidopsisgene family with similarity to floral homeotic and transcription factor genes[J].Genes Dev,1991,5(3):484-495.
[5] Kaufmann K,Melzer R,Theissen G.MIKC-type MADS-domain proteins:Structural modularity,protein interactions and network evolution in land plants[J].Gene,2005,347(2):183-198.
[6] Liu Y,Cui S J,Wu F,etal.Functional conservation of MIKC*-type MADS box genes inArabidopsisand rice pollen maturation[J].The Plant Cell,2013,25(4):1288-1303.
[7] Honma T,Goto K.Complexes of MADS-box proteins are sufficient to convert leaves into floral organs[J].Nature,2001,409(6819):525-529.
[8] Piwarzyk E,Yang Y,Jack T.Conserved C-terminal motifs of theArabidopsisproteins APETALA3 and PISTILLATA are dispensable for floral organ identity function[J].Plant Physiology,2007,145(4):1495-1505.
[9] 刘菊华,徐碧玉,张静,等.MADS-box转录因子的相互作用及对果实发育和成熟的调控[J].遗传,2010,32(9):893-902.
[10] Arora R,Agarwal P,Ray S,etal.MADS-box gene family in rice:Genome-wide identification,organization and expression profiling during reproductive development and stress[J].BMC Genomics,2007,8:242.
[11] Ma H,Depamphilis C.The ABCs of floral evolution[J].Cell,2000,101(1):5-8.
[12] Angenent G C,Franken J,Busscher M,etal.A novel class of MADS box genes is involved in ovule development inPetunia[J].Plant Cell,1995,7(10):1569-1582.
[13] Cui R,Han J,Zhao S,etal.Functional conservation and diversification of class E floral homeotic genes in rice(Oryzasativa)[J].Plant Journal,2010,61(5):767-781.
[14] Theissen G.Development of floral organ identity:Stories from the MADS house[J].Current Opinion in Plant Biology,2001,4(1):75-85.
[15] Ferrario S,Immink R G H,Shchennikova A,etal.The MADS box geneFBP2 is required for SEPALLATA function inPetunia[J].Plant Cell,2003,15(4):914-925.
[16] Pelucchi N,Fornara F,Favalli C,etal.Comparative analysis of rice MADS-box genes expressed during flower development[J].Sexual Plant Reproduction,2002,15(3):113-122.
[17] Kobayashi K,Yasuno N,Sato Y,etal.Inflorescence me-ristem identity in rice is specified by overlapping functions of threeAP1/FUL-like MADS box genes andPAP2,aSEPALLATAMADS box gene[J].Plant Cell,2012,24(5):1848-1859.
[18] Fornara F,Parenicova L,Falasca G,etal.Functional characterization ofOsMADS18,a member of the AP1/SQUA subfamily of MADS box genes[J].Plant Physiology,2004,135(4):2207-2219.
[19] Lee S,Kim J,Son J S,etal.Systematic reverse genetic screening of T-DNA tagged genes in rice for functional genomic analyses: MADS-box genes as a test case[J].Plant and Cell Physiology,2003,44(12):1403-1411.
[20] Nagasawa N,Miyoshi M,Sano Y,etal.SUPERWOMAN1 andDROOPINGLEAFgenes control floral organ identity in rice[J].Development,2003,130(4):705-718.
[21] Xiao H,Wang Y,Liu D F,etal.Functional analysis of the riceAP3 homologueOsMADS16 by RNA interference[J].Plant Molecular Biology,2003,52(5):957-966.
[22] Prasad K,Vijayraghavan U.Double-stranded RNA interference of a ricePI/GLOparalogOsMADS2,uncovers its second-whorl-specific function in floral organ patterning[J].Genetics,2003,165(4):2301-2305.
[23] Kang H G,Jeon J S,Lee S,etal.Identification of class B and class C floral organ identity genes from rice[J].Plant Mol Biol,1998,38(6):1021-1029.
[24] Li H,Liang W,Yin C,etal.Genetic Interaction ofOsMADS3,DROOPINGLEAF,andOsMADS13 in specifying rice floral organ identities and meristem determinacy[J].Plant Physiology,2011,156(1):263-274.
[25] Yamaguchi T,Nagasawa N,Kawasaki S,etal.The YABBY geneDROOPINGLEAFregulates carpel specification and midrib development inOryzasativa[J].Plant Cell,2004,16(2):500-509.
[26] Zheng M,Wang Y H,Wang Y L,etal.DEFORMED FLORAL ORGAN1 (DFO1) regulates floral organ identity by epigenetically repressing the expression ofOsMADS58 in rice (Oryzasativa)[J].New Phytologist,2015,206(4):1476-1490.
[27] Favaro R,Immink R G H,Ferioli V,etal.Ovule-specific MADS-box proteins have conserved protein-protein interactions in monocot and dicot plants[J].Molecular Genetics and Genomics,2002,268(2):152-159.
[28] Dreni L,Jacchia S,Fornara F,etal.The D-lineage MADS-box geneOsMADS13 controls ovule identity in rice[J].Plant Journal,2007,52(4):690-699.
[29] Dreni L A,Pilatone D P,Yun S,etal.Functional analysis of all AGAMOUS subfamily members in rice reveals their roles in reproductive organ identity determination and meristem determinacy[J].Plant Cell,2011,23(8): 2850-2863.
[30] Wang N,Li Y,Sang X,etal.Nonstopglumes(nsg),a novel mutant affects spikelet development in rice[J].Genes & Genomics,2013,35(2):149-157.
[31] Hu Y,Liang W,Yin C,etal.Interactions ofOsMADS1 with floral homeotic genes in rice flower development[J].Mol Plant,2015,8(9):1366-1384.
[32] Ciaffi M,Paolacci A R,Tanzarella O A,etal.Molecular aspects of flower development in grasses[J].Sexual Plant Reproduction,2011,24(4):247-282.
[33] Jeon J S,Lee S,Jung K H,etal.Production of transgenic rice plants showing reduced heading date and plant height by ectopic expression of rice MADS-box genes[J].Mol Breeding,2000,6:581-592.
[34] Gao X C,Liang W Q,Yin C S,etal.TheSEPALLATA-like geneOsMADS34 is required for rice inflorescence and spikelet development[J].Plant Physiology,2010,153(2):728-740.
[35] Kobayashi K,Maekawa M,Miyao A,etal.PANICLEPHYTOMER2 (PAP2),encoding a SEPALLATA subfamily MADS-box protein,positively controls spikelet meristem identity in rice[J].Plant Cell Physiol,2010,51(1):47-57.
[36] Li H,Liang W,Hu Y,etal.RiceMADS6 interacts with the floral homeotic genesSUPERWOMAN1,MADS3,MADS58,MADS13,andDROOPINGLEAFin specifying floral organ identities and meristem fate[J].The Plant Cell,2011,23(7):2536-2552.
[37] Ohmori S,Kimizu M,Sugita M,etal.MOSAICFLORALORGANS1,anAGL6-like MADS box gene,regulates floral organ identity and meristem fate in rice[J].Plant Cell,2009,21(10):3008-3025.
[38] Li H,Liang W,Jia R,etal.TheAGL6-like geneOsMADS6 regulates floral organ and meristem identities in rice[J].Cell Res,2010,20(3):299-313.
[39] Duan Y,Xing Z,Diao Z,etal.Characterization ofOsmads6-5,a null allele,reveals thatOsMADS6 is a critical regulator for early flower development in rice(OryzasativaL.)[J].Plant Mol Biol,2012,80(4/5):429-442.
[40] 宋远丽,栾维江.水稻开花的光温调控分子机制[J].中国水稻科学,2012,26(4):383-392.
[41] Komiya R,Ikegami A,Tamaki S,etal.Hd3aandRFT1 are essential for flowering in rice[J].Development,2008,135(4):767-774.
[42] Lu S J,Wei H,Wang Y,etal.Overexpression of a transcription factorOsMADS15 modifies plant architecture and flowering time in rice(OryzasativaL.)[J].Plant Molecular Biology Reporter,2012,30(6):1461-1469.
[43] Wang J D,Lo S F,Li Y S,etal.Ectopic expression ofOsMADS45 activates the upstream genesHd3aandRFT1 at an early development stage causing early flowering in rice[J].Botanical Studies,2013,54(1):12.
[44] Ryu C H,Lee S,Cho L H,etal.OsMADS50 andOsMADS56 function antagonistically in regulating long day (LD)-dependent flowering in rice[J].Plant Cell Environ,2009,32(10):1412-1427.
[45] Komiya R,Yokoi S,Shimamoto K.A gene network for long-day flowering activatesRFT1 encoding a mobile flowering signal in rice[J].Development,2009,136(20):3443-3450.
[46] Zhang H,Forde B G.AnArabidopsisMADS box gene that controls nutrient-induced changes in root architecture[J].Science,1998,279(5349):407-409.
[47] Remans T,Nacry P,Pervent M,etal.TheArabidopsisNRT1.1 transporter participates in the signaling pathway triggering root colonization of nitrate-rich patches[J].PNAS,2006,103(50):19206-19211.
[48] Puig J,Meynard D,Khong G N,etal.Analysis of the expression of theAGL17-like clade of MADS-box transcription factors in rice[J].Gene Expression Patterns,2013,13(5/6):160-170.
[49] Yu C,Liu Y,Zhang A,etal.MADS-box transcription factorOsMADS25 regulates root development through affection of nitrate accumulation in rice[J].PLoS One,2015,10(8):e0135196.
[50] Yu C Y,Su S,Xu Y C,etal.The effects of fluctuations in the nutrient supply on the expression of five members of theAGL17 clade of MADS-box genes in rice[J].PLoS One,2014,9(8):e105597.
[51] Wang H,Jiao X,Kong X,etal.A signaling cascade from miR444 to RDR1 in rice antiviral RNA silencing pathway[J].Plant Physiol,2016,170(4):2365-2377.
[52] Yan Y S,Wang H C,Hamera S,etal.miR444a has multiple functions in the rice nitrate-signaling pathway[J].Plant Journal,201478(1):44-55.
[53] Zhang J A,Nallamilli B R,Mujahid H,etal.OsMADS6 plays an essential role in endosperm nutrient accumulation and is subject to epigenetic regulation in rice(Oryzasativa)[J].Plant Journal,2010,64(4):604-617.
[54] Dreni L,Zhang D.Flower development:The evolutionary history and functions of theAGL6 subfamily MADS-box genes[J].J Exp Bot,2016,67(6):1625-1638.
[55] Opez-Dee Z P,Wittich P,Enrico Pè M,etal.OsMADS13,a novel rice MADS-box gene expressed during ovule development[J].Developmental Genetics,1999,25(3):237-244.
[56] Yang X,Wu F,Lin X,etal.Live and let die-the B(sister) MADS-box geneOsMADS29 controls the degeneration of cells in maternal tissues during seed development of rice(Oryzasativa)[J].PLoS One,2012,7(12):e51435.
[57] Chen C,Begcy K,Liu K,etal.Molecular characterization of rice endosperm development under heat stress identifiesOsMADS87 as a determinant of seed size and thermal sensitivity[J].Plant Physiology,2016,171(1):606-622.
[58] Khong G N,Pati P K,Richaud F,etal.OsMADS26 negatively regulates resistance to pathogens and drought tolerance in rice[J].Plant Physiology,2015,169(4):2935-2949.
Research Progress on MADS-box Genes in Rice(Oryzasativa)
SU Yali1,LIU Mengjia1,LI Haifeng1,2*
(1.College of Agronomy,Northwest A&F University/State Key Laboratory of Crop Stress Biology for Arid Areas,Yangling 712100,China; 2.Xinjiang Agricultural Vocational Technical College,Changji 831100,China)
MADS-box is important transcription regulatory factor in the process of rice growth and development,which participates in the regulation of plant growth and development and response to stress.In this paper,the structure,classification and distribution in chromosomes of the rice MADS-box transcription factors were stated,the biological functions of rice MADS-box transcription factors were reviewed in flower development,flowering time,root and seed development,and the development prospect and some problem existing in the study of rice MADS-box transcription factors function were described.
rice; MADS-box; transcription factors; flower development; flowering time
2016-03-27
国家自然科学基金项目 (31571657);新疆农业职业技术学院科研项目 (XJNZYKJ201501);高等学校中央基本科研业务费项目(2014ZZ009)
苏亚丽(1991-),女,河南新郑人,在读硕士研究生,研究方向:植物分子生物学。E-mail:354624492@qq.com
*通讯作者:李海峰(1970-),男,河南南阳人,副教授,博士,主要从事植物发育分子生物学方面的研究。 E-mail:lhf@nwsuaf.edu.cn
S511
A
1004-3268(2016)09-0001-07
