化能自养菌氨单加氧酶基因的克隆
2015-12-29龚国利史政豪魏选明
龚国利, 张 甜, 史政豪, 魏选明, 王 磊
(陕西科技大学 食品与生物工程学院, 陕西 西安 710021)
化能自养菌氨单加氧酶基因的克隆
龚国利, 张甜, 史政豪, 魏选明, 王磊
(陕西科技大学 食品与生物工程学院, 陕西 西安710021)
摘要:氨氧化细菌是一类革兰氏阴性的化能自养菌.也是生物脱氮工艺中不可缺少的一类细菌.本研究通过以土壤为材料富集氨氧化细菌,并从富集土样的全基因组中成功扩增到amoA全长基因,与NCBI标准菌株Nitrosomonas sp. GH22序列同源性达到99%,并用amoA全长基因构建得克隆载体,经菌落PCR和双酶切鉴定正确,为后期构建新型的生物脱氮基因工程菌奠定基础.
关键词:氨氧化细菌; 生物脱氮; 鉴定
0引言
自养细菌分为光能自养菌和化能自养菌,光能自养菌(微生物)一般都含有叶绿体,而化能自养菌又称为无机营养菌,是一类不依赖任何有机营养物即可生长、繁殖的微生物.这类微生物能氧化某种无机物并利用所产生的化学能还原二氧化碳和生成有机碳化合物.自然界中化能自养菌种类不多,并且氧化无机物的专性很强,例如硝化细菌只能氧化亚硝酸盐.化能自养菌在土壤中有相当数量,对物质转化有一定作用.
随着当代工业和社会的快速发展,向环境中排放大量生活污水及工业污水,而这些污水的主要污染成分又以氨态氮、分子氮、硝态氮、亚硝态氮等为主,如若这些污水不经处理直接排入江河中会造成水体富营养化,也会对人和生物体造成毒害作用[1].近年来世界各国人民的环保意识越来越强 ,对于水体污染处理也形成了许多新型工艺,如SHARON工艺、CANON工艺、OLAND工艺、单级自养脱氮工艺等[2,3].这些工艺的共同点就是在脱氮过程是基于氨氧化细菌存在的基础上进行的[4],可将氨氮转化成亚硝态氮,有氧条件下,反应方程为:2NH3+3O2→2HNO2+2H2O+能量.
氨氧化细菌的氨单加氧酶基因(amoA)[5],它可将铵氧化成羟胺,再通过氨氧化细菌的羟胺氧化酶基因(hao)使羟胺转化成亚硝酸来实现亚硝化作用[6],属革兰氏阴性专性化能自养细菌,以铵盐的氧化满足其能量需求,普遍分布在自然界的土壤、海洋及淡水中,但只占其中细菌总量的极小比例,且不同种属的氨氧化细菌所分布的环境不同.自养氨氧化细菌由于其生长缓慢和较难培养的缘故,因此直接从土壤中分离到纯种的氨氧化细菌有一定的难度.
本研究通过对采集的土壤样品[7]进行多次富集后提取土壤样品的全基因组扩增amoA基因,并构建克隆载体测序,为将来构建生物脱氮的基因工程菌提供了基础依据.
1材料与方法
1.1材料
1.1.1试剂及菌株
PMD18-T vector、限制性内切酶EcoRⅠ和SalⅠ均购自大连宝生物公司、细菌基因组提取试剂盒和胶回收试剂盒由北京天根生化有限公司提供、2×Power Taq PCR MasterMix购自北京百泰克生物技术有限公司、质粒提取试剂盒、PCR引物均由上海捷瑞生物公司提供、IPTG、X-Gal、氨苄青霉素(Sigma Cheminal Co.)、琼脂糖、大肠杆菌DH5α为本实验室保存.酵母浸粉、大豆蛋白胨(北京奥博星生物技术有限责任公司)、NaCl(天津市东丽区天大化学试剂厂)、(NH4)2SO4、KH2PO4、MgSO4·7H2O 、CaCl2均为分析纯.
1.1.2仪器
仪器:PCR仪、电泳仪、电泳槽(BIO-RAD)、BIS910凝胶成像系统(北京东胜创新生物科技有限公司)、TGL-16M高速冷冻离心机(长沙湘仪离心机有限责任)、立式压力蒸汽灭菌器(上海博讯实验有限公司医疗设备厂)、SHZ-82气浴恒温振荡器(金坛市荣华仪器制造有限公司)、移液枪(Eppendorf公司).
1.2方法
1.2.1氨氧化细菌的富集培养[8,9]
氨氧化细菌富集培养基 (NH4)2SO45 g,KH2PO40.7 g,MgSO4·7H2O 0.5 g,CaCl20.36 g,蒸馏水1 L,调节pH值为7.2,121 ℃高温灭菌30 min.
选取陕西科技大学花园土壤,称取1 g土壤样品于100 mL氨氧化细菌富集培养基中,磁力搅拌器搅拌使土样完全分散,摇床30 ℃、150 r/min富集15~30 d,每隔两天用格里斯试剂进行检验,待到富集液与格里斯试剂显粉色时即代表氨氧化细菌氧化铵盐生成一定数量的NO2-,培养液是否变红及其颜色深浅可指示NO2-的生成及其浓度,NO2-可与格里斯试剂共同作用生成橙红色的络合物[10].将生成红色较深的土样富集液继续转接至100 mL新鲜的氨氧化细菌富集培养基中培养,连续富集3~4次后即可提取富集土样的基因组进行后续实验.
1.2.2PCR扩增amoA全长基因
引物设计在NCBI Genbank上查找序列登录号L08050[11]amoA全长基因设计引物amof/amor、amo-1F/amo-2R,引物序列见表1所示.
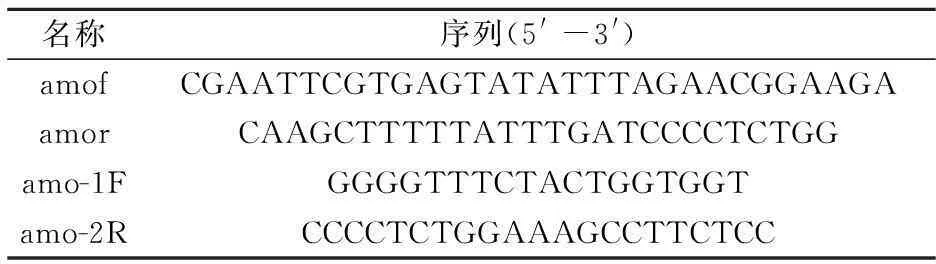
表1 amoA基因扩增的引物序列
(1)富集土壤总DNA的提取
取富集3次的土壤浑浊液2 mL,根据试剂盒提取土壤的总DNA.
(2)amoA全长基因的扩增
PCR反应体系50μL,DNA模板2μL、上游引物amof(10μmol/L)2μL、下游引物amor(10μmol/L)2μL、2×Power Taq PCR MasterMix 25μL、补水至50μL.
PCR反应条件:首先94 ℃变性3 min;然后30个循环:94 ℃ 30 s、48 ℃ 30 s、72 ℃ 1 min;最后是72 ℃延伸5 min,4 ℃保存.PCR产物经0.8%的琼脂糖凝胶电泳分析后进行切胶回收.
(3)以切胶回收的amoA全长基因组为模板进行套式PCR验证[12]
PCR反应体系20μL,DNA模板1μL、上游引物amo-1F(10μmol/L)1μL、下游引物amo-2R(10μmol/L)1μL、2×Power Taq PCR MasterMix 10μL、补水至20μL.
PCR反应条件:首先94 ℃变性3 min;然后30个循环:94 ℃ 1 min、55 ℃ 1 min 30 s、72 ℃ 1 min;最后是72 ℃延伸10 min,4 ℃保存.PCR产物经0.8%的琼脂糖凝胶电泳分析.
1.2.3amoA基因的克隆[13]
(1)LB液体培养基
酵母浸粉0.5 g,大豆蛋白胨1.0 g,NaCl 1 g,蒸馏水100 mL,121 ℃高温灭菌30 min.
(2)克隆载体PMD18-T-amoA的构建
amoA基因克隆载体PMD18-T vector连接,10μL反应体系:PMD18-T vector 1μL、amoA基因模板4μL、连接缓冲液SolotionⅠ 5μL ;16 ℃连接30 min.
PMD18-T-amoA转化感受态及PCR验证.将制备好的感受态细胞[14]从-80 ℃冰箱中拿出后置于碎冰上融解,然后加入10μL连接产物,混匀,冰上放置25 min;移入42 ℃水浴中放置90 s(秒表记)后,快速转移到冰浴中,冷却5 min,加入0.2 mL LB培养基,37 ℃、200 r/min振荡培养1 h后涂布含有Amp、IPTG和X-Gal的平板,过夜培养后挑取白色菌落进行菌落PCR验证,验证用M13通用引物.
1.2.4PMD18-T-amoA阳性菌落的筛选和鉴定
对于验证后的菌落挑选后进行扩大培养,抽质粒后进行双酶切验证,验证正确后送测序公司测序.
(1)PMD18-T-amoA阳性菌落PCR验证
PCR反应体系20μL,DNA模板1μL、上游引物M13f(10μmol/L)1μL、下游引物M13r(10μmol/L)1μL、2×Power Taq PCR MasterMix 10μL、补水至20μL.
PCR反应条件:首先95 ℃变性5 min;然后30个循环:95 ℃ 30 s、42 ℃ 30 s、72 ℃ 1 min;最后是72 ℃延伸10 min,4 ℃保存.PCR产物经0.8%的琼脂糖凝胶电泳分析.
(2)阳性菌落的双酶切鉴定
双酶切验证体系50μL.EcoRⅠ 1μL、SalⅠ 1μL、质粒30μL、10×H buffer 5μL、补水至50μL.
1.2.5测序及序列同源性分析
测序由北京奥科鼎盛生物科技有限公司完成.测序结果提交GenBank进行blast分析.
2结果与讨论
2.1amoA基因的扩增和套式PCR验证
2.1.1amoA基因的扩增
利用引物amof/amor对富集的土壤全基因组进行扩增,扩增到一段约830 bp的片段(见图1所示),与文献记载相符[13].
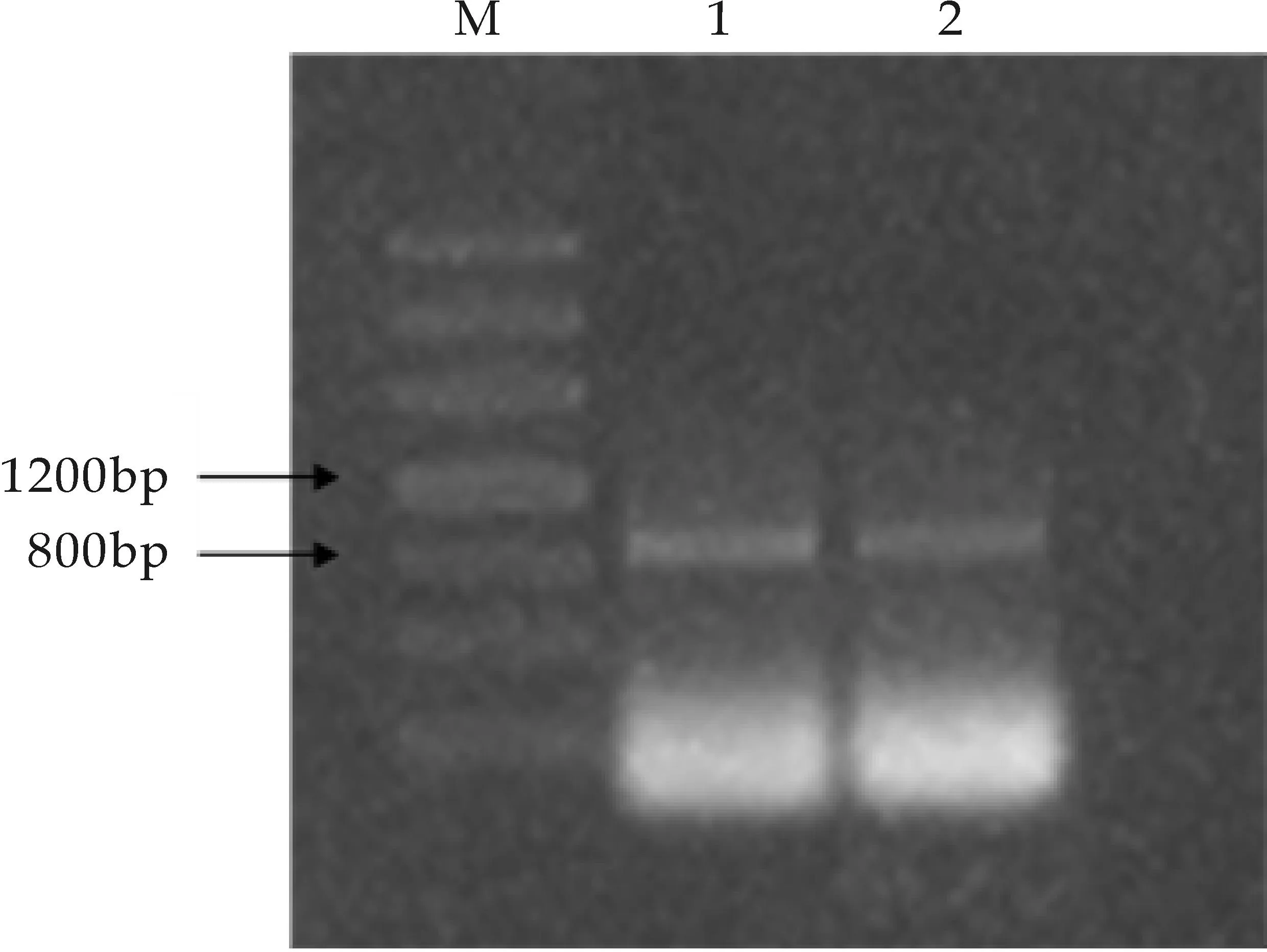
1、2为PCR扩增amoA全长基因图1 PCR扩增amoA全长基因
2.1.2 以扩增到的amoA基因为模板
利用引物amo-1F/amo-2R进行套式PCR,扩增到一段约500 bp左右的条带(见图2所示),与文献记载相符[12].
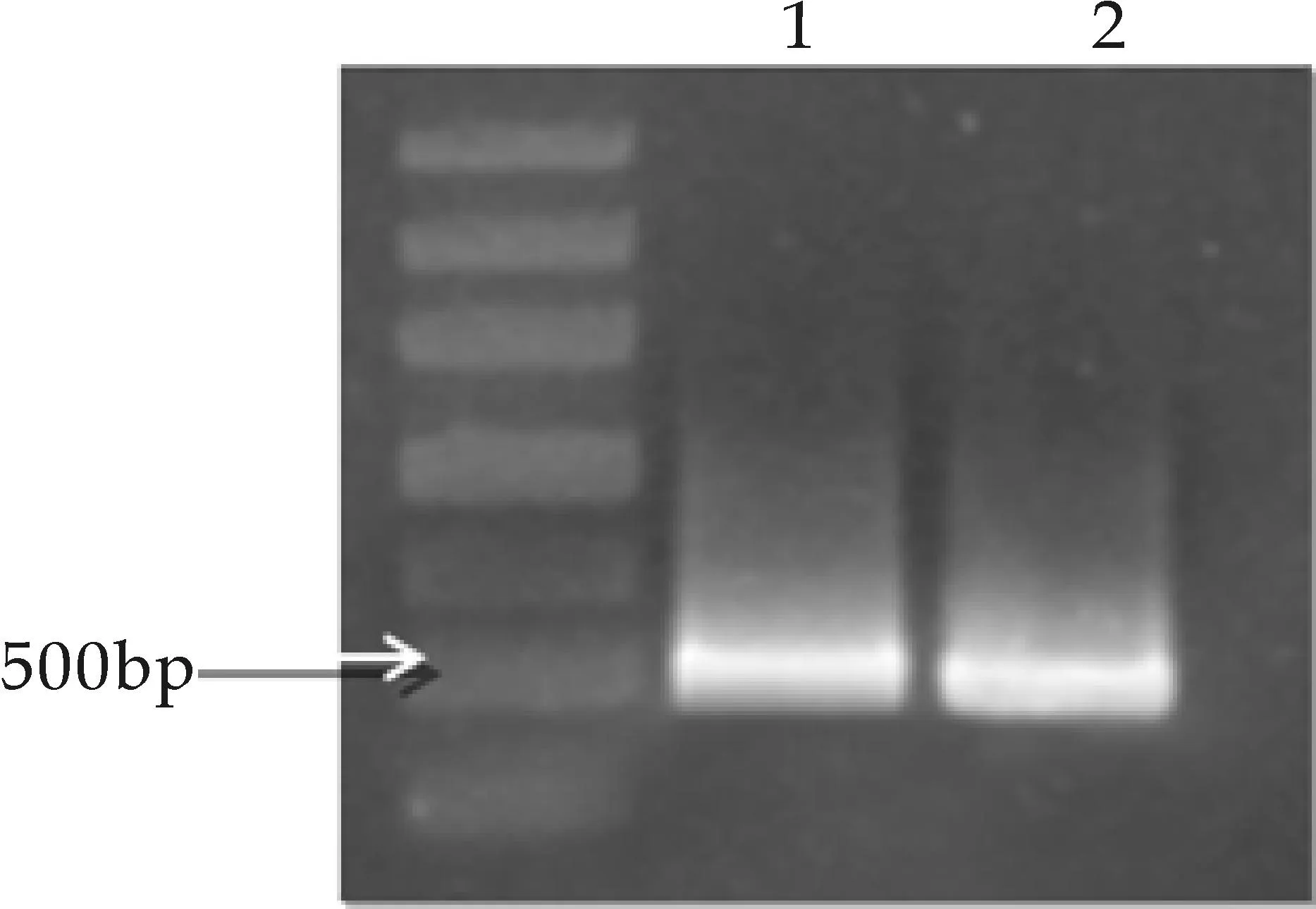
1、2为PCR扩增amoA基因片段图2 amoA基因套式PCR
2.2克隆载体PMD18-T-amoA挑选重组子进行菌落PCR验证
克隆载体PMD18-T-amoA挑选24个重组子进行菌落PCR验证,24个重组子依次命名为a1-a24(见图3所示).

1~24:a1~a24号重组子菌落PCR验证图3 克隆载体PMD18-T-amoA重组子验证
从图3可看出,除a2、a3号重组子之外,在830 bp附近都有有明亮的条带,初步说明已将目的片段连入克隆载体,挑选除a2、a3号外的任意两个重组子进行过夜培养后进行双酶切验证.
2.3克隆载体PMD18-T-amoA挑选重组子进行双酶切验证
克隆载体PMD18-T-amoA挑选a8、a11过夜培养抽质粒进行双酶切验证(见图4所示).

1:a8 EcoRⅠ/ SalⅠ双酶切;2:a8质粒;3:a11 EcoRⅠ/ SalⅠ双酶切;4:a11质粒图4 克隆载体PMD18-T-amoA重组子双酶切验证
双酶切下来一条约3 000 bp和830 bp的条带,进一步说明目的基因连接到克隆载体.
2.4amoA基因的测序结果
送a8和a11测序,其中a8的基因测序结果如下,a11测序结果未显示.
GTATATTTAGAACGGAAGAGATCCTGAAAGC
GGCCAAGATGCCGCCGGAAGCGGTCCATATG
TCACGCCTGGTTGATGCGGTTTATTTTCCGAT
TCTGGTTGTTCTGTTGGTAGGTACCTACCATA
TGCATTTCATGTTGTTGGCAGGTGACTGGGAT
TTCTGGATGGACTGGAAAGATCGTCAATGGT
GGCCTGTAGTAACACCTATTGTGGGCATTACC
TATTGCTCGGCAATTATGTATTACCTGTGGGT
CAACTACCGTCAACCATTTGGTGCGACTCTG
TGCGTAGTGTGTTTGCTGATAGGTGAGTGGC
TGACACGTTACTGGGGTTTCTACTGGTGGTC
ACACTATCCACTCAATTTTGTAACCCCAGGTA
TCATGCTCCCGGGTGCATTGATGTTGGATTTC
ACAATGTATCTGACACGTAACTGGTTGGTGA
CTGCATTGGTTGGGGGTGGATTCTTTGGCCT
GATGTTTTACCCGGGTAACTGGCCAATCTTTG
GCCCGACCCATCTGCCAATCGTTGTAGAAGG
AACACTGTTGTCGATGGCTGACTACATGGGT
CACCTGTATGTTCGTACGGGTACACCTGAGTA
TGTTCGTCATATTGAACAAGGTTCATTACGTA
CCTTTGGTGGTCACACCACAGTTATTGCGGC
ATTCTTCGCTGCGTTTGTATCCATGCTGATGTT
TGCAGTCTGGTGGTATCTTGGAAAAGTTTAC
TGCACAGCCTTCTTCTACGTTAAAGGTAAAA
GAGGACGTATCGTACAGCGCAATGATGTTAC
GGCATTTGGTGAAGAAGGGTTTCCAGAGGG
GATCAAATAA
2.5amoA基因在NCBI的同源性比对结果
a8序列通过NCBI在线blast分析,与a8序列同源比对后相似较高的基因序列见图5所示.
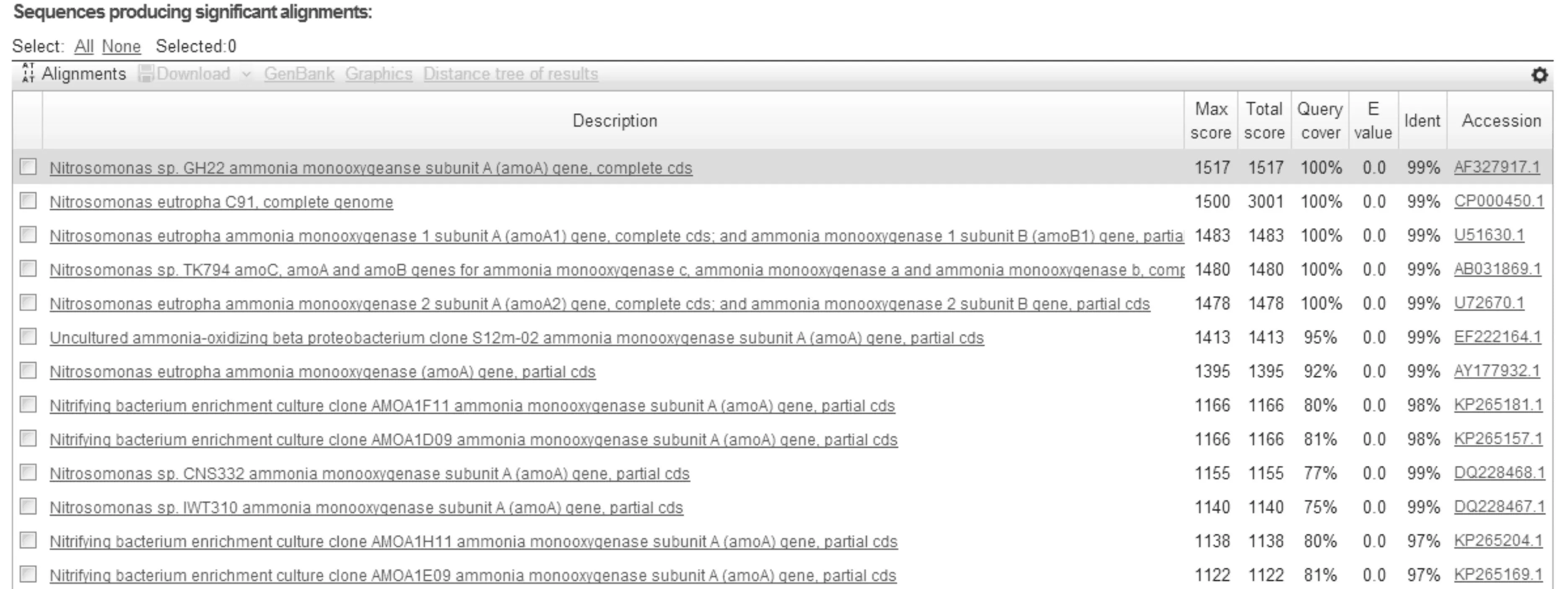
图5 a8序列在NCBI的同源性比对结果
2.6amoA基因进化树的构建
挑选序列同源相似性较高的序列使用MEGA6.0做进化树分析[15,16],见图6,分析结果说明扩增到的amoA基因确实是氨氧化细菌的amoA基因.
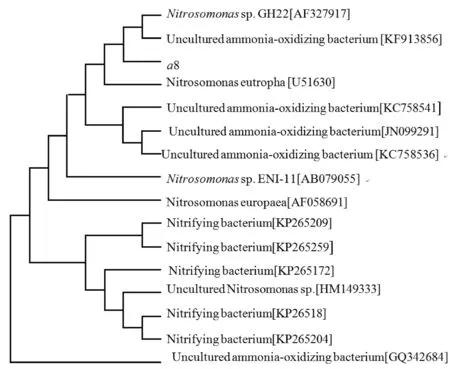
图6 a8序列进化树分析
2.7讨论
从富集土壤样品扩增得到的amoA基因有827 bp,从图5可看出在NCBI通过同源比对后,得到a8序列与Nitrosomonassp.GH22序列同源相似性达到99%;由图3可见,用M13通用引物进行菌落PCR验证,在830 bp附近有清晰地目标条带;双酶切下来两个条带,由图4可看出,一段约3 000 bp,为克隆载体的序列长度,一段约830 bp,为目的基因amoA序列长度,进一步证明了目的基因的正确性.
选择PMD18-T,带有Amp抗性,是一种高效连接的克隆载体,可以在30 min内与目标片段相连,转化的宿主菌选择E.coli DH5α,带有乳糖操纵子的调控区和连接目标片段的质粒在含有IPTG作为诱导物,以X-Gal为显色剂的固体培养基上,由于乳糖操纵子中的LacZ的释放的产物半乳糖苷酶,切割显色剂使吲哚基产生蓝色,被破坏的Lac菌株则产生白色菌落[16].因此把外源基因插入Lac的质粒上,就可通过菌落的颜色进行筛选.
3结束语
本研究无须长时间的分离培养纯种的氨氧化细菌,节省工作时间,通过富集土样后直接提取全基因组进行PCR扩增amoA基因,并且连接克隆载体进行测序,双酶切的基因可以进一步用于后续表达载体的构建,为构建新型的生物脱氮基因工程菌提供了依据.
参考文献
[1] 吴婉娥,葛红光,张克峰.废水生物处理技术[M].北京:化学工业出版社,2003:167-168.
[2] 吕锡武,李峰,稻森悠平,等.氨氮废水处理过程中的好氧反硝化研究[J].中国给水排水,2000,26(4):17-20.
[3] 郑兴灿,李亚新.污水除磷脱氮技术[M].北京:中国建筑工业出版社,1998:11-19.
[4] 郑平,徐向阳,胡宝兰.新型生物脱氮理论与技术[M].北京:科学出版社,2004.
[5] Klotz M G,Alzerreca J,Norton J M.A gene encoding a membrance protein exists upstream of the amoA/amoB genes in ammonia oxidizing bacteria:A third member of the amo operon[J].FEMS Microbiology Letters,1997,150:65-73.
[6] Rotthauwe J H,Deboer W,Liesack W.Comparative analysis of gene sequences encoding ammonia monooxygenase of Nitrosospira sp.AHB1 and Nitrosolobus multiformis C-71[J].FEMS Microbiology Letters,1995,133(1-2):131-135.
[7] 孙栋,唐莉丽,王倩倩,等.高盐极端环境土壤基因组DNA的分离纯化方法研究及基因文库的构建[J].广西农业生物科学,2006,25(1):24-29.
[8] 廖雪义,蓝荣.亚硝化细菌的分离及初步鉴定[J].生物技术,2008,18(6):50-52.
[9] 郭爱莲,李振海,黄淑菊.硝化细菌的分离研究[J].西北大学学报(自然科学版),1996,26(1):83-86.
[10] 张辉,李培军,胡筱敏,等.亚硝化细菌的筛选及培养条件的研究[J].化工环保,2006,26(5):366-369.
[11] McTavish H,Hopper A B,Fuchs J A.Sequence of the gene coding for ammonia monooxygenase in Nitrosomonas europaea[J].Journal of Bacteriology,1993,175:2 436-2 444.
[12] 陈春兰,陈哲,朱亦君,等.水稻土细菌硝化作用基因(amoA)和(hao)多样性组成与长期稻草还田的关系研究[J].环境科学,2010,31(6):1 624-1 632.
[13] 李君文,周娟,王新为,等.亚硝酸细菌amoA基因的克隆、测序与表达[J].应用与环境生物学报,2004,10(3):345-348.
[14] 杨坤,巩振辉,李大伟.大肠杆菌高效感受态细胞的制备及快捷转化体系的建立[J].北方园艺,2010,34(14):127-130.
[15] 黄建军,简纪常,吴灶和,等.一株海水氨氧化细菌的系统发育分析[J].广东海洋大学学报,2008,28(3):88-92.
[16] 陈岭.氨单加氧酶基因(amoA)在氨氧化细菌种群分析和定量检测中的应用研究[D].杭州:浙江大学,2003.
To clone the ammonia monooxygenase gene of autotrophic
bacteria ammonium oxidation capacity
GONG Guo-li, ZHANG Tian, SHI Zheng-hao, WEI Xuan-ming, WANG Lei
(School of Food and Biological Engineering, Shaanxi University of Science & Technology, Xi′an 710021, China)
Abstract:Ammonia oxidizing bacteria is a class of gram negative chemoautotrophic bacteria,and it is also a kind of indispensable in the process of biological removal of nitrogen.In this study,the amoA gene was successfully amplified from the whole genome of the enriched soil sample,then put this amoA gene sequence blasted in Genbank through Internet online;at last,we get a result that it has high homology with the amoA gene of Nitrosomonas sp. GH22(99%).According to the amoA full-length gene constructed a cloned vector,through the identification of colony PCR and restriction analysis were correct,to lay the foundation for the later construction of a new biological nitrogen removal genetically engineered bacteria.
Key words:ammonia oxidizing bacteria; biological nitrogen removal; identify
中图分类号:R285.5
文献标志码:A
文章编号:1000-5811(2015)05-0125-05
作者简介:龚国利(1976-),男,内蒙古丰镇人,教授,博士,研究方向:微生物发酵
基金项目:国家自然科学基金项目(20906085); 陕西科技大学学术骨干培育计划项目(XSG2010009)
收稿日期:*2015-05-19
