1株来自海洋的芽胞杆菌dhs-330转座突变库的构建及表面活性相关基因的克隆
2015-12-26张晓青王建艳郝建安张雨山
杜 瑾, 张晓青, 王建艳, 郝建安, 王 静, 张雨山
(国家海洋局 天津海水淡化与综合利用研究所,天津 300192)
1株来自海洋的芽胞杆菌dhs-330转座突变库的构建及表面活性相关基因的克隆
杜 瑾, 张晓青, 王建艳, 郝建安, 王 静*, 张雨山
(国家海洋局 天津海水淡化与综合利用研究所,天津 300192)
为研究产生物表面活性剂的海洋芽胞杆菌dhs-330合成活性产物的分子机制,应用质粒pIC333介导的mini-Tn10转座子随机突变技术,构建了芽胞杆菌dhs-330的突变体库。通过表面活性测定和反向PCR克隆,从300个突变株中筛选出产表面活性剂水平提高的突变株2株,分别在ycsG和yvkC基因发生插入突变;表面活性降低的突变株4株,分别在fenC、yrkF、kinE和sigD基因发生插入突变。这些基因可能与芽胞杆菌dhs-330中表面活性剂的合成代谢和调控有关。
转座突变;芽胞杆菌;生物表面活性剂
生物表面活性剂是微生物代谢合成的两亲化合物,不仅具有高效的起泡性和良好的破乳性,而且与化学合成的表面活性剂相比,具有无毒、易被生物降解、化学结构多样等优势,在化工、医药、农业和环境修复等领域具有良好的应用价值[1]。芽胞杆菌(Bacillussp.)是重要的生物表面活性剂产生菌,可合成脂肽类或糖脂类的表面活性剂。脂肽是由脂肪酸链与一个线状或环状的短肽相连而构成,不仅具有很强的表面活性,还具有抗菌性、抗肿瘤等生物活性[2-3]。1968年Arima等首次发现的BacillussubtilisIFO3039产生脂肽类物质,商品名为表面活性素Surfactin,是已知最有效的生物表面活性剂之一。芽胞杆菌还可合成丰原素(fengycin)、伊枯草菌素(iturin)、地衣素(lichenysin)等多种类型的脂肽类生物表面活性剂。糖脂(如鼠李糖脂、槐糖脂等)由亲水性糖基和疏水性脂肪酸链组成,具有突出的乳化性和增溶性,较好的洗油效率,在提高原油采收率[4]和修复海上溢油污染[5]等方面取得了很好的应用效果。Christova等[6]首次报道了1株具有鼠李糖脂合成能力的B.subtilis,并能够降解烃类物质。Cheng等[7]从胜利油田地下废水中分离的B.subtilisTU2能够同时生成脂肽和鼠李糖脂混合生物表面活性剂。然而,不同芽胞杆菌菌株中合成不同类型生物表面活性剂的代谢途径各异,相关代谢途径受到群体感应、环境因素等条件的调控,其分子机制和遗传背景十分复杂。因此,对相关功能基因和调控机制的解析已成为当前生物表面活性剂的研究热点[8]。海洋环境多变的温度、pH、盐度及局部污染等不利的自然条件,使海洋环境微生物往往具备优越的抗逆性、适应性,已成为人们筛选资源微生物的热点[9]。特别是在筛选产表面活性物质的海洋微生物方面,已发现的菌株包括芽胞杆菌、假单胞菌、红球菌、酵母菌等多个菌属[10],涉及的活性成分包括了脂肽类、糖脂类、胞外多糖等多种类型。本课题组前期的研究从渤海湾海水样品中筛选获得了1株产生物表面活性剂的芽胞杆菌dhs-330,本研究利用mini-Tn10转座系统构建dhs-330的基因突变库,筛选产表面活性剂性能显著变化的突变株并克隆其突变基因,为进一步阐明芽胞杆菌产表面活性剂的分子机制和遗传背景奠定基础。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒 芽胞杆菌(Bacillussp.)dhs-330由本课题组保存;大肠埃希菌(Escherichiacoli)DH5α购自北京博迈德科技发展有限公司;质粒pIC333由美国哈佛医学院Roberto Kolter教授惠赠。
1.1.2 培养基和溶液 ①M9-YE培养基:酵母提取物3 g,酸水解干酪素10 g,200 g/L葡萄糖溶液10 mL,1 mol/L MgSO42 mL,1 mol/L CaCl20.1 mL,5×M9盐溶液200 mL,定容至1 L并调整pH至7. 0。其中,5×M9盐溶液:Na2PO4·7H2O 64 g/L,KH2PO415 g/L,NaCl 2.5 g/L,NH4Cl 5 g/L。②LB培养基:蛋白胨10 g,酵母提取物5 g,NaCl 10 g,定容至1 L并调整pH至7. 0。固体培养基中添加琼脂粉2 g。筛选培养基中抗生素终浓度为红霉素1 μg/L,壮观霉素100 μg/L。③发酵培养基:葡萄糖31.5 g,酵母提取物1 g,尿素5 g,KH2PO40.8 g,Na2HPO4·12H2O 3 g,MgSO4·7H2O 0.15 g,微量元素溶液1 mL,定容至1 L并调整pH至7.0。其中,微量元素溶液:CaCl27.5 g/L,FeSO4·7H2O 2 g/L,MnSO4·H2O 2 g/L。④血琼脂平板培养基:购自天津润泰科技发展有限公司。⑤EP溶液:蔗糖0.625 mol/L,MgCl21 mmol/L。
1.1.3 试剂 细菌基因组提取试剂盒、质粒提取试剂盒、DNA产物纯化试剂盒均为天根生化科技有限公司生产;T4 DNA连接酶、限制性内切酶、DNA Marker为宝生物工程有限公司生产;壮观霉素和红霉素为Amresco公司生产;其他试剂为国产分析纯产品。
1.2 方法
1.2.1 感受态细胞制备及质粒转化 将LB平板划线活化的dhs-330单菌落接种于5 mL含0.4%甘氨酸的M9-YE培养基中,30 ℃、180 r/min振荡培养过夜;按照1%接种于50 mL含0. 4%甘氨酸的M9-YE培养基中,30 ℃、200 r/min振荡培养至OD600值达到0.4;4 ℃、5 000 r/min离心5 min收集菌体,用预冷的EP溶液洗涤细胞3次,最后一次用1/50初始培养物体积的预冷EP溶液重悬细胞,制成dhs-330的感受态细胞。每100 μL感受态细胞中加入10 μL脱盐后的质粒pIC333(约100 ng),轻轻混匀后加入到预冷的0.2 cm电转杯中;在1.5 kV电压、25 μF电容和200 Ω电阻条件下进行电击后,迅速加入900 μL 30 ℃预热的无抗M9-YE培养基;30 ℃、160 r/min复苏1 h,取100 μL培养物涂布于含红霉素和壮观霉素的双抗性LB固体平板上,30 ℃培养18 h获得转化子。
1.2.2 转座突变库的构建 挑取新鲜的dhs-330-pIC333转化子接种于含1 μg/L红霉素的3 mL LB液体培养基中,30 ℃、180 r/min振荡培养18 h;按照1%转接于3 mL不含抗生素的LB液体培养基中,30 ℃、200 r/min振荡培养3 h;再按照1%转接于3 mL无抗LB液体培养基中,42 ℃、200 r/min振荡培养4 h;培养物稀释涂布于含壮观霉素的LB固体平板上,30 ℃培养24 h;挑取单菌落分别点在互相对应位置的红霉素和壮观霉素抗性平板上,30 ℃培养24 h后,选择具有壮观霉素抗性且无红霉素抗性的单菌落即为突变子,获得dhs-330转座突变库。
1.2.3 转化子和转座突变株的检测 根据质粒pIC333中红霉素抗性基因、转座插入序列中的壮观霉素抗性基因分别设计引物:ErmF: 5′-ATGAACGAGAAAAATATAAAACAC-3′;ErmR: 5′-TTACTTATTAAATAATTTATAGCTATT-3′;SpcF: 5′-GCATTAATGAATCGGCCAACG-3′;SpcR: 5′-GTGGGTAAACCGTGAATATCG-3′。分别以转化子中的质粒、dhs-330基因组和突变子基因组DNA为模板进行PCR扩增,能够扩增出2.1 kb转座子片段、且不能扩增出750 bp红霉素抗性片段的转化子即为mini-Tn10插入突变子。PCR反应体系:10×Taqbuffer 2.5 μL,2.5 mmol/L dNTP 2 μL,上、下引物各1.5 μL,模板0.3 μL,Taq酶0.2 μL,ddH2O 17 μL,共25 μL。PCR反应条件:98 ℃ 2 min;98 ℃ 10 s、54 ℃ 30 s、72 ℃ 3 min进行30个循环;72 ℃延伸10 min,4 ℃保存。
1.2.4 产生物表面活性剂表型突变株的筛选 应用排油圈法对突变子库进行初筛,将突变子单菌落接种于20 mL发酵培养基中,30 ℃、180 r/min振荡培养24 h;取直径90 mm培养皿加入适量蒸馏水,在蒸馏水表面滴加3 mL液体石蜡形成油膜,取300 μL发酵培养液加入油膜中心,形成排油圈,测定排油圈的直径。比较表面张力值对突变株进行复筛,使用科诺全自动表面张力仪、采用白金板法测定发酵液稀释5倍后的表面张力值。
1.2.5 转座子插入位点的确定 提取突变株的基因组DNA,以Hind III对基因组进行酶切消化;酶切产物使用DNA产物纯化试剂盒纯化后自连,自连产物转化入大肠埃希菌DH5α感受态细胞中,涂布于含壮观霉素的LB固体平板筛选转化子。转化子提取质粒后,以引物Tn-F和Tn-R进行序列测定,在GenBank上进行Blast比对,确定mini-Tn10片段插入的基因位点。Tn-F:5′-GCCGCGTTGGCCGATTC-3′;Tn-R:5′-GATATTCACGGTTTAC-3′。引物合成及测序均由苏州金唯智生物科技有限公司完成。
2 结果与分析
2.1 海洋芽胞杆菌dhs-330突变体库的构建
本课题组前期从渤海湾海水样品中分离获得1株产生物表面活性剂的菌株,经初步鉴定为芽胞杆菌dhs-330。本研究利用携带转座子mini-Tn10的转座质粒pIC333成功转化进入该野生型菌株中,其优化的电转化条件为在添加0.4%甘氨酸的M9-YE培养基中培养dhs-330,当OD600达到0.4时收集菌体,用预冷的EP溶液洗涤细胞3次并悬浮菌体制成感受态细胞,每100 μL感受态细胞与100 ng pIC333质粒混合,在1.5 kV电压、25 μF电容和200 Ω电阻条件下进行电击转化,转化效率可达1.2×104cfu/μg。由于pIC333质粒为温度敏感型,转化子dhs-330-pIC333经42 ℃高温培养,可诱导转座子mini-Tn10随机插入dhs-330基因组,具有壮观霉素抗性且无红霉素抗性的菌株为转座突变株,初步获得300个突变株,转座效率约为80%。
在突变株中随机挑选3株以及出发菌株dhs-330基因组为模板,根据pIC333中红霉素抗性基因和插入序列中的壮观霉素抗性基因设计特异性引物,进行PCR扩增验证。电泳结果显示(图1),质粒pIC333可扩增到红霉素抗性片段(750 bp)和插入序列中的壮观霉素抗性片段(2.1 kb),dhs-330中2个片段都未扩增到;而3株突变株中仅扩增到插入序列(2.1 kb),未扩增到红霉素抗性片段,说明转座子mini-Tn10已成功插入dhs-330基因组中。
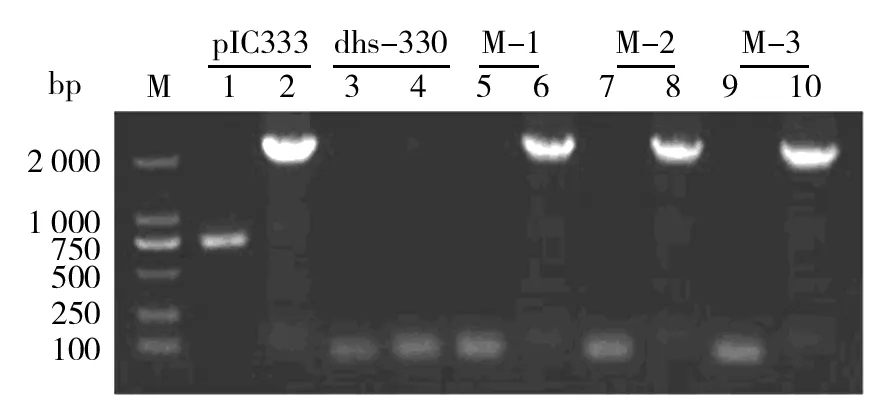
图1 转座子插入突变体的PCR鉴定Fig.1 PCR amplification of transposon insertion in mutants of Bacillus sp. dhs-330M:DL2 000;1、3、5、7、9:从pIC333质粒、dhs-330、3株随机突变子基因组中扩增红霉素抗性片段;2、4、6、8、10:相应菌株扩增的壮观霉素抗性基因M: DL2 000;PCR products of erythromycin resistance gene (1, 3, 5, 7,9) and spectinomycin resistance gene (2, 4, 6, 8,10) from pIC333, dhs-330, and 3 mutants, respectively
2.2 突变株特性及发酵液表面活性测定
测定dhs-330和突变株发酵液的排油圈大小,对菌株产生物表面活性剂的水平进行初筛;在300个突变株中,1-48和2-42发酵液排油圈分别达7.5 cm和7.0 cm,显著高于原始菌株dhs-330排油圈3.0 cm(如图2)。测定菌株发酵液稀释5倍时的表面张力,进行突变株的复筛;1-48和2-42可将发酵培养基的表面张力由70 mN/m降低至31.425 mN/m和32.783 mN/m,优于dhs-330的35.037 mN/m。经过初筛和复筛,还获得2-61、2-80、2-81和2-97共4株表面活性显著降低的负突变株(如图2),其排油圈分别为0.5、0.5、1.0和0.5 cm,显著低于原始菌株dhs-330。比较dhs-330和突变株的生长曲线表明,突变株较dhs-330的生长速率和最终生物量没有显著差异。观察菌株的形态及其在溶血平板上的溶血圈情况(如图3),可以看到正突变株1-48和2-42的溶血圈显著增大,负突变株2-61和2-80的溶血圈显著减小,而负突变株2-80、2-81和2-97较原始菌株dhs-330的菌落形态也有变化。
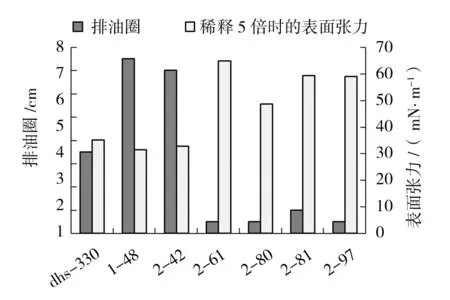
图2 dhs-330及突变株产表面活性剂水平比较Fig.2 Comparison of the biosurfactant production in dhs-330 and the mutants
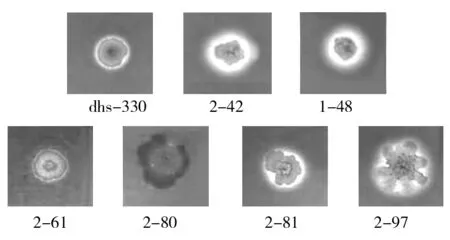
图3 dhs-330及突变株菌落形态及溶血圈Fig.3 Colonies with transparent circle of dhs-330 and the mutants on the blood plate
2.3 突变株中插入位点基因的反向PCR克隆
对2.2中产生物表面活性剂水平显著变化的6个突变株进行转座子mini-Tn10插入位点基因的反向PCR,对突变株基因组片段进行克隆和测序,根据测序结果进行Blast比对,结果见表1。其中所列基因与NCBI所公布的相应基因的同源性都在85%以上。
结果表明,产表面活性剂能力增强的突变株1-48中转座子插入位点的侧翼序列与枯草芽胞杆菌中ycsG基因同源性最高,该基因编码的YcsG与铜绿假单胞菌中的II型支链氨基酸转运系统BraB相似[11]。另一个正突变株2-42中的插入位点侧翼序列与枯草芽胞杆菌中yvkC基因同源性最高,该基因编码丙酮酸磷酸双激酶[12]。4株产表面活性剂能力降低的菌株中,突变株2-61中转座子插入位点位于fenC基因中,该基因编码肽单体酶FenC参与脂肽类物质fengycin的合成。突变株2-80中转座子插入位点为yrkF基因,编码硫氰酸酶YrkF。突变株2-81中转座子插入kinE基因,编码组氨酸激酶KinE;突变株2-97中插入突变的sigD基因编码sigma因子D,KinE与SigD均属于芽胞杆菌信号转导系统,参与调控芽胞杆菌中特定代谢途径。
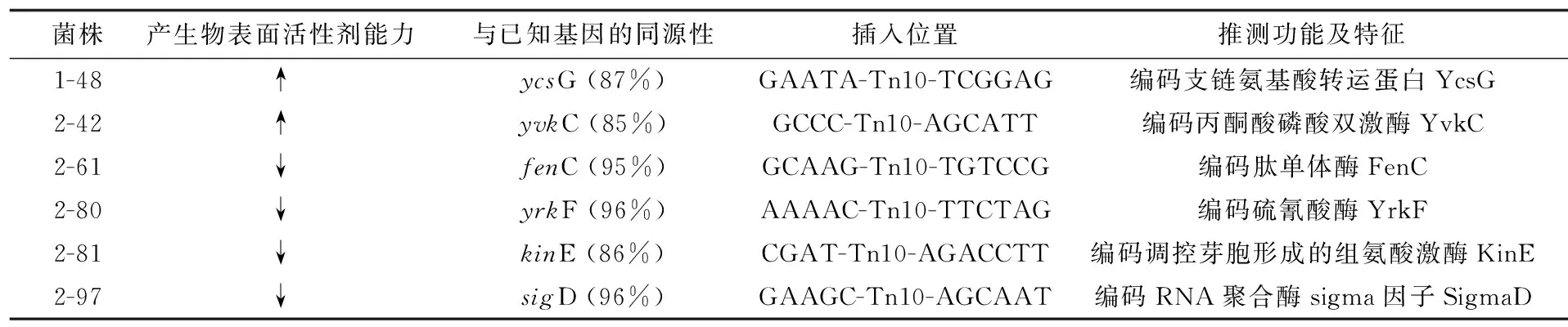
表1 转座子插入位点的基因
注:“↑”表示产生物表面活性剂能力上升;“↓”表示产生物表面活性剂能力下降
3 讨 论
携带转座元件mini-Tn10的穿梭质粒pIC333是芽胞杆菌属随机插入突变的重要工具,已成功应用于多种芽胞杆菌生理生化、代谢调控的功能基因研究,特别是在重要物质代谢途径及次级代谢产物相关调控基因的挖掘方面。如苏云金芽胞杆菌中生成对家蚕的毒力因子[13]、枯草芽胞杆菌中合成溶杆菌素[14]与核黄素[15]等活性物质的相关基因,都是通过pIC333介导构建的转座突变库筛选获得。本研究采用pIC333质粒构建了产生物表面活性剂菌株、海洋芽胞杆菌dhs-330的转座突变体库。
在成功构建dhs-330随机插入突变体库的基础上,本研究以突变株发酵液的排油圈为指标,从300个突变株中筛选到产表面活性剂显著变化的突变株6株,并确定了其基因组中转座子的插入位点。在4株表面活性剂合成能力显著降低的负突变株中,转座子分别插入突变了fenC、yrkF、kinE和sigD基因。其中,fenC是与表面活性剂合成直接相关的基因,其编码的肽单体酶FenC是fengycin合成的关键酶。Fengycin与surfactin、iturin是芽胞杆菌中3种常见的脂肽类产物,具有表面活性和抗菌活性;fengycin合成酶存在5个编码基因,依次为fenC、fenD、fenE、fenA和fenB,fengycin的合成(肽链延伸)过程是从FenC开始的[16]。kinE基因与sigD基因编码的调控因子,可能通过对生物表面活性剂合成途径的调控表达,从而影响活性产物的合成。组氨酸激酶KinE可感知细胞外信号,并转化为磷酸化信号启动芽胞调控蛋白Spo0A,Spo0A再调控下游SlrR/SlrA等途径,进而激活芽胞杆菌的芽胞生成和生物膜形成过程。SigmaD因子则可与SlrR/SlrA共同调控芽胞杆菌鞭毛生成、趋化运动等行为[12]。芽胞杆菌的运动行为和生物膜形成过程都需要合成表面活性物质辅助其进行,因此缺失kinE或sigD基因可能导致菌株启动表面活性剂合成途径的调控功能受阻,降低了活性产物合成水平。负突变株2-80发生插入突变的yrkF基因编码硫氰酸酶YrkF,参与含硫生物大分子的合成[17],但其对表面活性剂代谢合成的影响尚未见报道。
2株表面活性剂合成能力提高的正突变株中,插入位点分别位于ycsG和yvkC基因中。ycsG基因属于一种支链氨基酸的转运系统,在枯草芽胞杆菌基因组中一般以操纵子ycsFGI-kipIAR-ycsK的形式存在,受氮源调控并与产芽胞过程调控有关[11]。yvkC基因编码丙酮酸磷酸双激酶,参与糖酵解和丙酮酸代谢过程[12]。这些途径可能都参与了对表面活性剂合成的调控,这些基因的插入突变可能通过解除代谢抑制而使表面活性剂产量提高。本研究为利用芽胞杆菌合成生物表面活性剂的代谢调控、构建高产工程菌株等工作提供了参考。
[1] Banat IM, Franzetti A, Gandolfi I, et al. Microbial biosurfactants production, applications and future potential[J]. Appl Microbiol Biotechnol, 2010, 87(2): 427-444.
[2] Pirri G, Giuliani A, Nicoletto S, et al. Lipopeptides as anti-infectives: a practical perspective[J]. Cent Eur J Biol, 2009, 4(3): 258-273.
[3] Raaijmakers JM, De Bruijn I, Nybroe O, et al. Natural functions of lipopeptides fromBacillusandPseudomonas: more than surfactants and antibiotics[J]. FEMS Microbiol Rev, 2010, 34(6): 1037-1062.
[4] Wang Q, Fang X, Bai B, et al. Engineering bacteria for production of rhamnolipid as an agent for enhanced oil recovery [J]. Biotechnol Bioeng, 2007, 98(4): 842-853.
[5] Harvey S, Elashvili I, Valdes JJ, et al. Enhanced Removal of Exxon Valdez Spilled Oil from Alaskan Gravel by a Microbial Surfactant[J]. Nat Biotech, 1990, 8(3): 228-230.
[6] Christova N, Tuleva B, Nikolova-Damyanova B. Enhanced hydrocarbon biodegradation by a newly isolatedBacillussubtilisstrain[J]. Z NATURFORSCH C, 2004, 59(3/4): 205-208.
[7] Cheng FY, Yang H, Chen Y. Characterization of a blend-biosurfactant of glycolipid and lipopeptide produced byBacillussubtilisTU2 isolated from underground oil-extraction wastewater[J]. J Microbiol Biotech, 2013, 23(3): 390-396.
[8] Das P, Mukherjee S, Sen R. Genetic regulations of the biosynthesis of microbial surfactants: an overview[J]. Biotechnol Genet Eng Rev, 2008, 25: 165-185.
[9] Dash HR, Mangwani N, Chakraborty J, et al. Marine bacteria: potential candidates for enhanced bioremediation[J]. Appl Microbiol Biot, 2013, 97(2): 561-571.
[10]Satpute SK, Banat IM, Dhakephalkar PK, et al. Biosurfactants, bioemulsifiers and exopolysaccharides from marine microorganisms[J]. Biotechnol Adv, 2010, 28(4): 436-450.
[11]Wang L, Grau R, Perego M, et al. A novel histidine kinase inhibitor regulating development inBacillussubtilis[J]. Genes Dev, 1997, 11(19): 2569.
[12]Cozy LM, Phillips AM, Calvo RA, et al. SlrA/SinR/SlrR inhibits motility gene expression upstream of a hypersensitive and hysteretic switch at the level of σD inBacillussubtilis[J]. Mol Microbiol, 2012, 83(6): 1210-1228.
[13]Fedhila S, Guillemet E, Nel P, et al. Characterization of twoBacillusthuringiensisgenes identified byinvivoscreening of virulence factors[J]. Appl Environ Microbiol, 2004, 70(8): 4784-4791.
[14]Yazgan A, Ozcengiz G, Marahiel MA. Tn10 insertional mutations ofBacillussubtilisthat block the biosynthesis of bacilysin [J]. Biochim Biophys Acta, 2001, 1518(1-2): 87-94.
[16]Lin TP, Chen CL, Chang LK, et al. Functional and transcriptional analyses of a fengycin synthetase gene,fenC, fromBacillussubtilis[J]. J Bacteriol, 1999, 181(16): 5060-5067.
[17]Hunt J. Genetic and biochemical characterization of YrkF, a novel two-domain sulfurtransferase inBacillussubtilis[D]. Virginia Polytechnic Institute and State University, 2004.
Construction of Transposon Mutation Library from a Marine Bacillus sp. dhs-330 and The Cloning of Surface Activity Related Genes
DU Jin, ZHANG Xiao-qing, WANG Jian-yan, HAO Jian-an, WANG Jing, ZHANG Yu-shan
(Inst.ofSeawaterDesalinat'n&MultipurposeUtilizat'n,StateOc.Administ'n,Tianjin300192)
In order to study the molecular mechanism of bio-surfactant synthesis in a marine biosurfactant-producingBacillusdhs-330 strain, the dhs-330 mutation library was built via mini-Tn10 transposon random mutation technique mediated by plasmid pIC333 applied in this study. Through the measurement of surface activity and inverse PCR cloning, two strains that enhanced their production of surfactant were screened with insertion mutation onyesG andyvkC genes had happened respectively from 300 mutants; and four mutants that decreased their surface activity, with insertion mutation onfenC,yrkF,kinE andsigD genes had happened respectively. It was speculated that these genes may be related to the synthetic metabolism and regulation of biosurfactant inBacillussp. dhs-330.
transposon mutation;Bacillussp.; biosurfactant
国家自然科学基金青年项目(21406042);海洋公益性行业科研专项(201105026);海洋公益性行业科研专项(201305022-5)
杜瑾 女,工程师,博士。从事海水利用技术及合成生物学相关研究。Tel: 022-87898132, E-mail: dujin111@126.com
* 通讯作者。女,正高级工程师,硕士。从事海水利用技术及海洋化工相关研究。Tel: 022-87898178,E-mail: wang_nana@163.com
2014-12-08;
2015-02-12
Q75
A
1005-7021(2015)05-0079-06
10.3969/j.issn.1005-7021.2015.05.014