用rDNA-ITS方法鉴别内蒙古多种野生食用菌
2015-11-07刘晓婷郭九峰王淑妍李亚娇
刘晓婷 郭九峰王淑妍 李亚娇 那 日
(内蒙古大学物理科学与技术学院,内蒙古 呼和浩特 010021)
用rDNA-ITS方法鉴别内蒙古多种野生食用菌
刘晓婷 郭九峰*王淑妍 李亚娇 那 日
(内蒙古大学物理科学与技术学院,内蒙古 呼和浩特 010021)
采用rDNA-ITS分子标记对内蒙古地区市场11种常见野生食用菌,以2个已知长期人工栽培可食用菌种香菇和双孢蘑菇为比照进行分子鉴定;通过优化DNA提取方法及PCR反应条件,用通用引物 ITS1/ITS4扩增出650~800 bp的目的DNA片段,经 PCR 产物直接测序,然后与DNA 序列数据库中的信息资源进行比对,鉴定出11个样品分属于卷边网褶菌、香杏丽蘑、野蘑菇、马鞍菌、杨树口蘑、蒙古口蘑和白鳞蘑菇。利用DNAMAN8软件,分析13个材料rDNA中ITS区序列,做同源性矩阵,绘制NJ树,得出13种常见食用菌亲缘关系的结论。结果表明:rDNA-ITS分析方法可以有效地划分不同种属的菌种,可以区分出常见野生食用菌。
野生食用菌;DNA提取方法优化;rDNA-ITS分子鉴别;同源性分析;食品安全
1 引言
内蒙古地区野生食用菌资源丰富,野生食用菌在为人们提供丰富食物营养的同时,也为食用菌菌种市场混乱,假种、劣种充斥提供了条件,损害了生产者的利益。而误食“毒蘑菇”等事件的时有发生,也极大地影响了食用菌产业持续健康发展。为解决这一问题,食用菌的鉴别成为研究热点,相关报道逐年增加[1,2]。野生食用菌大多数属于担子菌门(Basidiomycoto),少数属于子囊菌门(Ascomycota)[3,4]。本试验涉及到的13种材料中,作为比照的香菇[5~7]和双孢蘑菇[8]为长期人工培育的食用菌,其他11种为内蒙古地区采集或购买的,多为人们常常食用但容易发生混淆引发食品安全问题的野生菌,有常见于报道的蒙古口蘑[9~11]、杨树口蘑[12,13]等。
传统的菌种鉴别以子实体形态、生长特性以及生理生化反应特征为依据,易受环境因素和生理状况的影响,对形态差异较小的种间及种内菌株难以鉴别。DNA分子标记技术具有形态学观察不可比拟的优越性,遗传物质不易受环境影响,而且在生活史任何阶段均可获得。通过DNA分子标记技术可以快速、有效地判别野生菌中的“相似蘑菇种”。近年来,DNA分子标记和应用分子标记鉴定食用菌菌种的技术得到了迅速发展[14~16],如rDNA-ITS方法、RAPD方法[17,18]、SSR方法[19,20]、AFLP[21~23]方法、SCAR方法[24]、SRAP方法[25,26]等。其中rDNA-ITS方法是目前采用较多的菌种鉴定方法,已有大量关于蒙古口蘑的鉴定报道,但未见对更多种的食用菌菌种鉴定的报道。
ITS(internal transcribed spacers)内部转录间隔区,包含2个不同的非编码区域的ITS1和ITS2。在ITS两侧的18S,5.8S,28S编码区具有非常保守的区域,便于设计引物[27,28]。由于真核生物核糖体DNA(rDNA)的重复片段中,ITS区进化速度较快且片段长度适中,该片段在基因组不同重复单元间十分一致。通过PCR扩增,克隆测序,最后利用测序结果来分析物种ITS区同源性关系,则可以找到物种种间相关系统进化关系。使得ITS成为高等真菌种间及种群系统性研究的重要核基因标记,具有很高的应用价值[29~31]。
2 试验材料与方法
2.1 供试材料
11种野生菌鉴别材料均采自或市购于内蒙古地区,编号为1~11号。其俗名,1~5号分别为油蘑、口蘑、黄盖子、黄蘑、白蘑;6~8号为黑里子;9~11号分别为地木耳、白蘑、黑蘑。另两个比照菌株香菇、双孢蘑菇编号为12、13。
2.2 样品处理
将供试菌的子实体置于烘干箱内,45 ℃烘干24 h。然后分别置于微量植物样品粉碎机内粉碎,分装于离心管内,标号1~13。置于4 ℃冰箱储存备用。
2.3 DNA提取与优化
(1)药品试剂。TIANGEN植物基因组DNA提取试剂盒,氯仿,乙醇。
(2)仪器设备。低温离心机,ABI梯度PCR仪,BIO-RAD电泳仪,超微量分光光度计,恒温水浴箱等。
(3) DNA提取操作步骤。①取事先处理好的食用菌样品约30 mg。②迅速转移到预先装有700 μL,65 ℃预热缓冲液GP1的离心管中,经迅速颠倒混匀后,将离心管65 ℃水浴20 min,水浴过程中颠倒离心管数次,以混合样品。③加入700 μL氯仿,充分混匀,12 000 rpm离心5 min。④小心地将上一步所得上层水相转入一个新的离心管中,加入700 μL缓冲液GP2,充分混匀。⑤将混匀的液体取700 μL转入吸附柱CB3中,12 000 rpm离心30 s,弃掉废液。⑥向吸附柱CB3中加入500 μL缓冲液GD(使用前已加入无水乙醇),12 000 rpm离心30 s,倒掉废液,将吸附柱CB3放入收集管中。⑦向吸附柱CB3中加入600 μL漂洗液PW(使用前已加入无水乙醇),12 000 rpm离心30 s,倒掉废液,将吸附柱CB3放入收集管中。⑧重复操作步骤⑦。⑨将吸附柱CB3放回收集管中,12 000 rpm离心2 min,倒掉废液。将吸附柱CB3置于室温下放置2~5 min,以彻底晾干吸附材料中残余的漂洗液。⑩将吸附柱CB3转入一个干净的离心管中,向吸附膜的中间部位悬空滴加30~50 μL纯水,室温放置2~5 min,12 000 rpm离心2 min。⑪将收集到离心管中的溶液重新滴加到吸附膜上,12 000 rpm离心2 min再次洗脱,将溶液收集到离心管中。⑫用微量分光光度计检测提取的DNA溶液,并记录。测量完毕后,置于4 ℃冰箱中短期保存。
2.4 ITS PCR体系建立
采用ITS通用引物ITS1和ITS4,碱基序列(5’-3’):ITS1,TCCGTAGGTGAACCTGCGG;ITS4,TCCTCCGCTTATTGATATGC。PCR反应体系为20 μL反应体系:Mix 10 μL,引物各1 μL,DNA模板2 μL,ddH20 6 μL。PCR反应程序:94 ℃预变性3 min;94 ℃变性40 s,45 ℃退火1 min,72 ℃延伸1 min,30个循环;72 ℃继续延伸10 min,4 ℃保存。电泳条件为:1.5%琼脂糖凝胶,电压100 V。
2.5 测序
将上步所得的1~13号DNA样品PCR扩增产物直接送有关公司测序,测序引物为PCR引物。
2.6 序列比对
(1)测序结果分析。NCBI在线BLAST,在GENBANK上分别输入13条测序结果进行BLASTn,在基因库中寻找与本次测序序列相似度高的序列,确定所鉴别的食用菌名称和相关菌种信息。根据测序图像与网上比对人工校对序列。
(2)运用DNAMAN软件序列分析。将校对好的13条DNA序列载入DNMMAN8软件,进行相关生物信息学分析。
2.7 生物信息学分析
利用DNAMAN8软件,比较载入的13个菌种rDNA-ITS序列,列出同源性矩阵,绘制同源树,读图,找到13个菌种之间的同源关系。
3 结果与分析
3.1 DNA提取优化
优化的DNA提取方法是将TIANGEN植物基因组DNA提取试剂盒提取植物基因组DNA的方法改进为:提取过程中省略说明书中加巯基乙醇的步骤,洗脱过程中用50 μL纯水代替说明书中50 μL洗脱缓冲液TE洗脱两次。
试验发现,对于干燥的真菌组织,是否加巯基乙醇对最后得到DNA的含量和质量没有明显影响,所以省略了此步。除此之外,对于洗脱过程,用洗脱缓冲液TE洗脱的效果明显不如纯水。改用纯水后,DNA含量明显增高,A260/A280值也回归在正常的1.7~1.9之间。
用微量分光光度计检测DNA提取结果可见,所提取的1~13号食用菌DNA含量均值在18.7~121 ng/μL,含量相对较高;A260/A280值均在1.7~1.9,属正常范围(表1)。
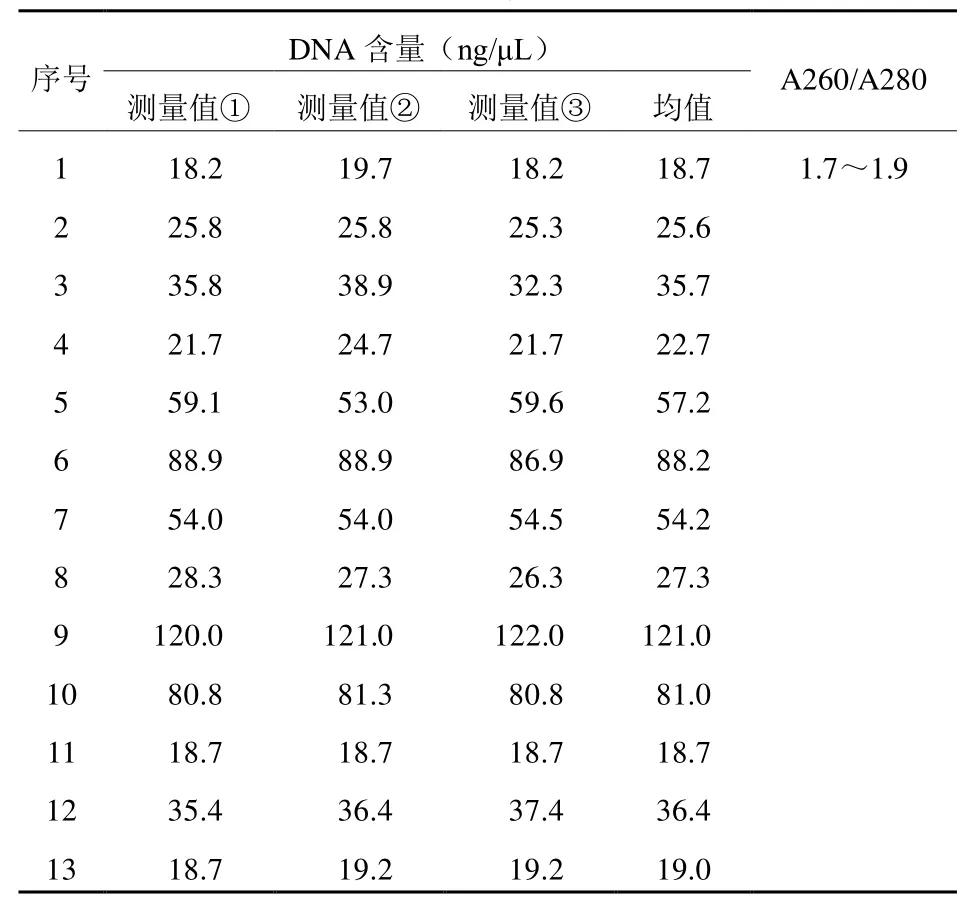
表1 DNA含量测定结果
3.2 ITS扩增结果
用通用引物ITS1和ITS4进行PCR扩增以后,将1~13号菌种样品扩增产物加入1.5%琼脂糖凝胶中,电泳35 min,紫外光下检测并拍照。13个常见食用菌菌种rDNA-ITS扩增产物为650~800 bp长度条带。7号、8号、11号条带长度比较一致,5号、10号条带长度比较一致,2号、4号条带长度比较一致,剩余的1号、3号、6号、9号、12号、13号也拥有各自长度的条带(图1)。可见,用rDNA-ITS方法可以有效地鉴别这13种常见食用菌。
2.3 鉴定结果
通过对13个菌种rDNA-ITS扩增产物测序,将序列数据进行网上BLAST,成功分辨出13份材料。1~13号菌种分别是:卷边网褶菌、杨树口蘑、香杏丽蘑、杨树口蘑、蒙古口蘑、野蘑菇、白鳞蘑菇、白鳞蘑菇、马鞍菌、蒙古口蘑、白鳞蘑菇、香菇、双孢蘑菇[32](表2)。
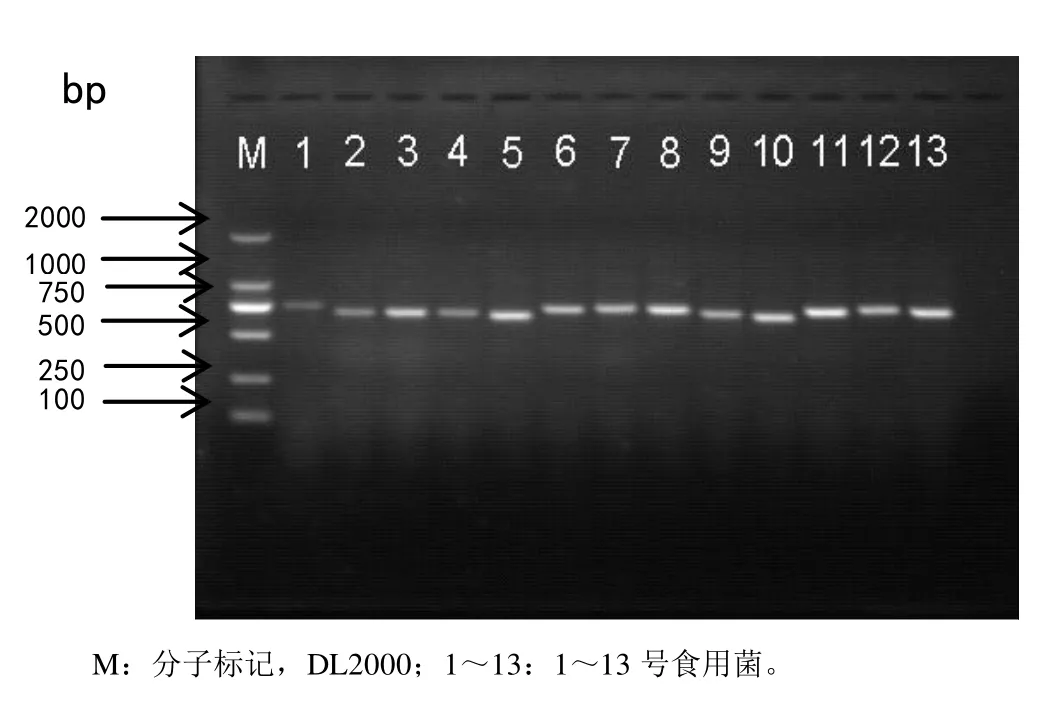
图1 供测菌种ITS序列PCR结果
3.4 同源树的建立及结果分析
通过对13个菌种rDNA-ITS扩增产物测序,用DNAMAN8软件对13条测序结果进行同源性分析,得到同源性矩阵,并做出NJ树(图2)。
不同的菌种间相似性的百分数有所不同。7号、8号、11号3种白鳞蘑菇,5号、10号2种蒙古口蘑,2号、4号2种杨树口蘑的相似性百分数在88.4%~100.0%,表现出较大的相似性。其他几种食用菌,1号卷边网褶菌,3号香杏丽蘑,6号野蘑菇,9号马鞍菌,12号香菇,13号双孢蘑菇两两之间的相似性百分数虽较低,但也在51.0%以上。可见13种常见食用菌,采用该方法可以有效地加以区分。
从NJ树中可见,属于子囊菌门的9号,最早与其他属于担子菌门的12个菌种得以明显分开;属于网褶菌科的1号,属于光茸菌科的12号,先后与其他属于口蘑科和蘑菇科的菌种也能分开;其中,口蘑属2号、4号和5号、10号聚为一支,再与丽蘑属3号聚在一起成为一簇,共属于口蘑科;蘑菇属8号、11号、13号与6号聚为一支,再与7号聚在一起成为一簇,共属于蘑菇科。总体来说,NJ树可以清晰辨别不同种属食用菌亲缘关系的远近,为这几种常见食用菌提供快速、有效的鉴别方法。
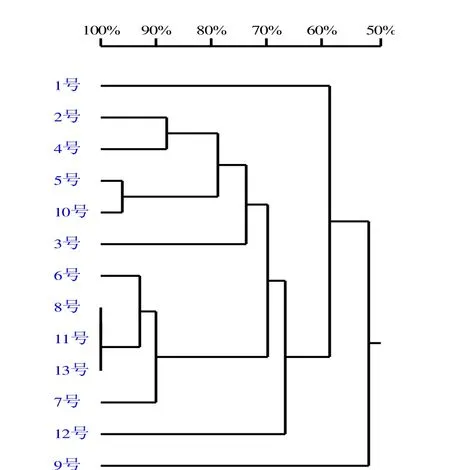
图2 NJ树
4 结论与讨论
4.1 结 论
从以上结果,可得出以下结论。
(1)TIANGEN植物基因组DNA提取试剂盒的操作步骤经过适当改进,可以用于高质量地提取市售蘑菇子实体DNA。
(2)通过rDNA-ITS序列分析,成功鉴别内蒙古地区13种不同科属常见食用菌,从而初步确立了快速、有效的鉴定技术。
(3)通过使用DNAMAN8软件对13条测序结果进行同源性分析,得到同源性矩阵,做出NJ树,明确了各种食用菌的亲缘关系。
(4)通过rDNA-ITS分子标记技术,可以快速、有效地辨别外形相似的几种野生菌,食用菌,特别是蘑菇市场销售产品的真假混乱等问题可以得到有效解决。
4.2 讨 论
(1)DNA提取方法优化。在改进TIANGEN植物基因组DNA提取试剂盒提取植物基因组DNA的方法试验中发现,30 mg食用菌风干样足够用以提取DNA,提取过程中可以省略加巯基乙醇的步骤,通过在洗脱过程中将50 μL洗脱缓冲液TE改用50 μL纯水代替,并洗脱两次,可以获得高含量、高质量的DNA。
操作过程中,需要注意的是:水浴过程中颠倒离心管以混合样品的动作要温和,颠倒次数越多混合越充分,DNA提取量也越高;在加入氯仿混匀取上层水相的步骤中,从离心机中拿取离心管要小心谨慎,这个步骤将直接影响提取的DNA溶液质量;在纯水洗脱的步骤中,要将50 μL纯水加到吸附柱底面的中部,避免纯水粘壁,且不能与DNA接触,影响洗脱效果,须静置2~5 min,使纯水充分溶解DNA,将其洗脱下来。
所提取的1~13号食用菌DNA含量均值在18.7~121 ng/μL,其中1号卷边网褶菌,11号白鳞蘑菇提取的DNA含量相对较低,9号马鞍菌较高,这可能与不同种蘑菇的内部性质有关。总体来说,通过改进DNA提取方法,整体水平上DNA含量、质量明显提高,达到预期的效果。
(2)rDNA-ITS分子鉴别。本试验通过测定常见野生食用菌菌株的rDNA中ITS区序列来探讨各个菌种的同源序列,通过rDNA-ITS序列分析,成功鉴别了内蒙古地区13种常见食用菌菌种,初步确立了快速、有效的菌种鉴定技术,初步探明rDNA-ITS方法是一种快速、有效、准确的鉴别常见食用菌的分子鉴定方法,也为今后食用菌的鉴别等相关研究提供一定参考。

表2 野生菌鉴定结果
[1] Das S. K., Mandal A., Datta A. K., et al. Nucleotide Sequencing and Identification of Some Wild Mushrooms[J]. Sci World J, 2013.
[2] 吴素蕊, 罗晓莉, 刘蓓, 等. 野生食用菌研究开发浅析及建议[J]. 食品科技, 2010(4): 100-103.
[3] S. Rajaratnam, T. Thiagarajan. Molecular characterization of wild mushroom[J]. European Journal of Experimental Biology, 2012, 2(2): 369-373.
[4] 李玉. 野生食用菌菌种分离与鉴定[D]. 福建农林大学, 2008.
[5] 秦莲花, 宋春艳, 谭琦, 等. 用ITS和ISSR分子标记技术鉴别香菇生产用种[J]. 菌物学报, 2006, 25(1): 94-100.
[6] Tanaka A., Miyazaki K., Murakami H., et al. Sequence characterized amplified region markers tightly linked to the mating factors of Lentinula edodes[J]. Genome, 2004, 47(1): 156-162.
[7] Mukhopadhyay K., Haque I., Bandopadhyay R., et al. AFLP based assessment of genetic relationships among shiitake (Lentinula ssp.) mushrooms[J]. Molecular Biology Reports, 2012, 39(5): 6059-6065.
[8] 许广波, 傅伟杰, 魏铁铮, 等. 双孢蘑菇的栽培现状及其研究进展[J]. 延边大学农学学报, 2001(1): 69-72.
[9] 张智毓, 姚庆智, 闫伟. 草原珍稀食用菌蒙古口蘑菌种的分子鉴定[J]. 生物技术, 2011(3): 40-43.
[10] 刘芳, 朱兵, 于景丽, 等. 分子标记对草原野生食用菌蒙古口蘑的鉴别分析[J]. 生物技术通报, 2010(6): 124-129.
[11] 吴恩奇, 图力古尔. 蒙古口蘑研究进展[J]. 中国食用菌, 2007, 26(4): 3-5.
[12] 卯晓岚. 介绍一种野生食菌——杨树口蘑[J]. 中国食用菌, 1987(6): 2.
[13] Moukha S., Ferandon C., Beroard E., et al. A molecular contribution to the assessment of the Tricholoma equestre species complex[J]. Fungal Biology, 2004, 117(2): 145-155.
[14] 吴学谦, 李海波, 魏海龙,等. DNA分子标记技术在食用菌研究中的应用及进展[J]. 浙江林业科技, 2004(2): 76-81.
[15] 黄映萍. DNA分子标记研究进展[J]. 中山大学研究生学刊(自然科学.医学版), 2010(2): 27-36.
[16] 刘华晶. 基于不同分子标记的黑龙江省野生黑木耳遗传多样性分析[D]. 东北林业大学, 2011.
[17] 邓旺秋, 杨小兵, 李泰辉, 等. 广东省草菇主要栽培菌株RAPD多态性分析[J]. 食用菌学报, 2004(3): 1-6.
[18] G. G. Fonseca, E. A. Gandra, L. F. Sclowitz, et al. Oyster mushrooms species differentiation through molecular markers RAPD[J]. International Journal of Plant Breeding and Genetics, 2008(2): 13-18.
[19] 王莹, 陈明杰, 汪虹, 等. 美味牛肝菌全基因组SSR位点的分布规律研究[J]. 菌物学报, 2015, 02: 204-214.
[20] H. S. Ro, S. S. Kim, J. S. Ryu, et al. Comparative studies on the diversity of the edible mushroom Pleurotus eryngii: ITS sequence analysis, RAPD fingerprinting, and physiological characteristics[J]. Mycological Research, 2007, 111(6): 710-715.
[21] K. H. Ma, G. A. Lee, S. Y. Lee, et al. Development and characterization of new microsatellite markers for the oyster mushroom (Pleurotus ostreatus)[J]. Journal of Microbiology and Biotechnology, 2009, 19(9): 851-857.
[22] A. Pawlik, G. Janusz, J. Koszerny, et al. Genetic diversity of the edible mushroom Pleurotus sp. By amplified fragment length polymorphism[J]. Current Microbiology, 2012, 65(4): 438-445.
[23] 许晓燕, 余梦瑶, 罗霞, 等. 利用AFLP和SRAP标记分析19株毛木耳的遗传多样性[J]. 西南农业学报, 2008(1): 121-124.
[24] 吴学谦, 李海波, 魏海龙, 等. SCAR分子标记技术在香菇菌株鉴定上的应用研究[J]. 菌物学报, 2005(2): 259-266.
[25] 朱坚, 高巍, 林伯德, 等. 金针菇种质资源的SRAP分析[J]. 福建农林大学学报(自然科学版), 2007(2): 154-158.
[26] 王守现, 刘宇, 耿小丽, 等. 应用SRAP标记对六个鸡腿菇菌株的多态性分析[J]. 江西农业大学学报, 2006(5): 753-757.
[27] 陈剑山, 郑服丛. ITS序列分析在真菌分类鉴定中的应用[J]. 安徽农业科学, 2007, 13: 3785-3786, 3792.
[28] 白树猛, 田黎. ITS序列分析在真菌分类鉴定和分子检测中的应用[J]. 畜牧与饲料科学, 2009, 30(1): 52-53.
[29] Conrad L. Schoch, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(16): 6241-6246.
[30] Hortal S., Pera J., et al. Molecular identification of the edible ectomycorrhizal fungus Lactarius deliciosus in the symbiotic and extraradical mycelium stages[J]. JOURNAL OF BIOTECHNOLOGY, 2006, 126(2): 123-134.
[31] Avin F. A., Bhassu S., Tan Y. S., et al. Molecular divergence and species delimitation of the cultivated oyster mushroom: integration of IGS1 and ITS[J]. Sci World J, 2014.
[32] 戴玉成, 周丽伟, 杨祝良, 等. 中国食用菌名录[J]. 菌物学报, 2010, 29(1): 1-21.
Identification of several edible mushrooms in Inner Mongolia by rDNA-ITS
Liu Xiaoting Guo Jiufeng* Wang Shuyan Li Yajiao Na Ri
(College of Physical Science and Technology, Inner Mongolia University, Huhhot 010021)
In this study, 11 common edible wild mushrooms from Inner Mongolia market were identified by rDNA-ITS molecular markers, and two known strains, Lentinula edodes (Berk.) Pegler and Agaricus bisporus (J.E.Lange) Imbach were used as a reference. 650~800 bp DNA fragments can be amplified by optimizing the DNA extraction method and PCR reaction system, using universal primers ITS1 / ITS4, The sequencing and blasting result showed that 11 mushrooms belong to Paxillus involutus (Batsch) Fr.,Calocybe gambosa (Fr.) Singer., Helvella elastica Bull., Agaricus arvensis Schaeff., Tricholoma populinum J.E. Lange, Tricholoma mongolicum S. Imai, and Agaricus bernardii Quél, respectively.
wild edible mushrooms; DNA extraction method optimization; rDNA-ITS molecular identification; homology analysis; food safety
S646
A
2095-0934(2015)05-301-06
国家自然科学基金(51267014)
刘晓婷(1991—),女,硕士研究生,研究方向为环境生物物理。E-mail:liuxiaoting0522@sina.com
郭九峰(1964—),男,研究员,博士,主要从事环境生物物理研究工作。E-mail:guojf101@sina.com
