植物长链非编码RNA功能研究进展
2015-10-27牛旭龙冯万军马金虎邢国芳
牛旭龙 冯万军 马金虎 邢国芳
(山西农业大学农学院,太谷 030801)
植物长链非编码RNA功能研究进展
牛旭龙 冯万军 马金虎 邢国芳
(山西农业大学农学院,太谷 030801)
长链非编码RNA(long non-coding RNA,lncRNA)是新近发现的基因表达调控因子,其广泛参与植物生长发育过程的调节,并可对环境胁迫作出响应。就lncRNA的产生与分类方法、植物中已报道的lncRNA及其对植物不同器官发育过程的影响、lncRNA与小RNA的关系、有关lncRNA的研究方法及目前研究中面临的问题进行了介绍。
植物lncRNA;生物学功能;研究方法;miRNA
经典的中心法则中认为基因一般的表达过程为经DNA转录生成RNA,再翻译为蛋白质,最终实现生物学功能。其中,RNA分子被认为是在这一过程中携带信息的唯一信使。但是,自克里克提出该理论以来,中心法则已经过了极大发展,RNA分子的角色也有了很大变化,研究证实在转录过程中它不仅是一个中间媒介,而且也是具有调节基因表达功能的分子。海量的基因组序列数据证实:DNA上编码蛋白质的区域(也就是通常说的基因)只占人类和其他高等动植物基因组的极小部分,在人体中仅有不到3%的序列编码蛋白质,基因组的绝大部分都不编码蛋白质和多肽[1]。此外,即使在具有较小基因组的模式植物拟南芥中,具有编码蛋白质功能的基因组序列也仅占整个基因组的50%以下[2]。在过去几十年的现代生物学研究中,人们的思路维持在一个“基因-mRNA-蛋白质-功能”的定式,从而有意无意地忽视了对基因组中非编码RNA的研究。
实际上,真核生物中存在多种非编码RNA,如转运RNA(tRNAs)、核糖体RNA(rRNAs)、小核RNA(snRNAs)、 小RNA(siRNAs和microRNAs)及长链非编码RNA(long non-coding RNA,lncRNAs)[3]。其中,lncRNA的长度一般在200-100 000 nt之间,占所有非编码RNA的80%以上[4]。目前,有关小RNA的研究报道较多,部分小RNA在转录和转录后的调控功能已基本清楚,然而有关lncRNA功能的研究仍较少。另外在技术上,由于lncRNA的构成单元是核苷酸,某些RNA的改变,不足以改变它的主要功能,因此使得lncRNA很难被传统的遗传学方法所发现。近年来,随着基因组测序和基因芯片技术的发展,更多新的lncRNA的发现及其调控功能的研究已经成为目前生命科学领域的研究热点之一。因此,本文将对这一领域的最新研究进展作一综述,旨在为后人的研究提供思路与方法。
1 lncRNA的产生及分类
类似于其他大多数RNA,lncRNA 也由RNA 聚合酶II 转录而来。然而,最新发现了一些lncRNA却由RNA 聚合酶 III转录,这与tRNA 和 5S RNA转录过程相似[5],另外还有一些lncRNA是经RNA 聚合酶V转录产生[6]。大量的动物基因组测序结果显示,lncRNA具有典型的RNA特征,如5'端帽子结构和多聚腺苷酸尾巴等。大多数lncRNA分布在细胞核内,也有少数位于细胞质组分里[7]。
lncRNA具有类型多、作用模式多和数量多的“三多”特点。因此,有关lncRNA的分类方法也有多种。根据其在基因组转录的位置lncRNA可分为3类:天然反义转录(NATs)、基因间lncRNA(lincRNA)和内含子lncRNA[8](图1)。天然反义转录(NATs)是一组编码蛋白质或非编码蛋白质的RNAs与其他(有义)转录物具有互补序列可以调节有义链的表达[9]。NATs最早在病毒和原核生物中发现。根据来源NATs可以分为两类:顺式NATs(cis-NATs)和反式NATs(trans-NATs)[10,11]。cis-NATs由基因组相同位点的反义链转录产生,互补区在两个基因的重叠部分,重叠长且完全互补,通常以“一对一”的方式调节有义链的表达[12]。而 trans-NATs则是在不同位点转录产生的正义和反义转录本,可与多个转录本部分互补,如部分trans-NATs后期加工可形成 microRNA或siRNA[13]。现在已有多种方法用于NATs的鉴定,如基因表达的连续分析、cDNA序列分析、Northern杂交、链特异性RT-PCR及电子杂交和基因芯片等,并对多种生物的NATs进行了较为详细的研究。NATs在基因组中普遍存在,在植物中占7%-9%,动物中除了线虫较少,在其他动物中可达到5%-30%[14]。基因间lncRNA(lincRNA)广泛参与哺乳动物细胞免疫监视、周期调控、胚胎干细胞多能性分化等多种生物学过程[15]。利用染色质分离技术(chromatin isolation by RNA purification,CHIRP)在全基因组内发现众多的lincRNA结合于特定的序列上,并且证实lincRNA在不同物种间具有高度的保守性,为进一步研究奠定了基础[16]。内含子lncRNA与表观遗传有密切联系,一个典型的内含子lncRNA- ANRASSF1通过招募PRC2到RASSF1A启动子上,降低RASSF1A的表达并增加细胞增殖,为描述其他内含子lncRNAs作用的位置特异性与表观遗传调节提供了依据[17]。
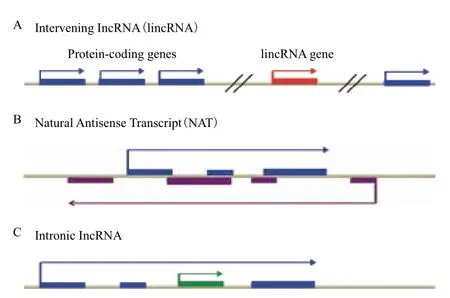
图1 LncRNA三种类型
如根据其功能差异将其分成两类:一类影响细胞发育进程,这类lncRNA通过反式作用、形成干扰RNA或直接与基因组DNA或蛋白互作调控细胞信号转导或蛋白/转录水平;第二类是通过调控转录、剪切或相邻基因的表达起作用[18]。而参照其与蛋白质编码基因的位置关系,可将它们分为正义lncRNA、反义lncRNA、双向lncRNA、基因内lncRNA和基因间lncRNA等5类[19]。
2 不同植物中筛选到的lncRNA
动物中的lncRNA研究主要集中在人、大鼠和线虫上,利用lncRNA沉默与定位分析(combined knockdown and localization analysis of noncoding RNAs,c-KLAN)、RNA免疫沉淀法(RNA-immunoprecipatation,RIP)、CHART(capture hybridization of RNA target)和ChRIP(chromatin RNA immunoprecipatation)等可对lncRNA进行功能缺失和细胞定位,通过研究lncRNA与蛋白质、DNA和RNA相互作用,探讨其功能与作用机制。
与动物相比,对于植物中lncRNA的研究才刚刚开始。现在仍主要依赖全基因组 cDNA文库、微阵列和RNA测序等高通量的技术对lncRNA进行全基因组范围的筛选。迄今为止,仅在少数几种植物中鉴定出lncRNA,涉及拟南芥、玉米、水稻和大豆等[20,21]。目前在植物中筛选到超过9 000个lncRNAs,占总lncRNA的1%,其中部分lncRNA的生物学功能已经相对清楚,如Liu等[22]采用tilling array技术,在拟南芥中发现了6 480个基因间转录本,其中2 708个lncRNAs可通过实验被检测到,一些lncRNA基因表现出较强的器官表达特异性,而其他的则与生物/非生物胁迫响应相关。与此同时,Zhang等[23]利用40个RNA-Seq数据集对水稻全基因组进行分析后,最终筛选到2 063个lncRNAs,其中大部分在水稻生殖过程中表现出特异性,进一步的功能分析显示,一些lncRNA可造成生殖缺陷。除拟南芥和水稻外,Zhang还对玉米自交系B73和Mo17进行正反交实验,分析了胚乳中所有等位基因表达情况,最终找到38个在胚乳中表达的lncRNA与基因印记有关。其中,有25个来源于母本,而其余13个来自父本。另外,Boerner等[24]鉴别出了玉米全长cDNA序列中包含的潜在lncRNA,结果显示,玉米基因组的基因和基因间位点序列中广泛存在非编码RNA的模板区域,同时推测它们可能通过多种RNA介导机制来调节其他基因的表达。综上所述,植物中存在有大量lncRNA,这可能与植物在生长发育和应激反应过程中存在复杂的分子调控网络有关。
3 植物中lncRNA的功能
3.1 lncRNA影响植物生长发育
在植物中,lncRNA广泛存在并参与生长发育进程的调节。研究表明,lncRNA 对植物发育的影响已渗透到根系、花器官建成及花粉发育等各个方面。研究表明很多lncRNA在生物体发育的特定阶段产生,具有组织或细胞特异性[25],在转录水平、转录后水平及表观遗传等方面调控基因的表达。
春化作用是环境控制的表观遗传学开关,研究证实,冬季的低温可以沉默开花阻遏基因FLC(FLOWERING LOCUS C)进而使植物在春季开花。原因在于长时间低温处理可诱导春化不敏感基因(VIN3)的表达,进而形成PRC2复合物,使开花阻遏基因H3K27me3处组蛋白大量被修饰,从而降低FLC基因表达水平。Swiezewski等[26]分析了拟南芥单核苷酸序列多态性,最终在基因组FLC基因所在的两条链50 kb的相邻区域找到两个lncRNA并分别将其命名为COLDAIR和COOLAIR。这两个lncRNA均可与PRC2蛋白复合体结合,从而造成FLC染色质组蛋白甲基化, 最终促进植物开花。这种低温春化过程发生在植株营养生长阶段, 而开花结实过程在其后, 因此春化效应通过细胞有丝分裂传递, 并不传递给子代。
Ding等[27]发现水稻光敏不育系(PGMS)受一个长为1 236个碱基的lncRNA调节,并称之为LDMAR(long-day-specific male-fertility-associated RNA ),长日照条件下,足够的LDMAR 转录量是维持正常花粉发育所必需的,但由于一个单碱基突变造成LDMAR 二级结构改变,导致LDMAR 在长日照下转录量降低,造成正处于发育的花药提前程序化死亡,出现不育性状。另外有研究表明,光温敏核不育系在P/TMS12-1位点编码一个独特的非编码RNA,进而加工成一个21 nt的小RNA——osa-smR5864w,从而导致粳稻农垦58S(NK58S)和籼稻培矮64S雄性不育性状的出现[28]。对比两个研究结果发现,在雄性不育系的12号染色体22258571位点存在C-G碱基颠换,这可能与自发突变引起的单核苷酸多态性(SNP)改变了LDMAR的二级结构,最终造成雄性不育性状的出现。另外,在玉米中也发现了一些参与生长发育的lncRNA。如Dai等和Ma等[29,30]从玉米成熟花粉cDNA文库中克隆到Zm401,该基因在花粉发育过程中特异表达,参与花药的发育进程。前期我们发现一个在玉米杂交种与亲本间差异表达的lncRNA-ZmUHR,其在根尖、幼穗等部位集中表达,并且表现为杂种上调,在47个与ZmUHR基因序列相一致的siRNAs中,有41个在杂种中特异表达。认为一些siRNAs在杂种中的差异表达可能是由于其前提转录水平的改变引起的[31]。
3.2 lncRNA在植物对环境胁迫响应中发挥作用
研究表明,许多lncRNAs对不同的逆境也有响应,包括一些生物和非生物胁迫[26,32-34]。如对拟南芥研究发现,经过2 h或10 h干旱、低温、高盐或脱落酸处理后,有1 832个lncRNAs表达水平发生明显变化。同时研究还发现,能提高植物抗病能力的延伸因子elf18基因(EF-Tu)能够促进一些lincRNAs(intergenic lncRNAs)的表达[22]。与拟南芥的研究结果相似,在小麦中有125个lncRNAs对白粉病和高温胁迫有响应[31]。上述研究结果表明,植物lncRNA可能通过多种途径来承受环境压力,进而使植物更好地应对生物和非生物胁迫,这为人们进一步了解它们的生物学功能积累了丰富的资料。
除了大规模鉴定胁迫响应lncRNAs外,其中几个lncRNAs的生物学功能及作用机理也相对清楚。如COLDAIR和COOLAIR这两个与生殖发育过程有关的lncRNAs,它们响应寒冷天气能诱导拟南芥开花[26,34]。此外,IPS1和AT4受磷饥饿诱导表达,通过阻碍miR399对其靶基因磷酸酯酶转录调控因子PHO2的抑制作用实现功能[35]。另有一个lncRNA——Npc536在拟南芥根和叶中受磷饥饿和盐胁迫后诱导上调表达,Npc536超表达后可以促进根系生长,从而使拟南芥适应磷饥饿和盐胁迫环境[36]。PHO1在植物体内起平衡体内磷酸盐的重要作用。研究发现在水稻基因组中含有3个PHO1的同源基因,与拟南芥中仅有的两个参与磷从根部向地上部运转的基因AtPHO1和AtPHO1;H1不同,水稻的3个PHO1能产生天然顺式反义转录本。在磷酸盐缺失的情况下,水稻PHO1;2表达量增加,PHO1;2蛋白浓度也随之增加,然而PHO1;2的mRNA丰度却没有变化。多聚核糖体分析显示,PHO1;2和反义NAT有激活核糖体转录的功能,揭示了lncRNA如何不依赖提高mRNA 表达量影响蛋白质水平[37]。
还需要注意的是,类病毒是一类使植物发病的lncRNAs,由一个246-400个核苷酸单链环状分子组成。由于类病毒进入宿主细胞后需要改变了lncRNA调控的区域化,了解类病毒条区域化的机制可能为深入了解细胞器如何运输外源和内源lncRNA提供思路[38]。
3.3 植物中lncRNA与小RNA的关系
作为一种新型的起调控作用的RNA,lncRNA可以以长链的形式直接在RNA水平上发挥功能,也可以形成短的microRNA(miRNA)和 small interfering RNA(siRNA)这两种主要的小分子RNA,影响生物体的各项生命活动[13]。长久以来,对于植物中lncRNA的功能研究主要集中在模式植物拟南芥上。lncRNA基因TAS3的转基因的结果表明,超表达TAS3使得拟南芥侧根伸长,转基因植株体内TAS3切割形成的miR390基因的靶基因,tasiARFs的转录水平显著提高,而miR390突变体中ta-siARFs表达则显著降低,表明这些lncRNAs是miRNA的前体。在人类和其他物种的基因组中一些lncRNA序列也是短非编码RNA的前体。通过分析拟南芥全长cDNA文库发现,76条新的lncRNAs和不同类型的小RNA前体npcRNAs,如miRNA、tasiRNA和24 nt的siRNA[32,39]。RT-PCR分析表明,npc83 是miR869a的前体,它在DCL4 突变体中积累。另外,在水稻中研究发现,p/tms12-1 是一个非编码RNA基因,它的原始转录本经过至少两次加工产生一个小RNA,与正常水稻品种相比,温敏不育水稻在该小RNA序列内存在一个单碱基突变,该突变影响了小RNA的表达水平,进而可能与靶基因互作而导致雄性不育[28]。
最新研究发现,在拟南芥和水稻中,一些lncRNA可以作为小RNA的内源诱捕靶标(eTM),如 miR160、miR166、miR156、miR159和 miR172,能够有效抑制这些小RNA的功能实现,进一步研究发现,miR160和miR166的内源诱捕靶标在调控植物发育方面发挥重要的作用。通过突变体研究发现拟南芥的lncRNA-AT4调控植物对低磷胁迫逆境的适应是通过长链的形式直接与miR399结合,降低miR399的表达,而 miR399通过靶向作用调控下游基因PHO2(UBC24)的表达,从而维持植物体的磷稳态[37]。在植物中,IPS1是一个广泛存在的受磷饥饿诱导的lncRNA,在植物的根和芽中特异表达,IPS1含有与miR399互补配对的基序,但是这种互补配对被一个位于miR399的剪切位点的错配环打断,从而阻碍miR399剪切IPS1,同时也使得miR399被螯合,并且两者的结合比miR399与其底物的结合更有竞争性。因此,当IPS1过量表达后,大量的miR399与IPS1配对,只有少数miR399与靶基因PHO2的mRNA配对,导致靶基因mRNA的大量积累,最终表现为枝条中磷含量的降低[35]。这种miRNA抑制机制称为靶基因模拟。靶基因模拟提供了一种新的分析miRNA功能的方法。
4 lncRNA的研究方法
近期,研究人员越来越重视lncRNA,认为lncRNA在基因表达调控中发挥重要作用,广泛参与生物体的生长、发育、衰老等生理过程[40]。lnc-RNAs调节机制多样,包括作为小RNA为前体[41],介导染色质重塑[17],顺式激活邻近基因[42,43],作为多蛋白复合物的支架[44]及反式作用于靶基因座[42]。为揭示lncRNA的分子作用机制,人们借助多种分子生物学研究方法,如微阵列(microarray)、RNA-seq、Northern 印迹(Northern blot)、实时荧光定量逆转录-聚合酶链反应(Real time quantitative reverse transcription-polymerase chain reaction,qRTPCR)、荧光原位杂交(Fluorescence in situ hybridization,FISH)和RNA干扰(RNA interference,RNAi)等分子生物学技术筛选到一些lncRNA[16]。然而对植物中lncRNA的研究仍处于初级阶段,目前lncRNA的研究主要集中在依赖生物信息学技术预测lncRNA,以挖掘其中lncRNA的序列、结构、表达及功能等信息。使用生物信息学方法对RNA-seq测序结果进行预测,根据预测结果进行RNAi和RIP等lncRNA功能验证实验,可以避免功能研究实验的盲目性,从而节约大量实验成本[45]。如Qi等[46]在模拟干旱条件下,利用RNA-seq技术并结合麦金尼斯筛选长链非编码RNA的方法对谷子进行全基因组系统地识别,最终确定了584条lncRNAs(494 lincRNAs和 90 NATS)。其中两个干旱调控NATs-Si003758m和Si038715m。Si003758m与植物氧化胁迫耐性相关,在PEG诱导的干旱条件下,Si003758m的正义和反义转录物均显著增加,Si038715m编码羟脯氨酸糖蛋白(HRGP),它是植物细胞壁的重要组成成分,在植物发育过程中起关键作用[47],也能提高植物的抗病性[48,49]
5 结语
借助于深度测序技术的进步和生物信息学手段,有关植物lncRNA的研究已经取得长足发展,可以预见,在不久的将来有关lncRNA的生物学功能鉴定的报道将出现井喷现象。但由于目前有关lncRNA的研究仍处于起步阶段,因此面临许多亟待解决的问题:(1)lncRNA的界定仍存在争议。目前仍然依赖于是否具有开放阅读框这一结构特征来判断转录产生的RNA是编码还是非编码RNA,实际上研究发现一些lncRNA包含ORF,而该ORF编码与某些已知基因编码蛋白质的同源序列。另外,从长度上对lncRNA进行划分的方法也存在较大争议,lncRNA 是一般被认为长度大于 200 nt,这一标准作为界定lncRNA过于武断,因为小于200 nt的非编码RNA中很多既不属于小RNA(small RNA)也不属于结构RNA(structural RNA),对其所属类别仍不是很清晰。(2)对其描述也不够全面,很难使初学者很快了解lncRNA的信息,交叉借鉴的可能性较小。lncRNA生物学功能的阐明并非易事。(3)lncRNA尚无统一的命名原则:一方面,如何区分功能性和非功能性非编码转录物依然存在困难;另一方面,由于lncRNA种类和功能的多样性,致使不同的lncRNA研究结果之间借鉴意义并不大。(4)对lncRNA的鉴定存在困难,在技术上,由于lncRNA的构成单元是核苷酸,某些RNA的改变,不足以改变它的主要功能,因此使得lncRNA很难被传统的遗传学方法发现。(5)用于 lncRNA 研究的技术多是一些高通量的方法,如何避免转录干扰是一个巨大挑战,而且大量的lncRNA信息获得后人们不能有效的进行结构和功能的分析。主要是因为相关数据库依然很少,有关算法也因对lncRNA信息的认识不够而在算法上有所匮乏。因此,需要建立更多更有效的方法用于系统地研究lncRNA的结构和功能。
综合来看,目前人们对lncRNA的了解只是冰山一角。人产对于植物中lncRNA的认识过程,目前已从lncRNA的筛选过渡到对其基因功能的注释和作用机制的研究,鉴于有关动物lncRNA的信息更加丰富,值得我们借鉴。此外,针对lncRNA 的结构特征和作用机制的数据信息,建立新的研究技术体系将是未来一个重要的研究方向。
[1] 陈润生. 非编码 RNA[J]. Progress in Biochemistry and Biophysics, 2013, 40(7):591-592.
[2]Matsui A, Ishida J, Morosawa T, et al. Arabidopsis transcriptome analysis under drought, cold, high-salinity and ABA treatment conditions using a tiling array[J]. Plant and Cell Physiology,2008, 49(8):1135-1149.
[3]Bai Y, Dai X, Harrison AP, Chen M. RNA regulatory networks in animals and plants:a long noncoding RNA perspective[J]. Briefings in Functional Genomics, 2015, 14(2):91-101.
[4]Esteller M. Non-coding RNAs in human disease[J]. Nature Reviews Genetics, 2011, 12(12):861-874.
[5] Dieci G, Fiorino G, Castelnuovo M, et al. The expanding RNA polymerase III transcriptome[J]. Trends Genet, 2007, 23:614-622.
[6]Wu J, Okada T, Fukushima T, et al. A novel hypoxic stressresponsive long non-coding RNA transcribed by RNA polymerase III in Arabidopsis[J]. RNA Biol, 2012, 9:302-313.
[7]Managadze D, Rogozin IB, Chernikova D, et al. Negative correlation between expression level and evolutionary rate of long intergenic noncoding RNAs[J]. Genome Biol Evol, 2011, 3:1390-1404.
[8] Moran VA, Perera RJ, Khalil AM. Emerging functional and mechanistic paradigms of mammalian long non-coding RNAs[J]. Nucleic Acids Research, 2012, 40(14):6391-6400.
[9]谢兆辉. 天然反义转录物及其调控基因的表达机制[J]. 遗传,2010, 32(2):122-128.
[10] Chen D, Yuan C, Zhang J, et al. PlantNATsDB:a comprehensive database of plant natural antisense transcripts[J]. Nucleic Acids Research, 2012, 40(D1):D1187-D1193.
[11] Yin Y, Zhao Y, Wang J, et al. antiCODE:a natural sense-antisense transcripts database[J]. BMC Bioinformatics, 2007, 8:319.
[12] Prasanth KV, Spector DL. Eukaryotic regulatory RNAs:an answer to the ‘genome complexity’conundrum[J]. Genes & Development, 2007, 21(1):11-42.
[13] Lindsey S, Raghavendra C, Sivalingam KM. Data gathering algorithms in sensor networks using energy metrics[J]. IEEE Transactions on Parallel and Distributed Systems, 2002, 13(9):924-935.
[14] Lapidot M, Pilpel Y. Genome-wide natural antisense transcription:coupling its regulation to its different regulatory mechanisms[J]. EMBO Reports, 2006, 7(12):1216-1222.
[15] 李灵, 宋旭. 长链非编码RNA在生物体中的调控作用[J].遗传, 2014, 36(3):228-236.
[16]夏天, 肖丙秀, 郭俊明. 长链非编码 RNA 的作用机制及其研究方法[J]. 遗传, 2013, 35(3):269-280.
[17] De Lucia F, Dean C. Long non-coding RNAs and chromatin regulation[J]. Curr Opin Plant Biol, 2011, 14(2):168-173.
[18]Tripathi V, Ellis JD, Shen Z, et al. The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation[J]. Molecular Cell, 2010, 39(6):925-938.
[19]Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4):629-641.
[20]Ponjavic J, Ponting CP, Lunter G. Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs[J]. Genome Research, 2007, 17(5):556-565.
[21] Struhl K. Transcriptional noise and the fidelity of initiation by RNA polymerase II[J]. Nat Struct Mol Biol, 2007, 14(2):103-105.
[22] Liu J, Jung C, Xu J, et al. Genome-wide analysis uncovers regulation of long intergenic noncoding RNAs in Arabidopsis[J]. The Plant Cell Online, 2012, 24(11):4333-4345.
[23]Zhang M, Zhao H, Xie S, et al. Extensive, clustered parental imprinting of protein-coding and noncoding RNAs in developing maize endosperm[J]. Proc Natl Acad Sci USA, 2011, 108(50):20042-20047.
[24]Boerner S, McGinnis KM. Computational identification and functional predictions of long noncoding RNA in Zea mays[J]. PloS One, 2012, 7(8):e43047.
[25]Lindberg D, de la Fuente Revenga M, Widersten M. Temperature and pH dependence of enzyme-catalyzed hydrolysis of transmethylstyrene oxide. A unifying kinetic model for observed hysteresis, cooperativity, and regioselectivity[J]. Biochemistry,2010, 49(10):2297-2304.
[26] Swiezewski S, Liu F, Magusin A, et al. Cold-induced silencing by long antisense transcripts of an Arabidopsis Polycomb target[J]. Nature, 2009, 462(7274):799-802.
[27] Ding J, Lu Q, Ouyang Y, et al. A long noncoding RNA regulatesphotoperiod-sensitive male sterility, an essential component of hybrid rice[J]. Proc Natl Acad Sci USA, 2012, 109(7):2654-2659.
[28] Zhou H, Liu Q, Li J, et al. Photoperiod-and thermo-sensitive genic male sterility in rice are caused by a point mutation in a novel noncoding RNA that produces a small RNA[J]. Cell Research,2012, 22(4):649-660.
[29] Dai XY, Yu JJ, Zhao Q, et al. Non-coding RNA for ZM401, a pollen-specific gene of Zea mays[J]. Acta Botanica Sinica-English Edition, 2004, 46(4):497-504.
[30] Ma J, Yan B, Qu Y, et al. Zm401, a short open reading-frame mRNA or noncoding RNA, is essential for tapetum and microspore development and can regulate the floret formation in maize[J]. Journal of Cellular Biochemistry, 2008, 105(1):136-146.
[31] Xin M, Wang Y, Yao Y, et al. Identification and characterization of wheat long non-protein coding RNAs responsive to powdery mildew infection and heat stress by using microarray analysis and SBS sequencing[J]. BMC Plant Biology, 2011, 11(1):61.
[32] Amor BB, Wirth S, Merchan F, et al. Novel long non-protein coding RNAs involved in Arabidopsis differentiation and stress responses[J]. Genome Research, 2009, 19(1):57-69.
[33] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs:insights into functions[J]. Nat Rev Genet, 2009, 10:155-159.
[34] Heo JB, Sung S. Vernalization-mediated epigenetic silencing by a long intronic noncoding RNA[J]. Science, 2011, 331(6013):76-79.
[35] Franco-Zorrilla JM, Valli A, Todesco M, et al. Target mimicry provides a new mechanism for regulation of microRNA activity[J]. Nature Genetics, 2007, 39(8):1033-1037.
[36] Wierzbicki AT, Haag JR, Pikaard CS. Noncoding transcription by RNA polymerase Pol IVb/Pol V mediates transcriptional silencing of overlapping and adjacent genes[J]. Cell, 2008, 135(4):635-648.
[37] Jabnoune M, Secco D, Lecampion C, et al. A rice cis-natural antisense RNA acts as a translational enhancer for its cognate mRNA and contributes to phosphate homeostasis and plant fitness[J]. The Plant Cell Online, 2013, 25(10):4166-4182.
[38] Gómez G, Pallás V. Viroids:a light in the darkness of the lncRNA-directed regulatory networks in plants[J]. New Phytologist, 2013, 198(1):10-15.
[39] Hirsch J, Lefort V, Vankersschaver M, et al. Characterization of 43 non-protein-coding mRNA genes in Arabidopsis, including the MIR162a-derived transcript[sJ]. Plant Physiology, 2006, 140(4):1192-1204.
[40] 祁云霞, 刘永斌, 荣威恒. 转录组研究新技术:RNA-Seq 及其应用[J]. 遗传, 2011, 33(11):1191-1202.
[41] Kim TK, Hemberg M, Gray JM, et al. Widespread transcription at neuronal activity-regulated enhancers[J]. Nature, 2010, 465(7295):182-187.
[42] Kim ED, Sung S. Long noncoding RNA:unveiling hidden layer of gene regulatory networks[J]. Trends in Plant Science, 2012, 17(1):16-21.
[43] Ørom UA, Derrien T, Beringer M, et al. Long noncoding RNAs with enhancer-like function in human cells[J]. Cell, 2010, 143(1):46-58.
[44] Tsai MC, Manor O, Wan Y, et al. Long noncoding RNA as modular scaffold of histone modification complexes[J]. Science, 2010,329(5992):689-693.
[45] 白晶, 李力恒, 孙尧, 等. 传统中草药高通量测序技术 RNA-seq 及 lncRNA 挖掘的应用策略[J]. 中医药信息, 2014(2):20-23.
[46] Qi X,Xie S,Liu Y, et al. Genome-wide annotation of genes and noncoding RNAs of foxtail millet in response tosimulated drought stress by deep sequencing[J]. Plant Mol Biol,2013,83(4-5):459-473.
[47] Hall Q, Cannon MC. The cell wall hydroxyproline-rich glycoprotein RSH is essential for normal embryo development in Arabidopsis[J]. Plant Cell, 2002, 14(5):1161-1172.
[48] Deepak S, Shailasree S, Kini RK, et al. Role of hydroxyproline-rich glycoproteins in resistance of pearl millet against downy mildew pathogen Sclerospora graminicola[J]. Planta, 2007, 226(2):323, 333.
[49] Carrieri C, Cimatti L, Biagioli M, et al. Long non-coding antisense RNA controls Uchl1 translation through an embedded SINEB2 repeat[J]. Nature, 2012, 491(7424):454-457.
(责任编辑 狄艳红)
Research Progress on Biological Functions of Long Non-coding RNA in Plants
Niu Xulong Feng Wanjun Ma Jinhu Xing Guofang
(College of Agriculture,Shanxi Agricultural University,Taigu 030801)
Long non-coding RNAs(lncRNAs)as newly discovered regulators for gene expression, participate in a wide range of biological processes in plants, and also could response to environmental stresses. In this review, we summarize the generation and classification of lncRNAs, the discovered lncRNAs in plants, their effects on the development processes of different organs in plants, as well as the relationship between lncRNA and small RNA, research methods, and issues in the current studies of lncRNAs.
plant lncRNA;biological function;research methods;miRNA
10.13560/j.cnki.biotech.bull.1985.2015.06.001
2014-06-06
国家自然科学基金项目(31200914),山西省青年科学基金项目(2012021023-4),中国博士后科学基金项目(2012M520600),山西省高等学校科技创新项目(2015145)
牛旭龙,男,硕士,研究方向:玉米功能基因组学;E-mail:ycyznxl@163.com
邢国芳,女,博士,副教授,研究方向:功能基因组及抗逆胁迫;E-mail:xingguofang9596@sina.com
