肺炎克雷伯菌超广谱β-内酰胺酶、头孢菌素酶和qnr基因检测及耐药分析
2015-10-25张欣欣王勇杨庆斌
张欣欣,王勇,杨庆斌
(安徽省合肥市第二人民医院呼吸内科,安徽合肥230001)
肺炎克雷伯菌超广谱β-内酰胺酶、头孢菌素酶和qnr基因检测及耐药分析
张欣欣,王勇,杨庆斌
(安徽省合肥市第二人民医院呼吸内科,安徽合肥230001)
目的检测痰标本中肺炎克雷伯菌超广谱β-内酰胺酶(ESBLs)、头孢菌素酶(AmpC酶)和qnr基因的存在情况及耐药性,为临床合理使用抗菌药物提供参考。方法收集痰标本中分离到的肺炎克雷伯菌114株,用琼脂稀释法检测耐药性,三维试验法和聚合酶链式反应(PCR)法检测ESBLs酶、AmpC酶及qnr基因存在情况。结果114株肺炎克雷伯菌中,ESBLs阳性58株(50.88%),产AmpC酶14株(12.28%),同时产ESBLs和AmpC酶10株(8.77%),qnr阳性2株;产酶株对抗菌药物的耐药率显著高于不产酶株。结论痰标本中肺炎克雷伯菌耐药基因的检测率较高,多重耐药明显;碳青霉烯类具有较好的抗菌活性。
肺炎克雷伯菌;细菌耐药;超广谱β-内酰胺酶;头孢菌素酶
肺炎克雷伯菌为呼吸道感染重要致病菌,患者病死率较高[1]。近年来,随着抗菌药物应用强度及程度的增加,肺炎克雷伯菌对抗菌药物的耐药率呈不断升高趋势。产超广谱β-内酰胺酶(ESBLs)、头孢菌素酶(AmpC酶)和qnr基因为肺炎克雷伯菌当前耐药的重要原因。笔者收集了114株痰标本中分离到的肺炎克雷伯菌,对其进行ESBLs,AmpC酶和qnr耐药基因检测及耐药性分析,为临床合理用药提供参考。
1 材料与方法
1.1 菌株来源
收集来自安徽省多家医院送检的痰标本中分离出的114株肺炎克雷伯菌,菌株鉴定均由Vitek全自动微生物分析系统完成。
1.2 药物敏感性试验
采用琼脂稀释法,操作和结果判读按照美国临床和实验室标准协会(CLSI)2012版标准进行。其中选择大肠埃希菌ATCC25922为不产酶株,阴沟肠杆菌029M为AmpC酶质控菌株,肺炎克雷伯菌ATCC700603为ESBLs质控菌株,菌株均由安徽省细菌耐药监控中心提供。氨苄西林(AMP,山东齐鲁制药有限公司),哌拉西林他唑巴坦(TZP,美国Wyeth-Ayerst公司),头孢呋辛(CXM,上海新亚药业有限公司),头孢噻肟(CTX,安徽威尔曼制药有限公司),头孢曲松(CRO,台湾泛生制药厂股份有限公司),头孢他啶(CAZ,葛兰素史克公司),头孢吡肟(FEP,中美上海施贵宝制药有限公司),头孢西丁(FOX,海口制药厂有限公司),氨曲南(ATM,江西制药有限责任公司),亚胺培南西司他丁(IMP,默沙东公司),美罗培南(MEM,日本住友制药株式会社),庆大霉素(GEN,上海第一生化药业有限公司),环丙沙星(CIP,广州南新制药有限公司),加替沙星(GAT,南京圣和药业有限公司),左氧氟沙星(LVX,江苏豪森药业股份有限公司),阿米卡星(AMK,上海旭东海普药业有限公司)。
1.3 Am pC酶及ESBLs检测
酶粗提物制备:参考文献[2]描述的方法培养细菌,获得菌体沉淀,反复冻融提取细菌酶液,在4℃以10 000 r/min离心10min提取上清液。上清液经MH琼脂平板细菌培养阴性后置-20℃冰箱保存,备用。
AmpC酶和ESBLs检测:参考文献[2]提供的方法行AmpC酶和ESBLs的三维试验检测。将受试菌液接种MH琼脂平板上,取头孢西丁纸片置平皿中心,余操作按步骤进行。如观察到狭槽与抑菌圈交接处出现扩大的长菌区,视为AmpC酶三维试验阳性。ESBLs检测时用头孢曲松取代头孢西丁,余操作相同,如在狭槽与头孢曲松交界处出现扩大的细菌生长区域,视为ESBLs阳性。
1.4 细菌质粒提取与聚合酶链式反应(PCR)扩增
对其中10株同时产ESBLs和AmpC酶的肺炎克雷伯菌行qnr基因PCR扩增,直接煮沸法提取细菌质粒。根据Genbank数据库中公布的qnr基因序列设计PCR扩增引物,引物由北京赛百盛公司合成。PCR反应体系(50μL)包含:10×buffer 5μL,dNTPS mixture(2.5mmol/L)4μL,上下游引物各1μL(10μmol/L),rTaq酶(5μ/L)0.25μL,质粒模板4μL,上述试剂均购自大连宝生物公司。引物序列P1:5′-TCAGCAAGAGGATTTCTCA3′,P2:5′-GGCAGCACTATTACTCCCA 3′。PCR反应条件:预变性94℃5min,变性94℃1min,退火温度48℃40 s,72℃延伸1min共35次循环,再72℃延伸5min。扩增片段长度为627 bp,扩增过程中以大肠埃希菌ATCC25922及空白为阴性对照,标准产酶株为阳性对照。选取PCR扩增阳性的产物送上海生工公司进行纯化测序,测序仪器为ABIPRISM3730,测序试剂为BigDye terminator v 3.1,测序结果由GenBank blast程序行比对确定。
2 结果
2.1 产ESBLs及Am pC酶检测
在114株肺炎克雷伯菌标本中,其中产ESBLs58株,阳性率达50.88%;产AmpC酶14株,阳性率达12.28%;同时产ESBLs和AmpC酶10株,阳性率为8.77%。
2.2 PCR扩增
10株肺炎克雷伯菌行PCR扩增后,2株qnr检测阳性。将测序结果上网比对,与GenBank数据库所提供的qnr基因序列一致。
2.3 肺炎克雷伯菌耐药试验
结果见表1。AmpC酶、ESBLs及同时产AmpC酶和ESBLs菌株对16种抗菌药的耐药率均高于阴性菌株,其中产ESBLs菌株仅对美罗培南、亚胺培南、哌拉西林他唑巴坦、头孢他啶敏感,对其余抗菌药物的耐药率均达50%以上;产AmpC酶菌株除对碳青霉烯类药物敏感外,其他抗菌药物耐药率基本在60%以上,且产酶肺炎克雷伯菌株多重耐药现象明显。
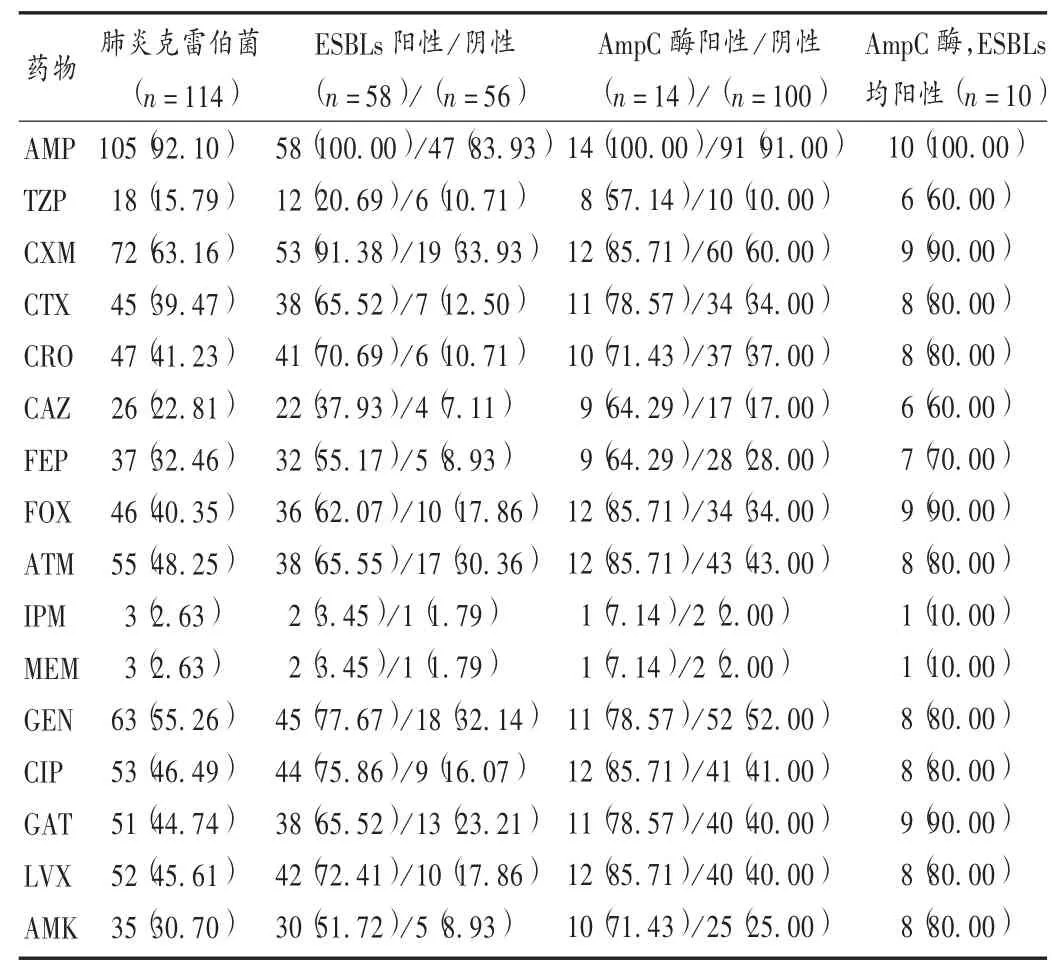
表1 肺炎克雷伯菌对抗菌药物的耐药率检测结果[株(%)]
3 讨论
肺炎克雷伯菌是医院获得性肺炎常见致病菌,分离率一直呈增多趋势[3]。随着抗菌药物的大量应用,肺炎克雷伯菌对药物的耐药性也呈现不断增强的趋势,耐药株不断增多,多重耐药现象也明显[4-5],这其中最主要的原因是其能产生大量ESBLs和AmpC酶。β-内酰胺酶主要通过水解β-内酰胺环,使β-内酰胺类抗生素丧失活性。肺炎克雷伯菌对β-内酰胺类抗生素耐药的重要机制就是产生ESBLs,ESBLs通过水解青霉素、广谱头孢菌素及单环β-内酰胺类药物的β-内酰胺酶,导致抗生素耐药。本研究中检测了114株痰标本中肺炎克雷伯菌,其ESBLs阳性率高,达50%左右,可能与本地区医院抗生素使用强度和种类有关,易诱导ESBLs产生。产ESBLs株仅对碳青霉烯类、哌拉西林他唑巴坦及头孢他啶较敏感,对其余抗菌药物的耐药率均达50%以上,耐药率较高。非产酶株对抗菌药物的耐药率几乎均小于35%(除氨苄西林),产酶株对抗菌药物的耐药率明显高于不产酶株。哌拉西林他唑巴坦较敏感,提示含有酶抑制剂的抗菌药物可提高对产酶菌株的敏感性。头霉素类抗菌药物如头孢西丁对产ESBLs细菌有较好的抗菌作用,但本试验结果显示,其耐药性高,且头霉素类易诱导细菌产生AmpC酶,故临床不宜应用。检测提示头孢他啶相对较敏感,可能与本地区头孢他啶的使用率较低及基因表型有关。因ESBLs基因存在多种表型,对头孢菌素的水解能力有差异。但因临床治疗效果不佳,故其使用仍需慎重。
目前对AmpC酶稳定的药物主要有碳青霉烯类和第4代头孢菌素类(如头孢吡肟)。本检测结果显示,AmpC酶株对包括头孢吡肟在内的抗菌药物耐药率基本在60%以上,可能与第4代头孢使用强度高及这14株中的10株同时产ESBLs酶有关,导致其耐药率较高,应引起重视。但碳青霉烯类药物仍具有较高的敏感度,故一旦发现AmpC酶阳性株,应首选。
肺炎克雷伯菌对抗菌药物耐药率高的主要原因是其能产生大量的ESBLs和AmpC酶,可通过质粒介导,导致耐药菌的传播[6-7]。肺炎克雷伯菌是多种耐药质粒主要的宿主之一,因整合子、转座子等作用,1种质粒可同时携带多个耐药基因,使得产ESBLs和AmpC酶的肺炎克雷伯菌表现为多重耐药。在114株肺炎克雷伯菌中发现有10株同时产ESBLs酶和AmpC酶,阳性率达8.77%,本检测结果显示,除亚胺培南及美罗培南敏感外,对其余抗菌药物药率均超过60%,多重耐药比较严重。
细菌对喹诺酮类药物的耐药机制主要有药物作用靶位的改变、膜通透性改变及主动外排机制,这些机制由染色体介导,为主动耐药机制[8-9]。qnr基因编码的蛋白可保护细菌免受喹诺酮类药物的攻击,从而增强细菌的耐药性[10-11]。qnr蛋白仅引起对喹诺酮类药物的较低水平耐药,但由于其具有对其他耐药突变的保护作用,使菌群能达到二次突变的浓度,从而使细菌发生更高程度的耐药[12-13]。本研究中检测出2株qnr阳性,均同时产ESBLs及AmpC株,除美罗培南、亚胺培南外,其他14种抗菌药均耐药。虽然qnr基因检测率较低,但由于qnr基因所在的质粒常携带其他耐药基因(如aac,bla等),可在同种或不同细菌之间传播,引起细菌耐药性的增加,故临床危害大。
综上所述,痰标本中分离的肺炎克雷伯菌产生ESBLs及AmpC酶、qnr基因存在可能是其对抗菌药物耐药或多重耐药的主要原因。产酶菌除碳青霉烯类敏感,对大多数抗菌药物耐药率高。我国耐药监测数据表明,肺炎克雷伯菌对碳青霉烯类药物的耐药率为10%左右,虽然大多数菌株仍对其敏感,但美国2011年发生了碳青霉烯类耐药的肺炎克雷伯菌流行[14],值得警惕。为防止肺炎克雷伯菌感染的发生和流行,只有合理应用抗菌药物,严格掌握用药原则;同时及时准确地进行细菌培养及ESBLs与AmpC酶、qnr基因的检测,才能为临床合理应用抗菌药物提供依据,以防止耐药菌产生。
[1]Clinical and Laboratory Standards Institute/NCCLS.Perfermance standards for antimicrobical susceptibility testing:Sixteenth informational supplement.CLSI/NCCLS document M100-S16[J].CL51 Wayne Pennaylvania,2006,26(3):31.
[2]吴伟元,陈民钧,王辉.阴沟肠杆菌去阻遏持续高产AmpC酶和超广谱β-内酰胺酶(ESBLs)的检测[J].中国临床药理学杂志,2001,17(2):104.
[3]文细毛,任南,吴安华,等.全国医院感染监控网医院感染病原菌分布及变化趋势[J].中华医院感染学杂志,2011,21(2):353.
[4]Nordman P,Cuzon G,Naas T.The real threat of Klebsiellapneumonia carbapenemase-producing bacteria[J].Lancet Infect Dis,2009,9(4):228-236.
[5]周蓉,朱卫民,黄文祥,等.855株肺炎克雷伯菌感染的临床分布及耐药性分析[J].中国抗生素杂志,2013,38(5):363-369.
[6]BabiniGS,Livermore DM.Antimicrobial resistance amongst Klehsiella spp collected from intensive care units in Southern and Western Europe in 1997-1998[J].JAntimicrob Chemother,2000,45(2):183-189.
[7]王寰,范晓磊.革兰阴性杆菌ESBLs和AmpC酶的检测及耐药分析[J].中国微生态学杂志,2008,20(6):591-593.
[8]Robicsek A,Jacoby GA,Hooper DC.The worldwide emergence of plasmid-mediated quinolone resistance[J].Lancet Infect Dis,2006,6(10):629-640.
[9]Chen FJ,Lo HJ.Molecularmechanism of fluoroquinolone resistance[J].J Microbiol Immunol Infect,2003,36:1-9.
[10]Martinez-Martinez L,Pascual A,Jacopy GA.Quinolone resistant from a transferable plasmid[J].Lancet,1998,351(9 105):197-199.
[11]马晓波,宋秀宇.质粒介导细菌对喹诺酮类抗菌药物的耐药机制[J].中华医院感染学杂志,2009,19(12):1 618-1 620.
[12]何菡,孙桂珍,王清涛.肺炎克雷伯杆菌质粒介导喹诺酮耐药基因qnr的研究[J].北京医学,2012,34(12):1 057.
[13]李涛,熊自忠,徐元宏.一个新qnr基因亚型的克隆表达及其多重耐药机制的研究[J].中华检验医学杂志,2006,29(6):546-549.
[14]DrorM,TeenaC,Pogue JM,etal.Outbreak of colistin-resistant,carbapenem-resistant klebsiella pneumoniae in metropolitan detroit,Michigan[J]. Antimicrobial Agents&Chemotherapy,2011,55(2):593-599.
Analysis of Drug Resistance and Detection of ESBLs,Am pCβ-lactamases and qnr in K lebsiella Pneum onia
Zhang Xinxin,Wang Yong,Yang Qingbin
(Department of Respiratory Medicine,Hefei Second People′s Hospital,Hefei,Anhui,China 230001)
Objective To investigate the antibiotic resistance and detection of ESBLs,Ampcβ-lactamases and qnr in Klebsiella pneumonia isolated from the sputum,and to provide reference for clinical rational use of antibiotics.Methods A total of 114 strains of Klebsiella pneumonia were selected,Agar dilution test was used in susceptibility testing.The Genotes of ESBLs,AmpC and qnr was detected and analyzed by three-dimensional test and PCR.Results In the 114 strains of Klebsielinsla pneumonia,58 strains(50.88%)produced ESBLs and 14(12.28%)produced AmpCβ-lactamases,10(8.77%)produced both ESBLs and AmpCβ-lactamases,2 strains carried qnr;The rates of resistance to most antibiotics in drug resistant enzyme strain were higher than negative counterparts.Conclusion The Klebsiella Pneurnonia Producing drug-resistant gene isolated from the sputum are common,most of them are multiple drug resistant.Almost all strains were susceptible to Carbapenem.
Klebsiella pneumonia;resistance to Drug;qnr;ESBLs;Ampc
R969.3;R915
A
1006-4931(2015)22-0054-03
2015-05-13)
