大肠杆菌topA-细胞中tetA-cfaABCE的高效转录促进DNA凝集*
2015-06-06赵舒怡张臻峰黄熙泰
赵舒怡,张臻峰,黄熙泰
(1.国家电网技术学院泰山校区,山东泰安271000;2.中国科学院微生物研究所微生物前期开发国家重点实验室,北京100101;3.南开大学生命科学院,天津300071)
DNA是细胞内最主要的遗传物质,其三维结构对细胞内DNA复制和RNA转录等生理活动有非常大的影响。因此,研究DNA的拓扑结构形式及其包装方式具有非常重要的意义。DNA超螺旋对于细胞活性来说是必需的,因此在细胞内是受到严密调控的[1]。胞内超螺旋程度受特定酶 (DNA拓扑异构酶和旋转酶)的控制,它们可以在DNA分子上引入或者去除超螺旋[2-5]。
基因的转录也对DNA的超螺旋状态也有很大的影响。在转录过程中,在转录复合体前方和后方DNA分别生成数量相当的正超螺旋和负超螺旋。而这些超螺旋可以通过多种途径进行消除,包括DNA拓扑异构酶的作用,DNA双螺旋与转录复合物的相互旋转使正超螺旋和负超螺旋互补消除等[6]。转录的过程中,在特定的拓扑异构酶失活的情况下,如果DNA双螺旋链与转录复合物的相互旋转同时也被阻止或者被限制,那么就会积累相应的超螺旋。因此,在拓扑异构酶I缺失的菌株中,tetA的共转译系统使其产物的氨基端的膜插入区结合到细胞质膜上,或者其他某些DNA结合蛋白 (如LacⅠ)结合到DNA双链上[7],均可阻止DNA链和转录复合物的相互旋转,在旋转酶 (gyrase)的作用下,引入负超螺旋,最终导致高度负超螺旋的产生[8-9]。通过对 pBR322质粒 DNA 详细的研究,Liu和Wang[10]认为这种负超螺旋增加是由于DNA模板在转录过程中的分子操作造成的,进而提出了“孪生超螺旋结构域”模型。而且孪生结构域导致的高度负超螺旋的形成依赖于膜结合蛋白如tetA蛋白,同时也依赖于转录复合物的数量以及转录的相对方向[8,10]。另外,Drolet和 Liu等[11]又提出了R-loop模型,即在转录延伸过程中形成的新生RNA链在转录复合物后方与其模板链形成RNA-DNA杂交双螺旋结构,该结构的形成导致了后续的超螺旋的产生,而高浓度的RNase H(特异性的降解RNA-DNA杂交链中的RNA)可以消除转录延伸过程中产生的负超螺旋[12-13]。在该假说中,DNA负超螺旋的形成依赖于转录的延伸,并且DNA拓扑异构酶在稳定该种结构方面起着重要的作用。R-loop可以稳定负超螺旋,同时高度的负超螺旋又促进R-loop的形成。
近十几年来,多个课题组利用体外[7,14]或体内[15-18]的转录体系证明,在体系中存在旋转酶(Gyrase)而缺失拓扑异构酶I(Topoisomerase I)时高效的转录可以促进质粒DNA负超螺旋的积累并形成高度负超螺旋结构 (Hypernegatively supercoiled DNA)。然而,需要指出的是这些研究中使用的均为单一的、相对较短的转录单元。而在细菌染色体上存在很多大的基因或基因簇,它们的转录单位通常都很大,这种长距离的转录可能会对DNA结构产生更大的影响。为了研究这种影响,我们使用pJGX15A作为报告质粒。该质粒的构建是直接将含有结构基因簇cfaABCE的DNA片段直接插入到tetA基因的下游[19]。由于插入片段上不带有启动子,该基因簇与tetA基因共转录并形成一个长的转录单位。此前的研究已证明在两种菌株中CFA/I均可以很好的表达并组装形成菌毛[19]。本课题的研究结果表明,在大肠杆菌topA-细胞中,tetA-cfaABCE的高效转录可以促使pJGX15A形成一种部分高度凝集的结构。该结构在氯喹-琼脂糖凝胶电泳具有较高的迁移率并表现为“弥散”状态。原子力显微镜的观察结构表明这种质粒DNA分子中含有不同程度的凝集结构且其结构参数明显有别于超螺旋质粒DNA。
1 材料与方法
1.1 材料
1.1.1 试剂 溶菌酶、SDS、PEG6000、溴化乙锭(EB)、氯喹等购自Sigma公司。胰蛋白胨、酵母提取物以及琼脂糖购自Oxoid公司。Tris、EDTA、NaOH、NaCl、MgCl2等常规化学试剂均为国产分析纯试剂。Taq酶、PCR产物回收试剂盒购自上海生工,PCR引物由上海生工合成。
1.1.2 质粒与菌株 大肠杆菌 HB101(topA+,gyrB+)与DM800(topA-,gyrB225)均由纽约公共卫生研究所Dr.Drlica提供,质粒pJGX15A(图1)则由中国医学科学院徐建国教授提供[19]。
1.2 方法
1.2.1 细菌培养及质粒提取 挑取大肠杆菌单菌落至5 mL LB试管中,37℃,200 r/min摇床震荡培养过夜。然后将菌液接种到200 mL新鲜的LB培养基中,在37℃下继续培养,并监测菌液在600 nm处的光吸收值。本研究中所有培养基中均添加了100 μg/mL氨苄青霉素;并根据需要分别添加0、6、12 μg/mL的四环素。当培养至所需的细胞生长时期后,离心 (6000 r/min,5 min)收集菌体,并提取质粒DNA。为避免菌体裂解过程中碱性试剂对质粒结构的损伤,采用温和的清亮裂解方法提取质粒 DNA[20]。
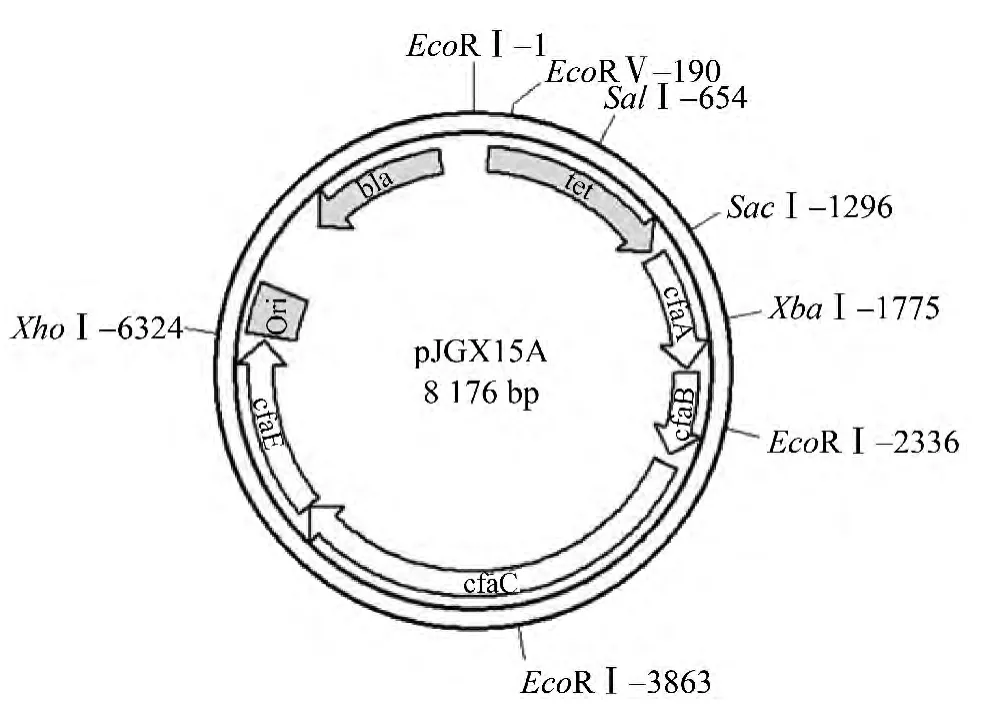
图1 质粒pJGX15A的限制图Fig.1 Restriction map of recombinant plasmid pJGX15A
1.2.2 氯喹-琼脂糖凝胶电泳 鉴于pJGX15A质粒较大,因此采用w=0.6%的琼脂糖凝胶电泳分析其DNA超螺旋状态。制备凝胶前,1×TAE缓冲液中加人适量的氯喹母液 (10 mg/mL)使其达到实验要求的浓度,再加人琼脂糖,加热制胶。电泳缓冲液中氯喹浓度应与琼脂糖凝胶中一致。电泳电压为75 V,时间为 12 h。电泳结束后,在 10 mmol/L MgCl2中脱色30 min,在0.5 μg/mL EB 溶液中染色1 h,再脱色30 min后,紫外灯下观察结果,拍照。在所有实验中,上样孔中均无残留DNA。
1.2.3 原子力显微镜成像 本实验利用二价离子将DNA桥联吸附在带负电的云母表面。将DNA样品稀释到1 μg/mL,向其中加入MgCl2至终浓度为1 mmol/L。取15 μL样品滴加到新剥离的云母片上,室温吸附5 min。100 μL去离子水润洗3遍,去除未吸附的DNA,然后用氮气吹干5 min。使用Nanoscope Ⅲ a Multimode-AFM instrument(Digital Instruments)在室温下利用轻敲模式 (Tapping mode)下进行扫描。探针使用共振频率为106 kHz的 Super-sharp silicon tips(Silicon-MDT Ltd.),扫描速度为1~2 Hz。开始扫描后调整设定的参数值以确保将干扰降至最低并保持图像清晰。
DNA分子的长度利用软件 ImageJ Ver1.33u(Wayne Rasband,National Institute of Health,USA)进行测量;高度与宽度则使用原子力显微镜自带的软件进行测量。
2 结果
2.1 pJGX15 A在两种宿主中的超螺旋水平
在添加了100 μg/mL氨苄青霉素和12 μg/mL四环素的LB培养基中,将含有pJGX15A的大肠杆菌HB101与DM800菌株分别培养到对数前期(A600约为0.4)和中期 (A600约为0.8),提取质粒DNA,并对其进行氯喹-琼脂糖凝胶电泳分析其拓扑结构。图2中A、B、C分别表示电泳体系中氯喹浓度逐渐增加时质粒pJGX15A的电泳情况。随着氯喹浓度的增加,质粒DNA的负超螺旋水平由于氯喹嵌入的逐渐增加而逐渐降低,产生从负超螺旋—松弛—正超螺旋的转变。
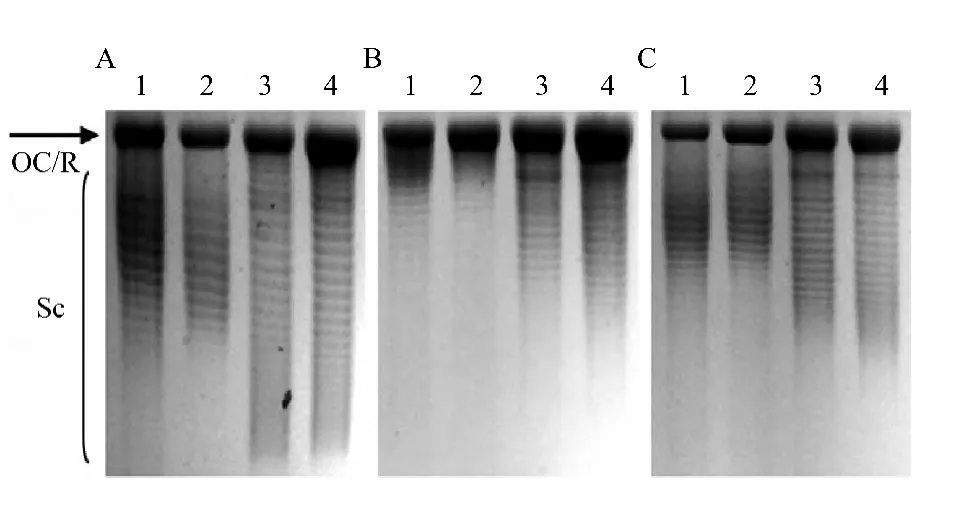
图2 质粒pJGX15A在HB101与DM800细胞中的超螺旋水平Fig.2 Agarose gel electrophoresis of pJGX15A DNA in the presence of 5 μg/mL(A),20 μg/mL(B)or 35 μg/mL(C)chloroquine The plasmid DNA samples were isolated from cells cultured in LB mediums containing 12 μg/mL tetracycline to the early-or mid-exponential phase.Lane 1:pJGX15A from HB101 cells in the early-exponential phase;lane 2:pJGX15A from HB101 cells in the mid-exponential phase;lane 3:pJGX15A from DM800 cells in the early-exponential phase;lane 4:pJGX15A from DM800 cells in the mid-exponential phase.OC/R:open circular or relaxed plasmid DNA;Sc:supercoiled plasmid DNA
结果显示,宿主DM800中的pJGX15A拓扑异构体分布范围较广,而宿主HB101中的pJGX15A的拓扑异构体则比较集中。这是由于DM800存在topA缺失以及gyrB225的补偿突变,使得其对胞内DNA超螺旋水平的控制能力较差所造成的。图2A中结果显示,低质量浓度氯喹 (5 μg/mL)中HB101和 DM800中的pJGX15A均为负超螺旋状态,且DM800中的pJGX15A泳动速度较快。图3B中结果显示,提高氯喹质量浓度至20 μg/mL时,HB101中pJGX15A大部分拓扑异构体由于氯喹的嵌入使其DNA结构接近于松弛环形DNA;而DM800中pJGX15A则仍有部分拓扑异构体保持较高的负超螺旋水平,因而泳动速度较快。图3C中结果显示,提高氯喹质量浓度至35 μg/mL时,两种宿主中pJGX15A大拓扑异构体由于氯喹的嵌入均变为正超螺旋,而DM800中pJGX15A部分拓扑异构体则形成高程度的正超螺旋,泳动速度较快。但需要注意的是,每个DNA样品中各拓扑异构体DNA的含量呈正态分布,因此比较种宿主质粒DNA拓扑异构体主体的超螺旋状态可以发现pJGX15A在二者中的平均超螺旋状态基本一致。综合以上结果,pJGX15A在宿主DM800中既含有高度负超螺旋的结构同时也含有低负超螺旋的结构,但其平均超螺旋水平与HB101中的质粒DNA相当。
2.2 DM800 菌株中pJGX15 A的特殊结构形式
早先的研究已经发现携带四环素抗性基因tetA的质粒pBR322在大肠杆菌topA-菌株DM800中具有高度的负超螺旋水平[8],这一点与我们上述的结果并不完全吻合。在实验过程中我们发现培养基中添加高浓度四环素后携带pJGX15A质粒的两种宿主菌均生长变缓,而携带pBR322质粒的宿主菌的生长则没有明显变化 (数据未给出),推测这一点可能是导致两种质粒DNA在DM800中的超螺旋水平有区别的原因,因此我们尝试降低培养基中的四环素质量浓度并重复以上实验。图3中的结果显示,当四环素质量浓度降低到 6 μg/mL时,pJGX15A在DM800菌株中的超螺旋水平明显高于HB101菌株;而当培养基中不添加四环素时,pJGX15A在DM800菌株中的超螺旋水平不仅明显高于HB101菌株,而且对数前期DM800细胞中提取的pJGX15A在电泳中表现出典型的“负超螺旋—高度负超螺旋”模式 (图3:3泳道)。仔细分析图3中pJGX15A的拓扑异构体在电泳中的分布模式可以发现,DM800细胞中的pJGX15A在电泳中不能被分离为清晰的拓扑条带,而是表现为泳动速度较快的“弥散”条带。这一点与高质量浓度四环素培养时的结果形成了鲜明的对比。这些结果表明,在四环素质量浓度较低时对数期DM800细胞中的pJGX15A形成了特殊的结构,这一结构有别于通常意义的超螺旋结构,可能是一种新的拓扑结构形式。
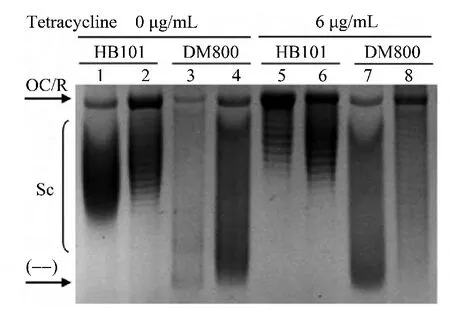
图3 DM800细胞中质粒pJGX15A形成特殊的拓扑结构形式Fig.3 Agarose gel electrophoresis of pJGX15A DNA in the presence of 5 μg/mL chloroquineThe plasmid DNA samples were isolated from cells in the early-(lanes 1,3,5 and 7)or mid-exponential phase(lanes 2,4,6 and 8).OC/R,open circular or relaxed plasmid DNA;Sc,supercoiled plasmid DNA;(--),hypernegatively supercoiled plasmid DNA
2.3 四环素质量浓度与细胞生长时期对pJGX15A特殊结构的影响
进一步对比图3中的结果发现,对数前期与中期的HB101细胞中的pJGX15A的结构基本一致,然而DM800细胞中提取的pJGX15A的结构在两个时期则有明显的不同——对数前期的pJGX15A超螺旋水平更高,也更易于形成“弥散”条带。这一点也促使我们进一步探讨细胞生长时期对pJGX15A拓扑结构的影响。在不同四环素质量浓度下培养HB101与DM800,分别收集对数期以及静止期的细胞并提取质粒DNA,然后利用氯喹-琼脂糖凝胶电泳分析DNA的拓扑结构 (如图4所示)。结果显示,四环素质量浓度与生长时期对pJGX15A在DM800中的拓扑结构均具有显著的影响。
当培养基中不添加四环素时,从DM800对数生长期细胞和静止期细胞中提取的质粒DNA样品之间有很大的区别 (图4:泳道3、4)。首先,从DM800对数生长期细胞中提取的质粒DNA的超螺旋程度很高,其中大部分都表现为类似高度负超螺旋的状态。其次,从DM800对数生长期细胞中提取的质粒DNA样品中的拓扑异构体不能被电泳分离,但静止期细胞中提取的质粒DNA样品那样。在topA突变体中tetA基因的转录已经被证实可以形成高度负超螺旋[9],然而,这种不规则凝集结构的形成说明串联基因tetA-cfaABCE长距离的转录能够促使DNA产生更加复杂的结构。不规则凝集结构的形成同样受到培养基中四环素质量浓度的影响。结果显示:当提高培养基中的四环素质量浓度时,质粒DNA的超螺旋程度随之下降;并且当四环素质量浓度达到12 μg/mL时,不规则凝集结构不能形成。由于四环素是一种转录抑制剂,因此可能是由于转录速度下降导致不规则凝集结构不能形成。
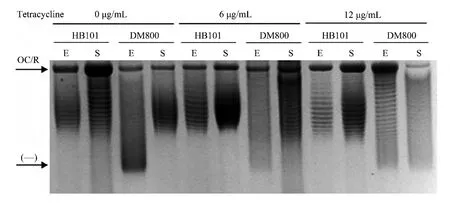
图4 四环素质量浓度与细胞生长时期对pJGX15A结构的影响Fig.4 Agarose gel electrophoresis of pJGX15A in the presence of 5 μg/mL chloroquineThe plasmid DNA samples were then isolated from cells in the exponential(E)or stationary(S)phase.OC/R,open circular or relaxed plasmid DNA;Sc,supercoiled plasmid DNA;(--),hypernegatively supercoiled plasmid DNA.
2.4 AFM观察pJGX15 A的特殊结构
为深入了解pJGX15A在DM800细胞中形成的特殊结构的结构特征及其与超螺旋DNA的区别,我们选择在不添加四环素的培养基中培养至对数期及静止期的DM800细胞中的质粒DNA样品,利用原子力显微镜技术对其结构进行观察与分析。其中使用的Mg2+浓度 (1 mmol/L)在细胞内Mg2+浓度的范围内,正如之前讨论过的一样,在这种Mg2+浓度下AFM可以给出可信的图片[21]。如图5所示,两种样品中几乎所有的DNA分子均为闭合环状分子,表明样品制备并未破坏DNA的结构。图5A中显示了DM800静止期细胞中提取的pJGX15A,DNA的结构非常均一,不含有复杂结构;大部分DNA分子呈现松弛或者1~2次互缠式结构,这可能是由于其超螺旋水平较低的缘故。而与此相反,DM800对数期细胞中提取的pJGX15A则表现出非常高的结构多样性 (图5B)。几乎所有DNA分子均表现出不同程度的凝集,大部分分子含有多个扭结 (图5B中箭头),甚至有些DNA分子完全压缩成了一团 (图5B中白色三角)。这种形态的多样性可能正是导致这些拓扑异构体不能被氯喹琼脂糖凝胶电泳分离形成梯状条带的原因。
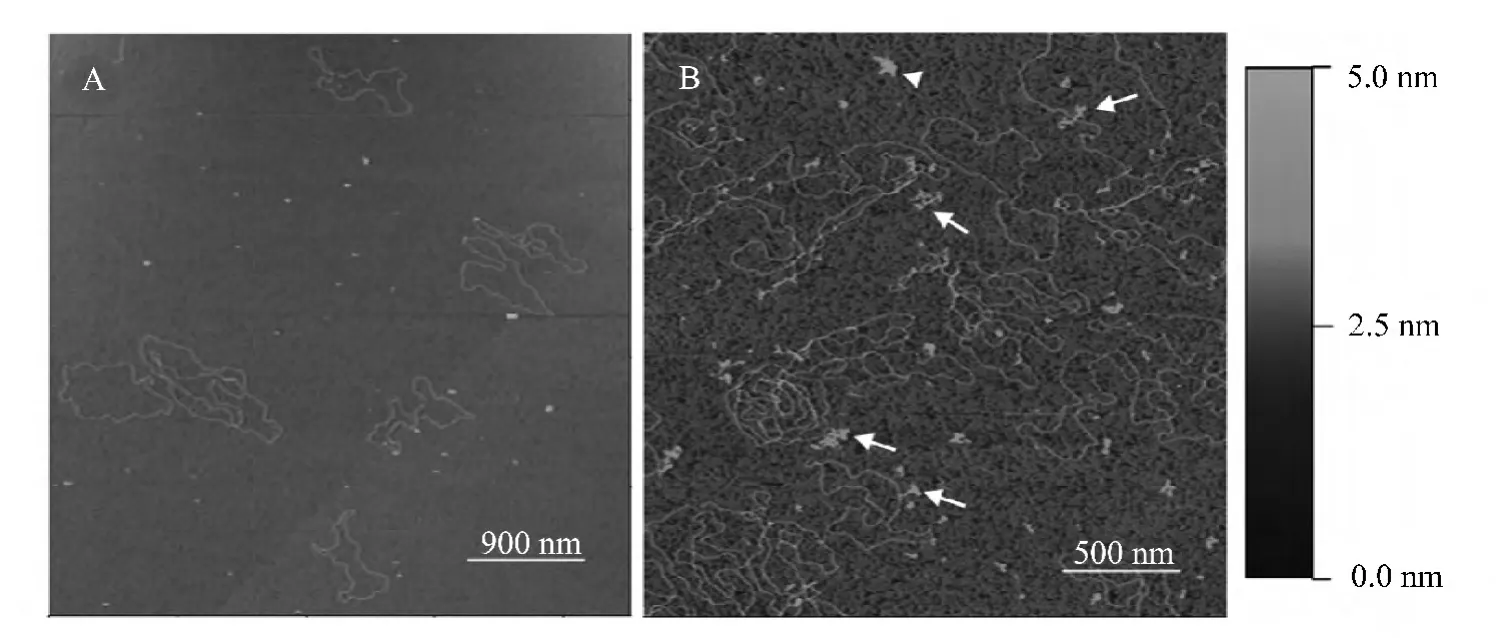
图5 AFM观察超螺旋与凝集pJGX15A的结构Fig.5 AFM images of supercoild(A)and condensed(B)pJGX15A from DM800 cells Grayscale represents 5 nm
图 6B-E中展示了几个凝集程度不同的pJGX15A分子,与图6A中的超螺旋DNA分子的结构相比较可以发现,这些发生凝集的DNA分子尺寸小于超螺旋DNA分子。图6A中超螺旋DNA分子的长度为2756 nm,与其理论计算长度2779 nm非常接近。而图6B-D中所示DNA分子的展开部分的链的长度则分别为2710、2640以及1394 nm;图6E中的结构不含有展开的DNA链且被完全压缩成了一团。这些数据表明,质粒DNA分子中的部分DNA双链确实在不同程度上被压缩成凝集结构,而且凝集程度越高,DNA分子的表观长度则越小。进一步对这些分子的高度进行了测量,结果显示所有DNA分子中展开的双链DNA的高度基本一致,约为1.1~1.3 nm,然而凝集部分的高度则有很大区别。图6E中的结构的平均高度约为3.8 nm,与图6D中DNA分子的凝集部分的高度(约为3.9 nm)相似,但是却大于图6C中DNA分子的凝集部分的高度 (约为2.1 nm)同时远远大于图6A中DNA链的高度 (约为1.2 nm)。

图6 几种典型的pJGX15A分子的结构Fig.6 AFM images of representative supercoiled(A)or condensed(B-E)structures of plasmid pJGX15A from DM800 Grayscale represents 5 nm
3 讨论
转录对DNA结构的影响已经研究了很长时间了。Liu 和 Wang[8,10]提出了转录的孪生结构域模型,而Drolet等[11-12]则提出DNA模板的高度负超螺旋结构的形成是与R-loop(s)的形成紧密联系在一起的。这些假说解释了转录是如何刺激DNA模版的高度负超螺旋结构的积累的,同时阐述了DNA拓扑异构酶I以及旋转酶在转录过程中的作用。然而,tetA基因的转录单位只有1190 bp,不足以促使DNA形成更加复杂的结构。在本实验中,我们使用大质粒pJGX15A来研究长距离转录对DNA结构的影响。该质粒包含一个tetA-cfaABCE共转录单位,这个共转录单位由tetA启动,并且新生的tetA多肽同样可以锚定在细胞膜上,这一点已经被证实对转录所引起的结构变化是非常重要的[22]。本实验发现质粒 pJGX15A在大肠杆菌topA-菌株DM800中形成一种特殊的凝集结构。这种凝集结构在氯喹-琼脂糖凝胶电泳中有着很高的迁移率而且表现为类似“弥散”的条带,这一点与正常超螺旋结构有很大的区别。AFM照片也显示这种不规则凝集结构不包含超螺旋。相反,它们包含分枝状的、高度凝集的结构 (图5B中箭号所示),部分分子甚至完全凝集成为一团 (图5B中三角所示)。这也许是这种不规则凝集结构有着很高的电泳迁移率并且拓扑异构体不能被电泳分离的原因。与此前发现的质粒pBR322所形成高负超螺旋结构相比,pJGX15A在DM800细胞中形成的凝集结构更加的复杂,说明长转录单元对DNA结构产生了巨大的影响。
本实验中这种凝集结构在大肠杆菌HB101细胞中并没有发现,说明pJGX15A凝集结构的产生是依赖于拓扑异构酶I的缺陷的。同时,这种凝集结构的形成在很大程度上受到细胞生长状态和培养基中四环素质量浓度的影响:①该凝集结构仅在DM800对数生长期细胞中形成,而当细胞进入静止期时,pJGX15A表现为超螺旋结构;②pJGX15A的凝集结构仅在低四环素质量浓度 (小于6 μg/mL)培养的DM800细胞中产生,高四环素质量浓度培养时,对数期与静止期DM800细胞中的pJGX15A均表现为超螺旋结构。由于进入静止期和增加培养基中的四环素质量浓度都会降低转录的效率,因此推测这种不规则凝集结构的生成依赖于高效的转录。
此前有实验室从大肠杆菌 HB101(topA+,gyrB+)细胞中发现了某种可变的不规则结构[23],具有这种结构的DNA同样不能被电泳分开成梯状拓扑异构体条带。AFM的结果显示该结构中也含有凝集的结构,与本实验中发现的凝集结构有相似之处。但进一步比较二者的结构参数可以发现这种不规则结构与本研究中发现的凝集结构相比,其凝集程度明显较低。这一点也与其超螺旋水平明显低于本研究中发现的凝集结构这一结果相符合。这些结果说明DNA凝集结构可以在大肠杆菌野生株中产生,而拓扑异构酶I的缺失可能促进了DNA凝集的累积。
本实验的结果表明,当大肠杆菌细胞中拓扑异构酶I缺失时,长转录单元的高效转录可能促使质粒DNA形成高度凝集的结构 (highly condensed structure)而非高度负超螺旋结构 (hypernegative supercoiled structure)。鉴于细菌染色体上存在很多大的基因或基因簇,这种结构的形成有可能参与细菌染色体DNA的组织包装。
[1]TSE-DINH Y C,QI H,MENZEL R.DNA supercoiling and bacterial adaptation:thermotolerance and thermoresistance[J].Trends in Microbiology,1997,5:323-326.
[2]CHRISTIANSEN K,WESTERJAARD O.Characterization of intra-and intermolecular DNA ligation mediated by eukaryotic topoisomerase I[J].Journal of Biological Chemistry,1994,269:721-729.
[3]FROELICH-AMMON S J,GALE K C,OSHEROFF N,et al.Site-specific cleavage of a DNA hairpin by topoisomerase II[J].Journal of Biological Chemistry,1994,269:7719-7727.
[4]GLLERT M,MIZUUCHI K,O’DEA M H,et al.DNA gyrase:an enzyme that introduces superhelical turns into DNA[J].Proceedings of the National Academy of Sciences USA,1976,73:3872-3876.
[5]PRUSS G J.DNA topoisomerase I mutants:increased heterogeneity in linking number and other replicon-dependent changes in DNA supercoiling[J].Journal of Molecular Biology,1985,185:51-63.
[6]THOMAS B,NIELSEN P E.In vitro transcription of a torsionally constrained template[J].Nucleic Acids Research,2002,30:803-809.
[7]LENG Fenfei,MCMACKEN R.Potent stimulation of transcription-coupled DNA supercoiling by sequence-specific DNA-bingding proteins[J].Proceedings of the National Academy of Sciences USA,2002,99:9139-9144.
[8]LODGE J K,KAZIC T,BERG D E.Formation of supercoiling domains in plasmid pBR322[J].The Journal of Bacteriology,1989,171:2181-2187.
[9]PHOENIX P,RAYMOND M A,MASSE E,et al.Roles of DNA Topoisomerases in the Regulation of R-loop formation in vitro[J].Journal of Biological Chemistry,1997,272:1473-1479.
[10]LIU L F,WANG J C.Supercoiling of the DNA template during transcription[J].Proceedings of the National A-cademy of Sciences USA,1987,84:7024-7027.
[11]MASSÉ E,DROLET M.R-loop-dependent hypernegative supercoiling in Escherichia coli topA mutants preferentially occurs at low temperatures and correlates with growth inhibition[J].Journal of Molecular Biology,1999,294:321-332.
[12]USONGO V,NOLENT F,SANSCARTIER P,et al.Depletion of RNase HI activity in Escherichia coli lacking DNA topoisomerase I leads to defects in DNA supercoiling and segregation[J].Molecular Microbiology,2008,69:968-981.
[13]BAAKLINI I,USONGO V,NOLENT F,et al.Hypernegative supercoiling inhibits growth by causing RNA degradation[J].The Journal of Bacteriology,2008,190:7346-56.
[14]LENG Fenfei,AMADO L,MCMACKEN R.Coupling DNA supercoiling to transcription in defined protein systems[J].Journal of Biological Chemistry,2004,279:47564-47571.
[15]BROCCOLI S,RALLU F,SANSCARTIER P,et al.Effects of RNA polymerase modifications on transcription-induced negative supercoiling and associated R-loop formation[J].Molecular Microbiology,2004,52:1769-1779.
[16]SAMUL R,LENG Fenfei.Transcription-coupled hypernegative supercoiling of plasmid DNA by T7 RNA polymerase in Escherichia coli topoisomerase I-deficient strains[J].Journal of Molecular Biology,2007,374(4):925-935.
[17]ZHI Xiaoduo,LENG Fenfei.Dependence of transcription-coupled DNA supercoiling on promoter strength in Escherichia coli topoisomerase I deficient strains[J].Gene,2013,514:82-90.
[18]DING Yue,MANZO C,FULCRAND G,et al.DNA supercoiling:a regulatory signal for the λ repressor[J].Proceedings of the National Academy of Sciences U S A,2014,111:15402-15407.
[19]张臻峰,于春风,万庆,等.产毒性大肠杆菌CFA/Ⅰ基因表达与DNA超螺旋状态的关系[J].南开大学学报:自然科学版,2004,37:7-12.
[20]HUANG Xitai,CHEN Xin.Supercondensed structure of plasmid pBR322 DNA in an Escherichia coli DNA topoisomerase II mutant[J],Journal of Molecular Biology,1990,216:195-199.
[21]ZHANG Pingcheng,BAI Chunli,CHENG Yingjun,et al,Study on the topological structure of pBR322 DNA by AFM[J].Science in China(Series C:Life Sciences),1996,39:1-7.
[22]LYNCH A S,WANG J C.Anchoring of DNA to the baterial cytoplasma memberane through cotranscriptional synthesis of polypeptide encoding membrane protein or proteins for export-a mechanism of plasmid hypernegative supercoiling in mutants deficient in DNA topoisomerase I[J].The Journal of Bacteriology,1993,175:1645-1655.
[23]ADAMĈÍK J,VÍGLASKY V,VALLE F,et al,Effect of bacteria growth temperature on the distribution of supercoiled DNA and its thermal stability[J].Electrophoresis,2002,23:3300-3309.
