基于实体词典与机器学习的基因命名实体识别*
2015-03-14夏光辉李军莲阮学平
夏光辉 李军莲 阮学平
(中国医学科学院医学信息研究所 北京 100020)
基于实体词典与机器学习的基因命名实体识别*
夏光辉 李军莲 阮学平
(中国医学科学院医学信息研究所 北京 100020)
将实体词典以特征的形式引入到机器学习模型中,提出一种基于实体词典与机器学习的基因命名实体识别方法,在GENIA 3.02语料上进行实验。测试结果表明引入实体词典特征后,在获得较高实体识别准确率的同时,优化CRFs识别模型的时间复杂度,提高系统识别效率。
实体词典; 机器学习; 基因命名实体; 命名实体识别
1 引言
现阶段计算机的广泛普及以及互联网技术的快速发展,使得信息的采集和传播变得简便、快捷,大量的信息开始以惊人的速度涌现,从而导致了“信息爆炸”现象产生。为了应对“信息爆炸”所带来的严峻挑战,人们迫切需要利用自动化工具以便于能够迅速而准确地从海量的信息资源中找寻最相关的信息,命名实体识别(Named Entity Recognition)正是为了应对这种挑战,满足信息处理时的需求而产生的。命名实体识别是自然语言处理中的核心技术,也成为自然语言处理的一个主要方向,在信息提取、信息检索、主题分类、知识发现等方面具有重要应用。生物医学的迅速发展,特别是2001年人类基因组工程草图的发表,与生物医学领域相关的科学数据呈指数级别增长,各种形式的生物医学文献和文本信息也迅速增长,这些文献数据隐藏着丰富的生物医学知识,因此,如何让生物医学研究人员从海量的相关文献中便捷地捕获生物医学信息变得迫在眉睫。基因、蛋白质等是生物体的主要组成部分,同时也是生命科学研究的主要对象,从医学文献中抽取基因、蛋白质等实体名称进一步发现它们之间的作用和关系具有非常重要的意义。基因命名实体是指遗传学领域具体的或抽象的实体,如基因名、DNA名、RNA名等。通常情况下,基因名称和蛋白质名称是一致的,只是具体的实例有区别;在文献中,作者经常也不会对基因和蛋白质作严格的区分;有的研究表明,当文献中出现的基因、蛋白质以及mRNA等名称时,即使是生物医学领域的专家,其正确区分基因和蛋白质实体的一致率也只有78%[1]。因此,本研究所指的基因命名实体实际上包括了基因和蛋白质两类命名实体。基因命名实体识别方法包括基于词典的方法、基于规则的方法等,由于基因命名实体名称的复杂性和多样性,目前基因命名实体识别的总体效果要比新闻领域等通用命名实体识别的准确性低很多。本文尝试基于词典与机器学习相结合的方法进行基因命名实体识别,以改进其准确性和实用性。
2 实体词典构建与机器学习实体特征构建
2.1 概述
基于词典的基因命名实体识别方法中,词典是核心,词典的完备程度对基因命名实体识别效果具有决定性作用;而基于机器学习的基因命名实体识别方法,需要构建基因命名实体的各种独特特征,通过统计语料中各种特征的出现频率,计算其作为基因命名实体的条件概率,最终对命名实体的类型做出预判。因此,实体词典生成与机器学习实体特征构建既是本文提出的基于词典与机器学习的基因命名实体识别方法的基础,也是基于词典与机器学习的基因命名实体识别过程的关键步骤。
2.2 实体词典构建
从美国国立医学图书馆研究和开发的医学一体化语言系统(Unified Medical Language System,UMLS)的133种语义类型中选择“Gene or Genome”、“Nucleic Acid, Nucleoside, or Nucleotide”、“Amino Acid, Peptide, or Protein”3种语义类型抽取与基因、蛋白质相关的术语作为基因实体词典的来源[2]。具体术语量,见表1。

表1 词典信息
2.3 机器学习实体特征构建
2.3.1 概述 实体特征是指基因文本中能正确区分基因实体的字符特征,特征构建是否合理、有效,直接关系到基因命名实体能否被正确地识别。实体特征能够准确地表征命名实体的特点,为命名实体的识别提供有效信息。由于基因命名实体的独特特点,当前已有很多研究者提出了各种各样的特征,而基于统计的机器学习模型的识别效果依赖于特征的质量和数量。本文通过对文献中基因命名实体的特点进行分析,结合目前在生物医学实体识别领域构建的特征类型[3-6],构建了13大类基因命名实体的特征。
2.3.2 单词特征(Word Features) 单词是文本自动分析和实体标注的基本单位,单词特征能够反映基因命名实体的语言信息,是基因命名实体识别最核心、最重要的特征。
2.3.3 构词特征(Word Structure Feature) 本文根据当前词是否由大小写字母、数字、连字符(-和/)、希腊字母、罗马数字、引号、括号等字符组成构建了构词特征,共包括18种子特征,以此来识别文本中当前词是否为基因命名实体。18种构词子特征,见表2。

表2 构词特征的18种子特征
2.3.4 关键词特征(Keywords Feature) 关键词是指在基因命名实体中出现频率较高的单词。通过判断当前词是否为关键词,可以识别可能出现在当前词附近的命名实体。
2.3.5 词缀特征(Affix Feature) 词缀是一种附着在词根或词干的语素,为规范词素,不能单独成字。黏附在词根前面的词缀称为前缀,黏附在词根后面的词缀称为后缀。在基因命名实体中,同一类物质一般会有相同的前后缀,如一般蛋白质名称都是以“ase”结尾。
2.3.6 词形特征(Morphlogy Feature) 基因命名实体是一类特异性非常高的命名实体,其通常具有相同的词形。因此,根据词形特征可以判别当前词是否属于基因命名实体。目前通用的词形特征表示方法是将大写字母替换为A,小写字母替换为a,数字替换为0,其他字符替换为x。
2.3.7 边界词特征(Boundary Word Feature) 边界词是指命名实体的第一个和最后一个单词。大部分基因命名实体是由多词组成的,利用边界词信息可以提高边界识别能力,减少复合性基因命名实体的识别错误率。
2.3.8 一元词特征(Unary Feature) 基因命名实体中存在大量仅由一个单词构成的实体,即一元词,如IGF2、IL-2A等。以一元词是否出现作为特征,可为当前词是否为基因命名实体提供准确、有效的信息。
2.3.9 嵌套词特征(Nested Feature) 词与语素按一定规则组合起来构成的合成词即为复合词。在本文中,包含了嵌套结构的基因实体都是复合词,即此类基因命名实体的组成部分也是一个独立的基因命名实体,如基因命名实体“NF-kappaB element”中包含基因命名实体“NF-kappaB”,这种嵌套结构增加了实体边界的识别难度。本文将基因命名实体中的嵌套结构单独标识出来,作为嵌套词特征识别基因命名实体,以减少命名实体边界识别的错误率。
2.3.10 停用词特征 (Stop Word Feature) 在信息检索中,为节省存储空间,提高搜索效率,在处理自然语言数据(或文本)之前或之后会自动过滤掉某些字或词,这些字或词即被称为停用词。在英文中,存在一部分单词是没有实际意义的,如“a”、“was”、“can”等这类词虽然出现频率较高,但是会严重影响搜索引擎的查准率,并降低搜索引擎的检索效率。在在遗传学领域,这类停用词对命名实体识别同样会带来负面影响,因此可以将文本中的停用词作为特征,减少识别过程中无用信息的干扰。
2.3.11 通用词特征(Common Word Feature) 通用词是指使用频率比较高、单词本身也具有实际意义,但是在各个专业领域都通用的单词。这类词不能反映基因领域的独特特点,也不是基因命名实体的组成部分,因此基因命名实体识别时意义不大,可以忽略这类词。
2.3.12 上下文特征(Context Feature) 上下文信息是指基因实体前一个词和后一个词的单词信息,利用上下文信息可以提高基因实体边界识别能力。
2.3.13 词性特征(Part of Speech Feature) 词性指作为划分词类的根据的词的特点,英语词汇可分为名词、动词、代词、形容词、副词、数词、冠词、介词、连词、感叹词等词性,通过词性特征有助于识别命名实体。自然语言处理中,一般利用词性标注器对文本进行词性标注,目前生物医学领域常用的词性标注器包括Stanford POS
tagger[7]、MedPost[8]、GENIA tagger[9]等,其中GENIA Tagger的训练语料由新闻领域的Wall Street Journal语料以及生物医学领域的GENIA语料和PennBiolE语料组成,对生物医学文献的词性标注效果较好,因此本文实验中也采用GENIA Tagger[10]工具包来获取单词的词性。
2.3.14 词典特征(Dict Feature) 传统基于词典的命名实体识别是在识别过程中完全依赖词典,一般使用不同的词典匹配方式在所构建的词典中查找字符串。本文是以机器学习模型作为基因命名实体识别的主要方法,而在识别过程中,将词典以特征的形式引入到机器学习模型当中。因此,本文基于基因实体词典构建了词典单词特征、词典一元词特征和词典嵌套词特征。
3 实体标注实现流程
本文是将外部词典以特征的形式引入机器学习方法中,基于词典和统计机器学习相结合的方法识别基因命名实体的实现流程,见图1。图1中上面的实框内表示的是构建词典特征的过程。首先构建基因实体识别所需要的词典资源;然后参照条件随机场(Conditional Random Fields, CRFs)识别模型的语料格式,对词典资源进行格式转换并提取特征,形成词典特征集合;最后将词典特征集合作为特征加入到训练语料进行训练获得识别模型。图1中下面的虚框表示的是基于CRFs的基因命名实体识别过程。首先将GENIA 3.02语料库转换为纯文本格式,按照特征规则提取语料的多维特征值;然后将词典特征集合加入训练语料中,结合语料中提取的特征生成多特征的基于CRFs的基因命名实体识别模型;最后用生成的模型标记测试语料完成基因命名实体识别任务。

图1 实体识别流程
4 结果与分析
4.1 评测指标
采用准确率P(Precision)、召回率R(Recall)和F测评值(F-measure) 对实验结果进行评估。准确率和召回率是命名实体识别领域常用的系统评测指标,其中准确率衡量正确识别的基因命名实体占所有识别出的基因命名实体的比例,召回率衡量正确识别的基因命名实体占评测语料中标注的所有命名实体的比例。准确率和召回率是相互矛盾、相互对立的两个评测指标,一般而言,准确率升高,召回率降低;召回率升高,准确率降低。因此,通常采用二者的综合加权指标F测评值来评估识别性能。准确率、召回率和F测评值的计算公式如下:

(1)

(2)
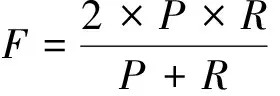
(3)
式中,P表示基因命名实体识别的正确率;R表示召回基因命名实体的能力;TP(True Positives)表示正确地识别为基因命名实体的数目;FP(False Positives)表示错误地识别为基因命名实体的数目;FN(False Negative) 表示错误地识别为非基因命名实体的数目。
4.2 基因实体识别的特征选择
命名实体识别系统需要构建丰富的特征集合以准确识别文本中的基因命名实体,选用的特征越具有基因命名实体的独特性,就越能提高基因命名实体识别系统的识别能力;但是选择的特征越多,系统识别的时间复杂度就越大。由于特征之间相互耦合,实际上并不是构建的所有特征都能够提高命名实体的识别能力,不合适的特征组合不仅无法区分基因命名实体和非基因命名实体,反而会降低单一特征对基因命名实体的识别能力,导致基因命名实体识别系统的识别性能下降。因此,本文尝试通过单独最优特征组合法,按识别性能的高低依次选取特征,构建一个数量少、质量高、时间复杂度合适的特征集合,以提高CRFs模型的识别效果。
4.3 基于机器学习的基因命名实体识别结果
本文实验中,依据单独最优特征组合法,选取 [F0(单词特征)、F33(词性特征)、F31(通用词特征)、F23(四字符后缀特征)、F22(三字符后缀特征)、F5(数字字母顺序组合)、F30(停用词特征)、F7(包含连字符)、F3(大小写字母组合)、F21(四字符前缀特征)]10个特征识别系统可以得到最大的F测评值(80.56%)。因此,由这10个特征构建的特征集合是单独最优特征组合法的最优特征集合,见表3。

表3 单独最优特征组合法的最优特征集合(%)
实验中,分别构建所有特征模板和最优特征模板,并分别处理训练语料和测试语料,用不同的特征集合构建的识别系统的时间复杂度,见表4。可知,利用构建的最优特征集合,不但系统性能提高了1.19%,达到了80.56%,而且时间复杂度大大降低,这充分体现了特征选择对机器学习识别模型的重要性。
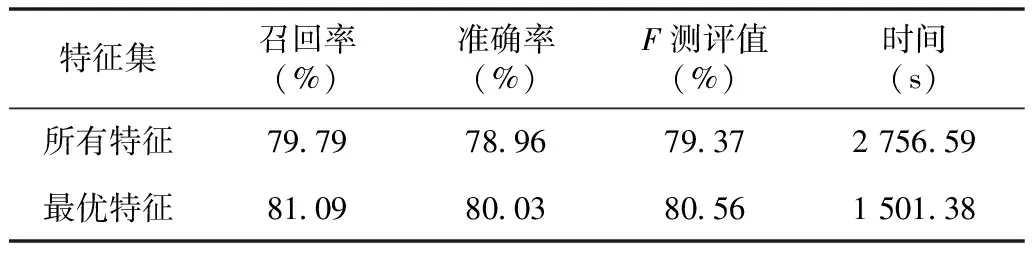
表4 不同特征集合的时间复杂度比较
4.4 基于实体词典和机器学习相结合的基因命名实体识别结果
本文主要研究的问题是将基于统计的机器学习方法和基于词典的方法相结合应用于基因命名实体识别领域。本文试验中,在特征集合中加入词典单词特征、词典一元词特征和词典嵌套词特征,分别计算单词特征与词典的3个特征联合的识别效果。各词典特征的实验结果,见表5。可见与单独考虑单词特征相比,3个词典特征加入后,都能在一定程度上提升基因命名实体识别的性能。本实验中,对新加入的词典特征仍按照单独最优特征组合法重新构建最优特征集合,最终构建的最优特征集合对应的6个特征为[F0(单词特征)、F33(词性特征)、F36(词典单词特征)、F31(通用词特征)、F23(四字符后缀特征)、F22(三字符后缀特征)]。加入词典特征训练得到的CRFs统计学习模型对测试语料做出预测,得到的实验结果,见表6。

表5 词典特征的识别结果(%)
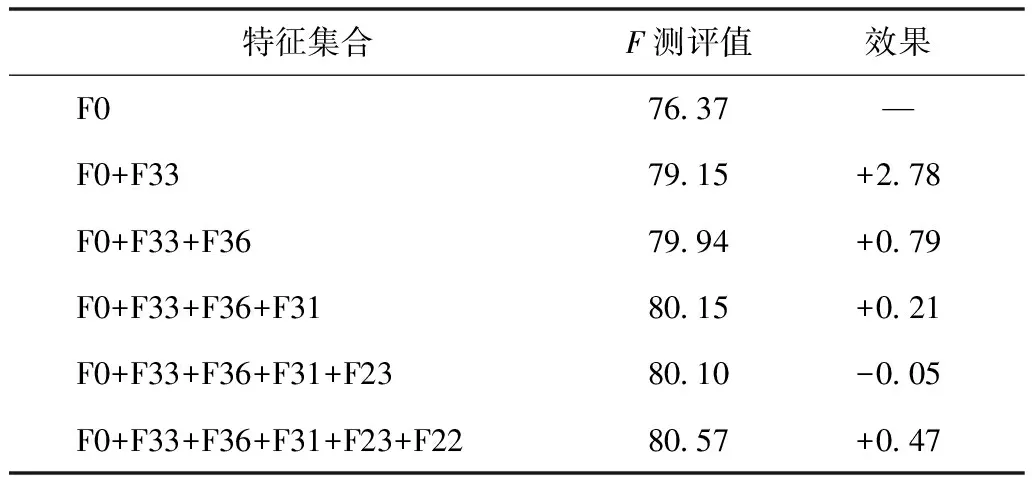
表6 加入词典特征的最优特征集合(%)
由表7可见,加入词典特征后,CRFs识别模型的识别收敛速度有明显的提升,只需要考虑[F0、F33、F36、F31、F23、F22]6个特征,CRFs识别模型就能获得较高的F测评值,超过了不加入词典特征时取得的最高F测评值,这在一定程度上优化了CRFs识别模型的时间复杂度,见表7,为CRFs识别模型从小规模的实验测试走向大规模工程化应用提供了条件。

表7 最优特征集合的时间复杂度比较
5 结语
近几年来,虽然基因命名实体识别在语料库构建、词典构建、特征构建、识别方法等方面取得了一定的进展,但由于基因命名实体的构词形式复杂多样,要使系统的识别性能达到可应用的程度仍面临着巨大挑战。因此,后续研究中可以在以下几方面进一步探讨:(1)构建规模更大、质量更高的语料库。机器学习方法主要是通过统计训练语料来得到相关参数并建立模型,因此语料库所含基因实体越多、语料库质量越高,建立的模型识别效果越好。 (2)提取深层次的实体特征,研究高效的特征表示方法。目前选取的单词特征、词性特征等只是对命名实体名称或语法成分的一种匹配,只用到了表层的文本信息,无法有效地识别句子中隐含的实体信息。在后续研究中,应更注重利用文本中的句法知识等深层次的信息,提取文本中命名实体的共指特征,从而提高系统识别命名实体的能力。(3)研究词典与机器学习方法更优的结合机制。基于词典是命名实体识别的一种比较简单的方式,完备的词典可提高系统识别己知命名实体的能力。因此,一方面可以通过词语原型化工具改进词典匹配算法,以降低英文单词的词形变化对词典特征构建的影响;另一方面还需要基于词典构建更多的特征加入到机器学习方法中,以减少机器学习模型对语料库的依赖,从而为基因命名实体识别系统从理论探索走向实际应用提供条件。
1 Hatzivassiloglou V, Duboue′ PA, Rzhetsky A. Disambiguating Proteins, Genes and RNA in text: a machine learning approach[J]. Bioinformatics, 2001, 1(1):1-10.
2 National Center for Biotechnology Information, U.S. National Library of Medicine. Semantic Network-UMLS®Reference Manual[EB/OL].[2015-02-10].http://www.ncbi.nlm.nih.gov/books/NBk9679/.
3 王琦.词典和机器学习相结合的生物命名实体识别[D].大连:大连理工大学,2009.
4 郑强.生物医学命名实体识别研究[D]. 长沙:国防科学技术大学,2009.
5 黄浩炜.SVM与基于转换的错误驱动学习方法相结合的生物实体识别[D]. 长沙:国防科学技术大学,2007.
6 周荣鹏. 生物医学文献中命名实体的识别[D]. 大连:大连理工大学,2009.
7 The Stanford Natural Language Processing Group. Stanford Log-linear Part-of-Speech Tagger[EB/OL].[2015-02-15].http://nlp.stanford.edu/software/tagger.shtml.
8 Smith L,Rindflesch T,Wilbur W J. MedPost: a part-of-speech tagger for bioMedical text[J].Bioinformatics,2004,20(14):2320-2321.
9 Tsuruoka Y,Tateisi Y,Kim J D, et al. Developing a Robust Part-of-Speech Tagger for Biomedical Text[J].Advances in Informatics Lecture Notes in Computer Science,2005, (374): 382-392.
10 Department of Information Science, Faculty of Science, University of Tokyo.GENIA Tagger:part-of-speech tagging, shallow parsing, and named entity recognition for biomedical text[EB/OL].[2015-02-15]. http://www.nactem.ac.uk/GENIA/tagger.
Gene Named Entity Recognition Based on Entity Dictionary and Machine Learning
XIA Guang-hui, LI Jun-lian, RUAN Xue-ping,
Institute of Medial Information, Chinese Academy of Medical Sciences, Beijing 100020, China
By introducing the entity dictionary into the model of machine learning in the form of characteristics, this article proposes a method of gene-named entity recognition based on entity dictionary and machine learning and experiments on corpus GENIT 3.02. As indicated by the test results, after the characteristics of the entity dictionary are introduced, while a higher accuracy rate of entity recognition is obtained, the time complexity of CRFs recognition model is optimized and the system's recognition efficiency is enhanced.
Entity dictionary; Machine learning; Gene named entity; Named entity recognition
2015-11-13
夏光辉,助理研究员,硕士,主要研究方向为医学知识组织建设与利用、医学文本信息检索与处理,发表论文20篇。
国家科技支撑计划项目(项目编号:2011BAH10B05)。
R-056
A 〔DOI〕10.3969/j.issn.1673-6036.2015.12.012
