质粒介导的氟喹诺酮类抗生素耐药机制研究进展
2014-11-23孔令聪刘树明马红霞
王 梓,孔令聪,高 铎,刘树明,马红霞,2*
(1.吉林农业大学动物科技学院,长春130118;2.吉林农业大学动物生产及产品质量安全教育部重点实验室,长春130118)
氟喹诺酮类药物作为广谱抗菌药自1962年投入临床使用至今已有50余年历史,已成为临床中最为常用的抗生素之一。氟喹诺酮类药物属于浓度依赖型抗生素,致病菌如果长期处于其亚抑菌浓度时极易产生突变成为耐药菌[1-3]。作为氟喹诺酮类药物的主要耐药机制之一,质粒介导的氟喹诺酮类药物耐药(Plasmid-mediated quinolone resistance,PMQR)具有传播速度快、基因亚型较多等特点,且其携带的耐药基因可对细菌染色体上氟喹诺酮类药物耐药基因进行靶位修饰,使靶位突变频率增加[4-5],严重影响氟喹诺酮类药物的使用寿命。故探索质粒介导的氟喹诺酮类耐药机制对于控制耐药基因的快速传播,减少耐药菌的危害具有重要意义。
1 质粒介导的氟喹诺酮类药物耐药基因的发现
现有研究表明,质粒携带的氟喹诺酮类药物耐药基因主要有3种,分别为包含有多种亚型的qnr基因、介导主动外排的基因、发生点突变的氨基糖苷乙酰转移酶基因。其中,Martínez-Martínez等在研究肺炎克雷伯菌天然携带的pMG252质粒时,首次发现了五肽重复序列qnr基因[6],随后陆续报道了多种qnr基因亚型[7-9]。此后,在对 qnr基因的作用机制进行研究时发现,qnr基因既能介导氟喹诺酮类药物耐药,还可以通过增加gyrA、parC等靶位基因的突变频率,使细菌耐药能力增强[10]。各qnr基因亚型概况如表1所示。随后,Haru Kato等[11]在日本确证了由质粒介导的外排基因QepA,在该试验中发现,QepA基因能够介导细菌对诺氟沙星的外排作用,且其外排可以被外排泵抑制剂CCCP(carbony cyanide m-chlorophenyl-hydrazone)抑制,为质子依赖型外排泵。由此推测,QepA基因所介导的外排作用可能是质粒介导的氟喹诺酮类药物耐药的重要途径之一。G.A.Jacoby等在研究中又发现了另一种PMQR基因aac(6')-Ib-cr,该基因由氨基糖苷乙酰转移酶基因发生Trp102Arg和Asp179Tyr点突变获得,其可介导大肠杆菌对环丙沙星的抗性[12],该基因自发现以来在氟喹诺酮耐药菌株中传播迅速,在三类PMQR基因中检出率较高[13-15]。
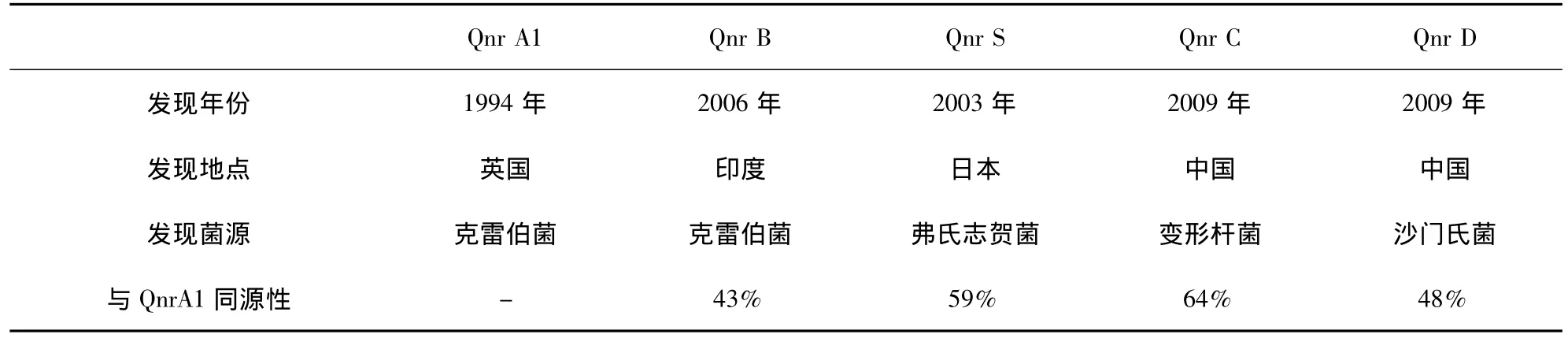
表1 qnr基因各亚型概况
2 质粒介导的氟喹诺酮类药物耐药基因的分布
现有流行病学调查结果发现,多种PMQR基因常共存于同一耐药质粒中。Kees Veldman等在欧洲13个国家分离出的沙门氏菌和大肠杆菌中分别检测出 qnrA1、qnrB、qnrD、aac(6’)-Ib-cr基因,其中 qnrD基因为在欧洲出现的首次报道[16];Ya-Hong Liu等在广东省分离的大肠杆菌中分别检测出 qnrB、qnrS、aac(6’)- Ib-cr、QepA 等基因[17];Patricia Andres等在南美洲分离的百余株肠杆菌中也扩增出 qnrB、qnrS、aac(6’)-Ib-cr等氟喹诺酮耐药基因[18]。由于单独的PMQR基因通常只介导低水平耐药,故耐药菌常通过多种耐药基因的共同作用以形成高水平耐药性。近年来,由于动物运输、耐药质粒的转移等因素,PMQR基因已经在世界范围内广泛分布,伴随着PMQR基因突变的不断发生,未来将会有更多的PMQR基因耐药亚型不断出现。同时,携带氟喹诺酮类药物耐药基因的质粒也可能携带其他多种耐药基因,报道最多的为与广谱β-内酰胺酶基因共存。通过多种类耐药基因在同一质粒上的整合即可形成多重耐药质粒,而且多重耐药质粒的分布范围也在逐步扩大,在沙门氏菌、克雷伯菌、变形杆菌中均发现多重耐药质粒的存在[19-20],但质粒携带的多种耐药基因的表达水平是否可以相互影响,还需进一步研究验证。
3 质粒介导的氟喹诺酮类药物耐药机制
3.1 PMQR基因的传播机制 携带PMQR基因的耐药质粒快速传播是目前氟喹诺酮类药物耐药性增强的重要因素之一。质粒上携带的PMQR基因可以直接通过质粒转移实现基因的相互传递,也可利用质粒中整合子、转座子的传递实现PMQR基因的快速传播。Humberto Barrios等将携带β-内酰胺耐药基因(CTX-M-15)和多个氟喹诺酮耐药基因(qnrA1、qnrB2、aac(6’)-Ib-cr)的质粒在不同温度下进行接合转移实验:结果在受体菌中发现了两个不同的质粒,其中大小为300 kb的质粒pSZ300中携带了 CTX -M -15、qnrA1、aac(6’)-Ib-cr基因,并产生氟喹诺酮类药物抗性;而大小为50 kb的质粒pSZ50中仅发现有qnrA1基因,测序结果显示 pSZ50包含于质粒 pSZ300中[21]。此试验证明,质粒在水平传播过程中能发生重组,从而导致耐药质粒多样化。Be'la Kocsis等首次发现在肺炎克雷伯菌的质粒ST147中qnrS1可与窄谱β-内酰胺酶LAP-2基因共存,并进一步通过质粒接合转移实验获得了一个97 kb质粒,其同时携带有供体菌上的上述两种耐药基因,进而使受体菌对头孢他啶、环丙沙星等药物的MIC值均提高了2倍以上[22]。如果细菌的染色体上存在耐药基因,则质粒介导的PMQR机制可促使细菌产生更高水平的耐药,给临床治疗带来困难。通过类似多药耐药转移质粒的横向传播,将会使不同菌属细菌间形成复杂的高水平多重耐药机制。而设法限制耐药质粒的快速传播,将成为减缓细菌对氟喹诺酮类抗菌药的主要途径之一。
3.2 PMQR基因表达的调控机制 目前有关QepA、aac(6’)-Ib-cr基因的研究多集中于流行病学调查中耐药基因的种类检测,关于影响PMQR基因调节和表达的报道还较少。最近的研究证实,细菌的SOS反应是调节qnr基因表达量的重要因素之一。SOS反应是细菌在DNA受到持续损伤压力下产生的一种自救反应,可以由LexA、RecA等基因调控,能够增加细菌耐药基因的突变频率。近年来已有学者相继报道了多种qnr基因可以在细菌发生SOS反应时发生表达量的改变。其中,David C.Hooper等通过改变温度增加细菌的生存压力使其产生SOS反应的实验结果发现,受试菌的qnrB基因表达量增加,进而对环丙沙星的耐受能力明显增强,对qnrB基因进一步基因环境研究表明,qnrB基因的上游序列中存在的LexA基因可对细菌DNA的持续损伤压力产生应答,从而引发细菌的SOS反应提高qnrB的表达量[23]。随后,A.Briales等发现qnrD基因以及一个新型染色体携带的Smaqnr基因都拥有相似的LexA结构,可以在不同浓度的环丙沙星、莫西沙星等药物作用下将上述qnr基因表达量提高 1.5 ~16 倍[23-24]。上述实验结果证明,通过温度改变、药物作用等方式增加细菌生存压力时,可以选择性的诱发细菌的SOS反应,继而使含有LexA结构的qnr基因发生表达量的改变,使细菌产生更强的耐药性。目前,有关三种PMQR基因表达机制的深入探索还有待进一步的研究。
3.3 PMQR基因的作用机制 在三种PMQR基因中,QepA的作用机制主要通过主动外排系统减少药物在细菌菌体内的聚集,其外排泵结构与MFS外排泵超家族极为相似,为质子依赖型外排泵,细菌可依靠其外排作用提高对诺氟沙星、环丙沙星等药物的MIC值2倍以上,并能在其他耐药基因共同作用下使细菌产生更强耐药性[11]。aac(6’)-Ib-cr的作用机制为,当 aac(6’)-Ib基因的102位色氨酸突变为组氨酸及179位天冬氨酸突变为酪氨酸共同发生时,即可将环丙沙星与细菌结合位点乙酰化,从而使其失去对细菌的抑制作用。细菌存在aac(6’)-Ib-cr基因的同时,还可增加染色体上相关耐药基因的突变频率,从而增强细菌的耐药性。而当QepA基因与aac(6’)-Ib-cr基因共存与耐药质粒上时,可使细菌对环丙沙星的耐药性较qnrA基因单独作用时提高4倍以上,达到药物敏感性折点[25]。而目前有关qnr基因作用机制尚未完全阐明,其机制的探索主要参考其他五肽重复蛋白(pentapeptide repeat-containing proteins),如染色体上同样可以介导氟喹诺酮类药物耐药的McbG和MfpA蛋白。编码McbG和MfpA的基因与qnr基因具有19.6%和18.9%的同源性,其主要耐药机制是通过保护DNA促旋酶和抑制药物对DNA的超螺旋作用[26-27]。近年来新发现了两种新型染色体携带的 qnr-like基因,即 EfsQnr和 Smaqnr。其作用机制与MfpA较相似,可以抑制ATP依赖型DNA超螺旋,从而产生对氟喹诺酮类药物的耐药性;而Smaqnr基因与qnrB1基因具有高达80%的同源性,是迄今为止发现的同源性最高的qnr-like基因[28-29]。通过对这些染色体上携带的qnr-like基因作用机制的深入研究,可以逐步探索质粒携带qnr基因的作用机制及遗传环境,为质粒介导的氟喹诺酮类药物耐药机制的进一步阐明及在根本上限制细菌的耐药性奠定基础。
4 展望
随着质粒介导的氟喹诺酮类药物耐药机制研究的不断深入,人们对细菌氟喹诺酮类耐药性的产生及传播有了更全面的认识。但同时,细菌的耐药性也日趋复杂,近期研究显示,质粒介导的 aac(6’)-Ib-cr基因在染色体中也被发现[25],这说明细菌正通过改变自身的耐药途径来增加人们控制其耐药性的难度。在未来的研究中,可能还会有更多耐药基因在染色体和质粒中相互整合,形成复杂的耐药环境。因此,在规范临床合理用药的同时,利用现有细菌耐药机制研究的深层成果,通过基因敲除、反义RNA等生物学方法控制耐药质粒的快速传播、抑制耐药基因的表达,将是未来控制细菌耐药性的有效途径之一。同时,对于细菌耐药机制的研究也应继续进行,阐明耐药基因在染色体与质粒中的整合途径及影响方式等未知耐药机制。相信经过广大科研工作者的不断努力,一定能够有效控制细菌耐药性,为人类的生命安全提供保障。
[1]Veldman K,Cavaco L M,Mevius D,et al.International collaborative study on the occurrence of plasmid-mediated quinolone resistance in Salmonella enterica and Escherichia coli isolated from animals,humans,food and the environment in 13 European countries[J].Journal of antimicrobial chemotherapy,2011,66(6):1278-1286.
[2]王小亮,徐立蒲,曹 欢,等.鱼源病原菌对氟氟喹诺酮类药物的耐药性分析[J].中国畜牧兽医,2013,40(3):195-198.
[3]Nozomi Aoike,Tomoo Saga,Sakata R,et al.Molecular Characterization of Extraintestinal Escherichia coli Isolates in Japan:Relationship between Sequence Types and Mutation Patterns of Quinolone Resistance-Determining Regions Analyzed by Pyrosequencing[J].Journal of Clinical Microbiology,2013,51(6):1692.
[4]Xiang Chen,Weiqiu Zhang,Pan W,et al.Prevalence of qnr,aac(6’)-Ib-cr,qepA,and oqxAB in Escherichia coli Isolates from Humans,Animals,and the Environment[J].Antimicrob Agents Chemother,2012,56(6):3423 -3427.
[5]Monika Dolejska,Laura Villa,Henrik Hasman et al.Characterization of IncN plasmids carrying blaCTX-M-1 and qnr genes in Escherichia coli and Salmonella from animals,the environment and humans[J].J Antimicrob Chemother,2013,68:333 -339.
[6]Martínez- Martínez L,Pascual A,Jacoby G A.Quinolone resistance from a transferable plasmid[J].The Lancet,1998,351(9105):797-799.
[7]Hata M,Suzuki M,Matsumoto M,et al.Cloning of a novel gene for quinolone resistance from a transferable plasmid in Shigella flexneri 2b[J].Antimicrobial agents and chemotherapy,2005,49(2):801-803.
[8]Jacoby G A,Walsh K E,Mills D M,et al.qnrB,another plasmid-mediated gene for quinolone resistance[J].Antimicrob Agents Chemother,2006,50:1178 -1182.
[9]Wang M,Q Guo,Xu X,et al.New plasmid-mediated quinolone resistance gene,qnrC1,found in a clinical isolate of Proteus mirabilis[J].Antimicrob.Agents Chemother,2009,53:1892 -1897.
[10]Cattoir V,P Nordmann.Plasmid-mediated quinolone resistance in gram-negative bacterial species:an update[J].Curr Med Chem,2009,16:1028 -1046.
[11]Yamane K,Wachino J,Suzuki S,et al.New plasmid-mediated fluoroquinolone efflux pump,QepA,found in an Escherichia coli clinical isolate[J].Antimicrobial agents and chemotherapy,2007,51(9):3354-3360.
[12]Robicsek A,Strahilevitz J,Jacoby G A,et al.Fluoroquinolone-modifying enzyme:a new adaptation of a common aminoglycoside acetyltransferase[J].Nat Med,2006,12:83 -88.
[13]Virginia Garcia- Fulgueiras,Luciana Robino,Gabriela Algorta,et al.Extended-spectrum β -lactamases and plasmid-mediated quinolone resistance in enterobacterial clinical isolates in the paediatric hospital of Uruguay[J].Antimicrob Chemother,2011,66:1725-1729.
[14]Fortini D,Fashae K,García-Fernández A,et al.Plasmid -mediated quinolone resistance and β-lactamases in Escherichia coli from healthy animals from Nigeria[J].Journal of antimicrobial chemotherapy,2011,66(6):1269-1272.
[15]Walsh F,Rogers T R.Comparison of plasmid-mediated quinolone resistance and extended-spectrum β-lactamases in third-generation cephalosporin-resistant Enterobacteriaceae from four Irish hospitals[J].Journal of medical microbiology,2012,61(1):142-147.
[16]Veldman K,Cavaco L M,Mevius D,et al.International collaborative study on the occurrence of plasmid-mediated quinolone resistance in Salmonella enterica and Escherichia coli isolated from animals,humans,food and the environment in 13 European countries[J].Journal of antimicrobial chemotherapy,2011,66(6):1278-1286.
[17]Liu B T,Liao X P,Yang S S,et al.Detection of mutations in the gyrA and parC genes in Escherichia coli isolates carrying plasmid-mediated quinolone resistance genes from diseased food-producing animals[J].Journal of medical microbiology,2012,61(Pt 11):1591-1599.
[18]Andres P,Lucero C,Soler-Bistué A,et al.Differential distribution of plasmid-mediated quinolone resistance genes in clinical enterobacteria with unusual phenotypes of quinolone susceptibility from Argentina[J].Antimicrobial agents and chemotherapy,2013,57(6):2467 -2475.
[19]Hu Y,Cai J,Zhang R,et al.Emergence of Proteus mirabilis Harboring blaKPC-2 and qnrD in a Chinese Hospital[J].Antimicrobial agentsand chemotherapy, 2012, 56(5):2278-2282.
[20]Herrera-León S,González- Sanz R,Herrera-León L,et al.Characterization of multidrug-resistant Enterobacteriaceae carrying plasmid-mediated quinolone resistance mechanisms in Spain[J].Journal of Antimicrobial Chemotherapy,2011,66(2):287-290.
[21]Ulises Garza- Ramos,Humberto Barrios,Maria Jose Hermandez- Vargas,et al.Transfer of quinolone resistance gene qnrA1 to Escherichia coli through a 50 kb conjugative plasmid resulting from the splitting of a 300 kb plasmid[J].Antimicrob Chemother,2012;67:1627-1634.
[22]Kocsis B,Kocsis E,Fontana R,et al.Identification of blaLAP-2 and qnrS1 genes in the internationally successful Klebsiella pneumoniae ST147 clone[J].Journal of medical microbiology,2013,62(Pt 2):269-273.
[23]Wang M,Jacoby G A,Mills D M,et al.SOS regulation of qnrB expression[J].Antimicrobial agents and chemotherapy,2009,53(2):821-823.
[24]Briales A,Rodriguez-Martinez J M,Velasco C,et al.Exposure to diverse antimicrobials induces the expression of qnrB1,qnrD and smaqnr genes by SOS-dependent regulation[J].Journal of antimicrobial chemotherapy,2012,67(12):2854-2859.
[25]Elena Ruiz,Yolanda Saenz,Myriam Zarazaga,et al.Qnr,aac(6’)-Ib- cr and qepA genes in Escherichia coli and Klebsiella spp.:genetic environments and plasmid and chromosomal location[J].Journal of antimicrobial chemotherapy,2012,67:886 -897.
[26]Rodríguez- Martínez J M,Velasco C,Briales A,et al.Qnrlike pentapeptide repeat proteins in gram-positive bacteria[J].Journal of antimicrobial chemotherapy,2008,61(6):1240-1243.
[27]Jacoby G A,Hooper D C.Phylogenetic analysis of chromosomally determined Qnr and related proteins[J].Antimicrobial agents and chemotherapy,2013,57(4):1930-1934.
[28]Hegde S S,Vetting M W,Mitchenall L A,et al.Structural and biochemical analysis of the pentapeptide repeat protein EfsQnr,a potent DNA gyrase inhibitor[J].Antimicrobial agents and chemotherapy,2011,55(1):110 -117.
[29]Velasco C,Rodriguez-Martinez J M,Briales A,et al.Smaqnr,a new chromosome-encoded quinolone resistance determinant in Serratia marcescens[J].Journal of antimicrobial chemotherapy,2010,65(2):239-242.
