枯草杆菌蛋白酶嗜热性改造单突变效果评判方法研究
2014-11-14李晓琴
夏 翾,马 帅,王 勤,李晓琴
(北京工业大学生命科学与生物工程学院,北京100124)
蛋白酶是一种天然的生物催化剂,具有较高的催化效率和底物特异性,被广泛应用于工业、食品业、医药业及人们的日常生活中[1-2]。蛋白酶发挥其高活性和高催化性是需要特定的条件作为保障的,如适当的PH值、温度、离子浓度等[3]。其中,温度这一因素尤为重要。蛋白酶只有在一定的温度范围内才能保持较高的活性,超出了这个温度范围,酶的活性就会降低甚至消失,严重影响它们的实际应用。因此,了解蛋白酶的热稳定机制进而提高蛋白酶的热稳定性[4],对扩大酶的应用范围与探究蛋白质结构和功能关系均具有非常重要的意义。
蛋白质的热稳定性是由多种作用共同参与的,不同的蛋白质可能有不同的耐热机制,在这些作用中比较重要的有:氨基酸组成、氢键、盐桥、疏水作用、包埋效应、环和N、C端的稳定性等[5]。目前,提高枯草杆菌蛋白酶热稳定性的方法主要有两种,一种是改变酶的催化环境,如调节PH值、增加有机溶剂、底物保护等,这种方法不能从本质上改变枯草杆菌蛋白酶的耐热性,因此在工业应用上会受到很多的限制;另一种是对蛋白酶进行残基突变,在蛋白质的薄弱环节引入有利残基,加强局部或整体的彼此相互作用,从而增加蛋白的热稳定性。
残基突变是提高蛋白酶热稳定性的有效方法,如何评判残基突变对蛋白酶热稳定性的影响?目前,对蛋白酶嗜热性突变效果的判定主要有两种方法,一种是生物实验的方法[6],即将突变成功的蛋白通过蛋白质合成技术制造出来,然后通过逐渐升温来验证其突变效果的优劣。该方法有较高准确性与可靠性,但是会耗费大量的时间和人力。另一种是生物信息学计算的方法[7-9],即计算ΔΔGu值来评定嗜热性突变的效果。ΔGu(蛋白质解折叠自由能)是蛋白质热动力学参数中最重要的参数,也是反应蛋白质热稳定性的一个通用指标,因此多数方法都是利用生物信息学计算的方法来模拟突变前后ΔGu的变化,即ΔΔGu(式1)。当ΔΔGu>0时,表明该突变对蛋白质的热稳定性有正效应(即嗜热性突变效果好),反之有负效应(即嗜热性突变效果差)。

2005年,Korkegian等直接从蛋白质结构入手预测蛋白质的ΔGu,该方法主要是利用一些经验数据或方法来分析蛋白质突变前后其结构或能量变化,来推断其 ΔGu的变化[10]。2006年,Saraboji等利用统计数据分析预测蛋白质的ΔGu,即用一些统计学的手段对突变体数据进行分析,得到一些统计学的规律,以此来判定突变前后的 ΔΔGu[11]。2008 年,Capriotti等提出机器学习方法,即利用蛋白质的序列或结构特征直接从数据入手,建立计算模型来预测ΔΔGu[12]。该方法较生物实验方法节约了时间和人力,但是其仍具有计算复杂、计算量大的缺点。
分子动力学(MD)模拟是研究蛋白质动力学性质进而揭示其生物学功能的重要工具,利用MD模拟结果提取有效参数、建立蛋白酶嗜热性改造突变效果评判方法是本文研究的重点。本文以研究资料颇为丰富的枯草杆菌蛋白酶BPN'为研究对象,选取实验上已证明的能提高其热稳定性的突变体来进行研究,运用分子动力学模拟的方法,通过对突变前后蛋白质多种能量参数的对比分析,提取了多个有效参数,并进一步建立了枯草杆菌蛋白酶嗜热性改造单突变效果评判方法。
1 材料
1.1 枯草杆菌蛋白酶
枯草杆菌蛋白酶是芽孢杆菌属细菌所分泌的胞外碱性蛋白酶,属丝氨酸蛋白水解酶类[13-16]。它催化水解蛋白质为氨基酸,在有机溶剂中也能催化多肽的合成。常见的枯草杆菌蛋白酶有 BPN'、Carlsbery、E、Savinase、BL 等,它们的一级氨基酸序列非常类似,晶体结构也基本相似[17]。枯草杆菌蛋白酶BPN'与E的区别是43个氨基酸残基发生替换,Carlsberg与E在序列上的区别是89个氨基酸残基替换再加上第56位残基缺失,BL与BPN'的区别是在275个氨基酸残基上有103个残基替换再加上4个残基(160~163)缺失。
枯草杆菌蛋白酶具有重要的应用价值,被广泛应用于洗涤剂、制革及丝绸等工业;此蛋白的结构与功能研究已有较长的历史,其蛋白结构、生化特性、催化机理等均有了较清晰的阐释。目前,在该酶的嗜热性改造方面有丰富的突变资料可供参考;另外,解析度为1.8 Å的该蛋白酶的晶体结构数据可以从PDB数据库中获取。基于以上原因,我们将枯草杆菌蛋白酶作为研究对象。
以枯草杆菌蛋白酶BPN'作为研究对象,它共有275个残基,空间结构如图1所示。
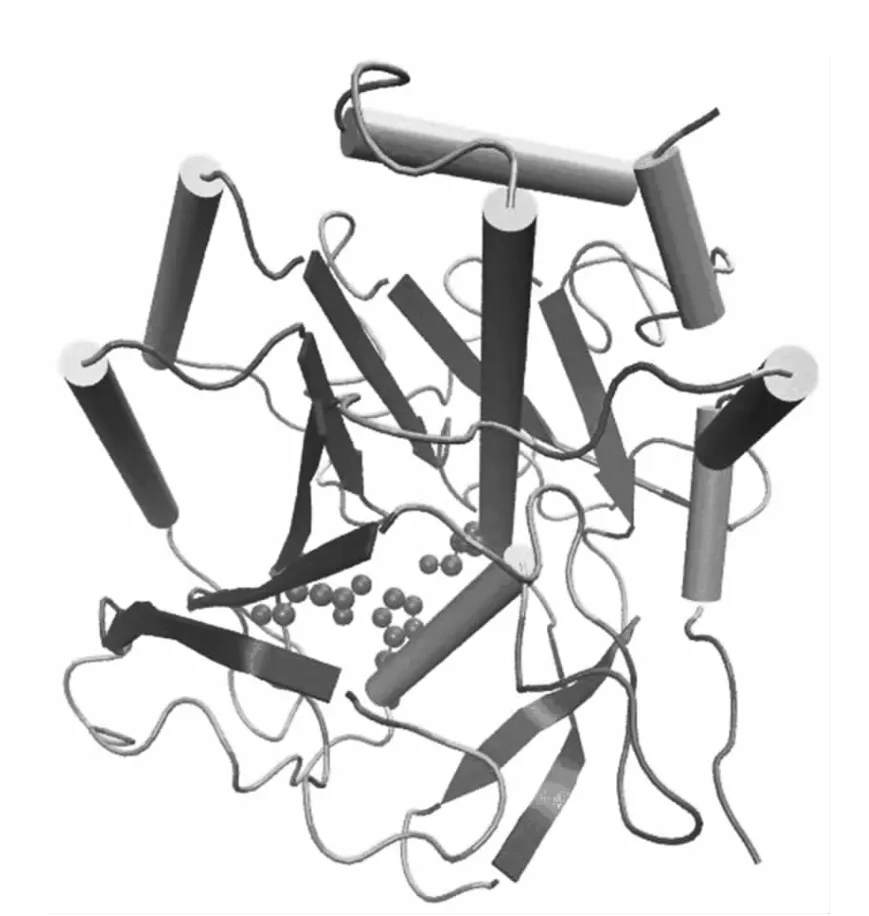
图1 枯草杆菌蛋白酶空间结构Fig.1 Spatial structure of Subtilisin BPN'
图中桶状结构代表α螺旋,固形箭头代表β折叠,小球代表催化三联体(Asp32、His64和Ser221)。图中可见β折叠位于蛋白质内部形成疏水核心,α螺旋位于结构表面,围绕在β折叠周围,整个蛋白质呈一个球状。
1.2 枯草杆菌蛋白酶突变位点的选取
选取6个实验上已证明的能提高蛋白质热稳定性的突变点作为研究对象,它们分别为:S161C、G166R[18]、G169A[19]、S188P[20]、K213R[21]以 及P239R[22]。表1为野生型枯草杆菌蛋白酶 BPN'及其6个突变体的Tm值及60℃下的半衰期时间,这两个值越大说明结构的热稳定性越强。由表中数据可以看出,六个突变体的热稳定性均强于野生型。表中Wild代表野生型枯草杆菌蛋白酶。
2 方法
2.1 枯草杆菌蛋白酶突变体的同源模建
从PDB数据库中下载解析度为1.8 Å的枯草杆菌蛋白BPN'晶体结构(PDB:2SIC),将此结构的E链作为野生型样本。利用SWISS-MODEL进行同源模建,得到表1所列的6个突变体的结构。

表1 枯草杆菌蛋白酶及其突变体的信息统计Table 1 Information statistics of subtilisin BPN'and its mutants
利用野生型及6个突变体的结构信息,分别计算主链结构的二面角,绘制图2所示的拉氏构象图。图A为野生型蛋白的拉氏构象图,图B、图C、图D、图 E、图 F、图 G 分别为 S161C、G166R、G169A、S188P、K213R、P239R突变体的拉氏构象图。图中黑色区域表示构象允许区域,在该区域内的任何成对二面角所规定的构象都是立体化学所允许的。灰色区域表示不完全允许区,表示在这个区域内的二面角所规定的主链构象虽然是立体化学可允许的,但是不够稳定,白色区域是不允许区域。在图中可以看到,构象角绝大部分都处于构象允许区域内,说明这些突变体的构象均处于合理的状态。

图2 枯草杆菌蛋白酶及其突变体所对应的拉氏构象图Fig.2 The ramachandran figure of subtilisin BPN'and its mutants
2.2 分子动力学模拟实验的设计
使用NAMD软件对野生型和6个突变体蛋白进行分子动力学模拟,选用CHARMM27全原子力场。预处理过程中,首先删掉晶体中的氢原子,再使用VMD软件自带的psfgen程序包补足氢原子,测得该晶体结构的净电荷为零;然后给该结构添加一个厚度为10 Å的TIP3P型水分子层,最终得到的水盒子的尺寸是 65 Å ×66 Å ×68 Å。
预平衡过程中,选用NPT系综。在1个标准大气压下,使用SHAKE算法约束氢键键长,截断半径为12 Å,非键相互作用在14 Å处截断。对1个野生型及6个突变体共进行了以下4次分子动力学模拟过程:
(1)温度恒定为370 K,步长为2 fs/step,运行100 000步,分子动力学模拟时间为0.2 ns,此过程命名为370 K_0.2 ns;
(2)温度恒定为370 K,步长为2 fs/step,运行1 000 000步,分子动力学模拟时间为2 ns,此过程命名为370 K_2 ns;
(3)温度恒定为370 K,步长为1 fs/step,运行100 000步,分子动力学模拟时间为0.1 ns,此过程命名为370 K_0.1 ns;
(4)初始温度为300 K,步长为2 fs/step,运行100 000步,每2 000步升高1 K,终止温度为350 K,分子动力学模拟时间为0.2 ns,此过程命名为300 K_0.2 ns。
其中370K_0.2ns作为标准组,其他3组作为对照组。
2.3 基于模拟结果的参数计算
每个野生型及其突变体分子动力学模拟完成后均输出一个“.log”日志文件,包含了模拟过程中的压力、温度以及本实验中需要用到的能量等信息。配置文件中设置每1 000步(即2 ps)输出一次能量信息,而我们需要计算的是在模拟的整个过程中或者某一特定时间段内的平均能量,因此具体方法如下:利用VMD软件的能量计算脚本 namdstats.tcl,结合输出的.log日志文件,提取蛋白体系的以下平均能量信息:BOND(键能)、ANGLE(键角能)、DIHED(二面角能)、ELECT(静电能)、VDW(范德华力能)、KINETIC(动能)。
3 结果与分析
3.1 模拟结果
依照上述方法,我们共得到4组动力学模拟后的能量结果(见表2~表5)。每组表格中上半部分为提取的能量值,下半部分为每个突变体与野生型能量的差值以及能量的变化趋势。
首先,以温度为1个标准大气压下温度为370 K、时间为0.2 ns的模拟作为标准组。表2是该组的模拟能量结果,我们提取的是每个结构从模拟第一步到最后一步的能量平均值。由表中数据可以看出,6个突变体与野生型酶相比,每种平均能量值均出现了相同的变化趋势,即 BOND增大、ANGLE增大、DIHED减小、ELECT减小、VDW 增大、KINETIC增大。
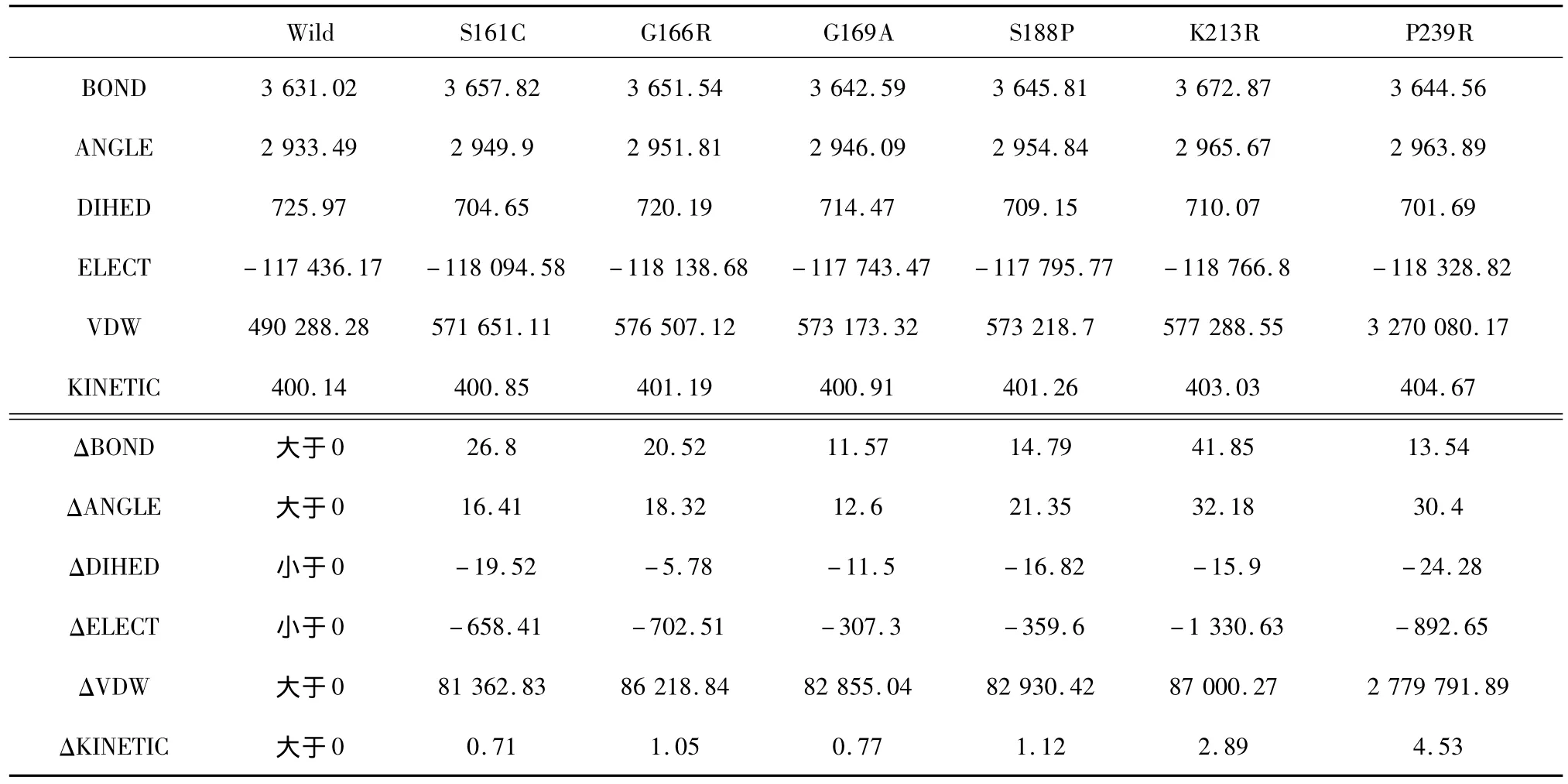
表2 370 K_0.2 ns模拟平均能量(kcal/mol)Table 2 Average energy data simulated-370 K_0.2 ns(kcal/mol)
标准组的模拟温度为370 K,模拟时间为0.2 ns,这样高温、短时间的模拟并没有使野生型及突变体动力学模拟达到平衡,为了检验非平衡态的能量统计结果的可靠性,我们在标准组370 K_0.2 ns模拟的基础上进行了较长时间的模拟,温度不变,模拟时间增加为标准组的10倍,即2 ns。图3为该模拟下野生型及其突变体的RMSD曲线图。可以看到,在1.0 ns~1.5 ns的时间段,7个结构的RMSD曲线均呈现平稳状态,说明在此时间段内达到了平衡状态。因此我们提取了这段时间内的能量平均值,并计算突变体与野生型的能量差值(见表3)。可以看到,表中突变体的6种平均能量值有着与标准组相同的变化趋势。
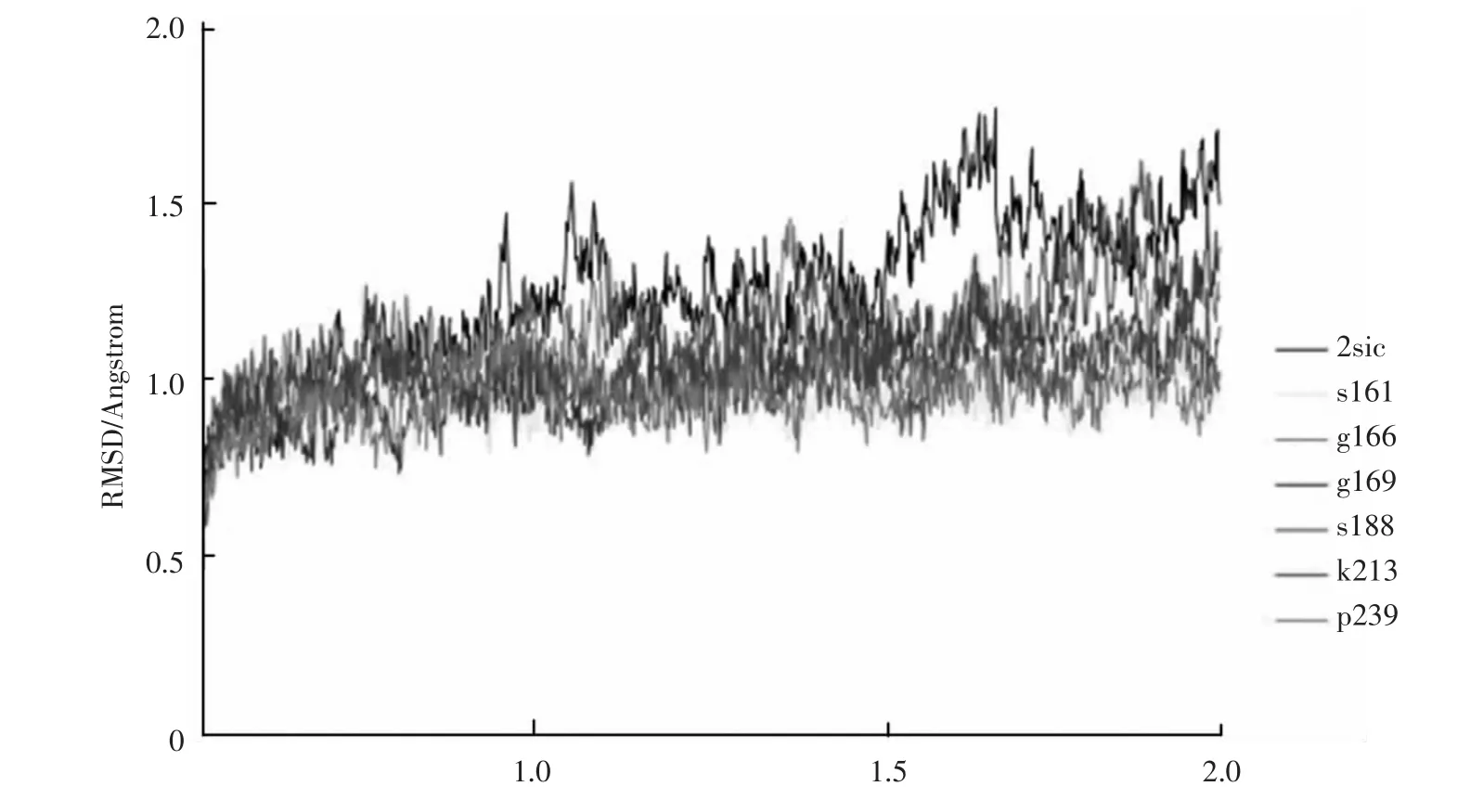
图3 枯草杆菌蛋白及其突变体2ns分子动力学模拟过程中的RMSD曲线Fig.3 The RMSD curves ofSubtilisin BPN'and its mutants in 2ns in the molecular dynamic simulation

表3 370 K_2 ns模拟平均能量差值(kcal/mol)Table 3 D-value of average energy data simulated-370 K_2 ns(kcal/mol)
从表2、表3的结果看,模拟时间的长短以及体系是否平衡对能量变化趋势并没有影响,为进一步检验模拟时间对结果的影响,我们在温度不变的情况下,进行了更短时间的模拟,模拟时间缩减为标准组的一半,即0.1 ns,其能量差值见表4。表4中所有能量的变化都较标准组呈现相同的趋势。
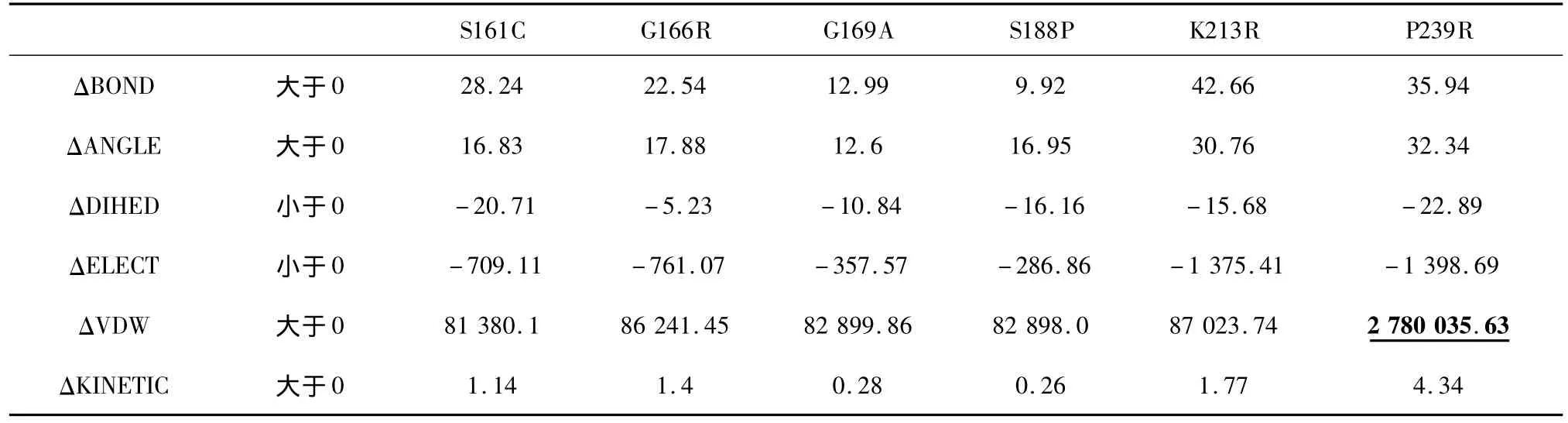
表4 370 K_0.1 ns模拟平均能量差值 (kcal/mol)Table 4 D-value of average energy data simulated-370 K_0.1 ns(kcal/mol)
最后,我们检验了温度对模拟结果的影响。在标准组的基础上我们做了一组温度均匀变化的模拟300 K_0.2 ns,时间同标准组设置一样,模拟过程中将温度从300 K均匀升高到350 K。能量差值结果见表5。由表中数据可以看出,在温度变化的模拟条件下突变体的平均能量还是出现了与标准组一样的变化趋势。

表5 300 K_0.2 ns模拟平均能量差值 (kcal/mol)Table 5 D-value of average energy data simulated-300 K_0.2 ns(kcal/mol)
3.2 结果分析
从上面4个表可以看出,突变体与野生型酶相比,均出现了BOND增大、ANGLE增大、DIHED减小、ELECT减小、VDW增大、KINETIC增大的趋势。研究表明,分子柔性的降低会带来嗜热性的增强[23],因此突变体的分子柔性均低于野生型,正是由于柔性降低、刚性增强,所以会使得BOND(键能)增大、ANGLE(键角能)增大以及 DIHED(二面角能)减小;而所有突变体的ELECT(静电能)都比野生型小,这与以前的实验中所说引入带电残基增强蛋白质热稳定性是相一致的,静电能越小稳定性越强[24];而VDW(范德华能)增大说明分子的极性也增大了,表明蛋白质的嗜热性与分子极性也密切相关;突变体的KINETIC(动能)虽然比野生型都有所增大,但变化范围很小,说明这个能量可能与热稳定性的关系并不大,但我们仍暂时保留这一项,等待后续实验进行进一步验证。
另外可以看到表中有一些数据与其他数据相差较大,我们将这些数据称为极端数据,并用特殊格式将这些极端数据标记出来。除去这些极端数据,我们得到以下突变体与野生型相比的能量变化范围(单位:kcal/mol):ΔBOND 为[ 2.66,8.57],ΔANGLE 为[3.32,32.18],ΔDIHED 为[-16.68,-2.66],ΔELECT 为[-1 375.41,-286.86],ΔVDW 为[81 362.83,87 027.74],ΔKINETIC 为[0.26,2.89]。
为进一步分析能量变化与蛋白嗜热性的关系,我们利用SPSS分析软件分别对以上4组模拟中每个突变体的6种能量差值与其对应的Tm或半衰期进行了斯皮尔曼(Spearman)相关性分析,99%置信度下的双侧检验的计算结果见表6,其中表的上半部分是G166R、G169A、S188P的Tm值与各个能量差值的绝对值之间的相关系数,下表下半部分是S161C、K213R、P239R的Half life与各个能量差值之间的相关系数。相关系数越大表示相关性越显著,正数表示正相关,负数表示负相关。
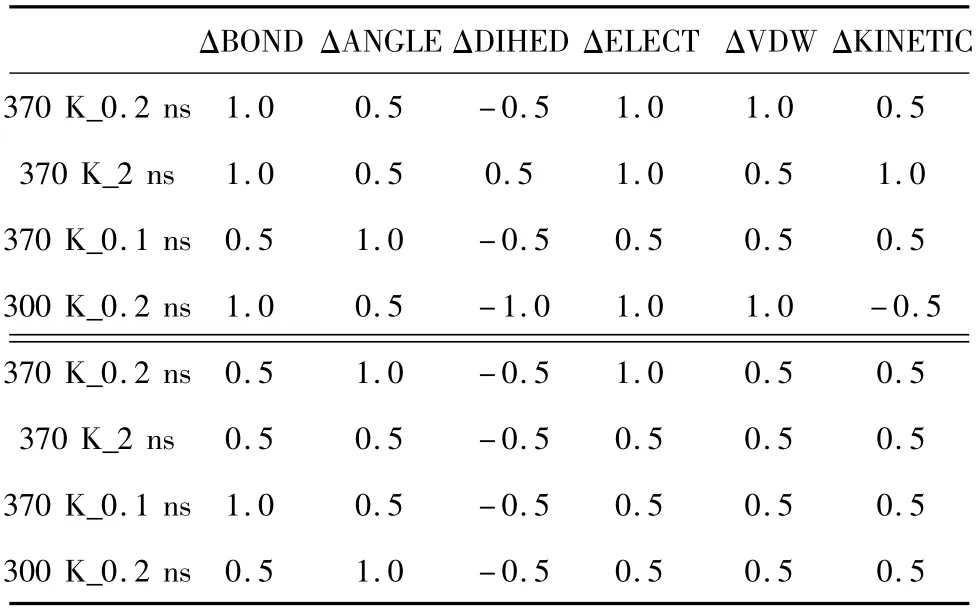
表6 能量差值与TmHalf life的相关系数Table 6 The correlation coefficient between the energy difference and TmHalf life
从表6可以看出:ΔBOND、ΔELECT与突变体的Tm相关性最强,3组模拟时间大于0.1 ns的相关系数均为1,同时与突变体的半衰期也分别有一组相关系数为1,总体平均值为0.75。说明 ΔBOND、ΔELECT对Tm及半衰期的变化最为敏感;ΔANGLE略低于ΔBOND、ΔELECT与突变体的Tm及半衰期的相关性,相关系数为1的共有 3组,平均为0.687 5;ΔVDW与突变体的Tm及半衰期的相关系数均为正值,有两组模拟时间大于0.1 ns的相关系数为1,总体均值为0.5,相关性一般;而ΔDIHED和ΔKINETIC与突变体的Tm及半衰期的相关性不稳定,说明ΔDIHED和ΔKINETIC对Tm及半衰期的变化趋势不敏感。
3.3 判定方法
从以上分析可以看出,在4组不同条件的模拟过程中,突变体的6个平均能量值与野生型相比均出现了相同的变化趋势,结合相关性的计算结果,ΔBOND、ΔELECT、ΔVDW、ΔANGLE、ΔDIHED、ΔKINETIC可以作为评判枯草杆菌蛋白酶嗜热性改造单突变效果评判的6个有效参数,其中ΔBOND、ΔELECT、ΔANGLE、ΔVDW 为特征有效参数,并采用370 K_0.2 ns模拟结果来提取上述参数。
枯草杆菌蛋白酶嗜热性改造单突变效果评判方法为:以标准组370 K_0.2 ns的模拟条件参数为准,分别对需要判定嗜热改造效果的突变体及野生型进行分子动力学模拟,利用模拟结果计算上述4个特征有效参数,如果ΔBOND、ΔVDW、ΔANGLE大于0,同时ΔELECT小于0,则可以判断该枯草杆菌蛋白酶突变体嗜热性提高,为有效突变,否则为无效突变。
4 检验与讨论
4.1 枯草杆菌蛋白酶其他突变点检验
我们从Protherm数据库中选定了2个实验上已经获取的枯草杆菌蛋白酶嗜热突变体作为检测样本,它们分别是Q206C和N218S。野生型及2个突变体在ProTherm数据库的记载见表7。表中2个突变体的Tm值都大于野生型,说明突变体的嗜热性要强于野生型。另外我们还选取了一个稳定性小于野生型的突变体P239G,实验测得此突变体在60℃时的半衰期比野生型减少了8.5 min。
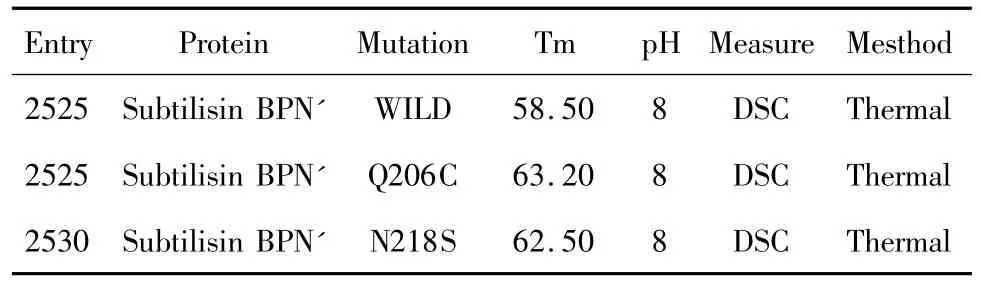
表7 枯草杆菌蛋白酶及其嗜热突变体在Protherm数据库中的记录Table7 Records of subtilisin BPN'and its mutants in protherm
根据3.3节提出的判定原则,我们对以上选取的两个有效突变体、一个无效突变体以及野生型进行分子动力学模拟,野生型命名为Wild。模拟结果见表8所示。

表8 检验样本模拟的平均能量Table 8 Average energy data simulated-test samples
表8下半部分第一列变化趋势为有效突变体Q206C及N218S的能量变化结果,第二列为无效突变体P239G的结果。从数据可以看出,Q206C和N218S这两个突变体的特征有效参数的取值满足有效突变条件,并且能量变化的数值也处于上文中所提到的范围之间,可以判断Q206C和N218S为有效突变;而P239G的能量变化趋势正好与之相反,为无效突变。
4.2 讨 论
本文以枯草杆菌蛋白酶BPN'为研究对象,利用4组不同条件分子动力学模拟结果,对其及其嗜热突变体的能量变化趋势进行对比分析,计算并提取了6个有效参数,并结合相关性分析结果,确定了4个特征有效参数,建立了判定枯草杆菌蛋白酶BPN'嗜热性突变效果的理论方法。研究发现,不同的模拟温度与时间对突变前后各个能量的变化趋势并没有影响。而且在较短的模拟时间条件下,即模拟没有达到平衡的状态下,有效参数及特征有效参数具有相同的变化趋势。本文提出的对枯草杆菌蛋白酶嗜热效果改造的判定方法非常简单有效,这将为以后筛选有效的突变体提供捷径。
利用短时间(时间小于等于0.2 ns)分子动力学模拟结果提取有效参数,在P239位点对应的两个突变体P239R和P239G上均出现了ΔVDW取值不同于正常ΔVDW取值范围的离群值,具体结果见表2、4、5、及表 8。其中 P239R 为有效突变,对应ΔVDW为正值;P239G为无效突变,对应ΔVDW为负值,尽管ΔVDW的变化趋势是对的,但P239R和P239G对应的ΔVDW明显超出ΔVDW的取值范围。这可能与脯氨酸(Pro)特殊的性质有关。由于脯氨酸是非极性氨基酸,而精氨酸(Arg)是极性带正电的氨基酸,因而引入带正电的Arg可以在增加蛋白的稳定性的同时使得分子极性大大增强,导致P239R的范德华能较其他几个能量呈现出更大的增长,造成极端数据的出现。而甘氨酸(Gly)是非极性不带电荷氨基酸,在P239G处为何也会出现极端数据,需要更多的实验来进行统计与验证。
此外,本文选取的试验样本仅为枯草杆菌蛋白酶,对其他蛋白酶是否具有普适性还需进一步研究与探讨。
References)
[1] NASCIMENTO W C A,MARTINS M L L.Production and properties of an extracellular protease from thermophilic Bacillussp [J]. Brazilian Journalof Microbiology,2004,35(1-2):91-96.
[2] ARGOS P,ROSSMAN M.G,GRAU U.M,ZUBER H.Thermal stability and protein structure [J].Biochemistry,1979,18(25):5698-5703.
[3] ARNOLD F H,GIVER L,GERSHENSON A,ZHAO H.Directed evolution ofmesophilic enzymesinto their thermophilic counterparts [J].Ann N Y Acad Sci,1999,870:400-403.
[4] LEHMANN M,LOCH C,MIDDENDORF A,et al.The consensus conceptforthermostability engineering of proteins:further proof of concept[J]. Protein Engineering,2002,15(5):403-411.
[5] SEELIGER D,GROOT B L.Proteinthermostability calculations using alchemical free energy simulations[J].Biophysical Journal,2010,98(10):2309-2316.
[6] 田健,王平.理性设计提高蛋白质热稳定性的研究进展[J].生物技术进展,2012,4(2):233-239.TIAN Jian,WANG Ping.Recent advances in the rational design to improve the proteinthermostability[J].Current Biotechnology,2012,4(2):233-239.
[7] GROMIHA M M,UEDAIRA H,AN J.ProTherm:Thermodynamic Database for Proteins and Mutants:developments in version 3.0[J].Nucleic Acids Res.,2002,30(1):301-302.
[8] GRIBENKO A V,PATEL M M,LIU J,et al.Rationalstabilization of enzymes by computational redesign of surface charge-charge interactions[J].Proc.Natl.Acad.Sci.,2009,106(8):2601-2606.
[9] JOO J C,PACK S P,KIM Y H,et al.Thermostabilization of bacillus circulans xylanase:Computational optimization of unstable residues based on thermal fluctuation analysis[J].Journal of Biotechnology,2011,151(1):56-65.
[10] KORKEGIAN A,BLACK M.E,BAKER D.Computational thermostabilization of an enzyme[J].Science,2005,308(5723):857-860.
[11] SARABOJI K,GROMIHA M.Average assignment method for predicting the stability of protein mutants[J].Biopolymers,2006,82(1):80-92.
[12] CAPRIOTTI E,FARISELLI P.A neural-network-based method for predicting proteinstability changes upon single point mutations[J].Bioinformatics,2004,20(Suppl 1):i63-68.
[13]毕汝昌,储乃明.枯草杆菌蛋白酶与蛋白质工程[J].生物化学与生物物理进展,1991,18(5):329-334.BI Ruchang,CHU Naiming.Subtilisin and protein engineering[J].Progress in Biochemistry and Biophysics,1991,18(5):329-334.
[14]王凡强,马美荣,王正祥,等.枯草杆菌蛋白酶基因工程的研究进展[J].生物工程进展,2000,20(2):41-44.WANG Fanqiang,MA Meirong,WANG Zhengxiang,et al.The research progress of Subtilisin gene engineering[J].Progress in Biotechnology,2000,20(2):41-44.
[15] MCKENNEY J M,KOREN M J,KEREIAKES D J.Safety and efficacy of a monoclonal antibody to proprotein convertase subtilisin/kexin type 9 serine protease,SAR236553/REGN727,in patients with primary hypercholesterolemia receiving ongoing stable atorvastatin therapy[J]. Journalofthe American College of Cardiology,2012,59(25):2344-2353.
[16] GUMBINER B,CHANDRASEKHAR U,JOH T.The effects of multiple dose administration of RN316(PF-04950615),a humanized IgG2a monoclonal antibody bindingproprotein convertase subtilisin kexin Type 9,in hypercholesterolemic subjects[J].Circulation,2012,126(suppl):A13524-A13524.
[17] KIMBER I,BASKETTER D A.Categorisation of protein respiratory allergens:The case ofSubtilisin[J].Regulatory Toxicology and Pharmacology,2014,68(3):488-492.
[18]ZHAO H M,ARNOLD F H.Directed evolution converts subtilisin E into a functional equivalent of thermitase[J].Protein Engineering,1999,12(1):47-53.
[19] PANTOLIANO M W,WHITLOW M,WOOD J F,et al.Large increases in general stability for subtilisin BPN'through incrementalchangesin the free energy of unfolding[J].Biochemistry,1989,28(18):7205-213.
[20] SÄTTLER A,KANKA S,MAURER K,et al.Thermostable variants of subtilisin selected by temperature-gradient gel electrophoresis[J].ELECTROPHORESIS,1996,17(4):784-92.
[21]CUNNINGHAM B C,WELLS J A.Improvement in the alkaline stability of subtilisin using an efficient random mutagenesisand screening procedure[J]. Protein Engineering,1987,1(4):319-325.
[22] TAKAGI H,MORINAGA Y,IKEMURA H,et al.The Role of Pro-239 in the Catalysis and Heat Stability of Subtilisin E[J].Journal of Biochemistry,1989,105(6):953-956.
[23] RADESTOCK S,GOHLKE H.Protein rigidity andthermophilic adaptation[J].Proteins:Structure,Function,and Bioinformatics,2011,79(4):1089-1108.
[24]CHAN C H,WILBANKS C C,MAKHATADZE G I,et al.Electrostatic contribution of surface charge residues to the stability of a thermophilic protein:Benchmarking experimental and predicted pKa values[J].PloS One,2012,7(1):e30296.
