乙肝病毒核心蛋白钉突部位基因工程改造对其功能的影响
2013-11-12陈江燕黄荣陶颖黄媛罗英英黄爱龙胡接力
陈江燕,黄荣,陶颖,黄媛,罗英英,黄爱龙,胡接力
重庆医科大学 感染性疾病分子生物学教育部重点实验室,重庆 400016
乙型肝炎病毒 (HBV) 是一种重要的病原体,可导致一系列慢性肝病[1-2]。HBV为直径42 nm的球形颗粒[3],呈双层壳结构,外壳主要由3种表面蛋白 (HBs) 构成[4],内层核衣壳由核心蛋白 (HBc) 组成,为正20面体结构[5],该结构包含90或120个HBc二聚体,每个二聚体由HBc单体通过二硫键连接[6-7]。HBc由4个α螺旋 (aa13-30,aa50-60-78,aa82-93-110,aa112-123-128) 和 C端精氨酸区组成[8]。第 2和第3个α螺旋转折形成钉状突起,aa80是尖突顶,位于核衣壳最表面 (图 1)[9-11]。HBc在HBV复制过程中必不可少,它所构成的核衣壳是包装前基因组 RNA (Pregenomic-RNA,pgRNA)和病毒聚合酶的场所[12],另外,HBc还介导病毒基因组DNA进入细胞核,继而生成共价闭合环状 DNA (Covalently closed circled DNA,cccDNA)作为复制的初始模板[13]。

图1 HBc蛋白二维结构示意图[10]Fig. 1 Diagram of HBc structure[10]. (A) HBc monomer, aa78-82 is at the spike. (B) HBc dimer.
HBc因其作用重要,一直是HBV复制研究中的重要对象,然而,由于缺乏稳定且特性好的抗HBc抗体,很多研究不便开展。本研究针对这一问题,拟利用绿色荧光蛋白 (EGFP) 对HBc进行基因工程改造,试图达到以下目的:1) 能对 HBc表达情况进行监控;2)使得用免疫学方法检测 HBc以及捕获核心颗粒 (含有HBV基因组 DNA及聚合酶的核衣壳)成为可能;3)与此同时,不影响HBc自身功能,即经过改造的HBc仍能支持HBV复制。为达到以上目的,我们认为,EGFP标签需表达于核衣壳表面,以便被抗 EGFP抗体识别和捕获。从核衣壳的冷冻电镜结构[8]可知,位于其最表面的部分是HBc钉突部位 (aa78~82),而不是C末端或N末端。因此,若在钉突位置插入EGFP,有可能使其暴露于核衣壳表面。但是,在一个蛋白的中间位置插入另一蛋白序列,有可能使这两种蛋白的三维结构都受到影响,不能正确折叠,从而影响甚至破坏二者的功能。Kratz等[14]的研究表明,对于截去C末端40 aa的HBc,当其钉突处aa79~80替换成GFP时,超过40%的融合GFP折叠正确,能产生绿色荧光,同时亦能形成与天然核衣壳类似的颗粒结构,然而,截去C末端的 HBc并不能支持 HBV复制[15],我们在本研究中将以完整HBc为基础进行改造,考察这些融合蛋白是否保留了HBc的正常功能,同时,我们还探讨了不同类型接头以及不同大小插入片段对HBc蛋白功能的影响。
1 材料与方法
1.1 材料
质粒 pCH9/3091由第三军医大学西南医院感染病研究所兰林博士馈赠,德国 Nassal[16]课题组构建;HEK293细胞、质粒pEGFP-N1为本实验室保存;Southern blotting检测试剂盒、Xteme HP转染试剂购自罗氏公司;内切酶DpnⅠ购自Promega公司。
1.2 方法
1.2.1 构建 HBV1.1c-质粒
采用不依赖酶切与连接的分子克隆方法(RLIC)[17-18]。以质粒pCH9/3091为模板,设计突变引物,使其HBc第40位氨基酸突变为终止密码,上游引物为:5'-GATACCGCCTCAG CTCTGTATCGGTAAGCCTTAGAGTCTCCTGA GCATTG-3',下游引物为:5'-CTCGTCGTCT AACAACAGTAGTCT-3',进行 PCR扩增。用QIAGEN 胶回收试剂盒回收扩增片段。回收片段带有突变碱基,作为大引物,用来替换模板质粒pCH9/3091中相应片段。反应体系:300 ng回收片段,50 ng pCH9/3091模板,0.5 μL PrimeSTAR 聚合酶等。反应条件:95 ℃预变性2 min;95 ℃ 15 s,55 ℃ 15 s,72 ℃ 3 min,18个循环。扩增产物用Roche PCR 产物纯化试剂盒纯化,取13 μL纯化产物用Dpn Ⅰ于37 ℃消化5 h,取2 μL消化后产物电转化JM109 感受态细胞,获得的克隆测序鉴定。
1.2.2 构建HBc质粒
以 pCH9/3091为模板,设计引物,上游引物为:5'-CAATCTCGGGAATCTCAATGTTAG GAGATTAGGTTAAAGGTCTTTGTAC-3';下游引物为:5'-GTACAAAGACCTTTAACCTAAT CTCCTAACATTGAGATTCCCGAGATTG-3',作反向替换 (等同大片段缺失突变),具体方法与前述类似。挑取克隆测序鉴定。
1.2.3 其他重组质粒的构建
将HBc aa79~80替换成EGFP有两种设计:1) HBc gfp-flex,由柔性接头连接EGFP两端并插入HBc中 (替换aa79~80)。首先以pEGFP-N1为模板,用引物FHBc gfp-flex1、RHBc gfp-flex1 PCR扩增,取0.1 μL扩增产物为模板,用引物FHBc gfp-flex2、RHBc gfp-flex2再次PCR扩增,得到两端加上柔性接头的 EGFP基因片段,其末端带有与 HBc aa79~80两侧序列同源的序列,然后以该片段为大引物,替换插入到 HBc中。方法类似 1.2.1。2)HBc gfp-heli,在 HBc gfp-flex基础上,EGFP两端各增加一段α螺旋的刚性接头,改变融合蛋白间的连接方式。构建方法大体与1)类似,但因该刚性接头序列较长且多重复碱基,难以直接用PCR将外侧序列加到上一轮PCR扩增片段两侧 (即heli2引物不能较好扩增heli1的扩增产物),因此需在每轮扩增后,增加一次克隆过程,共进行了3次克隆。
此外,我们还在HBc gfp-flex基础上,将插入的外源片段逐步缩短,构建 3种重组质粒:1) HBc gfp15-flex,为HBc gfp-flex 删除EGFP中间部分,仅保留 EGFP15个氨基酸的重组HBc。2) HBc flex,在HBc gfp15-flex的基础上进一步将重组蛋白减短,融合多肽为2个G4S。3) HBc 7980-为第79、80位氨基酸缺失的HBc蛋白。均采用与RLIC原理相同的反向替换法。
1.2.4 HEK293细胞培养及转染
细胞培养及转染:HEK293细胞使用含10%胎牛血清的 DMEM培养基,于 37 ℃,含 5%CO2,相对湿度大于95%的无菌孵箱中培养。转染质粒前,先将细胞接种于 6 孔板,密度约为1.5×105个/孔,培养约 20 h,换新鲜培养基。配制转染体系:质粒与脂质体以1 : 3的比例混匀 (2 μg DNA : 6 μL 脂质体),转染各孔,24 h后换液。继续培养4 d,收细胞。
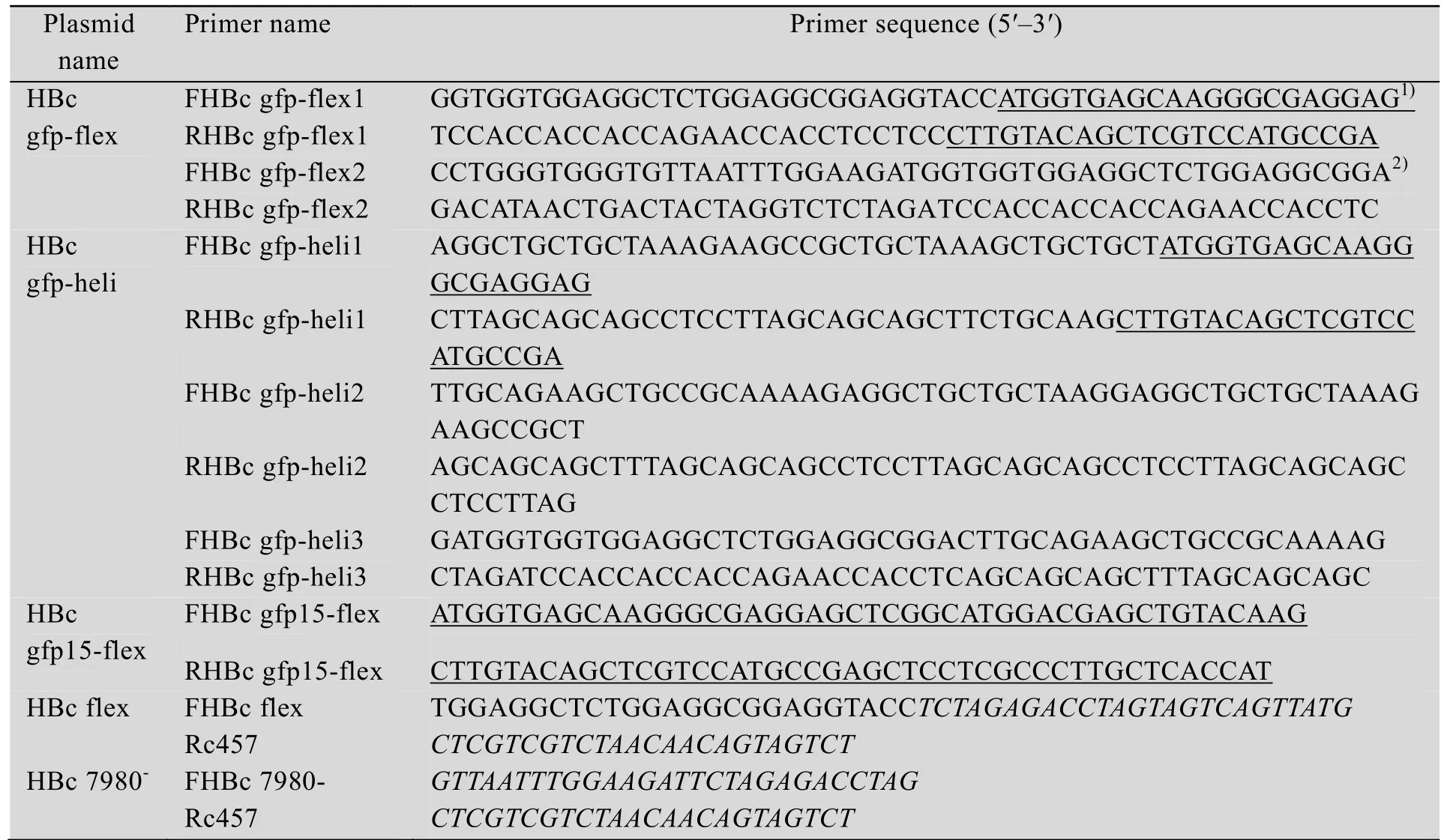
表1 其他重组质粒构建所用引物Table 1 Primers used in the construction of other recombinants
1.2.5 Southern blotting检测HBV DNA复制中间体
细胞转染5 d后,提细胞内核心颗粒HBV DNA:用500 μL细胞裂解液(10 mmol/L Tris-HCl pH 8.0,1 mmol/L EDTA,1% NP40,50 mmol/L NaCl,2%蔗糖)37 ℃孵育10 min,收集裂解物,15 000×g离心 4 min,取上清,加 5 μL 1 mol/L MgCl,50 U DNase I于 37 ℃孵育 5 h,加 200 μL沉淀剂(35% PEG 8000, 1.5 mol/L NaCl),置冰上40 min,13 000×g离心10 min后弃去上清,再用Proteinase K消化过夜,经酚氯仿抽提,异丙醇沉淀,将得到的产物溶于8 μL超纯水,用Southern blotting检测HBV DNA,具体操作步骤详见文献[19]。
2 结果
2.1 HBV1.1c-及HBc质粒的构建
构建HBc表达缺失的HBV 1.1倍体的目的,是为了后续检测各种经过改造的HBc是否保留正常功能,即支持HBV复制。我们在含1.1倍体 HBV基因组的 pCH9/3091基础上,将 HBc蛋白的第40个氨基酸突变成终止密码,同时不干扰HBV其他基因的表达[20](图2)。
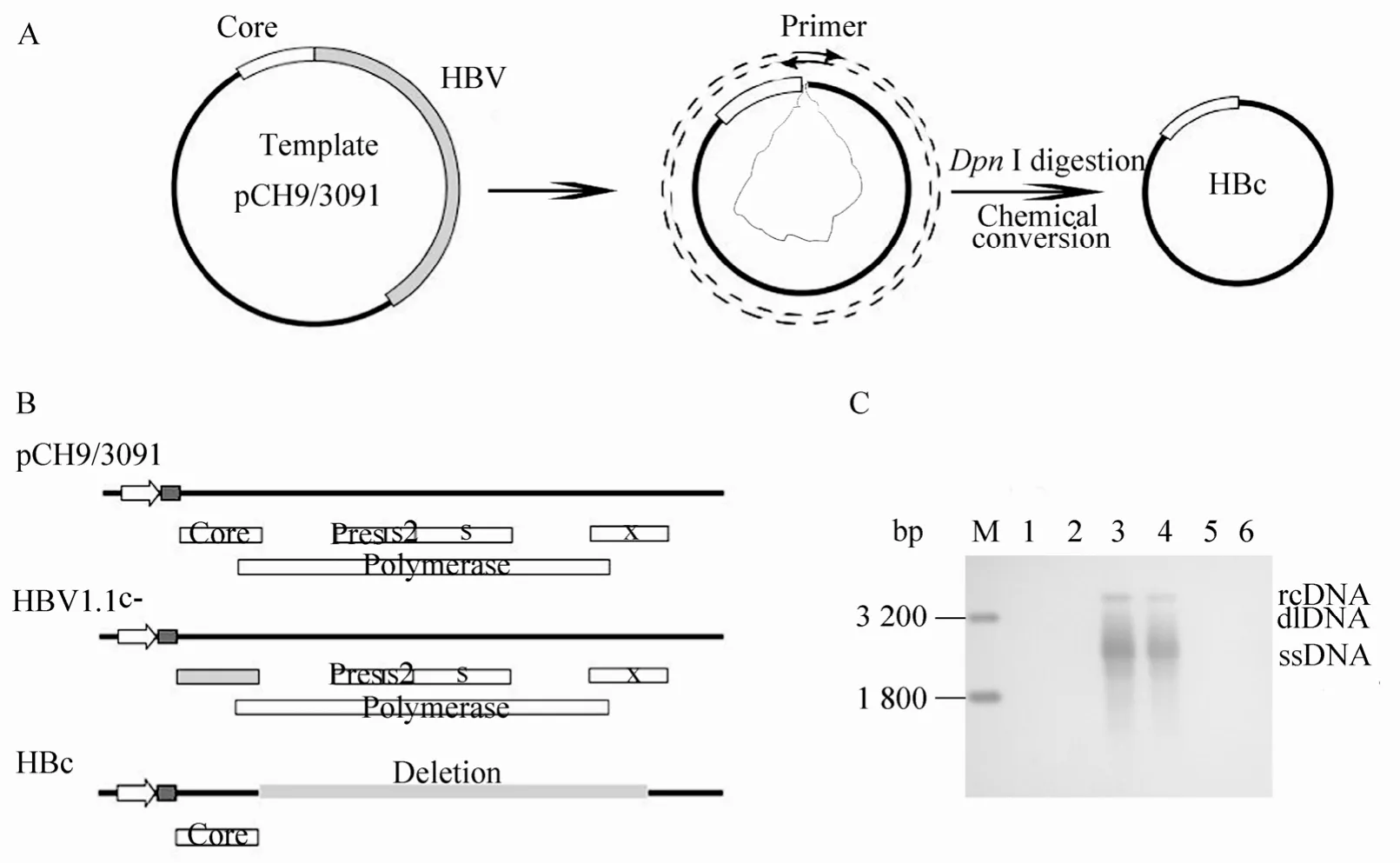
图2 HBV1.1c-及HBc的构建及功能鉴定Fig. 2 Construction of HBV1.1c- and HBc and functional characterization. (A)The principle of RLIC to delete a fragment from a plasmid. (B)Diagram of pCH9/3091, HBV1.1c- and HBc. (C)Detection of HBV DNA by Southern blotting. M: marker; 1–4: HBV DNA extracted from HEK293 cells cotransfected with different clones of HBc and HBV1.1c-. 1: HBc1+HBV1.1c-4; 2: HBc1+HBV1.1c-5; 3: HBc3+HBV1.1c-4; 4: HBc3+HBV1.1c-5; 5 and 6:negative control (transfection only with HBV1.1c-4 or HBV1.1c-5). HBc1 and HBc3 are two different clones of HBc. HBV1.1c-4 and HBV1.1c-5 are two different clones of HBV1.1c-. ssDNA: single stranded DNA.
HBc质粒仅需带有 HBVc基因及转录终止信号,我们以pCH9/3091为基础,使用类似RLIC原理的方法 (图2),具体为:设计一对完全互补的引物,引物两端序列分别与缺失目标序列两端同源,用该对引物进行替换反应,以去掉除了HBc及终止信号[21]以外的其他 HBV DNA。选择测序正确的HBc克隆质粒 HBc1、HBc3,分别与 HBV1.1c-克隆质粒HBV1.1c-4、HBV1.1c-5共转染HEK293细胞,对二者进行功能鉴定,二者具有正常功能的标准为:能支持 HBV复制。HBV复制中间体的Southern blotting 结果显示(图2),HBc3 与HBV1.1c-4/5共转染后,可观察到复制中间体形成[22],提示二者均保留了正常功能,支持HBV复制。
2.2 钉突部位融合EGFP的重组HBc功能鉴定

图3 HBc-EGFP的功能鉴定Fig. 3 Functional characterization of HBc-EGFP. (A)Structure of HBc, HBc gfp-flex and HBc gfp-heli. (B)Observation of green fluorescence in HEK293 cells transfected with different constructs (a, b: HBc gfp-flex; c, d:HBc gfp-heli). (C)Detection of HBV replication intermediates by Southern blotting: M: marker; 1: HBc+HBV1.1c-;2: HBc gfp-flex+HBV1.1c-; 3: HBc gfp-heli.+HBV1.1c-.
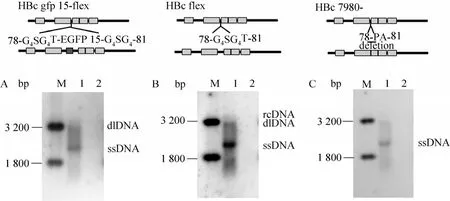
图4 HBc aa79~80短片段替换及缺失对其功能的影响Fig. 4 Impact of short insertion or deletion at aa79-80 of HBc on its function. (A) The structure and functional assay of HBc gfp15-flex. M: marker; 1: positive control HBc+HBV1.1c-; 2: HBc gfp15-flex+HBV1.1c-. (B) The structure and functional assay of HBc flex. (C)The structure and functional assay of HBc7980-.
在将HBc aa79-80替换成EGFP时,为了尽可能降低插入蛋白和本体蛋白在三维结构上的相互干扰,以减小对各自功能的影响,我们在插入位置设计了两种连接接头。一种是柔性接头,即在EGFP与HBc的两侧连接处,分别以多甘氨酸2G4S连接[14](HBc gfp-flex,图3),另一种是柔性+刚性接头,即在2G4S与EGFP之间,分别增加一段可形成 α螺旋的序列 A(EAAAK)4A[23](HBc gfp-heli,图3)。测序正确的HBc gfp-flex/HBc gfp-heli重组质粒,分别与HBV1.1c-以1 : 1 (摩尔比)共转染 HEK293细胞,转染24 h后开始观测到荧光,随时间延长,荧光增强。图3所示为48 h的荧光情况。HBc gfp-flex产生的荧光蛋白在细胞内分布均匀,强度较转染 pEGFP-N1弱,提示受融合蛋白间的相互影响,正确折叠的EGFP减少。HBc gfp-heli产生的荧光表现不同,在细胞内呈现分散的点状荧光。
2.3 aa79-80位置短片段替换影响 HBc的正常功能
由于钉突部位融合EGFP的重组HBc可产生绿色荧光,但不支持HBV复制,提示尽管至少部分 EGFP可进行正确折叠 (正确的三维结构是其发荧光的前提)[24],但至少部分HBc未获得正确折叠。我们接下来希望知道,是否是因为插入的EGFP片段过大,而影响了HBc的正常功能?为此,我们将插入片段逐步缩小,使替换的片段分别为EGFP的15个aa+柔性接头 (HBc gfp15-flex),10aa柔性接头 (HBc flex),以及仅缺失aa79~80(HBc 7980-)。将这些质粒分别与 HBV1.1c-共转染 HEK293细胞,然后提取细胞内核心颗粒DNA,Southern blotting检测HBV复制中间体。结果显示,这3种结构的质粒均未检测到病毒复制信号,而阳性对照 (野生型HBc+HBV1.1c-)可见明显复制。
3 讨论
HBc三维结构中的钉突部位 (aa78~82)位于核衣壳表面,其部位的抗原充分暴露,非常有利于与抗体结合,常作为植入外源抗原的部位[25],但通常植入这些抗原的目的,是为了利用HBc可聚合成较大颗粒的特性,获得更好的免疫原性,并不关心是否保留HBc的主要功能,即支持 HBV复制。本研究旨在对 HBc钉突aa79~80处进行改造,插入标记蛋白EGFP,希望在对 HBc进行标记的同时,保留 HBc支持HBV复制的正常功能。
在本研究中,我们首先采用类似RLIC原理的大片段缺失方法,构建了一系列重组质粒,包括HBc及3种HBc-EGFP截短质粒。该方法利用同源序列退火和线性扩增,原理本质上仍是RLIC,区别只是用短的片段替换目标载体上较长的片段,而RLIC是用大的片段替换载体上的较短区域,结果一个是基因缺失,一个是基因插入。该方法使用时,对缺失片段大小及缺失位置无限制,本研究中的几种缺失片段大小为数百或数千bp,这些结果显示,RLIC是一种基因工程改造的有力工具。
我们在插入蛋白两端设计了两种连接接头,一种柔性接头和一种刚性接头。两种重组HBc转染HEK293细胞后均能发出绿色荧光,提示至少部分EGFP均获得了正确折叠。然而,两种 HBc-EGFP重组体所呈现的荧光表现明显不同,柔性接头连接者荧光均匀,而刚性接头者荧光呈分散点状,我们推测,导致荧光点状分布的原因可能有:1) HBc-EGFP分子间自发聚集成团,导致荧光成点状;2) 重组蛋白滞留在某种细胞器,不能进行正常运转。究竟何种机制导致这种表型尚待进一步研究,无论如何,这种表型的改变应该与刚性接头的加入有关。在对 HBc-EGFP的功能进行检测时,我们未能检测到病毒复制信号,可能的原因是融合的EGFP破坏了 HBc的正确结构,或者仅有少部分保留了正确结构而不足以被检测到。为了验证是否因为EGFP为较大的分子 (238 aa)而影响了HBc蛋白的结构与功能,我们缩短融合蛋白分子,其中一种仅为缺失aa79~80,发现这些分子仍不支持HBV复制,这提示aa79~80对于维持HBc的正常功能较为重要。Matthias等[26]证明,HBc蛋白缺失第80位氨基酸可支持HBV复制,因此我们认为aa79可能为HBc支持HBV复制不可缺失的氨基酸。
致谢 感谢美国 Drexel 大学医学院 Dr. Guo Haitao对本文的审阅。
[1]Liaw YF, Chu CM. Hepatitis B virus infection.Lancet, 2009, 373(9663): 582–589.
[2]McMahon B. Epidemiology and natural history of hepatitis B. Semin Liver Dis, 2005, 25(Suppl 1):3–8.
[3]Dane DS, Cameron CH and Briggs M. Virus-like particles in serum of patients with Australia-antigen- associated hepatitis. Lancet,1970, 295(7649): 695–698.
[4]Heermann KH, Goldmann U, Schwarz W, et al.Large surface proteins of hepatitis B virus containing the pre-s sequence. J Virol, 1984, 52(2):396–402.
[5]Steven AC, Conway JF, Cheng NQ, et al. Structure,assembly and antigenicity of hepatitis B virus capsid proteins. Adv Virus Res, 2005, 64:125–164.
[6]Chang C, Zhou S, Ganem D, et al. Phenotypic mixing between different hepadnavirus nucleocapsid proteins reveals C protein dimerization to be cispreferential. J Virol, 1994,68(8): 5225–5231.
[7]Pumpens P, Grens E. Hepatitis B core particles as a universal display model: a structure-function basis for development. FEBS Lett, 1999, 442(1):1–6.
[8]Crowther RA, Kiselev NA, Bottcher B, et al.Three-dimensional structure of hepatitis B virus core particles determined by electron cryomicroscopy. Cell, 1994, 77(6): 943–950.
[9]Konig S, Beterams G, Nassal M. Mapping homologous interaction sites in the hepatitis B virus core protein. J Virol, 1998, 72(6):4997–5005.
[10]Wynne SA, Crowther RA, Leslie AGW. The crystal structure of the human hepatitis B virus capsid. Molecular Cell, 1999, 3(6): 771–780.
[11]Conway JF, Cheng N, Zlotnick A, et al. Hepatitis B virus capsid: localization of the putative immunodominant loop (residues 78 to 83)on the capsid surface, and implications for the distinction between c and e-antigens. J Mol Biol, 1998,279(5): 1111–1121.
[12]Pollack JR, Ganem D. An RNA stem-loop structure directs hepatitis B virus genomic RNA encapsidation. J Virol, 1993. 67(6): 3254–3263.
[13]Yeh CT, Liaw YF, Ou JH. The arginine-rich domain of hepatitis B virus precore and core proteins contains a signal for nuclear transport. J Virol, 1990, 64(12): 6141–6147.
[14]Kratz PA, Bottcher B, Nassal M. Native display of complete foreign protein domains on the surface of hepatitis B virus capsids. Biochemistry, 1999,96(5): 1915–1920.
[15]Gallina A, Bonelli F, Zentilin L, et al. A recombinant hepatitis B core antigen polypeptide with the protamine-like domain deleted selfassembles into capsid particles but fails to bind nucleicacids. J Virol, 1989, 63(11): 4645–4652.
[16]Nassal M. The arginine-rich domain of the hepatitis B virus core protein is required for pregenome encapsidation and productive viral positive! strand DNA synthesis but not for virus assembly. J Virol, 1992, 66(7): 4107–4116.
[17]Huang Y, Tao Y, Zhang WL, et al. A new method for DNA molecular cloning. Sci Sin Vitae, 2011,41(9): 722–729 (in Chinese).黄媛, 陶颖, 张文露, 等. 一种新的 DNA 分子克隆方法. 中国科学, 2011, 41(9): 722–729.
[18]Aslanidis C, de Jong PJ. Ligation-independent cloning of PCR products (LIC-PCR). Nucleic Acids Res, 1990, 18(20): 6069–6074.
[19]Cui J, Hu JL, Zhang WL, et al. Analysis of hepatitis B virus replication in vitro by chemiluminescent detection method.Biotechnology, 2009, 19(2): 44–46 (in Chinese).崔静, 胡接力, 张文露, 等. 化学发光法检测体外HBV复制. 生物技术, 2009, 19(2): 44–46.
[20]Seeger C, Mason WS. Hepatitis B virus biology.Microbiol Mol Biol, 2000, 64(1): 51–68.
[21]Paran N, Ori A, Haviv I. A composite polyadenylation signal with TATA box function.Mol Cell Biol, 2000, 20(3): 834–841.
[22]Rieger A, Nassal M. Specific hepatitis B virus minus-strand DNA synthesis requires only the 5′encapsidation signal and the 3′-proximal direct repeat DR1. J Virol, 1996, 70(1): 585–589.
[23]Arai R, Ueda H, Kitayama A, et al. Design of the linkers which effectively separate domains of a bifunctional fusion protein. Protein Eng, 2001,14(8): 529–532.
[24]Niwa H, Inouye S, Hirano T, et al. Chemical nature of the light emitter of the aequorea green fluorescent protein. Proc Natl Acad Sci USA,1996, 93(24): 13617–13622.
[25]Borisova G, Arya B, Dislers A, et al. Hybrid hepatitis B virus nucleocapsid bearing an immunodominant region from hepatitis B virus surface antigen. J Virol, 1993, 67(6): 3696–3701.
[26]Koschel M, Oed D, Gerelsaikhan T, et al.Hepatitis B virus core gene mutations which block nucleocapsid envelopment. J Virol, 2000, 74(1):1–7.
