浙江沿海斑尾刺虾虎鱼的DNA条形码
2013-10-21顾翠平陈永久
顾翠平,高 鹏,陈永久
(浙江海洋学院海洋与科学学院,浙江舟山 316022)
虾虎鱼隶属鲈形目Perciformes、虾虎鱼亚目Gobioidei,是鲈形目中最大的亚目。据不完全统计,虾虎鱼亚目鱼类,全世界约有2 211种,分属于9科270属[1]。虾虎鱼广泛分布于亚洲、大洋洲、非洲、美洲和欧洲的热带、亚热带与温带水域中[2]。中国虾虎鱼的种类丰富,现有9科5亚科106属307种,其中淡水66种,海水241种,占世界虾虎鱼类总数的13.9%[3]。根据伍汉霖和钟俊生[3]记载,刺虾虎鱼Acanthogobius是虾虎鱼科中的一个属,已经报道的种类包括长体刺虾虎鱼A.elongata、黄鳍刺虾虎鱼A.flavimanus、乳色刺虾虎鱼A.lactipes、棕刺虾虎鱼A.luridus、斑尾刺虾虎鱼A.ommaturus和斑鳍刺虾虎鱼A.stigmothonus。
斑尾刺虾虎鱼A.ommaturus Richardson,又名矛尾刺虾虎鱼A.hasta、斑尾复虾虎鱼Synechogobius ommaturus。它们曾经认为是不同物种,但现在普遍接受的观点是,这些物种都为同种异名[3-5]。本文中涉及的A.hasta、A.ommaturus和S.ommaturus都称斑尾刺虾虎鱼。斑尾刺虾虎鱼是暖温性近岸底层中大型虾虎鱼类,主要分布在中国、朝鲜半岛及日本的沿海、港湾及河口咸淡水交混处,偶尔也进入淡水,喜欢栖息于底质为淤泥或泥沙的水域[6]。由于个体较大、生长较快,且肉质鲜美,有一定的经济价值,斑尾刺虾虎鱼越来越受到人们的关注,因此它们承受的捕捞压力也逐渐增大。
DNA条形码(DNA barcoding)通过已有的、短的、标准的参考物种DNA序列来鉴定新标本的种类,可以用来寻找新物种和隐种、区别形态难以辨别的姐妹种等。加拿大分类学家HEBERT等[7]首次提出DNA条形码的概念,之后越来越多的研究证明DNA条形码技术可以准确地鉴别物种[8]。采用COI基因序列,WARD等[9]发现澳大利亚207种的海洋鱼类均能得到有效的区分;JEON等鉴别出形态上难以辨别的虾虎鱼近缘种[10];此外,SIN等[11]利用COI基因和16S rRNA基因序列变异清楚地将形态相似的中国枪乌贼Loligo chinensis和剑枪乌贼L.edulis区分开。本文采用线粒体COI基因片段序列研究浙江沿岸刺虾虎鱼物种的DNA条形码、多样性以及系统关系,为科学合理地开发和保护刺虾虎鱼资源提供理论基础。
1 材料与方法
1.1 样本采集和保存
一共30尾刺虾虎鱼于2012-2013年夏天采自舟山群岛的泗礁(简称SJ)、大长涂(简称CT)、岱山(简称 DS)、舟山(简称 ZS)、朱家尖(简称 ZJJ)、桃花岛(简称 TH)、虾峙(简称 XZ)、象山旦门(简称 XS)、临海红脚岩(简称HJY)和苍南渔寮(简称YL)。详细信息见表1。取其腹鳍于95%的乙醇中,保存于-20℃冰箱中以备用。
1.2 DNA的提取、扩增及测序
基因组DNA的提取使用Promega公司Wizard○RGenomic DNA Purification Kit试剂盒(具体操作方法参考Promega的手册),DNA样本于-20℃保存备用。用1.2%琼脂糖凝胶电泳检测DNA质量,DNA浓度用紫外分光光度计检测。COI基因扩增引物来自WARD等[12],为FishF1:5'-TCAACCAACCACAAAGACATTGGCAC-3',FishR1:5'-TAGACTTCTGGGTGGCCAAAGAATCA-3'。引物由上海生工生物股份有限公司合成。
PCR扩增在一个25 μL的反应体系中进行,包括DNA Tag聚合酶,4种dNTP,以及1对核苷酸引物和DNA模板。PCR反应程序为95℃预变性2 min;95℃变性40 s,52℃退火 60 s,72℃延伸90 s,共35个循环;72℃最后延伸5 min。PCR产物用1.5%的琼脂糖凝胶电泳分离,检测阳性后送至上海生工股份有限公司,采用ABI遗传分析系统进行测序。
1.3 数据分析
DNA序列使用BioEdit v.7.0.9.0[13]和ClustalX 2.0[14]软件进行比对,并辅以人工校对。单倍型数、单倍型多样性指数(Hd)、核苷酸多样性指数(π)、核苷酸多态位点、简约信息位点数的计算使用DnaSP 5.0[15]软件。Kimura双参数遗传距离计算和聚类分析采用MEGA 5.0[16]软件;单倍型网络图使用Network[17]软件计算;刺虾虎鱼系统关系的分析以阿部鲻虾虎鱼为外群,采用NJ方法;聚类树各分支的置信度检验采用10 000重复次数的Bootstrap。贝叶斯(Bayesian)系统关系的构建来采用MrBayes v.3.1[18]。MrBayes采用jModeltest v.0.1.1[19]选出的最佳核苷酸替代突变模型,运行两个同步搜索,每个搜索4个MCMC(Markov Chain Monte Carlo;马尔可夫链蒙特卡洛)链和1 000万重复采样。采样间隔为1 000;最初的2 500个树被丢弃,链间分裂频率的平均标准差用来推断MCMC运行的完善程度。

表1 浙江沿海刺虾虎鱼的样本信息Tab.1 Sample information of Acanthogobius from coastal areas of Zhejiang.
2 结果
2.1 线粒体COI基因序列特征、单倍型和遗传距离
从30尾刺虾虎鱼样本中,获得长度为615 bp的COI DNA片段序列,共检测到10个核苷酸多态位点、简约信息位点4个、单倍型多样性指数(Hd)为0.69;核苷酸多样性指数(π)为0.0033。COI基因片段中 T、C、A、G 的含量分别为 30.5%、27.7%、23.7%、18.1%。可以看出,A+T含量(54.2%)稍高于G+C含量(45.8%)。
根据DNA条形码数据库(http://www.barcodinglife.org/)的建议,24条来自GenBank的刺虾虎鱼COI DNA序列被用于遗传距离和系统关系分析。为了让所有Acanthogobius的DNA序列长度一致,用BioEdit编辑后使其最终长度的都为552 bp。所有54条COI基因序列共定义了15个刺虾虎鱼单倍型(表1)。Kimura双参数模型计算的遗传距离(表2)显示,刺虾虎鱼物的遗传距离分为两类,种内和属内种间。其中,种内的距离为0.002~0.013(平均值0.007),属内种间的距离为0.158~0.217(平均值0.186),属内种间的距离显著大于种内的距离(约27倍)。浙江沿海的30个刺虾虎鱼样本属于7个单倍型,都为斑尾刺虾虎。将舟山群岛各点作为整体,由单倍型网络图可以看出,舟山群岛、象山旦门、临海红脚岩及苍南渔寮分享了同一单倍型H4,共16个个体,占总数的53%,H1-H3、H6只分布在舟山。舟山群岛和临海红脚岩分享了单倍型H5,H7只在象山旦门和苍南渔寮发现(单倍型网络如图1)。
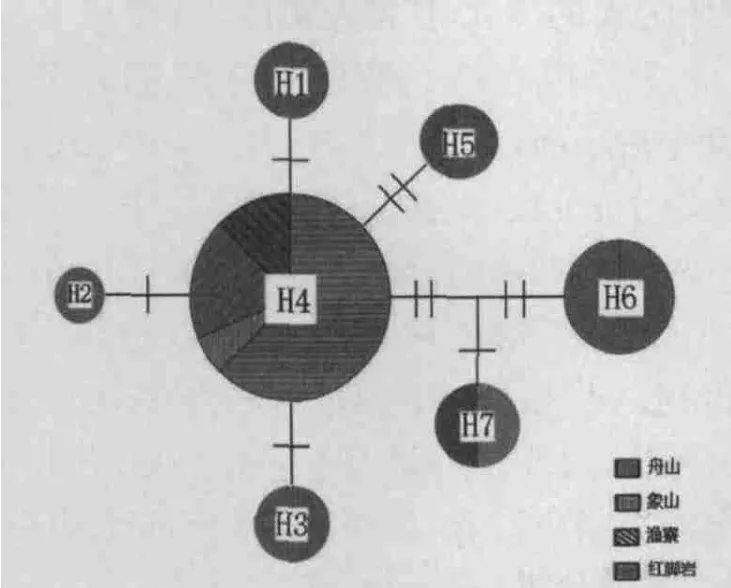
图1 基于COI序列浙江沿海刺虾虎鱼单倍型网络关系图圆圈面积与单倍型频率成正比,短划线代表单倍型间的核苷酸替换数目Fig.1 Haplotype network for COI of Acanthogobius from the coastal areas of Zhejiang
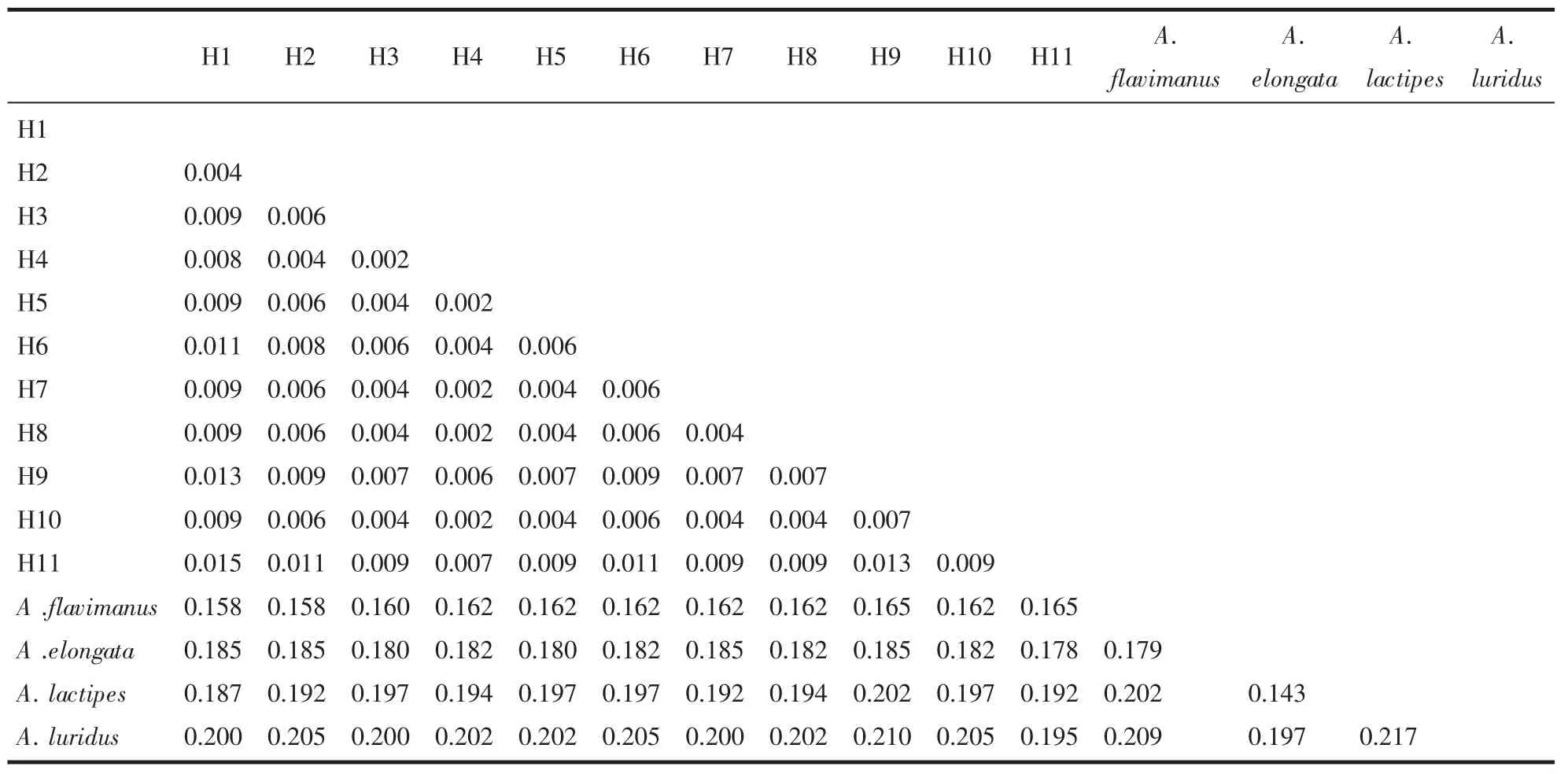
表2 基于COI基因片段序列的刺虾虎鱼的种间和种内遗传距离Tab.2 Intraspecific and interspecific genetic distances among samples of Acanthogobius based on COI gene sequences
2.2 刺虾虎鱼的系统发生关系
贝叶斯法系统关系分析采用jModeltest软件建议的最佳核苷酸替换模型TPM2uf+G。以阿部鲻虾虎鱼为外群,获得的刺虾虎鱼NJ和Bayesian系统关系的拓扑结构基本一致(Bayesian系统树如图2)。可以看出,所有A.ommaturus、A.hasta和S.ommaturus的单倍型共同形成一支亲缘关系很近的单系群,它们与黄鳍刺虾虎鱼A.flavimanus聚成一支;与之平行的一支则由长体刺虾虎鱼A.elongata和乳色刺虾虎鱼A.lactipes组成;此外,棕刺虾虎鱼A.luridus为单独一支。
3 讨论
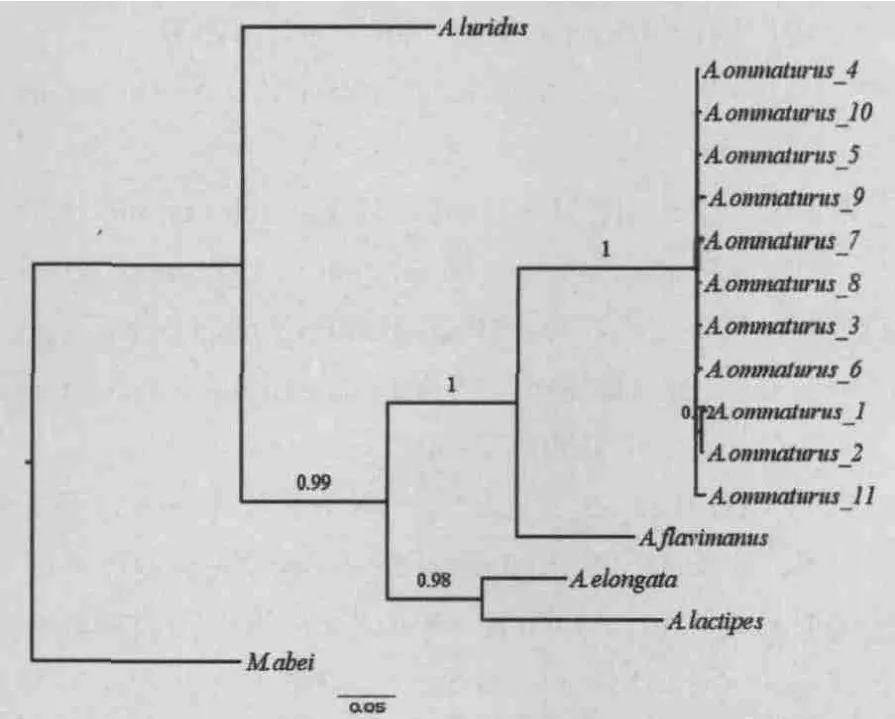
图2 基于COI基因单倍型序列Bayesian系统树(单倍型信息详情见表1)Fig.2 Bayesian phylogeny of Acanthogobius based on COI gene sequences(See Table 1 for haplotype information).
本研究建立了浙江沿海刺虾虎鱼线粒体COI基因片段615 bp的DNA条形码。COI碱基A+T含量(54.2%)高于G+C含量(45.8%),基本符合4种核苷酸在线粒体基因组中分布不均一性的特点,与在其它鲈形目中的结果类似[20]。
HEBERT等[7]提出利用COI基因序列进行物种鉴定的关键在于属内种间的遗传距离必须大于种内的遗传距离,而且种间距约为种内距离的10倍以上。通过对动物界13 320个物种进行分析,HEBERT等认为大部分动物种内距离小于0.01,很少大于0.02。我们的DNA条形码中,刺虾虎鱼种内距离绝大多数小于0.01,最大为0.013,与Hebert得出的结论一致。刺虾虎鱼属内种间的平均距离远远大于种内平均距离,约27倍左右,也与HEBERT的结果相吻合。
单倍型网络关系结果显示,舟山群岛、象山旦门、临海红脚岩及苍南渔寮都分享了单倍型H4,共16个个体,占总数的53%,推测单倍型H4很可能是较原始的单倍型类型。
DNA条形码分析结果显示,浙江沿海的斑尾刺虾虎与斑尾复虾虎鱼和矛尾刺虾虎鱼具有很近的亲缘关系,与目前普遍接受的观点吻合,即它们为同种异名的[3-5]。长期以来,斑尾刺虾虎鱼和矛尾刺虾虎鱼的分类地位存在一定的争议。矛尾刺虾虎鱼是Temminck和Schlegel根据采自日本长崎的标本命名[3],斑尾刺虾虎鱼是RICHARDSIN在1845年以采自长江口和广州的标本命名。1845年RICHARDSON还命名了S.ommaturus(http://fishdb.sinica.edu.tw/)。GÜNTHER和Bleeker认为这斑尾刺虾虎鱼和矛尾刺虾虎鱼是不同物种[21-23],而TOMIYAMA、秦克静等和倪勇等发表论文建议它们属于同一个物种[4-5,24]。由于A.ommaturus和A.hasta都在1845年发表,而后者发表的月份未注明,因此只能认为是在1845年的最后一个月,即12月。根据命名法则的优先规律,A.hasta只能是A.ommaturus的异名[3]。目前,伍汉霖等和台湾鱼类资料库(http://fishdb.sinica.edu.tw/)中均使用的是A.ommaturus;而中国海洋生物名录[25]中还采用的是A.hasta。
[1]NELSON J S.Fishes of the world[M].4th ed.New York:John Wiley and Sons,2006.
[2]THACKER C E.Molecular phylogeny of the gobioid fishes(Teleostei:Perciformes:Gobioidei)[J].Molecular Phylogenetics and Evolution,2003(26):354-368.
[3]伍汉霖,钟俊生.中国动物志.硬骨鱼纲.鲈形目.虾虎鱼亚目[M].北京:科学出版社,2008.
[4]倪 勇.上海鱼类志[M].上海:上海科学出版社,1990.
[5]秦克静.辽宁动物志.虾虎鱼亚目.[M].沈阳:辽宁科学技术出版社,1987.
[6]成庆泰,郑葆珊.中国鱼类检索[M].北京:科学出版社,1987.
[7]HEBERT P N D,CYWINSKA A,BALL S L,et al.Biological identifications through DNA barcodes[J].Proceedings of the Royal Society of London Series B,Biological Sciences,2003a(270):313-321.
[8]TAVARES E S,BAKER A J.Single mitochondrial gene barcodes reliably identify sister-species in diverse clades of birds[M].BMC Evolutionary Biology,2008,8(1):81-81.
[9]WARD R D,HANNER R,HEBERT P D N.The campaign to DNA barcode all fishes[J].Journal of Fish Biology,2009,74(2):329-356.
[10]JEON H B,CHOI S H,SUK H Y.Exploring the utility of partial cytochrome c oxidase subunit 1 for DNA barcoding of gobies[J].Animal Systematics,Evolution and Diversity,2012(28):269-278.
[11]SIN Y W,YAU C,CHU K H.Morphological and genetic differentiation of two loliginid squids,Uroteuthis (Photololigo)chinensis and Uroteuthis(Photololigo)edulis(Cephalopoda:Loliginidae)in Asia[J].Journal of Experimental Marine Biology and Ecology,2009(369):22-30.
[12]WARD R D,ZEMALK T S,INNES B H,et al.DNA barcoding Australia's fish species[J].Philosophical Transactions of the Royal Society B,Biological Sciences,2005(360):1 847-1 857.
[13]HALL T A.BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J].Nucleic Acids Symposium Series,1999(41):95-98.
[14]CHENNA R,SUGAWARA H,KOIKE T,et al.Multiple sequence alignment with the Clustal series of programs[J].Nucleic Acids Research,2003(31):3 497-3 500.
[15]LIBRADO P,ROZAS J.DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J].Bioinformatics,2009(25):1 451-1 452.
[16]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular Biology and Evolution,2011(28):2 731-2 739.
[17]BANDELT H J,FORSTER P,R?HL A.Median-joining networks for inferring intraspecific phylogenies[J].Molecular Biology and Evolution,1999(16):37-48.
[18]RONQUIST F,HUELSENBECK J P.MrBayes 3:Bayesian phylogenetic inference under mixed models、[J].Boformatics,2003(19):1 572-1 574.
[19]POSADA D.jModelTest:Phylogenetic Model Averaging[J].Molecular Biology and Evolution,2008(25):1 253-1 256.
[20]杨 帆,何利军,雷光春,等中国东南沿海弹涂鱼科常见鱼类的遗传多样性和DNA条形码[J].生态学杂志,2012,3 1(3):676-683.
[21]GÜNTHER A.Catalogue of the fishes in British Museum.Catalogue of the acanthopterygain fishes in the collection of the British Museum[M].London:British Museum,1861(373):21-142.
[22]GÜNTHER A.Report on a collection of fishes made by Mr.Swinhoe in China[R].The Annals and Magazine of Natural History,1874,13(4):154-159.
[23]BLEEKER P.Memoire sur la fanua ichthyologique de Chine[M].Ned Tijdschr Dierk,1873(4):113-154.
[24]TOMIYAMA I.Gobiidae of Japan[J].Japan J Zool,1936,7(1):37-112.
[25]刘瑞玉.中国海洋生物名录[M].北京:科学出版社,2008.
