E R I C-P C R技术对单增李斯特菌的溯源分析
2013-05-18刘海泉姜文洁孙晓红吴启华潘迎捷
刘海泉,朱 颖,姜文洁,孙晓红,吴启华,潘迎捷,赵 勇,*
(1.上海海洋大学食品学院,上海201306;2.农业部水产品贮藏保鲜质量安全风险评估实验室(上海),上海201306;3.上海水产品加工及贮藏工程技术研究中心,上海201306;4.美国缅因大学食品科学与人类营养系,美国缅因州04469-5735)
单增李斯特菌(Listeria monocytogenes)是一种重要的食源性致病菌[1],因感染后引起的李氏杆菌病死亡率约为20%~30%[2]。根据李斯特菌具有特定的表面蛋白,结合玻片凝集实验,单增李斯特菌至少有13种血清型[3]。目前,单增李斯特菌流行病学调查的传统方法多以血清分型、噬菌体分型等表型分析方法为主[4-6],这些传统的分型方法常常存在较高的误差。分子分型是一种新兴的流行病学调查手段,不仅克服了血清分型等传统方法的不足,还能从遗传学角度进行流行规律探讨。其中,ERIC-PCR是基于“肠道细菌基因间重复序列”(Enterobacterial Repetitive Intergenic Consensus,ERIC)具有分型率较高、准确性好、简便快捷、无需特殊仪器等优点,首先在大肠杆菌中应用[7];随后扩展到其他人类病源菌,包括沙门氏菌[8-9]、副溶血性弧菌[10-12]等,以及动物及植物病原菌[13-15]。本研究采用ERIC-PCR法对从三个农贸市场猪肉样品分离到的17株菌株进行了基因分型,并进行了多样性分析,探讨了基因型与区域性分布的关联性,以期为单增李斯特菌的溯源和流行性分析提供指导。
1 材料与方法
1.1 材料与仪器
菌株 根据国家标准[16]从上海地区三个农贸市场的猪肉样品中分离到17株单增李斯特菌菌株(表1),这些菌株分别从市场中不同摊位的样品中分离出;单增李斯特菌ATCC 19115(简写为LM) 作为阳性对照;Taq DNA聚合酶、dNTP 大连TaKaRa公司;Biospin细菌基因组DNA提取试剂盒 杭州Bioer公司;DNA Marker、溶菌酶、蛋白酶K 北京天根生化科技有限公司;李斯特菌选择性增菌液(LB1)、胰蛋白胨大豆肉汤(TSB)、胰蛋白胨大豆琼脂(TSA) 北京陆桥技术责任有限公司;李斯特菌选择性琼脂(PALCAM)、脑心浸液(BHI)、抗生素 英国OXOID公司;Milli-Q水。

表1 菌株来源及背景Table 1 Source and background of the strains
离心机、PCR仪、微量移液器 德国Eppendorf;电泳仪、凝胶成像分析系统 美国Bio-Rad公司。
1.2 实验方法
1.2.1 细菌DNA提取 取于-80℃、25%甘油保存的菌种,接种于BHI肉汤在37℃下复苏后,划线接种于TSA琼脂,挑取单菌落接种于营养肉汤中,在37℃下培养至对数期(OD600=0.8)。再划线接种于PALCAM琼脂上于37℃培养24h,连续活化2次。挑取单菌落,接种于LB1中,37℃ 170r/min振荡培养,培养至OD600=0.8提取DNA[17]。使用Biospin细菌基因组DNA提取试剂盒。按照说明书提取DNA。
1.2.2 PCR方法 ERIC-PCR引物参照Jersek等[18]的设计。正向引物Eric1R:ATGTAAGCTCCTGGGGATT CAC,反向引物Eric2:AAGTAAGTGACTGGGGTGAG CG,引物由上海生物工程技术有限公司合成。
PCR扩增体系溶液体积为25μL,其中ddH2O 16.2μL、10×缓冲液(+Mg2+)2.5μL、dNTPs(2.5mmol·L-1)2.0μL、引物10μmol·L-1各1μL、Taq酶(5U·μL-1)0.3μL、模板DNA 2.0μL。
PCR程序如下:95℃预变性2min;94℃ 30s,92℃30s,50℃ 30s,52℃ 1min,65℃8min,共循环35次;最后65℃维持8min。PCR扩增产物在1.5%琼脂糖凝胶120V电压,电泳30min。
1.2.3 ERIC-PCR分型结果分析 经BioRad Molecular Imager® Gel Doc TM XR凝胶拍照后,使用自带的ImageLab Version 3.0分析软件中的“分析工具箱”-“泳带和条带”进行泳带分析;采用“1”和“0”的方式记录在Excel中,每个样品的扩增带存在时赋值为“1”,不存在时赋值为“0”。最后,通过NTsys 2.10e进行分析,得到Jaccard的遗传相似性系数矩阵和遗传距离矩阵,采用非加权配对法(UPGMA法)做遗传分析的树状图。凡相似度大于0.8者为同一亚型,小于0.8者为不同的基因型[19]
2 结果与讨论
2.1 单增李斯特菌的ERIC-PCR分型
实验结果表明,17株单增李斯特菌和质控菌株ATCC 19115(LM)经ERIC-PCR扩增,产生了12种不同的指纹图,且指纹图谱呈现出多态性(图1)。PCR产物为3~8条,主带为2条。
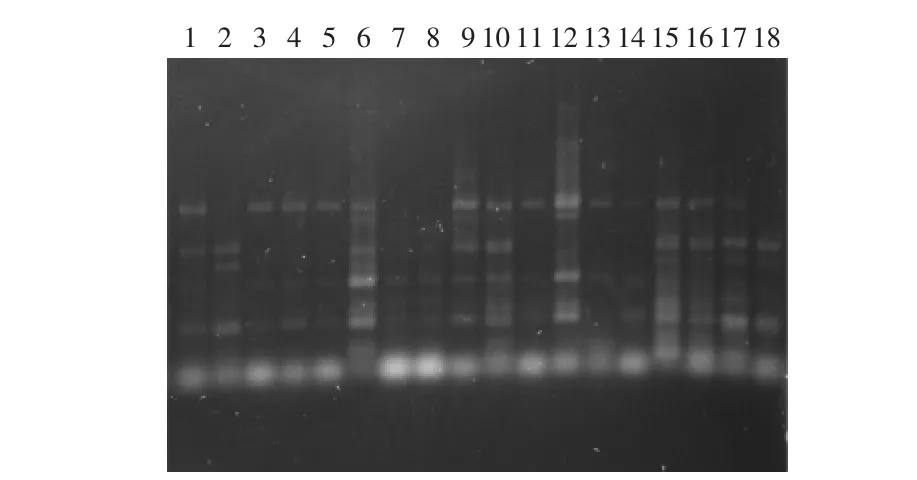
图1 单增李斯特菌菌株ERIC-PCR指纹图Fig.1 ERIC-PCR fingerprints of Listeria monocytogenes strains注:1.ATCC 19115;2~18,LM1~LM17。
2.2 单增李斯特菌ERIC-PCR指纹图谱聚类分析
聚类分析结果表明,17株单增李斯特菌分为了至少6个基因类群,分别标记为Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ及Ⅵ型(图2)。其中Ⅳ型菌株最多,达11株;Ⅴ型略少,为2株;Ⅰ、Ⅱ、Ⅲ和Ⅵ型最少,均只有1株,其中LM1与质控菌株ATCC 19115最近。
从图2可以看出,菌株LM2、LM10、LM12和LM13,LM5和LM8,LM6和LM7可能为同一菌株。大部分菌株可能为LM9菌株的相似菌株,而菌株LM1与菌株LM16和LM17位于另外的分枝。菌株LM2、LM3、LM4、LM5、LM6、LM7、LM8、LM10、LM11、LM12和LM13为同一分支,说明它们可能为同类菌株,但来源不同,说明该类菌污染了多个不同样品,是重要的污染食品的菌株类型;菌株LM12/LM13、LM14/LM15、LM16与LM17为同一批次、同一采集点、不同样品分离到的,它们处于不同分支,说明它们不是同一菌株,这揭示在同一地点有可能污染多种单增李斯特菌。菌株LM1、菌株LM9、菌株LM14/LM15、LM16和LM17分别代表了不同菌株,而它们分别只在一个地点分离得到,说明它们是在本实验采样范围内,污染食品较少的一株单增菌株。这17株菌株中至少包含了6种血清型。

图2 17株单增李斯特菌指纹图谱聚类分析图Fig.2 Dendrogram based on UPGMA cluster analysis of ERIC-PCR data from 17 Listeria monocytogenes strains
此外,从不同地点分离到的单增李斯特菌呈现出不同的聚类特征。其中,市场3中分离出的菌株表现为最多的基因型类群,而市场1和2中分离到的基因型类群主要是Ⅰ和Ⅳ类群;Ⅳ类群是主要类群(图3)。
通过分析可以发现ERIC-PCR基于基因组上的特定重复序列能够很好把来源不同的单增李斯特菌菌株进行区分、归类,进而达到溯源目的。

图3 单增李斯特菌ERIC-PCR基因型类群与分离点间的关系Fig.3 Relationship between Listeria monocytogenes strains’sources and ERIC-PCR genotypes
单增李斯特菌是一种食源性、细胞内寄生致病菌,可引起严重的李氏杆菌病,已引起了公众和食品安全专家的关注。由于其天然抗性,单增李斯特菌在自然界和食品加工厂中广泛存在,99%的人类李氏杆菌病是因食用受污染的食品而引起[2,20]的。虽然单增李斯特菌不形成芽孢,它可以在食品加工环境中广泛存在,并能坚持较长的时间,甚至数年之久[21]。单增李斯特菌已成为一个重大的公共卫生问题。由于单增李斯特菌菌株的毒力和致病性存在差异,具有多样性[22]。而亚型分型可以准确、迅速地确定或追踪单增李斯特菌单个菌株,这在控制和预防李氏杆菌病以及菌株的流行病学和群体遗传学研究是必不可少的,这样可以阻止李氏杆菌病的蔓延和减少不必要的食品召回。分子示踪能够准确地评价单增李斯特菌特定的污染来源[23]。
基因分型的方法包括了基于基因片段的亚型技术[脉冲场凝胶电泳(pulsed-field gel electrophoresis,PFGE)、扩增片段长度多态性(amplified-fragment length polymorphism,AFLP)、核糖体分型、质粒分型(plasmid typing)、多位点可变数目串联重复分析(multilocus variable-number tandem-repeat analysis,MLVA)和可变位点或缺检测(detection of loci that are variably absent or present,VAP)];基于PCR的分型技术[随机扩增多态性DNA(random amplification of polymorphic DNA,RAPD)、扩增片段长度多态性(amplified fragment length polymorphism,AFLP),PCR-限制性片段长度多态性(PCR-restriction fragment length polymorphism,PCR-RFLP)和重复元素PCR(repetitive element PCR,REP-PCR技术)]和DNA测序为基础的亚型技术[多位点序列分型(multi-locus sequence typing,MLST)、多毒力位点序列分型(multiple-virulence-locus sequence typing,MVLST)和单位点的测序和单核苷酸多态性(SNP)][24-25]。虽然PFGE法准确性最高,但由于操作步骤繁杂,且需要特殊仪器,在普通实验室很难开展;而ERIC-PCR法操作简便,无需特殊仪器,且重复性和区分能力同PFGE非常相近,更易于在普通实验室中应用[26]。虽然根据Hunter等[27]介绍的计算方法,该分型方法的辨别指数值只有60%,但通过金莉莉等[28-30]从2002年使用ERICPCR来鉴定食品中的单增李斯特菌,以及本研究进一步表明ERIC-PCR能够很好的区分不同来源单增李斯特菌菌株来看,ERIC-PCR为单增李斯特菌的流行病学调查以及有效防控单增李斯特菌的污染,提供了适用于普通实验室的简单、方便、快捷、准确的方法。
3 结论
ERIC-PCR技术能够区分、鉴别不同来源的单增李斯特菌菌株,实现实验室对食品中单增李斯特菌溯源及示踪,为控制食品中单增李斯特菌的污染及防控李氏杆菌病提供了切实可行的工具。
[1]Norton D M,Braden C R.Foodborne Listeriosis.[M].Boca Raton:CRC Press,2007:305-306.
[2]Mead P S,Slutsker L,Dietz V,et al.Food-related illness and death in the United States[J].Emerging Infectious Diseases,1999,5(5):607-625.
[3]Kathariou S. Listeria monocytogenes virulence and pathogenicity,a food safety perspective[J].J Food Prot,2002,65(11):1811-1829.
[4]Liu D Y,Lawrence M L,Gorski L,et al.Listetia monocytogenes serotype 4b strains belonging to lineages I and III possess distinct molecular features[J].Journal of Clinical Microbiology,2006,44(1):214-217.
[5]Loessner M J.Improved procedure for bacteriophage typing of Listeria strains and evaluation of new phages[J].Applied and environmental microbiology,1991,57(3):882-884.
[6]Graves L M,Swaminathan B,Reeves M W,et al.Comparison of ribotyping and multilocus enzyme electrophoresis for subtyping of Listeria monocytogenes isolates[J].Journal of Clinical Microbiology,1994,32(12):2936-2943.
[7]Versalovic J,Koeuth T,Lupski J R.Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes[J].Nucleic acids research,1991,19(24):6823-6831.
[8]Burr M D,Josephson K L,Pepper I L.An evaluation of ERIC PCR and AP PCR fingerprinting for discriminating Salmonella serotypes[J].Letters in Applied Microbiology,1998,27(1):24-30.
[9]张宏梅,石磊,李琳.沙门氏菌ERIC指纹分析[J].云南农业大学学报,2007(4):467-470.
[10]金莉莉,董雪,王秋雨,等.沈阳市副溶血弧菌重复序列PCR分型[J].中国公共卫生,2008(3):351-353.
[11]Khan A A,McCarthy S,Wang R F,et al.Characterization of United States outbreak isolates of Vibrio parahaemolyticus using enterobacterial repetitive intergenic consensus(ERIC) PCR and development of a rapid PCR method for detection of O3:K6 isolates[J].Fems Microbiology Letters,2002,206(2):209-214.
[12]丁久法,潘迎捷,陈洪友,等.副溶血性弧菌ERIC-PCR分型及毒力基因检测研究[J].食品工业科技,2010(8):137-141.
[13]Louws F J,Rademaker J L W,de Bruijn F J.The three Ds of PCR-based genomic analysis of phytobacteria:Diversity,detection,and disease diagnosis[J].Annual Review of Phytopathology,1999,37:81-125.
[14]肖丹,曹海鹏,胡鲲,等.淡水养殖动物致病性嗜水气单胞菌ERIC-PCR分型与耐药性[J].中国水产科学,2011(5):1092-1099.
[15]李鹏,李军星,李玉峰,等.中国东南部地区副猪嗜血杆菌分离株ERIC-PCR指纹图谱分析[J].中国兽医学报,2009(12):1566-1570.
[16]刘秀梅,杨洋,陈伟伟,等.GB/T4789.30-2010,食品安全国家标准食品微生物学检验单核细胞增生李斯特氏菌检验[S].北京:中国标准出版社,2010.
[17]刘海泉,赵强,孙晓红,等.多重PCR快速检测食品中的单核细胞增生性李斯特菌[J].中国农业科学,2010(23):4893-4900.
[18]Jersek B,Gilot P,Gubina M,et al.Typing of Listeria monocytogenes strains by repetitive element sequence-based PCR[J].Journal of Clinical Microbiology,1999,37(1):103-109.
[19]伍晓锋,黎毅敏,卓超,等.18株铜绿假单胞菌的耐药谱和ERIC-PCR分型[J].中国抗生素杂志,2007(9):560-563.
[20]Jalali M,Abedi D.Prevalence of Listeria species in food productsin Isfahan,Iran[J].InternationalJournalofFood Microbiology,2008,122(3):336-340.
[21]Lunden J M,Autio T J,Korkeala H J.Transfer of persistent Listeria monocytogenes contamination between food-processing plants associated with a dicing machine[J].J Food Prot,2002,65(7):1129-1133.
[22]Liu D,Ainsworth A J,Austin F W,et al.Characterization of virulent and avirulent Listeria monocytogenes strains by PCR amplification of putative transcriptional regulator and internalin genes[J].J Med Microbiol,2003,52(Pt 12):1065-1070.
[23]Meinersmann R J,Phillips R W,Wiedmann M,et al.Multilocus sequence typing of Listeria monocytogenes by use of hypervariable genes reveals clonal and recombination histories of three lineages[J].Appl Environ Microbiol,2004,70(4):2193-2203.
[24]Boxrud D.Advances in subtyping methods of foodborne disease pathogens[J].Curr Opin Biotechnol,2010,21(2):137-141.
[25]Liu D.Identification,subtyping and virulence determination of Listeria monocytogenes,an important foodborne pathogen[J].J Med Microbiol,2006,55(6):645-659.
[26]Liu P Y-F,Shi Z-Y,Lau Y-J,et al.Comparison of different PCR approaches for characterization of Burkholderia(Pseudomonas) cepacia isolates[J].Journal of Clinical Microbiology,1995,33(12):3304-3307.
[27]Hunter P R,Gaston M A.Numerical index of the discriminatory ability oftyping systems:an application of Simpson’s index of diversity[J].Journal of Clinical Microbiology,1988,26(11):2465-2466.
[28]金莉莉,王秋雨,李万杰,等.ERIC-PCR方法鉴定单核细胞增生性李斯特氏菌研究[J].辽宁大学学报:自然科学版,2002(2):155-158.
[29]金莉莉,王秋雨,侯潇.ERIC-PCR技术在李斯特氏菌种、菌株鉴定中的应用[J].遗传,2003(2):195-197.
[30]金莉莉,王秋雨,王芳,等.食源性单核增生李斯特氏菌重复序列-PCR分型与鉴定研究[J].中国人兽共患病学报,2008(8):712-717.
