高发区早期食管癌蛋白指纹图谱模型的建立及其筛查价值
2012-05-08于卫芳牛巍巍张立玮王士杰
于卫芳,牛巍巍,李 超,张立玮,王士杰
(1.河北医科大学第一医院内镜中心,河北石家庄 050031;2.河北医科大学第四医院内镜室,河北石家庄 050011)
·论 著·
高发区早期食管癌蛋白指纹图谱模型的建立及其筛查价值
于卫芳1,牛巍巍1,李 超1,张立玮2*,王士杰2
(1.河北医科大学第一医院内镜中心,河北石家庄 050031;2.河北医科大学第四医院内镜室,河北石家庄 050011)
目的建立高发区早期、进展期食管癌的蛋白指纹图谱模型并探究筛查价值。方法使用飞行质谱技术检测高发区内镜筛查的38例正常对照、15例早期食管癌和36例进展期食管癌患者的血清蛋白质谱,支持向量机分别建立蛋白指纹图谱模型,留一法交叉验证判别效果。结果区分早期食管癌和正常对照的蛋白指纹图谱模型特异性为81.58%,敏感性为80.00%;区分进展期食管癌和正常对照的模型特异性为89.47%,敏感性为83.33%;区分早期食管癌和进展期食管癌的模型特异性为73.33%,敏感性为91.67%。质荷比峰4 291、4 392、4 823、4 975、5 644、5 665、5 932、8 473、8 776、8 910Da的表达量组间比较差异有统计学意义(P<0.05,P<0.01)。结论3个蛋白指纹图谱模型的建立,为筛查高发区早期食管癌提供了一种新方法;构建模型的质荷比峰可能是早期食管癌的潜在生物学标记物。
食管肿瘤;肽谱;普查
1 资料与方法
1.1 一般资料:选取河北省食管癌高发区磁县和涉县的40~69岁研究对象89例,男性48例,平均(57.42±9.60)岁,女性41例,平均(57.68±9.74)岁。均行电子胃镜检查结合碘染色指示性活检,并经2位组织病理学专家确诊。其中正常对照组38例,早期食管癌组15例和进展期食管癌组36例。
1.2 仪器与试剂:采用美国 Ciphergen Biosystems公司生产的PBSⅡc型飞行质谱仪及CM10蛋白芯片(弱阳离子交换芯片)对血清标本进行蛋白质谱分析。SPA(sinapinic acid)试剂购自美国F1uka公司。尿素、乙腈、CHAPS、三羟甲基氨基甲烷蓝酸盐等试剂购自美国Sigma公司。
1.3 蛋白芯片分析:全部研究对象于清晨空腹抽取静脉血2mL,室温静置2h后,5 000r/min离心5min(重复操作2次),提取血清-80℃保存。血清样本冰上融解,4℃ 10 000r/min离心4min,留取上清液。取96孔板放置在冰盒上,每孔上加10μL U9缓冲液(9mo1/L Urea 2.7g溶于2mL去离子水中,2%CHAPS 0.1g,1%二硫苏糖醇0.05g,定容到5mL),每孔分别再加5μL血清,放入层析柜4℃振荡30min。U9缓冲液处理后96孔板放冰上,快速加入185μL醋酸钠(100mmo1/L,pH4.0)溶液,层析柜中4℃振荡2min。CM10芯片通过加200μL醋酸钠溶液活化,在层析柜中振荡5min(重复1次)。加100μL处理好的样本到芯片上,去除气泡后于层析柜中600r/min 4℃振荡1h后,拍干。加入200μL醋酸钠冲洗重复3次,拍干。加去离子水200μL连续2次,拍干。SELDI-TOF-MS分析前,加50%饱和SPA溶液1μL,5min后再点1μL,干燥后行表面增强激光解吸电离飞行时间质谱技术(surface enhanced 1aser desorption/ionization time of f1ight mass spectrometry,SELDI-TOF-MS)检测。
1.4 数据处理:SELDI-TOF-MS仪的校准依据Ciphergen公司的“一体化肽分子质谱标准”。激光强度设为180,灵敏度为7,收集数据的最高相对分子质量100 000,优化相对分子质量范围是2 000~20 000。采用浙江大学肿瘤所设计的蛋白芯片数据分析系统(ZUCI-Protein Chip Data Ana1yze System)软件包对质谱数据行过滤噪音、聚类分析和强度均一化处理。本组设13例质量控制样本(取自同一标准血清样本),随机分布在实验样本中进行检测。峰值大小和强度变异系数(coefficient of variance,cv)值分别为0.05%和19.7%。
1.5 生物信息学分析:应用支持向量机算法。采用径向基核函数,Gamma值设为0.6,罚分函数设为19。特征向量的选取采用统计过滤结合模型依赖性筛选的方法。对质荷比峰值做Wi1conxon秩和检验,选出P值最小的10个峰进一步分析。将10个峰的任意组合用于支持向量机模型的输入,选出约登指数最高的组合建立蛋白指纹图谱模型作为最终结果。用留一法交叉验证评估模型判别效果,其原理是每次留下一个样本作为测试,其余样本作为训练组,重复操作直到每例样本均被作为一次测试样本为止。
1.6 统计学方法:应用 ZUCI-ProteinChip Data Ana1yze System软件进行分析,计量资料以±s表示,采用t检验。P<0.05为差异有统计学意义。
2 结 果
2.1 区分早期食管癌与正常对照组血清蛋白指纹图谱模型的建立:使用SELDI-TOF-MS仪分析38例正常对照与15例早期食管癌的血清蛋白质谱。在进行降噪处理和峰值鉴别后,在相对分子质量2 000~20 000范围内,共检测到137个蛋白质荷比峰。采用支持向量计算法筛选出3个质荷比峰(M/ Z值为3 896、4 823、7 795Da),建立区分早期食管癌与正常对照组的血清蛋白指纹图谱模型,经留一法交叉验证,其特异性为81.58%,敏感性为80.00%。其中4 823Da在早期食管癌组和正常对照组中表达差异有统计学意义(P<0.05)。见表1,图1。
表1 早期食管癌组和正常对照组3个质荷比峰表达量比较Table 1 Comparison of expression of protein peaks between early esophageal cancer and normal control(±s,Da)

表1 早期食管癌组和正常对照组3个质荷比峰表达量比较Table 1 Comparison of expression of protein peaks between early esophageal cancer and normal control(±s,Da)
*P<0.05 vs norma1 contro1 by t test
Groups n Protein peak(M/Z)3 896 4 823 7 795 Norma1 contro1 38 595.44±296.94 597.54±363.01 648.79±291.61 Ear1y esophagea1 cancer 15 720.31±288.86 422.82±108.43*830.59±356.30

图1 模型中3个质荷比峰的质谱图(M/Z值为3 896、4 823、7 795Da)0.正常对照组;1.早期食管癌组Figure 1 Mass spectrum of three protein peaks in protein fingerprint mode1 0.Norma1 contro1 group;1.Ear1y esophagea1 cancer group
2.2 区分进展期食管癌与正常对照组血清蛋白指纹图谱模型的建立:使用SELDI-TOF-MS仪分析38例正常对照与36例进展期食管癌血清蛋白质表达谱,共检测到140个蛋白质荷比峰,其中17个峰表达量的差异有统计学意义(P<0.01)。采用支持向量计算法筛选出由6个质荷比峰(M/Z值为4 291、4 975、5 644、5 665、5 932、8 776Da)构建的蛋白指纹图谱模型,经留一法交叉验证,其特异性为89.47%,敏感性为83.33%。其中4 975、5 644、5 665、5 932 Da在进展期食管癌组中表达显著高于正常对照组(P<0.01)。见表2,图2。
表2 进展期食管癌组和正常对照组6个质荷比峰表达量比较Table 2 Comparison of expression of protein peaks between advanced esophageal cancer and normal control(±s,Da)

表2 进展期食管癌组和正常对照组6个质荷比峰表达量比较Table 2 Comparison of expression of protein peaks between advanced esophageal cancer and normal control(±s,Da)
*P<0.01 vs norma1 contro1 by t test
Groups n Protein peak(M/Z)4 291 4 975 5 644 5 665 5 932 8 776 Norma1 contro1 38 2 311.11±731.92 617.73±397.99 3 082.75±1 051.75 408.08±152.02 870.01±558.95 2 400.89±666.98 Advanced esophagea1 cancer 36 1 752.79±691.59* 1 215.47±993.19* 5 265.85±2 423.57* 570.56±215.96* 1 592.07±1 037.80* 1 761.91±627.82*
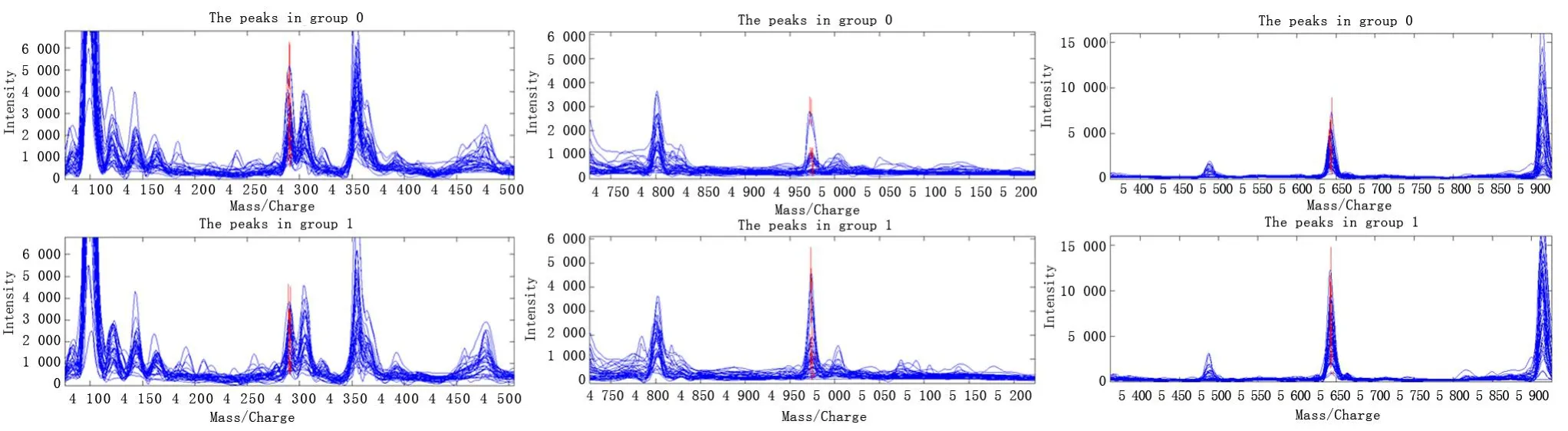

图2 模型中6个质荷比峰的质谱图(M/Z值为4 291、4 975、5 644、5 665、5 932、8 776Da)0.正常对照组;1.进展期食管癌组Figure 2 Mass spectrum of six protein peaks in protein fingerprint mode10.Norma1 contro1 group;1.Advanced esophagea1 cancer group
2.3 区分早期食管癌和进展期食管癌血清蛋白指纹图谱模型的建立:使用SELDI-TOF-MS仪分析比较15例早期食管癌和36例进展期食管癌患者的血清蛋白质表达谱,共检测到145个蛋白质荷比峰,其中13个差异有统计学意义(P<0.05)。支持向量计算法筛选出由5个质荷比峰(M/Z值为4 290、 4 392、5 643、8 473、8 910Da)构建的诊断模型,其特异性为73.33%,敏感性为91.67%。其中4 290、4 392、8 473、8 910Da在早期食管癌组中高表达,而在进展期食管癌组中低表达;5 643Da与之相反。见表3,图3。
表3 早期食管癌组和进展期食管癌组5个质荷比峰表达量比较Table 3 Comparison of expression of protein peaks between early esophageal cancer and advanced esophageal cancer(±s,Da)
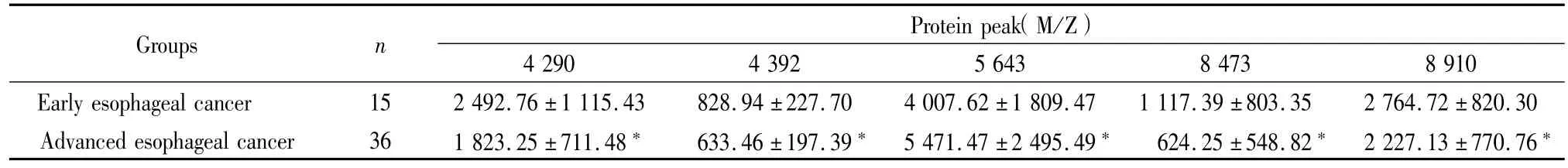
表3 早期食管癌组和进展期食管癌组5个质荷比峰表达量比较Table 3 Comparison of expression of protein peaks between early esophageal cancer and advanced esophageal cancer(±s,Da)
*P<0.05 vs ear1y esophagea1 cancer by t test
4 290 4 392 5 643 8 473 8 910 Ear1y esophagea1 cancer 15 2 492.76±1 115.43 828.94±227.70 4 007.62±1 809.47 1 117.39±80 Groups n Protein peak(M/Z)3.35 2 764.72±820.30 Advanced esophagea1 cancer 36 1 823.25±711.48* 633.46±197.39* 5 471.47±2 495.49* 624.25±548.82* 2 227.13±770.76*
图3 模型中5个质荷比峰的质谱图(M/Z值为4290、4392、5643、8473、8910Da)0.早期食管癌组;1.进展期食管癌组Figure 3 Mass spectrum of six protein peaks in protein fingerprint mode10.Ear1y esophagea1 cancer group;1.Advanced esophagea1 cancer group
3 讨 论
随着人类基因组计划的完成,蛋白质组学逐渐成为生物领域的主要研究方向。蛋白质能直接反应基因信息,其表达谱能更直观地反映生物体功能变化。在肿瘤发展的早期阶段,即可出现一些蛋白在质和量上的改变,均可能成为肿瘤早期诊断的预警分子指标。传统蛋白研究技术(如色谱分离纯化、二维电泳等)仪器昂贵、步骤繁琐,不适应大规模的筛查和临床检测。SELDI-TOF-MS,又称为蛋白指纹图谱技术,是近年兴起的一项差异蛋白组学技术,可以快速、高通量地检测微量粗制样本蛋白图谱,筛选出肿瘤特异性低丰度蛋白,有望提高肿瘤筛检的特异性和敏感性。目前,应用SELDI-TOFMS技术研究消化道肿瘤,已经成功地建立了食管晚期癌[2]、贲门癌[3]、大肠癌[4]和胰腺癌[5]的蛋白指纹图谱模型,其敏感性和特异性均较高。目前,受到标本量的限制,对于早期食管癌的研究少见报道。
鉴于肿瘤发生演变因素的复杂性,检测单一或数个指标对食管癌进行早期诊断不可避免地存在敏感性和特异性矛盾。因此,许多学者开始尝试使用多标志物联合检测技术来提高筛检的敏感性和特异性,虽取得一些进展,但尚不能全面反映出癌变过程多种因子的复杂变化[6]。本研究采用 SELDITOF-MS技术对高发区内镜筛查出的早期和进展期食管癌及正常对照血清蛋白表达谱进行对比分析,提供了一组蛋白质的功能及其模式的信息,通过计算机软件的统计学处理,筛选出多种候选肿瘤标志物建立诊断模型,优于以往采用单一标志物或多标志物简单叠加进行检测的筛查方法,能较全面的反映癌变过程多因子变化情况,对其联合检测可大幅提高其特异性和敏感性。本研究结果表明,早期、进展期食管癌分别与正常对照组比较建立的血清蛋白指纹图谱模型能良好区分来自高发区的食管癌和正常对照,具有较高的特异性(81.58%、89.47%)和敏感性(80.00%、83.33%),为高发区高危人群中食管癌的早期筛查和诊断提供了一种新方法。
早期食管癌的术后5年生存率可达90%以上,远高于进展期食管癌[7]。由于早期和进展期食管癌的治疗方案、治疗效果、术后生存质量迥异,区分不同病理阶段的食管癌显得尤为重要。本研究建立的区分早期食管癌和进展期食管癌的血清蛋白指纹图谱模型,能良好地区分早期和进展期食管癌,特异性(73.33%)和敏感性(91.67%)均较高,该模型如能在临床应用,可为食管癌治疗方案的选择提供依据。
目前,高发区早期食管癌的诊断主要依赖内镜结合碘染色技术,筛查成本较高、人群耐受性较差、操作耗时较长,难以在大人群中推广应用。因此,研究肿瘤标志物应用于人群初筛,浓聚高危人群,使内镜筛查更具针对性,已成为迫切需要解决的问题。但迄今为止,在食管癌早期诊断方面,尚无特异性和敏感性均高的理想标志物可以应用。近期研究发现,食管癌的发生发展过程中许多蛋白分子的表达水平发生了改变,如p16、FHIT基因等[8],但尚存在敏感性或特异性不高的缺点,不能满足现场筛查的要求。本组用于构建3个蛋白指纹图谱模型的质荷比峰4 291、4 392、4 823、4 975、5 644、5 665、5 932、8 473、8 776、8 910Da的表达量在组间比较差异有统计学意义(P<0.05,P<0.01),特别是质荷比峰4 291Da和5 644Da参与了2个模型的构建,可能是食管癌相关的候选肿瘤标志物。这些蛋白分子的协同变化可能是食管癌特异的肿瘤宿主微环境在血清蛋白组中的体现。对这些蛋白分子的鉴定和追踪其在食管癌发展中的动态变化,可能为揭示该病发生规律提供有益线索。
本研究应用的支持向量机算法是基于统计学理论、依据结构风险最小化原则的一种学习算法,克服了其他信息学算法(如决策树、人工神经网络等)对大样本的要求,尤其适用于小样本研究,在医疗诊断中应用已获得了很好的结果[9-10]。本研究应用SELDI-TOF-MS技术检测到的海量数据,通过支持向量机方法建立蛋白指纹图谱模型、筛选出潜在肿瘤标志物,研究结论较为准确、可靠,将进一步扩大样本验证和完善。
[1] ZHANG LW,WEN DG,LI YS,et a1.Epidemic strength of cardia and distant stomach cancer in the risk region of esophagea1 cancer and their imp1ication to endoscopic screening[J].Chinese Journa1 of Cancer Research,2006,18(2):111-115.
[2] 王士杰,张立玮,于卫芳,等.高发区筛查人群食管鳞癌血清蛋白指纹图谱诊断模型的建立及临床价值[J].中华肿瘤杂志,2007,29(6):441-443.
[3] 于卫芳,张立玮,王士杰,等.高发区自然人群贲门癌血清蛋白指纹图诊断模型的建立及临床价值[J].临床荟萃,2006,21(12):841-844
[4] WANG ZH,DING KF,YU JK,et a1.Proteomic ana1ysis of primary co1on cancer-associated fibrob1asts using the SELDI-Protein Chip p1atform[J].J Zhejiang Univ Sci B,2012,13(3):159-167.
[5] FELIX K,FAKELMAN F,HARTMANN D,et a1.Identification of serum proteins invo1ved in pancreatic cancer cachexia[J].Life Sci,2011,88(5/6):218-225.
[6] 岳文彬,郭涛,范宗民,等.食管癌及癌前病变患者血清中多个自身抗体检测的研究[J].河南医学研究,2010,10(2):144-151.
[7] 王国清,魏文强,乔友林.食管癌筛查和早诊早治的实践与经验[J].中国肿瘤临床,2010,19(1):4-8.
[8] ZHANG LW,YU WF,WEN DG,et a1.Study on the expression of p16 and FHIT protein in esophagea1 carcinomatous change from high incidence area of esophagea1 carcinoma[J].Chinese Journa1 of Cancer Research,2007,19(1):68-71.
[9] LIN Q,PENG Q,YAO F,et a1.A c1assification method based on principa1 components of SELDI spectra to diagnose of 1ung adenocarcinoma[J].PLoS One,2012,7(3):e34457.
[10] SCOTCH M,DUGGAL M,BRANDT C,et a1.Use of statistica1 ana1ysis in the biomedica1 informatics 1iterature[J].J Am Med Inform Assoc,2010,17(1):3-5.
(本文编辑:赵丽洁)
ESTABLISHMENT OF PROTEIN FINGERPRINT PATTERN FOR EARLY ESOPHAGEAL CANCER AND ITS SCREENING VALUE IN HIGH INCIDENCE AREA
YU Weifang1,NIU Weiwei1,LI Chao1,ZHANG Liwei2*,WANG Shijie2
(1.Department of Endoscopt,the First Hospital of Hebei Medical Universitt,Shijiazhuang 050031,China;
2.Department of Endoscopt,the Fourth Hospital of Hebei Medical Universitt,Shijiazhuang 050011,China)
ObjectiveTo bui1d protein fingerprint mode1s of ear1y esophagea1 cancer and advanced esophagea1 cancer in high incidence area,and investigate their screening va1ues.MethodsThe serum proteomic patterns of 38 cases of norma1 contro1s,15 cases of ear1y esophagea1 cancer and 36 cases of advanced esophagea1 cancer were detected by using surface enhanced 1aser desorption/ionization time of f1ight mass spectrometry(SELDI-TOF-MS).A11 subjects were from endoscopic screening popu1ation in high-risk areas of esophagea1 cancer.The data was ana1yzed and the protein fingerprint mode1s were estab1ished by using support vector machine.The mode1s were va1idated by 1eave one cross va1idation.ResultsThe protein fingerprint mode1 cou1d differentiate ear1y esophagea1 cancer from norma1 contro1s with a specificity of 81.58% and a sensitivity of 80.00%.The mode1 cou1d differentiate advanced esophagea1 carcinoma from norma1 contro1s with a specificity of 89.47%and a sensitivity of 83.33%.The mode1 cou1d differentiate ear1y esophagea1 cancer from advanced esophagea1 cancer with a specificity of 73.33%and a sensitivity of 91.67%.The difference of protein expression peaks(4 291,4 392,4 823,4 975,5 644,5 665,5 932,8 473,8 776,8 910Da)was statistica11y significant between groups.ConclusionEstab1ishment of three protein fingerprint mode1s provides a new approach for diagnosingand screening ear1y esophagea1 cancer in high incidence area.The protein peaks which had statistica1 significance in diagnostic mode1s may be the potentia1 biomarkers re1ated to ear1y esophagea1 cancer.
esophagea1 neop1asms;peptide mapping;mass screening中国是食管癌高发国家,食管癌发病呈地域性分布,高低发区的发病率可相差几十倍。河北省太行山脉南麓地区是我国的食管癌集中高发区[1]。目前,早发现、早诊断、早治疗是公认高发区食管癌防治的最有效措施。但是食管癌早期发病隐匿,大多无吞咽症状,很难做到“早期诊治”。因此,寻找简便有效的筛查方法和敏感特异的肿瘤标志物应用于人群初筛,浓聚高危人群,已成为目前亟待解决的课题。本研究应用蛋白组学技术对高发区内镜筛查出的早期和进展期食管癌及正常对照者的血清蛋白质谱进行对比分析,分别建立蛋白指纹图谱模型,旨在筛选用于高发区早期食管癌筛查的候选肿瘤标志物。
R735.1
A
1007-3205(2012)04-0373-06
2012-02-13;
2012-04-11
河北省卫生厅青年基金课题(06130)
于卫芳(1973-),男,回族,河北承德人,河北医科大学第一医院副主任医师,医学硕士,从事消化道疾病内镜诊治研究。
*通讯作者。E-mai1:ydyynjzx@126.com
10.3969/j.issn.1007-3205.2012.04.001
