基于线粒体16S r RNA与COI基因序列的刻肋海胆属系统发育研究*
2012-01-10曾晓起张文峰高天翔
曾晓起,张文峰,高天翔
(中国海洋大学水产学院,山东青岛266003)
刻肋海胆属(Temnopleurus)隶属于海胆纲(Echinoidea),拱齿目(Camarodonta),刻肋海胆科(Temnopleuridae)[1]。刻肋海胆属常见有5个种:哈氏刻肋海胆(Temnopleurus hardwickii)、细雕刻肋海胆(T.toreumaticus)、芮氏刻肋海胆(T.reevesii)、T.alexandri和T.michaelseni。哈氏刻肋海胆为中国、韩国和日本沿岸特有种,在我国黄、渤海沿岸很常见,并向南分布到舟山群岛和台湾海峡;细雕刻肋海胆分布于非洲东海岸、波斯湾、大洋洲东海岸以及东亚,为我国南北各海区的广分布种;芮氏刻肋海胆广泛分布于印度-西太平洋沿岸,为南海和东海的常见种[2];T.alexandri和T.michaelseni主要分布于大洋洲东海岸。目前国内外对刻肋海胆属生物学方面的研究主要集中在发育生物学和形态学[3-5],而对我国刻肋海胆属系统发育学方面的研究尚未见报道。
线粒体DNA(mt DNA)由于其具有遵循严格的母系遗传、几乎无重组、结构简单以及在不同的区域进化速度存在差异等优点,逐渐成为种群遗传学和分子系统发育研究的重要标记[6-8]。16S r RNA基因是非编码蛋白质基因,其大部分区域发生的突变为中性突变,加之其进化速度适中,因此常被应用于不同阶元物种的系统进化和分类研究[9]。刘晓慧等[10]对我国5种经济海胆(光棘球海胆、中间球海胆、马粪海胆,海刺猬和紫海胆)线粒体16S r RNA基因片段的序列进行了分析,并得到了较为可信的种间遗传距离和系统进化关系。细胞色素氧化酶I(Cytochrome oxidase subunit I,COI)基因是线粒体氧化呼吸链的重要成员,其序列已被应用于贝类、海参等无脊椎动物的系统发育学研究[11-12]。本研究对我国分布的3种刻肋海胆(哈氏刻肋海胆、细雕刻肋海胆、芮氏刻肋海胆)的线粒体16S r RNA和COI基因部分序列进行比较分析;并进一步探讨了刻肋海胆属内5种不同种海胆的遗传分化程度以及种间系统进化关系,以期为我国海胆的种质鉴定和系统进化研究提供理论依据。
1 材料与方法
1.1 实验材料
实验所用哈氏刻肋海胆(缩写为HS)样品于2010年10~11月分别采自辽宁大连黑石礁(1个)、山东青岛大公岛(2个)和浙江温州南麂列岛(4个);细雕刻肋海胆(缩写为XD)样品于2010年10~11月分别采自辽宁大连黑石礁(3个)、浙江温州南麂列岛(1个)和福建泉州崇武(2个);芮氏刻肋海胆(缩写为RS)于2010年10月采自福建漳州东山(2个)。采集后,用95%酒精固定,保存备用。T.alexandri、T.michaelseni和外群杂色角孔海胆(Salmacis sphaeroides)的序列信息来自GenBank(见表1)。
表1 本研究所用海胆基本信息Table 1 List of sea urchins in this study
1.2 实验方法
1.2.1 基因组DNA提取 取海胆样品的齿间肌,采用标准的酚-氯仿法[13]提取基因组DNA,将乙醇沉淀后的基因组DNA溶解于100μL TE溶液,4℃保存备用。
1.2.2 目的片段的PCR扩增和测序
用于扩增线粒体16S r RNA片段的引物[12]为:
16Sar:5′-CGCCTGTTTAACAAAAACAT-3′,
16Sbr:5′-CCGGTCTGAACTCAGATCATG-3′。
用于扩增线粒体COI片段的引物[14]为:
COIef:5′-ATAATGATAGGAGGGTTTGG-3′,
COIer:5′-GCTCGTGTGTCTACGTCCAT-3′。
16S r RNA和COI 2个基因片段的PCR过程,分别参照李颖[12]和Kerry L.Howell[14]的反应体系和反应条件进行。以上反应均设阴性对照以排除DNA污染的情况。取2μL PCR扩增产物进行1%琼脂糖凝胶电泳检测(U=5 V/cm)。
用UNIQ-10柱式DNA胶回收试剂盒(上海华舜)进行PCR产物的回收纯化。纯化后的产物送上海桑尼公司进行双向测序。
1.3 数据分析
应用DNAStar软件(DNASTAR,Inc)中的Seq-Man软件对哈氏刻肋海胆、细雕刻肋海胆和芮氏刻肋海胆的正反链序列进行组装,分别得到16S r RNA和COI基因片段的部分序列。用Clustal X对序列进行比对,用MEGA4[15]软件统计3种海胆的变异位点(variable sites)和简约信息位点(parsimony infoanative site)数,转换(Ts)颠换(Tv)位点数,再计算转换颠换比率(Ts/Tv),计算3种海胆的单倍型数,并将单倍型序列提交到Genbank。
结合从Gen Bank中获得的刻肋海胆属另外2种海胆(Temnopleurus alexandri,Temnopleurus michaels-eni)的16S r RNA和COI基因片段序列,基于Kimura-2-parameter模型计算这5种海胆的种间平均遗传距离。用PAUP4.0[16]和Modeltest 3.7[17]软件对联配的同源序列进行核苷酸替代模型筛选,得到2个目的片段碱基替代最适模型及其相关参数。使用MEGA4和PAUP4.0软件,应用距离法(NJ)、最大简约法(MP)、最大似然法(ML),分别构建5种海胆16S r RNA和COI 2个基因片段的系统发育树。
2 结果
2.1 基因片段序列多样性
16S r RNA基因片段:3种海胆共存在9个插入或缺失位点,89个变异位点,包含54个简约信息位点。发生碱基转换和颠换位点数平均为24和17个,转换/颠换平均值为1.4(见表2)。哈氏刻肋海胆6个个体共有3种单倍型,分别是:HSA(HS1、HS4、HS6、HS7)、HSB(HS2)、HSC(HS5),所对应的GenBank登录号是:JN128612~JN128614;细雕刻肋海胆6个个体共有4种单倍型,分别是:XDA(XD1)、XDB(XD2、XD4、XD6)、XDC(XD3)、XDD(XD5),所对应的Gen-Bank登录号是:JN128615~JN128618;芮氏刻肋海胆2个个体共有2种单倍型,分别是:RSA(RS1)、RSB(RS2),所对应的GenBank登录号是:JQ065692和JQ065693。
COI基因片段:3种海胆无插入或缺失位点,157个变异位点,包含155个简约信息位点。发生碱基转换和颠换位点数平均为38和32个,转换/颠换平均值为1.2(见表2)。哈氏刻肋海胆7个个体共有6种单倍型,分别是:HSCA(HS1)、HSCB(HS2)、HSCC(HS3、HS6)、HSCD(HS4)、HSCE(HS5)、HSCF(HS7),所对应的GenBank登录号是:JN128619~JN128624;细雕刻肋海胆5个个体共有5种单倍型,分别是:XDCA(XD1)、XDCB(XD2)、XDCC(XD 4)、XDCD(XD5)、XDCE(XD6),所对应的GenBank登录号是:JN128625~JN128629;芮氏刻肋海胆2个个体共有2种单倍型,分别是:RSCA(RS1)、RSCB(RS2),所对应的GenBank登录号是:JN128630和JN128631。

表2 3种刻肋海胆16S r RNA和COI基因片段序列多样性Table 2 Sequence variability for 16S rRNA and COI of 3 species of genus Temnopleurus
2.2 遗传分化
结合在GenBank中获得刻肋海胆属另外2种海胆的16S r RNA和COI片段序列,用Mega4.0软件,基于Kimura-2-parameter模型计算得到5种海胆种间平均遗传距离(见表3)。从结果来看在16S r RNA水平上各种间的遗传距离在0.078~0.185之间,在COI水平上各种间的遗传距离在0.158~0.206之间。在16S r RNA和COI 2个基因片段上,均表现为芮氏刻肋海胆和T.michaelseni遗传距离最小,分别为0.078和0.158。16S r RNA基因片段上,T.alexandri和芮氏刻肋海胆遗传距离最大,为0.185;COI基因片段上,哈氏刻肋海胆和芮氏刻肋海胆遗传距离最远,为0.206(见表3)。

表3 5种海胆种间平均K2-P遗传距离(左下角:16S r RNA,右上角:COI)Table 3 Average K2-P genetic distance based on 16S rRNA region(below diagonal)and COI region(above diagonal)of 5 different species genus Temnopleurus

图1 基于16S r RNA基因片段构建NJ、MP和ML系统进化树(后验概率高于50%标注在分支上)Fig.1 Phylogenetic tree of 5 species constructed with neighbor-joining,maximum parsimony and maximum likelihood method based on 16S r RNA(numbers on the tree representposterior probability values)
2.3 系统发育分析
基于5种海胆16S r RNA和COI基因片段序列的数据,用MEGA4和PAUP4.0软件,以杂色角孔海胆为外类群,应用距离法(NJ)、最大简约法(MP)、最大似然法(ML)分别重建了2个片段各种单倍型的系统发育树,得到的系统发育树拓扑结构基本一致(见图1,2)。在2个基因片段上哈氏刻肋海胆和细雕刻肋海胆均先聚为一支,再与T.alexandri聚类;芮氏刻肋海胆和T.michaelseni聚为一支。
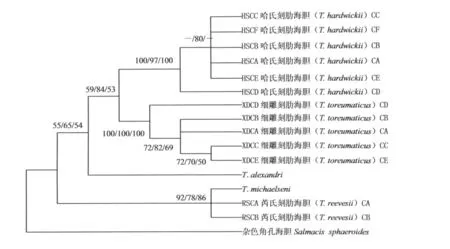
图2 基于COI基因片段构建NL、MP和ML系统进化树(后验概率高于50%标注在分支上)Fig.2 Phylogenetic tree of 5 species constructed with neighbor-joining,maximum parsimony and maximum likelihood method based on COI(numbers on the tree representposterior probability values)
3 讨论
张文峰等[18]对哈氏刻肋海胆、细雕刻肋海胆和芮氏刻肋海胆,3种海胆的形态学进行了定量研究。其中聚类分析研究结果为哈氏刻肋海胆和细雕刻肋海胆先聚为一支,再与芮氏刻肋海胆聚类。这一形态学结果和本研究中利用16Sr RNA和COI基因片段进行的聚类分析结果是一致的。
由表2可以看出,16Sr RNA和COI基因片段的变异位点数存在差异。mt DNA中不同区域的核苷酸突变速率不同,或不同的基因片段受到的选择压力不同,是造成线粒体不同基因片段变异存在差异的主要原因[19]。
McCartney等[20]以上新世中期(约350万年前)巴拿马地峡(Isthmus of Panama)首次开放作为分子钟标准,估算出长海胆属内COI基因核苷酸分歧速率为3.49%/百万年。Jeffery[21]认为长海胆科和刻肋海胆科亲缘关系很近,可以将长海胆属的COI基因核苷酸分歧速率应用于刻肋海胆属。因此,本研究以3.49%/百万年的核苷酸分歧速率应用于5种海胆的COI基因片段。推断,哈氏刻肋海胆先与芮氏刻肋海胆在约590万年前分化,然后再与T.michaelseni在约570万年前分化,最后与细雕刻肋海胆和T.alexandri在约500万年前分化。分化事件主要发生在中新世晚期(Late Miocene)至上新世早期(Early Pliocene)。这与Hewitt认为物种的形成过程主要发生在上新世和更新世[22]是一致的。
[1] Liao Y L,Clark A M.The Echinoderms of Southern China[M].California:Science Press,1995:347-351.
[2] 张凤瀛,廖玉麟.中国动物图谱-棘皮动物[M].北京:科学出版社,1964:83-84.
[3] Kitazawa C,Nishimura H,Yamaguchi T,et al.Novel morphological traits in the early developmental stages of Temnopleurus toreumaticus[J].Biological bulletin,2009,217:215-221.
[4] Okada K,Miyauchi H.Figures of the standard early development of Temnopleurus toreumaticus[J].Journal Gakugei Bull,Tokushima University,1958,8:59-71.
[5] Osanai K.Behavior of pigment granules during early development in eggs of a sea urchin Temnopleurus hardwickii[J].Annual Report Faculty of Education Iwate University,1969,29:35-38.
[6] Brown W M.Evolution of Animal Mitochondrial DNA[M].Sunderland:Evolution of Genes and Proteins,1983:62-88.
[7] Avise J C.Phylogeography[M].London:Harvard University Press,2000:1-36.
[8] Moritz C,Dowling T E,Brown W M.Evolution of animal mitochondrial DNA:relevance for population biology and systematics[J].Annual Review of Ecology and Systematics,18:269-292.
[9] 蒙子宁,庄志猛,丁少雄,等.中国近海8种石首鱼类的线粒体16S r RNA基因序列变异及其分子系统进化[J].自然科学进展,2004,14(5):514-521.
[10] 刘晓慧,黄佳琪,周遵春,等.5种经济海胆线粒体16S r RNA基因片段的序列分析[J].水产科学,2007,26(6):331-334.
[11] 毛阳丽,蔡厚才,李成九,等.基于线粒体COI与16S r RNA基因序列探讨贻贝属的系统发育[J].南方水产,2010,6(5):27-36.
[12] 李颖,刘萍,孙慧玲,等.仿刺参(Apostichopus japonicus)mt DNA三个基因片段的序列分析[J].海洋与湖沼,2006,37(2):143-153.
[13] Sambrook J,Fritsch E F,Maniatis T,et al.Molecular Cloning:A Laboratory Manual[M].New York:Cold Spring Harbor Laboratory Press,1996:464-468.
[14] Kerry L.Howell,Alex D.Rogers,Paul A.Tyler,et al.Reproductive isolation among morphotypes of the Atlantic seastar species Zoroaster fulgens(Asteroidea:Echinodermata)[J].Marine Biology,2004,144:977-984.
[15] Kumar S,Tamura K,Nei M,et al.MEGA 3:integrated software for molecular evolutionary genetics analysis and sequence alignment[J].Briefings in Bioinformatics,2004,5(2):150-163.
[16] Swofford D L.PAUP*Phylogenetic Analysis Using Parsimony(*and other methods),Version 4[M].Massachusetts:Sinauer Associates,2002.
[17] Posada D,Crandall K A.Modeltest:Testing the model of DNA substitution[J].Bioinformatics,1998,14:817-818.
[18] 张文峰,曾晓起,王长云.对三种刻肋海胆分类的数量分析研究[J].中国海洋大学学报:自然科学版,2011,41:169-174.
[19] ZhengX D,WangR C,WangX F,et al.Genetic variation in population of the common chinese cuttle fish Sepiella Maindroni(Mollusca:Cephalopoda)using allozymes and mitochondrial DNA sequence analysis[J].Journal of Shellfish Research,2001,20(3):1159-1165.
[20] McCartney M A,Keller G,Lessios H A.Dispersal barriers in tropical oceans and speciation in Atlantic and eastern pacific sea urchins of the genus Echinometra[J].Molecular Ecology,2000,9:1391-1400.
[21] Charlotte H J,Richard B E.Phylogeny and evolution of developmental mode in Temnopleuridae(Echinoids)[J].Molecular Phylogenetics and Evolution,2003,28:99-118.
[22] Hewitt G M.Some genetic consequences of ice ages and their role in divergence and speciation[J].Biological Journal of the Linnean Society,1996,58(3):247-276.
