HCV非结构蛋白NS5A基因的斑马鱼肝脏表达模型
2011-04-11赵晔胡占英佟军威赵立勋蒋建东张靖溥
赵晔,胡占英,佟军威,赵立勋,蒋建东,张靖溥
·论著·
HCV非结构蛋白NS5A基因的斑马鱼肝脏表达模型
赵晔,胡占英,佟军威,赵立勋,蒋建东,张靖溥
目的建立 HCV 非结构蛋白 NS5A 基因在斑马鱼肝脏特异表达的模型,研究 HCV NS5A 的体内作用机制并初步探讨 HCV NS5A 对肝病理标志基因表达的影响。
肝炎病毒属; 斑马鱼; 疾病模型,动物;基因表达
www.cmbp.net.cn 中国医药生物技术, 2011, 6(6):437-444
丙型肝炎病毒(hepatitis C virus,HCV)感染引发的肝炎、肝硬化和肝细胞癌(hepatocellular carcinoma,HCC)严重威胁着人类健康[1-2]。据WHO 统计,目前全世界约有 1.7 亿人感染 HCV,每年约有 300 万 ~ 400 万新增感染者[3]。我国属于 HCV 感染的高发病区,感染率约为 1.5% ~3.5%,约有 4000 万感染者[4]。迄今为止,HCV 感染的防治仍缺乏有效的方法。目前临床用于治疗HCV 感染主要的方案是采用聚乙二醇干扰素联合利巴韦林,但这种治疗手段只针对不足半数的患者起作用,治疗效果个体差异性较大,且具有反复性[5-6]。由于缺少稳定可靠的 HCV 感染细胞模型和动物模型,人们对 HCV 致病机制、抗病毒药物筛选和疫苗等方面的研究进程受到严重阻碍。现阶段只有黑猩猩是公认的易感性强的 HCV 天然宿主模型,该模型能够用于 HCV 整个生命周期的研究[7],但是复杂的伦理问题及成本问题严重制约了该模型的应用。人肝嵌合小鼠模型也被证实可以感染 HCV[8],然而,由于该模型操作复杂,重现性较差,不能大规模运用于科研和药效筛选。因此,开发新型 HCV 小动物模型,成为 HCV 研究中亟待解决的一个问题。
丙型肝炎病毒自 1989 年被 Choo 等[9]首次成功克隆至今,其分子生物学特性研究取得了很大的发展。HCV 病毒的基因组为一条线性单股正链RNA,全长约为 9600 个核苷酸,包含 5′ 端非翻译区(5′-non translation region,5′-NTR)、一个约为9.4 kb 的开放式阅读框(ORF)和 3′ 非翻译区(3′-non translation region,3′-NTR)。其中 ORF 编码一条约含 3000 个氨基酸残基的多肽链,经过宿主信号肽酶和病毒编码的蛋白酶切割加工后,产生至少 10 种蛋白,包括结构蛋白(核心蛋白 Core、包膜糖蛋白 E1 和 E2、离子通道蛋白 P7)和非结构蛋白(NS2-3、NS4A、NS4B、NS5A 和NS5B)[10]。其中,核心蛋白 Core 被证实与肝脏的病理过程直接相关,但近年研究表明,NS5A 与病毒的致病过程和细胞内信号传导也存在密切关系,包括干扰素抵抗和肝癌形成[11-12]、抑制 ERK MAPK 途径和抑制 EGF 激活的 p38MAPK 途径[13],以及激活 JNK 信号途径[14]。NS5A 蛋白还可以通过多种机制,抑制细胞凋亡,与细胞凋亡调控相关的因子 TNF-α,Bcl2 蛋白家族以及 P53 等均被证实能够与 NS5A 相互作用[14-16]。NS5A 是一种 447 个氨基酸的磷蛋白,具有基本的磷酸化(56 kD)和高度磷酸化(58 kD)两种存在形式,磷酸化位置通常为丝氨酸残基,少数苏氨酸残基也可被磷酸化[17]。Lemm 等[18]通过 HCV 复制的细胞模型筛选出潜在作用于 NS5A、抑制 HCV 复制的小分子化合物 BMS-824,为 NS5A 作为药物靶点的研究提供了有力证据。
近年来,斑马鱼作为研究脊椎动物发育的模型已被普遍接受,在医药领域利用斑马鱼研制疾病模型也正愈来愈受到重视[19]。斑马鱼作为新型脊椎类模式生物,具有很多优于其他传统模式生物的特点,如个体小、通体透明、繁殖量大、生长发育较快、养殖成本低、操作简便等,并具备高通量药物筛选的优势。其生长发育过程和组织系统结构与人类有较高的相似性[19-20]。在 HCV 研究方面,利用转基因的技术手段,发现肝脏毒性化合物硫代乙酰胺(TAA)能够诱发 Core 转基因斑马鱼产生肝硬化及肝细胞癌的症状[21-22]。该模型为本实验运用斑马鱼建立 HCV 非结构蛋白病理模型提供了依据。
本研究以斑马鱼作为模式生物,构建了肝脏特异表达 HCV NS5A 的基因表达载体,并首次建立了肝脏表达 HCV NS5A 的斑马鱼模型,对 NS5A潜在作用的基因进行了探讨。该模型可以用于HCV NS5A 的体内病理机制的研究。
1 材料与方法
1.1 材料
1.1.1实验动物 成年斑马鱼 AB 系(Danio rerio,AB line),清华大学生命科学院孟安明教授惠赠,在(28 ± 1)℃,14 h 光照/10 h 黑暗条件下正常喂养。
1.1.2质粒和细胞系 质粒 pDsRed-N1 和pIRES2-DsRed 由杭州师范大学张亮惠赠;HCV J4L6S(1b) 质粒为本实验室保存;pMD18-T 和pMD19-T 购自日本 Taraka 公司;Huh7 细胞由本所病毒室惠赠。
1.1.3试剂及工具酶 DNA 片段凝胶回收试剂盒、Taq DNA 聚合酶、限制性内切酶 BamH I、Sal I、Not I、Xho I、Nde I、EcoR I 等、二硫苏糖醇 (DTT)、dNTP、T4 DNA 连接酶、碱性磷酸酶(CIAP)、异丙基-β-D-硫代吡喃半乳糖苷(IPTG)、均购自日本 Takara 公司;Long AMP Taq 聚合酶、限制性内切酶 Rsr II,Ase I,BsrG I 购自美国 NEB公司;AMV 反转录酶、Pfu DNA 聚合酶购自美国Promega 公司;DNA 片段凝胶回收试剂盒购自美国 Qiagen 公司;抗 HCV NS5A 兔源单克隆抗体购自美国 Abcam 公司;辣根过氧化物酶标记的羊抗兔抗体 HRP-IgG 购自北京中杉金桥公司;DIG RNA Labelling Kit 购自瑞典罗氏公司;Supersignal® West Pico 化学发光底物购自美国 Thermo 公司。
1.1.4仪器 MG96G 型 PCR 扩增仪购自杭州朗基公司;BINTA 2020D 凝胶成像系统购自我国BINTA 公司;SZX16 型体式显微镜与 IX51 型荧光显微镜购自日本奥林巴斯公司;Basic164-5050型伯乐电泳仪购自美国伯乐公司;500D 型数码相机购自日本佳能公司;AlphaEase® FC 成像系统购于美国 Alpha Innotech 公司。
1.2 方法
1.2.1肝脏特异表达 HCV NS5A 载体的构建 以本室保存的 HCV J4L6S(1b)质粒为模板,在引物中分别引入 Xho I 与 Sal I 的酶切位点,并在其首尾分别加入起始密码子和终止密码子,PCR 方法扩增 HCV NS5A 的片段。上游引物:5′ AACTGCAGA TGTCCGGCTCGTGGCTAAGGG 3′;下游引物:5′CCAATGCATTGGTTCTGCAGCTAGCAGCAGA CGACATCCT 3′。PCR 反应条件:94 ℃ 预变性5 min;94 ℃ 变性 1 min,55 ℃ 退火 1 min,72 ℃延伸 2 min,循环 30 次;最后 72 ℃ 延伸 10 min。所得片段大小约为 1400 bp。利用内切酶 Xho I、Sal I 将片段切下,连入相同位点酶切后的载体pIRES2-DsRed 中。将斑马鱼肝脂肪酸结合蛋白基因(L-FABP)增强子序列(由上海生工公司合成)用Ase I 切出,并与上述构建质粒在 Ase I 酶切位点拼接,形成肝脏特异表达的 pFC-5AiR 质粒。经上海生工科技有限公司测序鉴定上述基因改造位点和表达载体拼接正确。
1.2.2细胞转染 采用 LipofectamineTM 2000 Reagent 转染试剂盒进行 Huh7 细胞转染实验。基本实验程序:在 24 孔板进行转染,50 μl/孔无血清DMEM 高糖培养基稀释 1 μg DNA(根据需要合并稀释);25 μl/孔无血清 DMEM 高糖培养基稀释2 μl LipofectamineTM 2000 Reagent(根据需要合并稀释),室温孵育 5 min 后与稀释的 DNA 合并混匀,室温孵育 20 min。按照每孔 100 μl DNA-脂质体复合物溶液加入 80% ~ 90% 汇合度且生长状态良好的 Huh7 细胞中,置于二氧化碳培养箱内37 ℃ 培养约 6 h,待细胞完全贴壁后,吸出转染培养基;每孔加入 500 μl DMEM 高糖培养基,继续培养至 24 h,在显微镜下观察转染结果。实验以转染 pIRES2-DsRed 的结果作为阳性对照,未转染质粒组作为阴性对照。
1.2.3斑马鱼的显微注射和荧光显微镜观察 将pFC-5AiR 载体线性化后,注射到 1 ~ 8 细胞期的斑马鱼胚胎中,待发育至 8 dpf(days postfertilization),用荧光显微镜在激发光 556 nm、发射光 586 nm 下观察斑马鱼身体上红色荧光蛋白的表达及分布情况。以注射 pIRES2-DsRed 线性片段的斑马鱼为阳性对照。
1.2.4RT-PCR 检测 取注射 pFC-5AiR 后10 dpf 荧光检测阳性幼鱼 50 条,用 Trizol方法,提取的幼鱼总 RNA,通过逆转录试剂盒合成cDNA。以 cDNA 为模板进行 PCR,检测靶基因NS5A表达情况。同法,反转录 6 dpf 和 9 dpf 荧光阳性幼鱼 cDNA,扩增相应的肝病理标志基因。均以同批野生型幼体为正常对照组;以β-actin基因的扩增为内对照。引物序列见表 1 和表 2。

表1 RT-PCR 所用引物序列Table 1 Primers for RT-PCR

表2 病理标志基因引物Table 2 Primers of liver-disease associated marker genes
1.2.5Western blot 检测 HCV NS5A 蛋白的表达 用 RIPA 组织裂解液处理 10 dpf 荧光阳性幼鱼,每组 100 条,分别提取总蛋白,经 12% SDS-PAGE 电泳分离后转移到硝酸纤维素膜(NC膜),用5% 脱脂奶粉的 TBS(20 mmol/L Tris,pH 7.4,150 mmol/L NaCl)室温封闭 1 h 或 4 ℃ 封闭过夜。将该硝酸纤维素膜浸入用 1% 脱脂奶粉的TBS 液稀释 1000 倍的 NS5A 抗体中,于 4 ℃ 温育过夜,充分漂洗后,加入 HRP 交联的羊抗兔二抗,室温处理 1 h,充分漂洗,加入 Supersignal® West Pico 化学发光底物,用 AlphaEase® FC Imaging System成像系统拍照。
1.2.6整体原位杂交 主要参照文献[23]进行。以克隆的 HCVNS5A(nt 6524-6712)基因序列作为模板,利用 DIG RNA Labeling Kit 分别合成NS5A的正、负链 RNA 探针。取 4% 多聚甲醛固定的 10 dpf 幼鱼,用 1 × PBST 溶液洗去多余的多聚甲醛溶液,分别用蛋白酶 K 和 DNA 酶 I 处理,将幼鱼置于 65 ℃ 水浴进行预杂交 4 h,然后加入合成的 RNA 探针,65 ℃ 水浴杂交过夜。用0.2 × SSC 溶液洗去未结合的探针,加入anti-Dig-AP 与 RNA 探针 4 ℃ 结合过夜。用 1 × PBST 溶液洗去未结合的抗体,再加入 BCIP/NBT溶液显色 30 min,迅速用 1 × PBST 溶液洗去多余的显色液,在显微镜下观察并记录结果。
1.2.7斑马鱼肝脏脂肪代谢及纤维化相关基因及HCV 感染相关的宿主信号基因检测 对于肝脏脂肪代谢及纤维化相关的基因,本文选择了脂肪细胞特异性标志物脂联素(Adiponectin)、转化生长因子-β(transforming growth factor-β,TGF-β)、戊二酰辅酶 A 合成酶(HMGS)、3-羟基-3-甲基酰辅酶 A还原酶(HMGR)、低密度脂蛋白受体(LDLR)进行了检测。通过 RT-PCR 检测这些基因的转录水平,与未注射 pFC-5AiR 的野生型斑马鱼幼鱼的基因水平进行对比,以β-actin为内参。
对于与丙型肝炎病毒感染相关的宿主基因,本文选取了精胺基琥珀酸盐合成酶(Argininosuccinate synthetase,Argsyn)、溶质载体家族成员 2(Solute carrier family 2,ScarF2)、致癌基因(Ras-related GTP binding D,Rasgbd)、趋化因子-1(Chemokine-1)、电子传递黄素蛋白多肽α(electron transfer flavoprotein alpha polypeptide,ETFA)进行了检测,以β-actin为内参。
2 结果
2.1 基因构件 pFC-5AiR 的靶基因表达检验
为了构建可在斑马鱼肝脏表达的基因载体,我们将斑马鱼肝脂肪酸结合蛋白(L-FABP)基因的增强子序列插入到真核表达载体 pIRES2-DsRed 的CMV启动子上游,以增强肝组织表达的选择性,构建了一种红色荧光蛋白报告基因DsRed和HCVNS5A基因双顺反子的 pFC-5AiR 表达载体,结构如图1A 所示。为了检验该基因构件是否可正确进行靶基因的表达,首先在 Huh7 细胞水平进行了DsRed报告基因的表达观察。如图1B 所示,在荧光显微镜下可清晰地观察到 pFC-5AiR 转染细胞与阳性对照组均有红色荧光蛋白的表达;而未转染的对照组细胞则未见红色荧光。结果说明,所构建的 pFC-5AiR 基因表达载体具有表达功能。
2.2 HCV NS5A 在斑马鱼幼体肝脏的表达
2.2.1报告基因红色荧光蛋白DsRed基因在斑马鱼肝脏的表达 以 pIRES2-DsRed 为阳性对照组,将注射 pFC-5AiR 的斑马鱼幼体,在荧光显微镜下观察,如图2所示,斑马鱼肝区出现明显的红色荧光。而注射 pIRES2-DsRed 的对照组中,红色荧光集中在腹腔,在肝区未见红色荧光。这表明L-FABP基因的增强子序列能够调控其下游基因在斑马鱼肝脏的特异表达。
2.2.2HCVNS5A基因在斑马鱼体内的转录和翻译 RT-PCR 结果显示,注射 pFC-5AiR 质粒的斑马鱼幼体检测到较高的NS5A转录水平(图3Aa)。同样,用抗 HCV NS5A 抗体进行 western blot 检测,检测到大小约 56 kD 的蛋白,与 NS5A的大小一致(图3Ab)。而野生型对照组中未出现对应的 HCV NS5A 的转录产物和蛋白条带。结果表明 pFC-5AiR 表达构件能够在斑马鱼幼体中表达 HCV NS5A 蛋白。

图1 pFC-5AiR 基因表达载体在 Huh7 细胞的表达[A:基因构件 pFC-5AiR 结构示意图;B:荧光显微镜观察基因构件pFC-5AiR 荧光蛋白的表达(400 ×)]Figure 1 Expression of pFC-5AiR in Huh-7 cells [A: Schematic diagram of pFC-5AiR; B: Fluorescence observation of pFC-5AiR expression in Huh-7 cells (400 ×)]

图2 荧光显微镜观察基因构件 pFC-5AiR 在斑马鱼肝区特异表达(8 dpf)Figure 2 Observation of pFC-5AiR injected zebrafish by fluorescence microscopy (8 dpf)
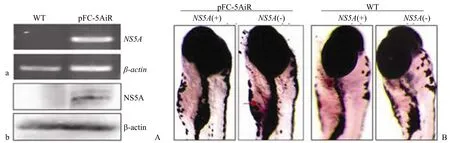
图3 HCVNS5A基因在斑马鱼体内的表达与定位Figure 3 Expression and localization of HCVNS5Agene in zebrafish larvae
2.2.3HCVNS5A基因在斑马鱼肝脏中表达的定位情况 为了进一步验证 pFC-5AiR 中的 HCVNS5A基因在斑马鱼体内的表达定位情况,采用整体原位杂交技术,选取在荧光显微镜下表达红色荧光的 8 dpf 斑马鱼幼鱼,分别用正义和反义的NS5A探针进行原位杂交实验。结果显示,用NS5A反义链探针杂交后在斑马鱼肝脏区域可以观察到阳性信号,而用相应的正义探针杂交的对照组无阳性结果(图3B)。说明 HCVNS5A基因可在斑马鱼的肝区表达。野生组(WT)也分别用正、反义探针杂交,结果均为阴性。
2.3 HCVNS5A基因表达对斑马鱼肝脏病理基因表达的影响
为了探讨 HCV NS5A 对肝脏的病理影响,本研究分析了斑马鱼胚胎注射 pFC-5AiR 后肝脏脂肪代谢和纤维化的标志基因表达的变化。通过RT-PCR 检测这些基因的转录水平,结果显示,Adiponectin、TGF-β、HMGS、HMGR和LDLR在斑马鱼发育 6 d 和 9 d 的表达量较野生型均有所升高,提示 HCVNS5A的表达影响了斑马鱼幼鱼肝脏脂代谢水平,可能引发肝脏内脂肪代谢功能异常及肝脏纤维化(图4)。

图4 注射 pFC-5AiR 基因构件对斑马鱼肝脏病理标志基因转录的影响Figure 4 Changes of liver pathological gene expression in 6 dpf and 9 dpf zebrafish injected with pFC-5AiR
另外,本实验还检测了以往文献报道的与丙型肝炎病毒感染相关的宿主信号基因,结果显示,Argsyn的表达较野生型在 6 dpf 和 9 dpf 时均显著升高,ScarF2和Rasgbd在 6 dpf 较野生型更高,而 9 dpf 时这两个基因的表达较野生型对照组无明显升高趋势;Chemokine1和ETFA的表达量均无明显的变化(图4)。
综上所述,在 HCV NS5A 模型中,脂代谢及纤维化的相关因子Adiponectin、TGF-β、HMGR、HMGS,LDLR以及 HCV 感染相关因子Argsyn、ScarF2、Rasgbd的表达水平均有不同程度的上调趋势。
3 讨论
3.1 HCV 非结构基因NS5A斑马鱼肝脏表达模型的鉴定
基因构件 pFC-5AiR 质粒利用斑马鱼脂肪酸结合蛋白增强子(eFABP)与载体CMV启动子配合,共同调控下游基因的转录。这一思路是受到Her 等[24]运用eFABP与广泛性SV40启动子配合的基因构件的启示。本研究通过荧光显微镜观察到红色荧光蛋白的表达均集中在斑马鱼肝脏区域,并通过 RT-PCR 和 western blot 方法证实了 HCV非结构基因NS5A在体内的表达。原位杂交结果显示了NS5A基因的肝脏特异性表达。这是目前已知的第一个 HCV NS5A 的斑马鱼模型。
3.2 HCV NS5A 表达导致斑马鱼模型的病理变化
研究表明 HCV 的感染导致肝脏脂肪变性,可能源于 HCV 利用宿主细胞脂肪的合成系统,上调胆固醇和脂肪酸合成相关的基因表达[20-21, 25]。羟甲基戊二酰辅酶 A 还原酶(HMGR)和羟甲基戊二酰辅酶 A 合成酶(HMGS)控制胆固醇的合成,两者在 HCV 感染的患者肝脏中均被证实上调[21]。在本研究中对斑马鱼胚胎注射 HCVNS5A基因表达构件 pFC-5AiR 后,HMGR与HMGS的转录水平均被上调,这与 HCV 感染导致激活宿主细胞的胆固醇合成途径相吻合。文献报道称,在 HCV 病毒感染宿主后,宿主细胞内 TGF-β 信号途径均有所改变,上调的TGF-β...与肝脏纤维化相关[25-26];低密度脂蛋白受体 LDLR 作为 HCV感染的细胞膜受体[27],其突变与家族性高胆固醇血症密切相关[28],本研究显示这两个基因在 NS5A 表达后均有所上调,推测 NS5A 可能参与了肝脂代谢和纤维化的途径。
3.3 斑马鱼肝脏模型的优势
除了具有胚胎学和遗传学的优势以外,斑马鱼的肝脏不同于哺乳动物之处在于胚胎期的肝脏不是血管生成的器官,因此肝脏的大小或结构的改变与造血功能障碍导致的变化无关。斑马鱼的肝脏在受精发育 5 d 后结构和功能趋向成熟,并可以在显微镜下直接观察。Passeri 等[29]利用斑马鱼幼鱼建立了酒精脂肪肝模型,通过幼鱼肝脏内与脂代谢相关的细胞色素 p450 2e1(cyp2e1)、超氧化物歧化酶(SOD)和免疫球蛋白结合蛋白 BIP 基因表达量的上调,证明酒精与肝脏纤维化的关系。Matthews 等[30]运用 S-腺苷水解酶(Ahcy)突变的手段建立了肝脏再生和脂肪变性的斑马鱼 ducttrip突变体,并推断出肿瘤坏死因子的表达与 S-腺苷的相关性。运用斑马鱼突变体基因筛选的方法,Sadler研究小组[19]筛选得到 3 种与肝病相关的新基因。这些研究为利用斑马鱼研究肝脏发病的分子机理、以及对肝脏发育机制的研究提供了有力证明。本研究建立的 HCV NS5A 斑马鱼肝脏模型,也为运用斑马鱼模式生物研究 HCV NS5A 致肝脏病变提供了新的思路。
志谢感谢王为先老师提供显微注射技术支持;孟杰老师在斑马鱼材料和管理方面的大力支持。
[1] Alter MJ, Margolis HS, Krawczynski K, et al. The natural history of community-acquired hepatitis C in the United States. The Sentinel Counties Chronic non-A, non-B Hepatitis Study Team. N Engl J Med, 1992, 327(27):1899-1905.
[2] Martins T, Narciso-Schiavon JL, Schiavon LL. Epidemiology of hepatitis C virus infection. Rev Assoc Med Bras, 2007, 57(1):107-112.
[3] Alter MJ. Epidemiology of hepatitis C virus infection. World J Gastroenterol, 2007, 13(17):2436-2441.
[4] Gao X, Cui Q, Shi X, et al. Prevalence and trend of hepatitis C virus infection among blood donors in Chinese mainland: a systematic review and meta-analysis. BMC Infect Dis, 2011, 11(1):88.
[5] Hadziyannis SJ, Sette H Jr, Morgan TR, et al. Peginterferon-alpha2a and ribavirin combination therapy in chronic hepatitis C: a randomized study of treatment duration and ribavirin dose. Ann Intern Med, 2004, 140(5):346-355.
[6] Tsubota A, Fujise K, Namiki Y, et al. Peginterferon and ribavirin treatment for hepatitis C virus infection. World J Gastroenterol, 2011, 17(4):419-432.
[7] Yanagi M, Purcell RH, Emerson SU, et al. Transcripts from a single full-length cDNA clone of hepatitis C virus are infectious when directly transfected into the liver of a chimpanzee. Proc Natl Acad Sci U S A, 1997, 94(16):8738-8743.
[8] Mercer DF, Schiller DE, Elliott JF, et al. Hepatitis C virus replication in mice with chimeric human livers. Nat Med, 2001, 7(8):927-933.
[9] Choo QL, Kuo G, Weiner AJ, et al. Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome. Science, 1989, 244(4902):359-362.
[10] Pozzetto B, Bourlet T, Grattard F, et al. Structure, genomic organization, replication and variability of hepatitis C virus. Nephrol Dial Transplant, 1996, 11 supp 4:2-5.
[11] Hidajat R, Nagano-Fujii M, Deng L, et al. Cleavage of the hepatitis C virus NS5A protein by caspase-3 in the interferon sensitivitydetermining region in a sequence-dependent manner. Kobe J Med Sci, 2004, 50(5-6):153-166.
[12] Tan SL, Katze MG. How hepatitis C virus counteracts the interferon response: the jury is still out on NS5A. Virology, 2001, 284(1):1-12.
[13] Georgopoulou U, Caravokiri K, Mavromara P. Suppression of the ERK1/2 signaling pathway from HCV NS5A protein expressed by herpes simplex recombinant viruses. Arch Virol, 2003, 148(2):237-251.
[14] Park KJ, Choi SH, Choi DH, et al. 1Hepatitis C virus NS5A protein modulates c-Jun N-terminal kinase through interaction with tumor necrosis factor receptor-associated factor 2. J Biol Chem, 2003, 278(33):30711-30718.
[15] He Y, Tan SL, Tareen SU, et al. Regulation of mRNA translation and cellular signaling by hepatitis C virus nonstructural protein NS5A. J Virol, 2001, 75(11):5090-5098.
[16] Ghosh AK, Steele R, Meyer K, et al. Hepatitis C virus NS5A protein modulates cell cycle regulatory genes and promotes cell growth. J Gen Virol, 1999, 80(5):1179-1183.
[17] Tellinghuisen TL, Foss KL, Treadaway J. Regulation of hepatitis C virion production via phosphorylation of the NS5A protein. PLoS Pathog, 2008, 4(3):e1000032.
[18] Lemm JA, O'Boyle D, Liu M, et al. Identification of hepatitis C virus NS5A inhibitors. J Virol, 2010, 84(1):482-491.
[19] Sadler KC, Amsterdam A, Soroka C, et al. A genetic screen in zebrafish identifies the mutants vps18, nf2 and foie gras as models of liver disease. Development, 2005, 132(15):3561-3572.
[20] Lam SH, Wu YL, Vega VB, et al. Conservation of gene expression signatures between zebrafish and human liver tumors and tumor progression. Nat Biotechnol, 2005, 24(1):73-75.
[21] Rekha RD, Amali AA, Her GM, et al. Thioacetamide accelerates steatohepatitis, cirrhosis and HCC by expressing HCV core protein in transgenic zebrafish Danio rerio. Toxicology, 2008, 243(1-2):11-22.
[22] Amatruda JF, Shepard JL, Stern HM, et al. Zebrafish as a cancer model system. Cancer Cell, 2002, 1(3):229-231.
[23] Thisse C, Thisse B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nat Protoc, 2008, 3(1):59-69.
[24] Her GM, Yeh YH, Wu JL. 435-bp liver regulatory sequence in the liver fatty acid binding protein (L-FABP) gene is sufficient to modulate liver regional expression in transgenic zebrafish. Dev Dyn, 2003, 227(3):347-356.
[25] Jármay K, Karácsony G, Nagy A, et al. Changes in lipid metabolism in chronic hepatitis C. World J Gastroenterol, 2005, 11(41):6422-6428.
[26] Fujino T, Nakamuta M, Yada R, et al. Expression profile of lipid metabolism-associated genes in hepatitis C virus-infected human liver. Hepatol Res, 2010, 40(9):923-929.
[27] Monazahian M, Böhme I, Bonk S, et al. Low density lipoprotein receptor as a candidate receptor for hepatitis C virus. J Med Virol, 1999, 57(3):223-229.
[28] Soutar AK, Naoumova RP. Mechanisms of disease: genetic causes offamilial hypercholesterolemia. Nat Clin Pract Cardiovasc Med, 2007, 4(4):214-225.
[29] Passeri MJ, Cinaroglu A, Gao C, et al. Hepatic steatosis in response to acute alcohol exposure in zebrafish requires sterol regulatory element binding protein activation. Hepatology, 2009, 49(2):443-452.
[30] Matthews RP, Lorent K, Mañoral-Mobias R, et al. TNFalphadependent hepatic steatosis and liver degeneration caused by mutation of zebrafish S-adenosylhomocysteine hydrolase. Development, 2009, 136(5): 865-875.
A zebrafish model for liver-specific expression of HCV NS5A gene
ZHAO Ye, HU Zhan-ying, TONG Jun-wei, ZHAO Li-xun, JIANG Jian-dong, ZHANG Jing-pu
ObjectiveTo establish a zebrafish model for liver-specific expression of HCV non-structural protein NS5A, and to explore the effects of HCV NS5A on liver pathological marker genes in vivo.MethodsLiver-specific gene expression regulating element zebrafish fatty acid binding protein enhancer (eFABP) sequence and HCV NS5A was cloned. By ligating the eFABP, CMV promoter, NS5A, the IRES of encephalomyocarditis virus (EMCV) and DsRed in turn, a liver-specific gene expression vector pFC-5AiR was constructed. Reporter gene DsRed was detected in Huh7 cells transfected with pFC-5AiR. The linearized vector was subsequently microinjected into zebrafish embryos. Liver-specific expression of pFC-5AiR was detected by observation under fluorescence microscopy and whole mount in situ hybridization methods. Moreover, the transcriptional and translational levels of NS5A were investigated by RT-PCR and western blot. Finally, expression level of some liver pathology associated genes was investigated by RT-PCR.ResultsCell transfection results showed that red fluorescent protein gene DsRed in pFC-5AiR is able to be expressed in Huh7 cells. After microinjection of the vector into zebrafish embryos, red fluorescence was observed to be located in the zebrafish liver under fluorescent microscopy. RT-PCR and western blot results indicated that NS5A is able to be expressed in zebrafish. In situ hybridization results further confirmed that the expression of NS5A in zebrafish liver. In addition, some pathological marker genes, such as Adiponectin, Argsyn, LDLR, TGF-β and HMGR etc., were up-regulated in response to HCV NS5A expression, suggesting that NS5A may be involved in liver lipid metabolism disorder and fibrosis.ConclusionA zebrafish model for HCV NS5A expression in liver has been successfully established. Preliminary investigation shows that HCV NS5A expression in vivo is associated with aberrant genes expression involved in liver pathological process. This model can be used to study the pathological role of HCV NS5A in vivo.
Hepatitis; Zebrafish; Disease models, animal; Gene expression
ZHANG Jing-pu, Email: zjp5577@126.com
10.3969/cmba.j.issn.1673-713X.2011.06.010
国家自然科学基金面上项目(30772681);“重大新药创制”科技重大专项(2009ZX09301-003-6-2)
100050 北京,中国医学科学院医药生物技术研究所药理室
张靖溥,Email:zjp5577@126.com
2011-10-28
方法克隆肝基因表达调控序列——斑马鱼脂肪酸结合蛋白基因增强子序列(eFABP)和 HCV 非结构蛋白基因NS5A 序列,利用 CMV 启动子序列,构建 HCV NS5A 基因和报告基因绿色荧光蛋白基因 eGFP 在肝脏共表达的基因构件 pFC-5AiR。经细胞转染实验验证所建构件的报告基因 DsRed 表达红色荧光蛋白后,将该基因构件线性化并经显微注射导入斑马鱼胚胎;通过荧光显微镜观察和整体原位杂交技术对目的基因 NS5A 的表达进行定位,验证其肝脏特异性表达。采用 RT-PCR 方法和 western blot 方法对HCV NS5A 基因的转录和翻译水平进行检测;并检测肝脏病理相关的标志基因的转录水平变化。
结果细胞转染实验证明基因构件 pFC-5AiR 的报告基因DsRed 能够在肝癌细胞 Huh7中表达;斑马鱼胚胎注射该基因构件后在肝脏能够观察到红色荧光蛋白基因 DsRed的表达;RT-PCR 和 western blot 结果显示 HCV 基因 NS5A能够在斑马鱼体内正确表达;原位杂交结果进一步证明了NS5A 的表达集中在斑马鱼肝脏内。进一步 RT-PCR 检测表明 HCV NS5A 在斑马鱼体内的表达可导致一些肝代谢和纤维化相关基因的上调,如Adiponectin、LDLR、Argsyn、TGF-β 和 HMGR 等,推测 NS5A 在肝脏脂肪病变和纤维化中具有一定作用。
结论成功建立了斑马鱼肝脏表达 HCV NS5A 的模型;初步数据表明 HCV NS5A 在斑马鱼体内的表达与部分肝代谢和纤维化标志基因的异常表达相关;该模型可以用于HCV NS5A 的体内病理机制的研究。
Author Affiliation:Department of pharmacology, Institute of Medicinal Biotechnology, Chinese Academy of Medical Sciences, Beijing 100050, China
www.cmbp.net.cn Chin Med Biotechnol, 2011, 6(6):437-444
