一种表征蛋白质可分泌性的结构融合度特征
2010-10-11高翠芳吴小俊田丰伟夏雨陈卫
高翠芳,吴小俊,田丰伟,夏雨,陈卫
1 江南大学信息工程学院,无锡 214122
2 江南大学食品学院 食品科学与技术国家重点实验室,无锡 214122
生物技术与方法
一种表征蛋白质可分泌性的结构融合度特征
高翠芳1,吴小俊1,田丰伟2,夏雨2,陈卫2
1 江南大学信息工程学院,无锡 214122
2 江南大学食品学院 食品科学与技术国家重点实验室,无锡 214122
选择适宜的信号肽是实现外源蛋白高效分泌表达的一个重要因素。本研究利用生物信息学方法分析信号肽与外源蛋白之间的相容程度,将其定义为结构融合度,并从数学角度分析拼接信号肽与目的蛋白邻近残基之间的相互作用,提出了信号肽拼接区域与目标蛋白之间的数学模型,利用该模型进行结构融合度特征提取,以此来表征外源蛋白质的可分泌性。模拟结果显示结构融合度特征能有效区分枯草芽胞杆菌宿主的可分泌和不可分泌蛋白。研究结果有助于信号肽的选择,对目的蛋白分泌表达的优化具有一定的指导意义。
信号肽,蛋白质分泌,结构融合度,特征提取
Abstract:Selection of suitable signal peptides is an important factor for efficient secretion of heterologous proteins. We defined structural fusion degree (SFD) as the compatibility degree of target proteins and signal peptides by a bioinformatics approach. We mathematically analyzed the interaction of fused signal peptides and adjacent residues of proteins, and proposed a mathematical model of extended signal region and the protein. SFD Features was extracted from this model to characterize the secretability of heterologous proteins. Simulation tests showed that SFD features can effectively discriminate high secretory proteins from poor ones in the hostBacillus subtilis. Results from this research will be useful in signal peptide selection and have a better guiding significance for the optimization of heterologous protein secretion.
Keywords:signal peptide, secretion of protein, structural fusion degree, feature extraction
能穿过合成所在的细胞位置转移到其他细胞组织中去的蛋白质,统称为分泌性蛋白质。分泌性蛋白的分泌依赖于信号肽的存在 (一般位于蛋白质链的 N-端),新合成的蛋白质在信号肽的引导下实现转移以后,信号肽序列则在信号肽酶的作用下被切除[1-2],释放出成熟蛋白质。采用分泌方式将目标蛋白质直接输出到发酵液可大大提高工业生产率[3-4]。生物工程中采用基因技术分泌表达外源蛋白时,主要策略是推测目标蛋白质可能具有的分泌途径,将该途径可识别的信号肽融合到目标蛋白质,尝试其分泌表达水平。但是该方法通常带有较大程度的盲目性[5],因为外源蛋白质的来源菌种与宿主菌在进化关系上可能差别很大,不能预知蛋白质本身与它所融合的信号肽之间的相容程度。通过不断更换信号肽反复尝试或者对信号肽进行优化虽然方法可行,但试验费用较高且研究所包涵的共性结果较少,对其他外源蛋白质分泌表达的指导意义不大。生物信息学中常用的模式识别方法是分析和处理生物数据的重要手段[6-7],如果采用该智能预测技术对外源蛋白质的可分泌性进行预分析,可大大减少不必要的生物试验。
模式识别方法用于分泌性蛋白的研究,目前主要集中在识别给定蛋白中信号肽的存在以及识别信号肽的切割位点[8-12]。其中滑动窗体 (以切割位点为基准,包括上下游部分氨基酸) 的方法较适用于识别信号肽的切割位点[8-9],序列中相邻氨基酸的环境信息也有助于提高信号肽切割位点的预测精度[9-10]。SignalP3.0是目前准确率较高的信号肽预测软件[11],它采用固定长度滑动窗体与氨基酸组分特征相结合的方法可有效提高信号肽识别率。这些研究都是以独立的蛋白序列作为识别对象,没有替换和拼接新的信号肽,因此不必考虑蛋白质本身与信号肽之间的相容问题。而外源蛋白拼接了新的信号肽以后,蛋白主链仍与原蛋白质相同,拼接前后序列的相似程度很高,但分泌水平可能变化很大。目前已开发的蛋白序列特征提取方法,如:氨基酸组分[13]、序列顺序关系[14]、序列小波能量[9,15]等,直接用来识别外源蛋白质的分泌性难度很大,而且也不能揭示人工信号肽与目标蛋白质之间的相容程度。为了提取有效的特征向量来表征蛋白质的分泌特性,本研究建立了信号肽扩展序列与目标蛋白之间的数学关系模型,从该模型提取了结构融合度特征 (SFD),并利用这些特征来识别可分泌和不可分泌外源蛋白质。
1 材料:构建蛋白质样本数据集
枯草芽胞杆菌Bacillus subtilis属于革兰氏阳性真细菌,是食品安全菌种,能够将大量蛋白质以分泌的方式输出到细胞外,可作为分泌表达外源蛋白的良好受体菌[16-17]。本研究中采用的高分泌量天然样本来自枯草芽胞杆菌N-端融合了Sec途径信号肽的目标蛋白质,共有 4种:AmyE、AprE、NprB、SacB。外源蛋白质所融合的可识别信号肽来源于这些天然样本。另外根据研究要求,从已报道的在枯草芽胞杆菌中进行过分泌尝试的样本中,选择可分泌蛋白质和不可分泌 (极低分泌量) 蛋白质作为外源蛋白样本 (表1~2)。蛋白原始序列 (氨基酸序列)可在UniProtKB/Swiss-Prot数据库(http://www.uniprot.org/uniprot/) 中查询获得。信号肽序列位于蛋白质主链的前面 (也就是N端),数据库中用signal记录说明。
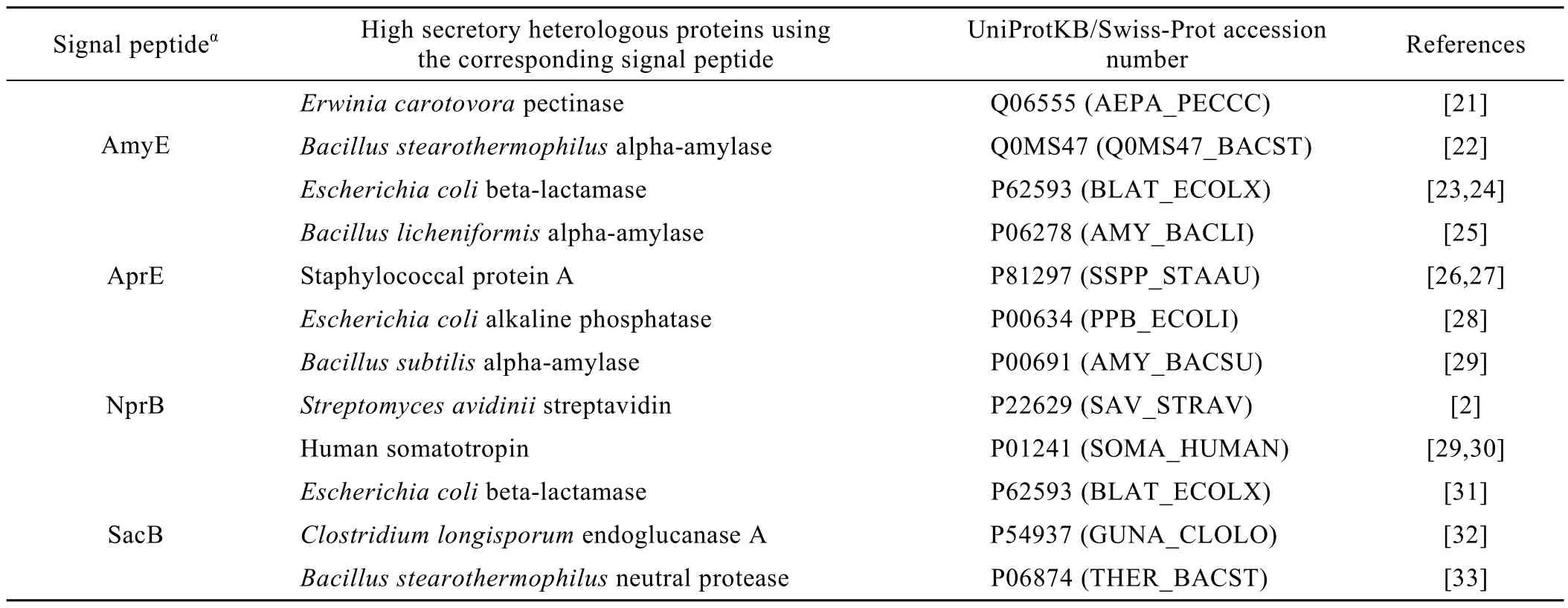
表1 高分泌外源蛋白样本信息Table 1 Information of high secretory heterologous protein samples
用枯草芽胞杆菌中可识别的高分泌蛋白质AmyE、AprE、NprB、SacB的信号肽作为人工信号肽 (表3),根据表 1~2中信号肽与各外源蛋白质的对应关系,若外源蛋白质是不可分泌蛋白质,直接将外源蛋白质与相应人工信号肽进行融合。若外源蛋白质是可分泌蛋白质且有自己的信号肽,则切除其原始信号肽,再与相应人工信号肽进行融合,过程如图1所示,其中 (a) 为从天然样本中获得可识别的信号肽序列,(b) 为切除外源样本的原始信号肽序列,(c) 为将天然可识别信号肽融合到外源蛋白质的N-端。生物方法即为采用基因技术将可识别的天然信号肽融合到外源蛋白质的N-端。
对表 1~2中的所有外源蛋白样本,利用计算机技术切除原始信号肽序列,然后再拼接人工信号肽序列,得到各个外源蛋白质的新序列样本,从而构建人工研究样本集。

图1 将可识别的天然信号肽融合到外源蛋白质的N-端Fig.1 Model for recognized natural signal peptide in-frame fuse to N-terminal of heterologous protein chain.
2 方法:特征提取
2.1 氨基酸组分特征
提取蛋白质特征的典型方法是根据序列中氨基酸的组成成分。蛋白质链通常用一条氨基酸序列来描述,链上的元素就是氨基酸的名称。按照字母排列顺序,构成蛋白质序列的20种氨基酸的基本字符集为:

表2 低分泌外源蛋白样本信息Table 2 Information of poor secretory heterologous protein samples
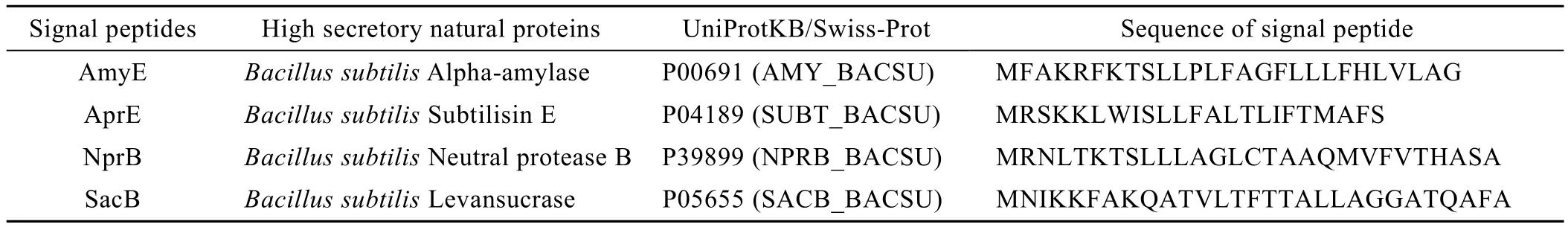
表3 信号肽序列信息Table 3 Information of signal peptides

Θ中每个字符代表一种氨基酸,给定一个包含n个氨基酸残基的蛋白质序列Q:

其中 Ri(i=1,2,3…n) 为集合 Θ中的字符。为了能用计算机处理蛋白质样本,需要把字符序列表示成向量数据,传统的离散化方法是用20维向量来表示蛋白质的20种氨基酸组成[13]:
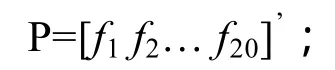
与集合Θ中的字符排列顺序对应,其中f1表示丙氨酸(A) 在整条蛋白质序列中出现的频率,f2表示半胱氨酸(C) 在整条蛋白质序列中出现的频率,以此类推。这样可以提取蛋白质基本组成的20维特征向量。
2.2 结构融合度特征 (SFD)
上述向量是对整个蛋白质序列的特征提取,无法直接用来区分外源蛋白质的可分泌性。希望提取的特征向量能揭示人工信号肽与目标蛋白之间所蕴藏的内在关系,因此提出在信号肽拼接区与目标蛋白质之间建立数学关系模型,从模型得到的特征向量能客观地反映人工信号肽和目标蛋白质之间的融合程度,进而反映拼接后目标蛋白质的可分泌性。
2.2.1信号肽扩展序列信息集
为了研究信号肽拼接以后与邻近残基之间的相互作用,也就是拼接区域的局部融合程度,对信号肽序列进行了扩展,使其延伸到包括部分外源蛋白序列,称为信号肽扩展序列。例如信号肽序列的长度为l,延伸长度为15(与信号肽相邻的15个氨基酸残基),则扩展序列的长度为l+15,如图2。
设S是信号肽扩展序列,则有:

其中Ri(i=1,2,3…l+15) 为集合Θ中表示氨基酸名称的字符。根据子序列分布集合的描述方法[18],构建了信号肽扩展序列 S的子序列分布集合,集合中是序列 S的所有包含信号肽片段的子序列。由于信号肽序列一般包括3个功能区域[19],分别由不同极性的氨基酸组成,考虑到3个功能区域的相互作用,子序列应包含完整的信号肽序列。例如:第k(k≤16)条子序列是Uk={R1R2... RlRl+1... Rl+k-1}。按此规则,可以得到唯一的子序列分布集合:Ω=(U1U2... U15U16)。其中:

图 3的例子显示了信号肽扩展序列通过窗体拉伸得到的子序列分布集合 (即框中的子序列)。其中原信号肽长度为l=23 (即灰色区域内的序列)。显然,序列集合Ω中共有16条子序列,其中U1就是信号肽序列,U16就是信号肽扩展序列。每条序列都比前一条序列多一个氨基酸残基。子序列长度的延伸包含了信号肽与蛋白主链邻近残基之间的相互作用,因此扩展序列信息集一定程度上蕴含了拼接区域的局部特征,也蕴含了局部结构的融合信息。

图2 信号肽扩展序列Fig.2 Extended signal peptide sequence.
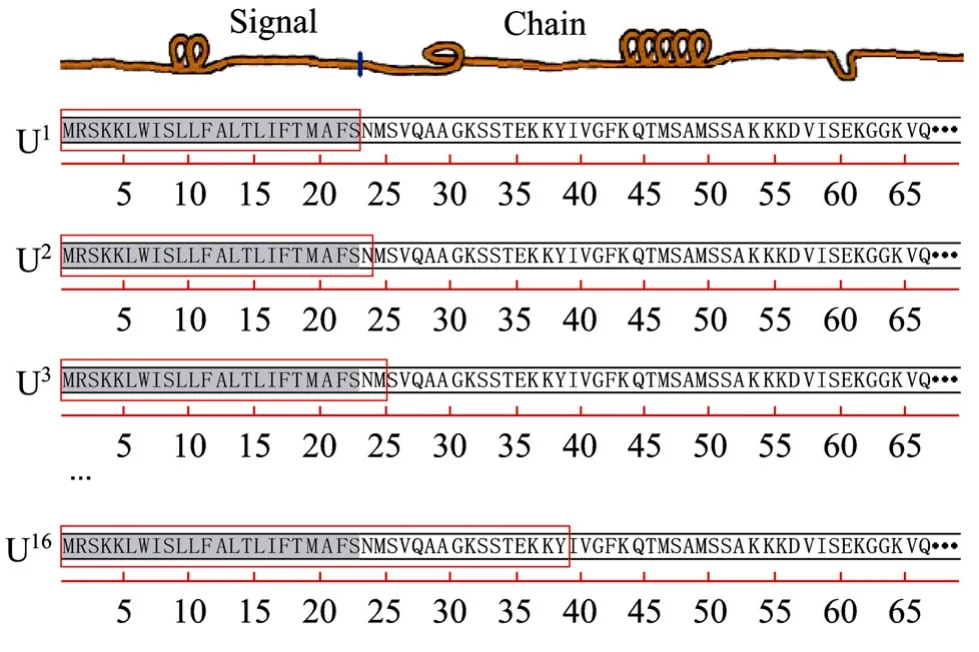
图3 信号肽扩展序列的子序列分布集合Fig.3 Distribution of sub sequence set of extended signal peptide sequence.
2.2.2提取结构融合度特征的数学模型
对于集合Ω中的每个子序列,均提取20维的氨基酸组分特征向量,共得到16个特征向量,组成信号肽拼接区域的特征矩阵A:

其中V1=[v1,1v1,2…v1,20]’ 是子序列U1的特征向量,V2=[v2,1v2,2…v2,20]’ 是子序列 U2的特征向量,以此类推。按照同样方法提取整个蛋白质链的特征向量B=[b1b2…b20]’。
集合Ω中各子序列之间存在部分重叠,因此矩阵A中各向量之间存在一定的相关信息。协方差是用来描述两个变量之间相互关系的数字特征,利用多个不同变量的协方差组成的协方差矩阵,可进行相应的总体相关性分析。设C是矩阵A中各维分量的协方差矩阵:

矩阵C是对称阵,其中第(i,j) 个元素是矩阵A中第i维分量与第j维分量 (第i行与第j行) 的协方差。设D是由C的特征向量组成的矩阵,由于矩阵的特征向量既能描述矩阵本身的特征又不损失矩阵信息,还能方便数学上的处理,因此在矩阵D与向量B之间建立数学关系:

矩阵D蕴含了信号肽拼接区域的特征,向量B则蕴含了整个蛋白质链的特征,因此未知向量X=[x1x2…x20]’可以体现信号肽拼接区与目标蛋白质之间的内在关系,这里称为结构融合度特征 (SFD),这种特征向量可用来描述外源蛋白质的分泌特征。
若D是满秩矩阵,则D−1存在,方程组(1)的解向量为X=D−1B。当矩阵D是奇异矩阵时,D−1不存在,可用最小二乘法得到方程组(1) 的解向量。这种基于融合程度的方法只依赖于序列本身,不涉及任何主观因素,可以快速对人工信号肽与蛋白主链建立内在关系分析,避免了不断更换信号肽进行反复尝试的盲目性。
3 模拟实验与结果分析
在第 1部分构建的人工蛋白序列集上,分别取信号肽扩展序列的延伸长度为 5、10、15、20,对不同的延伸长度,每个蛋白序列均提取20维的结构融合度特征,加上氨基酸组分的基础特征,这样共得到 5个分别用不同特征向量表示的数据集。为了直观观察各数据集中的样本分布情况,同时尽量保持原样本间的相互距离关系,用线性映射的方法[20]将特征向量投影到二维平面,结果如图4。
对于以上 5种特征,用下面的指标来检验其有效性:即类内距离tr(Sw) 尽量小,类间距离tr(Sb) 尽量大的准则。因此类间距与类内距的比值tr(Sb)/tr(Sw) 越大,聚类效果越好。
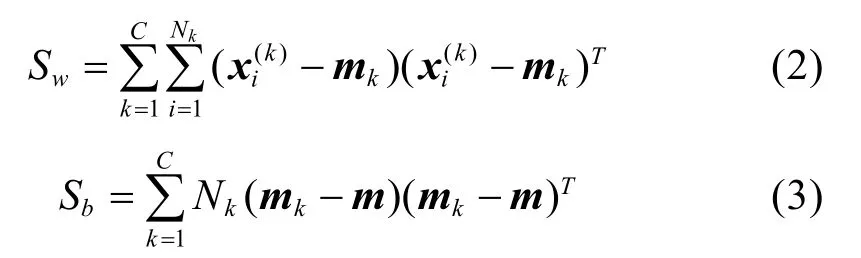
其中C是分类数,Nk是第k类的样本数,mk是第k类样本的均值向量,m是所有样本的均值向量。

表4 不同特征的有效性指标Table 4 Validity indexes of different features
在表4显示的5种特征中,当信号肽延长10或15个氨基酸残基时,结构融合度特征的类间距与类内距比值较大。图4中的二维特征分布图也显示,此时特征的类内紧致性和类间可分性较好。这说明信号肽拼接以后与蛋白主链中较邻近的15个氨基酸残基之间的相互作用较大,因此根据局部相互作用提取的融合度特征可以用来描述蛋白质的分泌性能。
采用FCM模糊聚类算法,分别用上述5种特征向量对表 1~2中的各蛋白样本进行聚类分析,划分为可分泌和不可分泌两类 (其中天然可分泌样本和人工可分泌样本属于同一类),聚类结果如表5。
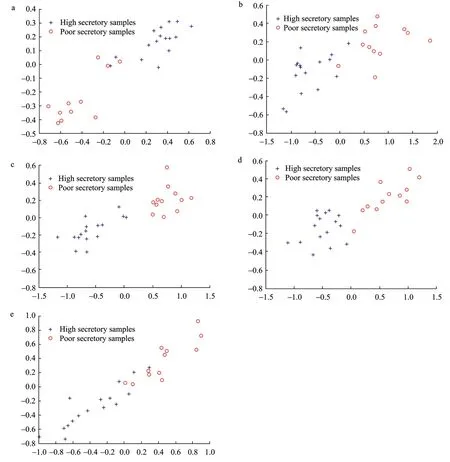
图4 不同特征集的二维分布效果Fig.4 2-D mapped distribution of different features. (a) Features of amino acid composition. (b) Features of SFD (prolongation is 5)(c) Features of SFD (prolongation is 10). (d) Features of SFD (prolongation is 15). (e) Features of SFD (prolongation is 20).
通过模拟实验发现,信号肽延长的氨基酸个数较少 (<5) 时,信号肽本身的特征信息影响较大,聚类结果基本上按照信号肽的种类划分,也就是同一种信号肽会划分为一类,不能区分可分泌和不可分泌蛋白质。相反,当信号肽延长的氨基酸个数较多 (>30) 时,信号肽本身的特征信息被淡化,结构融合度特征与整个蛋白质链的特征向量很接近。
为进一步测试利用SFD特征对外源蛋白分泌性的识别效果,与目前常用的信号肽预测软件进行了实验比较,分别是 SignalP3.0 (http://www.cbs.dtu.dk/services/SignalP/),TargetP(http://www.cbs.dtu.dk/services/TargetP/),PrediSi (http://www.predisi.de/),Phobius(http://www.ebi.ac.uk/Tools/phobius/)。其中 SignalP3.0包括神经网络(SignalP3-NN)和隐马尔科夫模型(SignalP3-HMM)两种预测结果,SignalP3-NN中用3种分值:max S、mean S、D-score预测给定蛋白中是否存在信号肽,其中max S是预测信号肽存在的粗测值; mean S可预测信号肽长度,也是SignalP2.0中识别信号肽的标准; D-score是识别信号肽的高级标准。对于 4种信号肽来源的天然样本,实验所涉及的几种算法均能给出正确预测结果,无需再做比较,故这里只列出各算法对人工样本的预测结果(表 6~7)。
目前的预测软件仅局限于识别蛋白序列中是否存在信号肽,无法判断给定蛋白属于天然样本还是人工拼接样本。对于拼接后的人工样本,信号肽序列的存在并不能表示分泌水平高,只有两者真正相容才能实现高分泌。表中结果显示,预测软件虽然能识别信号肽的存在,却几乎把所有拼接天然信号肽的外源蛋白预测为可分泌蛋白,只有少数几种不可分泌蛋白被识别出。本研究的特征提取方法与以前的研究不同,是从两者的融合程度出发,揭示人工信号肽与目标蛋白之间蕴藏的内在关系,因此能较好地区分拼接样本中的可分泌和不可分泌外源蛋白。

表5 采用不同特征得到的聚类精度Table 5 Clustering accuracy obtained by different features

表6 高分泌外源蛋白样本的预测结果Table 6 Prediction results of high secretory heterologous protein samples
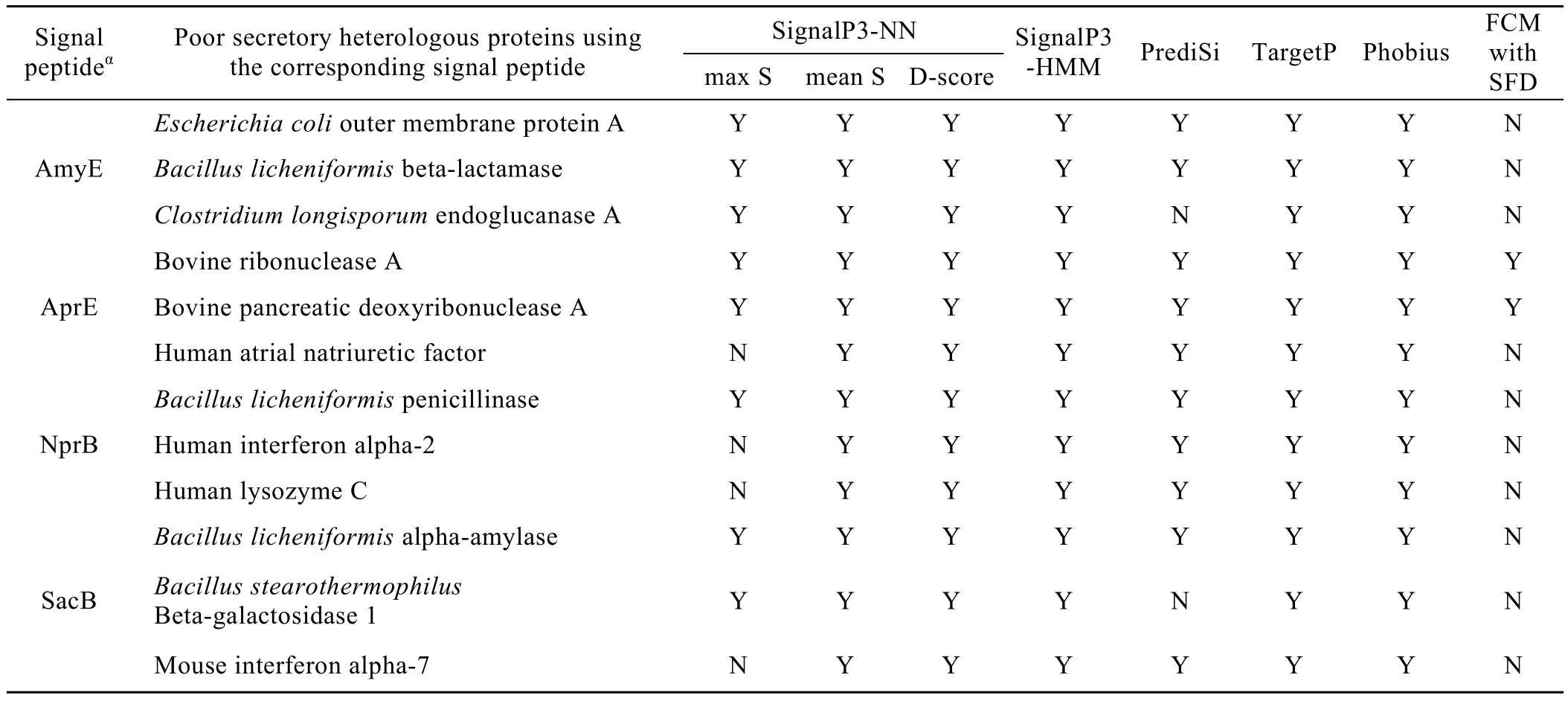
表7 低分泌外源蛋白样本的预测结果Table 7 Prediction results of poor secretory heterologous protein samples
4 结论
实现外源蛋白高效分泌的一个重要因素是选用适宜的信号肽。本研究根据人工信号肽与目标蛋白质的融合程度进行前期分析预测,能为外源蛋白质的信号肽选择提供理论参考,减少目标蛋白质进行生物试验的分泌尝试次数。文章中是基于氨基酸组分的结构融合度特征提取,对于其他不同角度的蛋白序列特征,如序列相关系数特征和序列小波能量特征等,也可用文中的数学模型得到结构融合度特征,但具体特征的有效性如何,有待继续研究。模型中对信号肽拼接区的特征矩阵A的相关信息分析可以有多种方法,除了文章中所利用的协方差矩阵以外,还有矩阵的自相关分析、矢量间的信息增量等方法。另外,信号肽扩展序列的延伸长度不同,所提取的特征效果也不同,文章对此作了实验对比和初步分析,但是拼接后信号肽与邻近残基之间的更密切的相互作用,有待于更深入的研究。对于文章中的理论研究结果,将来需采用生物试验做进一步验证,结合生物验证结果,对文章中所提出的理论进行调整和完善。
REFERENCES
[1] Tjalsma H, Antelmann H, Jongbloed JDH,et al.Proteomics of protein secretion byBacillus subtilis:separating the “Secrets” of the secretome.Microbiol Mol Biol Rev, 2004, 68(2): 207–233.
[2] Wei XF, Wang DM, Liu S,et al. Signal sequence and its application to protein expression.Biotechnol Bull, 2006,6: 38–42.
韦雪芳, 王冬梅, 刘思, 等. 信号肽及其在蛋白质表达中的应用. 生物技术通报, 2006, 6: 38–42.
[3] Schallmey M, Singh A, Ward OP. Developments in the use ofBacillusspecies for industrial production.Can JMicrobiol, 2004, 50(1): 1–17.
[4] Zhang XZ, Cui ZL, Hong Q,et al. High-level expression and secretion of methyl parathion hydrolase inBacillus subtilisWB800.Appl Environ Microbiol, 2005, 71(7):4101–4103.
[5] Fu LL, Xu ZR, Li WF,et al. Protein secretion pathways inBacillus subtilis: implication for optimization of heterologous protein secretion.Biotechnol Adv, 2007,25(1): 1–12.
[6] Liew AWC, Yan H, Yang M. Pattern recognition techniques for the emerging field of bioinformatics: a review.Pattern Recognition,2005, 38(11): 2055–2073.
[7] Keedwell E, Narayanan A. Intelligent Bioinformatics: the Application of Artificial Intelligence Techniques to Bioinformatics Problems. Chichester, West Sussex,England: John Wiley & Sons Ltd, 2005: 101–218.
[8] Chou KC. Prediction of protein signal sequences.Curr Protein Pept Sci, 2002, 3(6): 615–622.
[9] Li YZ, Wen ZN, Zhou CS,et al. Effects of neighboring sequence environment in predicting cleavage sites of signal peptides.Peptides, 2008, 29(9): 1498–1504.
[10] Käll L, Krogh A, Sonnhammer ELL. A combined transmembrane topology and signal peptide prediction method.J Mol Biol, 2004, 338(5): 1027–1036.
[11] Bendtsen JD, Nielsen H, Heijne G,et al. Improved prediction of signal peptides: SignalP 3.0.J Mol Biol,2004, 340(4): 783–795.
[12] Liu H, Yang J, Liu DQ,et al. Using a new alignment kernel function to identify secretory proteins.Protein Pept Lett, 2007, 14(2): 203–208.
[13] Shen HB, Chou KC. Ensemble classifier for protein fold pattern recognition.Bioinformatics, 2006, 22(14): 1717–1722.
[14] Chou KC. Prediction of protein cellular attributes using pseudo amino acid composition.Proteins: Struct, Funct,Genet, 2001, 43(3): 246–255.
[15] Liò P. Wavelets in bioinformatics and computational biology: state of art and perspectives.Bioinformatics,2003, 19(1): 2–9.
[16] Sonenshein AL, Hoch JA, Losick R.Bacillus subtilisand Its Closest Relatives. Washington DC: ASM Press, 2001.
[17] Xia Y, Chen W, Zhao JX,et al. Construction of a new food-grade expression system forBacillus subtilisbased on theta replication plasmids and auxotrophic complementation.Appl Microbiol Biotechnol, 2007, 76(3):643–650.
[18] Zhang M, Fang WW, Zhang JH,et al. MSAID: multiple sequence alignment based on a measure of information discrepancy.Comput Biol Chem, 2005, 29(2): 175–181.
[19] Tjalsma H, Bolhuis A, Jongbloed JDH,et al. Signal peptide-dependent protein transport inBacillus subtilis: agenome-based survey of the secretome.Microbiol Mol Biol Rev, 2000, 64(3): 515–547.
[20] Bian ZQ, Zhang XG. Pattern Recognition. 2nd ed. Beijing:Tsinghua University Press, 2000: 185–198.
边肇祺, 张学工. 模式识别. 2版. 北京: 清华大学出版社, 2000: 185–198.
[21] Hemila H, Pakkanen R, Heikinheimo R,et al. Expression of theErwinia carotovorapolygalacturonase-encoding gene inBacillus subtilis: role of signal peptide fusions on production of a heterologous protein.Gene, 1992, 116(1):27–33.
[22] Tsukagoshi N, Iritani S, Sasaki T,et al. Efficient synthesis and secretion of a thermophilic alpha-amylase by protein-producingBacillus brevis47 carrying theBacillus stearothermophilusamylase gene.J Bacteriol, 1985,164(3): 1182–1187.
[23] Palva I, Sarvas M, Lehtovaara P,et al. Secretion ofEscherichia colibeta-lactamase fromBacillus subtilisby the aid of alpha-amylase signal sequence.Proc Natl Acad Sci USA, 1982, 79(18): 5582–5586.
[24] Palva I, Sarvas M, Lehtovaara P,et al. Secretion ofEscherichia colibeta-lactamase fromBacillus subtilisby the aid of alpha-amylase signal sequence.Biotechnology,1992, 24: 344–348.
[25] Sloma A, Pawlyk D, Pero J. Development of an Expression and Secretion System inBacillus subtilisUtilizing sacQ//Ganesan AT, Hoch JA, ed. Genetics and Biotechnolog ofBacilli. San Diego: Academic Press,1988: 23–26.
[26] Vasantha N, Thompson LD. Fusion of pro region of subtilisin to staphylococcal protein A and its secretion byBacillus subtilis.Gene, 1986, 49(1): 23–28.
[27] Fahnestock SR, Fisher KE. Expression of the staphylococcal protein A gene inBacillus subtilisby gene fusions utilizing the promoter from aBacillus amyloliquefaciensalpha-amylase gene.J Bacteriol, 1986,165(3): 796–804.
[28] Payne MS, Jackson EN. Use of alkaline phosphatase fusions to study protein secretion inBacillus subtilis.J Bacteriol, 1991, 173(7): 2278–2282.
[29] Nakayama A, Shimada H, Furutani Y,et al. Processing of the prepropeptide portions of theBacillus amyloliquefaciensneutral protease fused toBacillus subtilisalpha-amylase and human growth hormone during secretion inBacillus subtilis.J Bacteriol, 1992, 23(1): 55–69.
[30] Franchi E, Maisano F, Testori SA,et al. A new human growth hormone production process using a recombinantBacillus subtilisstrain.J Bacteriol, 1991, 18(1/2): 41–54.
[31] Edelman A, Joliff G, Klier A,et al. A system for the inducible secretion of proteins fromBacillus subtilisduring logarithmic growth.FEMS Microbiol Lett, 1988,52(1/2): 117–120.
[32] Petit MA, Joliff G, Mesas JM,et al. Hypersecretion of a cellulase fromClostridium thermocelluminBacillus subtilisby induction of chromosomal DNA amplification.Biotechnology, 1990, 8(6): 559–563.
[33] Zhang M, Zhao C, Du LX,et al. Expression, purification,and characterization of a thermophilic neutral protease fromBacillus stearothermophilusinBacillus subtilis.Sci China Series C: Life Sci, 2008, 51(1): 52–59.
[34] Simonen M, Tarkkaa E, Puohiniemia R,et al.Incompatibility of outer membrane proteins OmpA and OmpF ofEscherichia coliwith secretion inBacillus subtilis: fusions with secretable peptides.FEMS Microbiol Lett, 1992, 79(1/3): 233–241.
[35] Imanaka T, Tanaka T, Tsunekawa H,et al. Cloning of the genes for penicillinase, penP and penI, ofBacillus licheniformisin some vector plasmids and their expression inEscherichia coli,Bacillus subtilis, andBacillus licheniformis.J Bacteriol, 1981, 147(3): 776–86.
[36] Soutschek-Bauer E, Staudenbauer WL. Synthesis and secretion of a heat-stable carboxymethylcellulose fromClostridium thermocelluminBacillus subtilisandBacillus stearothermophilus.Mol Gen Genet, 1987, 208(3):537–541.
[37] Vasantha N, Filpula D. Expression of bovine pancreatic ribonuclease A coded by a synthetic gene inBacillus subtilis.Gene, 1989, 76(1): 53–60.
[38] Wang LF, Wong SL, Lee SG,et al. Expression and secretion of human atrial natriuretic alpha-factor inBacillus subtilisusing the subtilisin signal peptide.Gene,1988, 69(1): 39–47.
[39] Takagi M, Imanaka T. Role of the pre-pro-region of neutral protease in secretion inBacillus subtilis.J Ferment Bioeng, 1989, 67(2): 71–76.
[40] Palva I. Construction of aBacillussecretion vector.University of Helsinki, 1983.
[41] Yoshimura K, Toibana A, Kikuchi K,et al. Differences betweenSaccharomyces cerevisiaeandBacillus subtilisin secretion of human lysozyme.Biochem Biophys Res Commun, 1987, 145(2): 712–718.
[42] Ganesan AT, Hoch JA.BacillusMolecular Genetics and Biotechnology Applications. San Diego: Academic Press,1986: 479–491.
[43] Dion M, Rapoport G, Doly J. Expression of the MuIFN alpha 7 gene inBacillus subtilisusing the levansucrase system.Biochimie, 1989, 71(6): 747–755.
Characterization of protein secretion based on structural fusion degree
Cuifang Gao1, Xiaojun Wu1, Fengwei Tian2, Yu Xia2, and Wei Chen2
1School of Information Engineering,Jiangnan University,Wuxi214122,China
2State Key Laboratory of Food Science and Technology,School of Food Science and Technology,Jiangnan University,Wuxi214122,China
Received:November 19, 2009;Accepted:February 2, 2010
Supported by:Program for New Century Excellent Talents in University of China (No. NCET-06-0487), National Natural Science Foundation of China (Nos. 60572034, 60973094, 30670065), Natural Science Foundation of Jiangsu Province (No. BK2006081), Program for Innovative Research Team of Jiangnan University (No. JNIRT0702).
Corresponding author:Xiaojun Wu. Tel: +86-510-85912139; Fax: +86-510-85912136; E-mail: wu_xiaojun@yahoo.com.cn
教育部新世纪优秀人才计划项目 (No. NCET-06-0487),国家自然科学基金 (Nos. 60572034, 60973094, 30670065),江苏省自然科学基金 (No.BK2006081),江南大学创新团队计划项目 (No. JNIRT0702) 资助。
