基于线粒体Cytb基因的江淮下游6个湖泊翘嘴鲌群体遗传多样性分析
2024-04-08李大命杨子萍刘燕山唐晟凯谷先坤殷稼雯
李大命 杨子萍 刘燕山 唐晟凯 谷先坤 殷稼雯
摘要:以线粒体Cytb基因作为分子标记对长江、淮河下游6个湖泊翘嘴鲌群体共262尾个体的遗传多样性、遗传分化及历史动态进行分析。结果显示,翘嘴鲌Cytb基因序列全长为1 141 bp,碱基A+T的含量(55.9%)高于G+C含量(44.1%)。262条Cytb基因序列检出42个变异位点,定义46个单倍型,总群体单倍型和核苷酸多样性分别为0.916±0.010和0.00 256±0.00 008;6个群体的单倍型多样性为(0.798±0.059)~(0.927±0.019),核苷酸多样性为(0.002 04±0.000 27)~(0.002 85±0.000 18),表明翘嘴鲌遗传多样性属于高Hd和低Pi模式。基于单倍型构建的系统发育树和进化网络图显示,6个群体的单倍型未形成明显的地理格局。分子方差分析(AMOVA)显示,遗传变异主要来自于群体内部(91.64%),群体间遗传分化系数(Fst)为0.083 62(P<0.01);6个群体间的Fst为 -0.004 27~0.155 29,其中,长江群体和淮河群体间均具有显著中度或高度遗传分化(P<0.05)。中性检验结果显示,Tajimas D和Fus Fs均为负值(P<0.05),核苷酸不配对分布曲线呈现单峰型,表明翘嘴鲌群体在历史上经历过显著的种群扩张。整体来看,江淮下游湖泊的翘嘴鲌野生种质资源遗传多样性较低,长江水系群体和淮河水系群体间有显著的遗传分化,应采取措施恢复翘嘴鲌资源量及提高其遗传多样性,并将长江流域和淮河流域湖泊群体划分为不同的管理单元进行保护。
关键词:翘嘴鲌;Cytb基因;遗传多样性;遗传分化;种群历史动态
中图分类号:S917 文献标志码:A
文章编号:1002-1302(2024)03-0213-07
翘嘴鲌(Culter alburnus)俗称白鱼、翘嘴白丝、大白鱼,隶属鲤形目(Cypriniformes)鲤科(Cyprinidae)鲌亚科(Culterinae)鲌属(Culter),广泛分布于我国各江河湖泊[1]。翘嘴鲌为凶猛肉食性鱼类,它能有效控制小型鱼类种群数量,减轻小型鱼类对浮游生物的压力,在优化鱼类群落机构及维持水域生态系统稳定方面发挥了重要作用[2]。翘嘴鲌肉味鲜美、肉质细嫩洁白、营养丰富、经济价值高,且生长迅速,是我国常见的淡水养殖品种之一[3]。多年来,由于过度捕捞、生态环境污染及栖息地破坏多种因素的影响,翘嘴鲌野生资源遭到严重破坏,种群数量急剧衰退,遗传多样性下降,个体小型化、低龄化趋势严重,因此加强翘嘴鲌种质资源保护成为亟待解决的问题[4]。
迄今,有关翘嘴鲌的研究主要集中在年龄、生长、繁殖、营养、养殖等方面[5-9],而有关翘嘴鲌资源的遗传多样性则需加强研究。鱼类遗传多样性是鱼类物种生存与进化的基础,是评价自然资源的重要依据,开展鱼类遗传多样性研究,对渔业资源管理与养护、人工增殖放流等工作具有重要的指导意义[10]。线粒体DNA(mitochondrial DNA,mtDNA)作為核外遗传物质,具有分子小、结构简单、进化快、严格的母系遗传等特点,已成为群体遗传学和系统发育研究的重要分子标记[11]。细胞色素b基因(cytochrome b,Cytb)是mtDNA上的蛋白质编码基因之一,其结构和功能较为清楚,进化速度适中,有通用引物便于扩增和测序,因此在鱼类群体遗传多样性和遗传结构研究方面有着广泛应用[12-14]。目前,已有利用mtDNA的不同区域和基因序列开展翘嘴鲌群体遗传多样性评价的一些报道。杨子拓和黄小彧等采用D-loop控制区序列分别研究了珠江水系和长江水系翘嘴鲌的遗传多样性及遗传分化[15-16]。王伟等利用COⅡ序列分析了6个水域的翘嘴鲌群体遗传多样性,结果显示翘嘴鲌遗传多样性较低(Hd:0.604 8,Pi:0.001 63)[17]。伊西庆基于ND2序列探究了我国东部6个湖泊翘嘴鲌遗传多样性和遗传结构,结果表明部分群体间出现高度遗传分化[18]。江淮下游是我国淡水湖泊的集中分布区之一,但对于江淮水系内部及水系间不同湖泊的翘嘴鲌群体遗传多样性及分化尚缺乏了解。
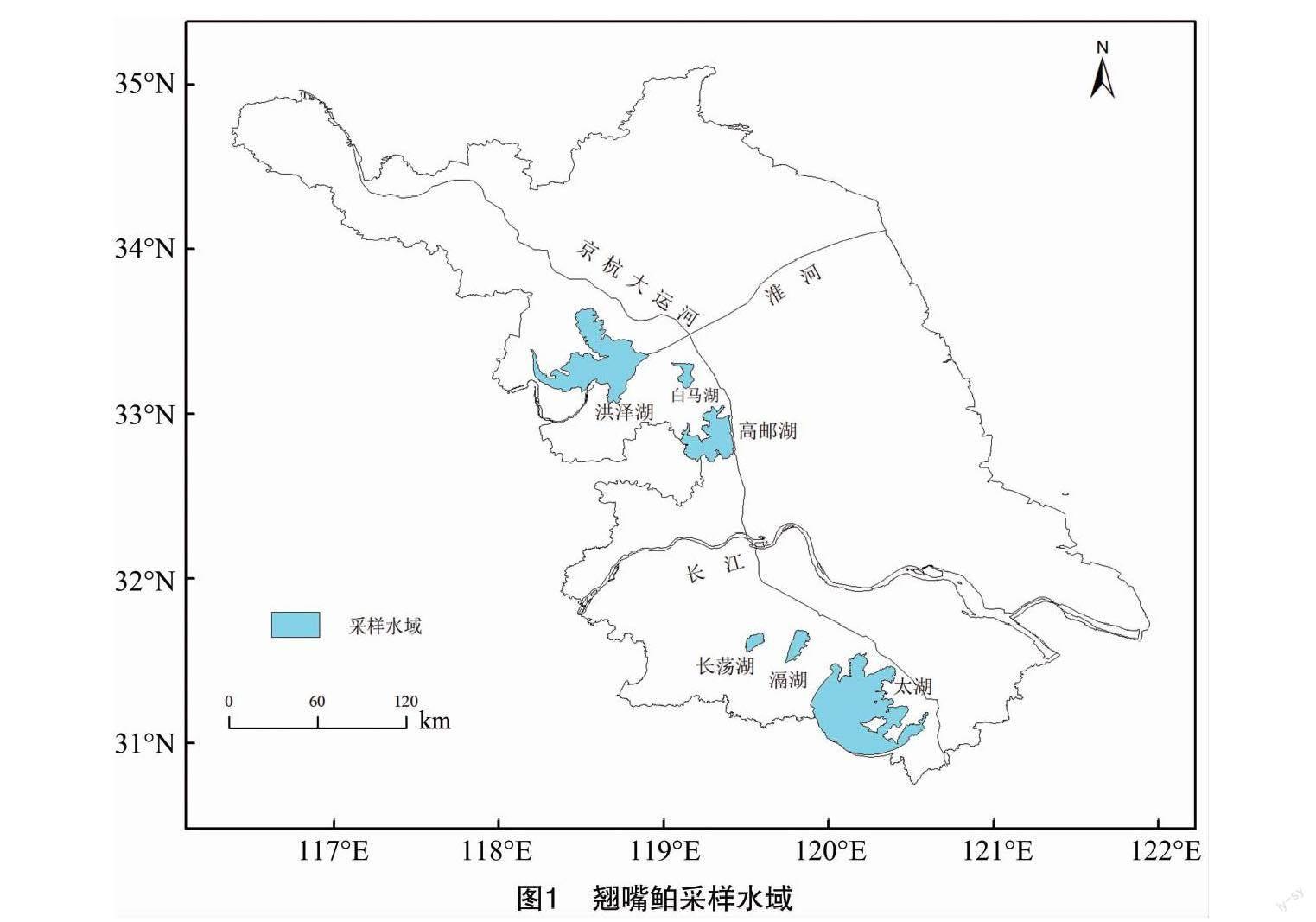
本研究采集长江下游的太湖、滆湖及长荡湖和淮河下游的高邮湖、白马湖及洪泽湖6个湖泊翘嘴鲌群体,通过mtDNA Cytb基因序列的比较分析,探究江淮下游6个湖泊的翘嘴鲌群体遗传多样性和进化关系,期望深入了解6个湖泊翘嘴鲌群体的遗传背景和种质资源现状,以便于为翘嘴鲌的野生资源科学保护、合理利用及增殖放流提供基础数据。
1 材料与方法
1.1 样本采集
2021年5月至2022年10月,在长江下游的太湖、滆湖、长荡湖和淮河下游的高邮湖、白马湖、洪泽湖采集翘嘴鲌样品。各群体的采样水域和样本数见图1和表1,测量翘嘴鲌的的体长、体质量,剪取尾鳍组织放入无水乙醇中固定保存。
1.2 DNA提取、PCR扩增和测序
采用TaKaRa公司的DNA提取试剂盒从尾鳍组织提取翘嘴鲌的基因组DNA,通过1%琼脂糖凝胶电泳验证DNA的完整性,并使用分光光度计测定DNA浓度后,于-20 ℃保存备用。
采用通用引物L14724(5′-CGTCAGTCCTTTACTTCGCA-3′)和H15915(5′-AGGGCATACTCACGGGGTTG-3′)[19]扩增翘嘴鲌Cytb基因序列,引物由生工生物工程(上海)股份有限公司合成。PCR反应体系共50.0 μL,其中,包括:2×Premix TaqTM 25.0 μL(TaKaRa Taq 1.25 U/25 μL、0.2 mmol/L dNTP、1.5 mmol/L Mg2+、色素Marker、比重增加物和稳定剂)、上下游引物各2.0 μL(10 μmol/L)、DNA模板2.0 μL(40 ng/μL)和ddH2O 33.0 μL。PCR扩增程序为:94 ℃预变性4 min;94 ℃变性 40 s,55 ℃复性30 s,72 ℃延伸1 min,35 个循环;72 ℃ 延伸5 min。PCR完成后取4.0 μL经1%琼脂糖凝胶电泳检测,剩余的PCR产物送至生工生物工程(上海)股份有限公司,进行纯化和双向测序,测序引物与扩增引物一致。
1.3 数据处理和分析
使用BioEdit 7.0软件读取序列,并参照测序图谱人工校对序列中可能的错误。使用CLUSTALX 2.0软件对序列进行多重比对和排序。使用MEGA 7.0软件统计序列的碱基组成、突变位点数和突变类型,计算群体间的Kimura双参数遗传距离,利用邻接法(neighbor-joining,NJ)构建单倍型系统发育树,系统树中节点的自举置信水平应用自引导估计,循环次数为1 000次。采用Network软件构建单倍型网络图,用以展示单倍型之间的进化关系。使用DanSP 6.0软件计算群体的单倍型数量、单倍型多样性(haplotye diversity,Hd)、核苷酸多样性(nucleotide diversity,Pi)等遗传多样性参数。采用Arlequin 3.5软件进行分子方差分析(analysis of molecular variance,AMOVA)和计算群体间遗传分化指数(Fst),开展Tajimas D及Fus Fs中性检验和构建核酸错配分布曲线,评估翘嘴鲌群体历史动态。
2 结果与分析
2.1 翘嘴鲌Cytb序列变异和遗传多样性
本研究获得6个翘嘴鲌群体262条Cytb基因序列,其长度为1 141 bp,碱基平均含量分别为A(29.3%)、T(26.6%)、C(29.4%)和G(14.7%),其中A+T的含量(55.9%)高于G+C的含量(44.1%),具有明显的碱基组成偏倚性。
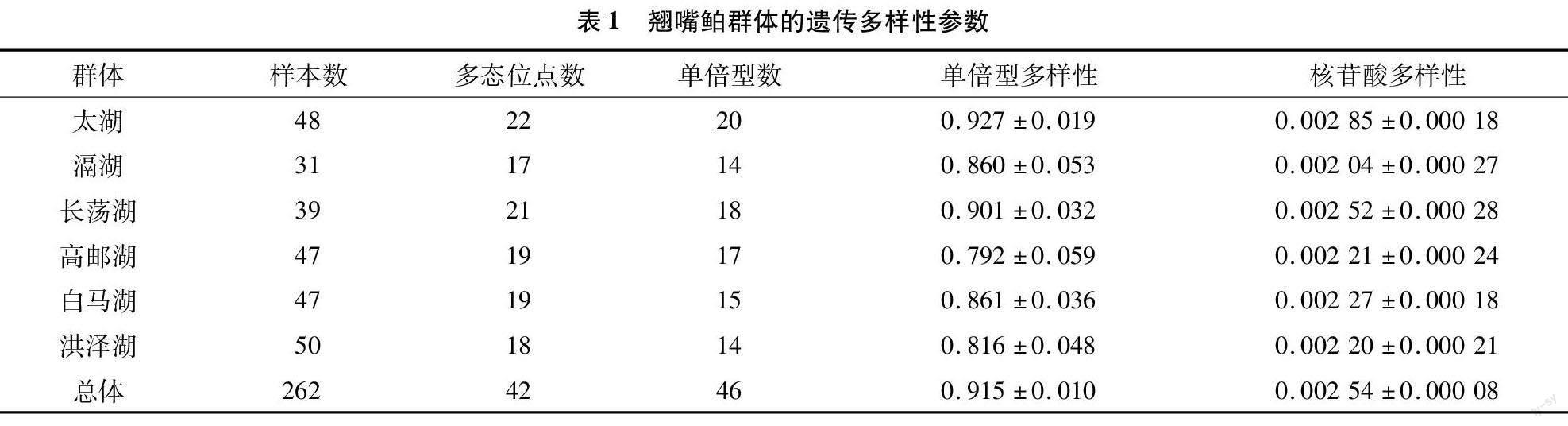
由表1可知,262条Cytb序列检出42个变异位点,其中,有33个简约信息位点和9个单一信息位点,没有插入或缺失位点。262条序列定义46种单倍型,整个群体的单倍型多样性和核苷酸多样性分别为0.915±0.010和0.002 54±0.000 08;6个群体的单倍型多样性为(0.792±0.059)~(0.927±0.019),核苷酸多样性为(0.002 20±0.000 21)~(0.002 85±0.000 18),其中,太湖群体的单倍型多样性和核苷酸多样性最大,高邮湖群体的单倍型多样性最小,滆湖群体的核苷酸多样性最小。
2.2 群体单倍型组成及系统发育关系
由表2可知,6个群体262个个体共定义46种单倍型,其中,太湖群体拥有的种类数最多(20种),滆湖和洪泽湖群体拥有的种类数最少(均为14种),长荡湖、高邮湖和白马湖群体各拥有18、17、15种单倍型。
46种单倍型有26种共享单倍型,其中,单倍型Hap2、Hap8和Hap41为6个群体共享,个体数量分别有36、15、25个,合计占比为28.8%;其他共享单倍型为部分群体拥有,单倍型Hap22为高邮湖、白马湖和洪泽湖群体共享,其个体数量最多(56个),占比为21.2%。独享单倍型有20种,其中,单倍型Hap3、Hap4、Hap18和Hap20为太湖群体独有,单倍型Hap6、Hap34、Hap37和Hap38为滆湖群体独有,单倍型Hap13、Hap31、Hap33和Hap43为长荡湖群体独有,单倍型Hap5、Hap23、Hap29和Hap30为高邮湖群体独有,单倍型Hap14、Hap27为白马湖群体独有,单倍型Hap26和Hap28为洪泽湖群体独有。
以蒙古鲌(Culter mongolicus)为外类群,采用邻接法构建单倍型系统进化树。由图2可知,6个群体形成了单系类群,各群体的单倍型呈混杂分布模式,没有特定的地理群体谱系。由单倍型网络结构图(图3)可知,群体间拥有多个共享单倍型,各单倍型相互散布在不同的地理群体中,未形成明显的系统地理格局,这与系统发育树结果相一致。
2.3 群体遗传分化
由表3可知,6个群体间的遗传距离为0.002 2~0.003 0,其中,太湖和洪泽湖群体间遗传距离最大,洪泽湖和高邮湖群体间遗传距离最小。由6个群体的UPGMA聚类结果(图4)可知,洪泽湖、高邮湖和白马湖群体聚为一支,滆湖和长荡湖群体聚为一支,最后与太湖群体聚在一起。
由AMOVA结果(表4)可知,6个群体间的遗传变异为8.36%,群体内变异为91.64%,整体遗传分化系数Fst值为0.081 68(P<0.01),分化达中等水平。将6个群体分为长江水系组(太湖、滆湖和长荡湖群体)和淮河水系组(高邮湖、白马湖和骆马湖)进行分子方差分析,结果显示,组间遗传变异为12.04%,组内群体间变异为0.77%,群体内变异为87.19%,组间的遗传分化指数Fct值为0.120 39(P<0.01),达到中等分化水平。
两两群体间的遗传分化指数Fst值为 -0.004 27~0.155 29,Fst值统计结果(表3)表明,长江水系和淮河水系群体内的Fst值为 -0.004 27~0.023 37(P>0.05) 均小于0.05,表明群体间遗传分化较弱;2个水系群体间的Fst值为0.060 83~0.155 29(P<0.01),表明群體间具有极显著的中等水平遗传分化。
2.4 群体历史动态
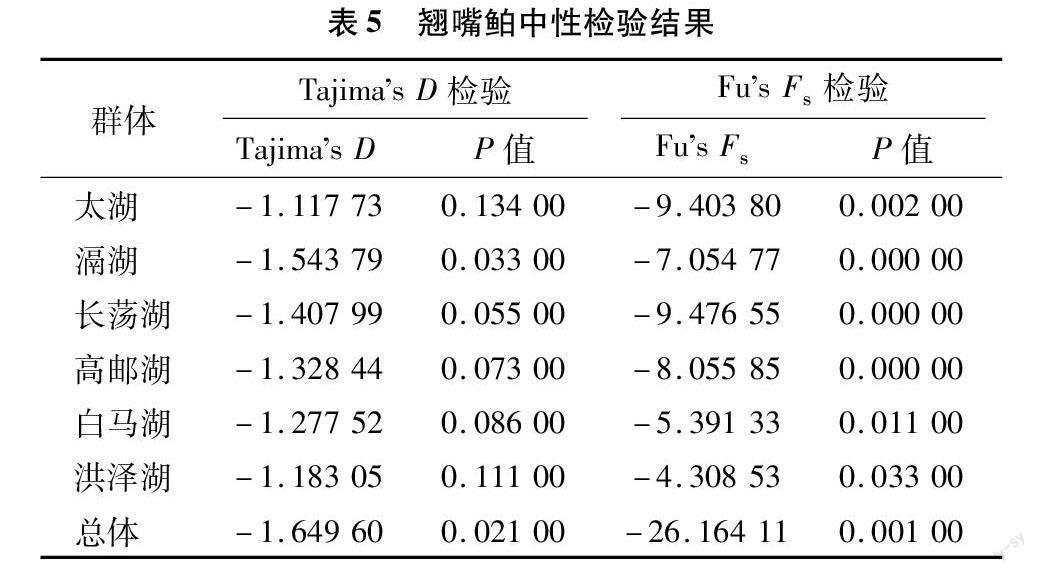
翘嘴鲌群体的Tajimas D和Fus Fs中性检验结果(表5)显示,6个群体的Tajimas D为 -1.543 79~-1.117 73,但仅有滆湖群体的统计具有显著性差异(P<0.05);6个群体的Fus Fs为-9.476 55~-4.308 53,统计均具有显著性差异(P<0.05)。将6个群体作为一个整体进行Tajimas D和Fus Fs中性检验,结果显示Tajimas D和Fus Fs均为负值,统计具有极显著性差异(P<0.01),且核苷酸歧点分布曲线具有典型的单峰模式,表明翘嘴鲌群体在历史上经历了显著的种群扩张过程。
3 讨论
3.1 翘嘴鲌群体遗传多样性
遗传多样性是种群生存和进化的基础,遗传多样性越丰富,种群生存、繁衍和扩张的潜力越大,对环境变化的抵抗和适应能力就越强。单倍型多样性(Hd)和核苷酸多样性(Pi)是评价种群遗传多样性的重要指标,其值越大,则种群的遗传多样性就越高[20]。相比较而言,核苷酸多样性考虑各种单倍型在群体中所占的比例,反映的群体多态性程度更为精确[21]。本研究通过Cytb基因探究了6个湖泊的翘嘴鲌群体遗传多样性,结果显示,整个翘嘴鲌群体的单倍型多样性和核苷酸多样性分别为0.916±0.010和0.002 56±0.000 08;6个群体的单倍型多样性为(0.798±0.059)~(0.927±0.019),核苷酸多样性为(0.002 04±0.000 27)~(0.002 85±0.000 18)。
由此可知,6个湖泊的翘嘴鲌群体遗传多样性有较大差异,其中,太湖群体的遗传多样性最高,高邮湖群体的遗传多样性最低,这可能与湖泊的生态环境、捕捞强度及渔业管理模式等多种因素有关[22-23]。根据GRANT等划分的遗传多样性类型(高Hd高Pi、高Hd低Pi、低Hd高Pi和低Hd低Pi)[24],翘嘴鲌群体的遗传多样性属于高Hd和低Pi类型,提示翘嘴鲌种质资源遗传多样性較低。近几十年来,受过度捕捞、环境污染、涉水工程、非法采砂等不利因素的影响,导致湖泊生态环境质量下降和栖息生境缩减,翘嘴鲌资源量下降和遗传多样性丧失。已有很多研究显示,我国的野生翘嘴鲌种质资源均具有高Hd低Pi现象[15-18,25-27],说明翘嘴鲌野生种质资源遗传多样性普遍较低,急需采取措施恢复翘嘴鲌资源量及提高其遗传多样性。
鱼类的遗传多样性模式与进化历史密切相关。当Hd≥0.5、Pi<0.005时,通常认为鱼类是受瓶颈效应后种群数量的迅速扩张而导致,因核苷酸多样性的积累时间比单倍型多样性的积累时间更漫长得多,从而形成高Hd、低Pi模式[24]。中性检验和核苷酸歧点分布是评估群体历史动态的常用方法,若Fus Fs和Tajimas D检验结果为负值且具有显著性差异,歧点分布曲线为单峰型,说明DNA序列中含有比中性进化模型更多的核苷酸位点变化,可能预示种群经历过一个扩张的历史[28-29]。从整体观察(表5、图5)可知,翘嘴鲌群体的Fus Fs和Tajimas D检验均为负值,且具有显著性差异,核苷酸歧点分布曲线呈现典型的单峰型,表明翘嘴鲌在历史上经历过显著的群体扩张过程,从而形成了高Hd、低Pi的遗传模式,这与已有研究结果[18,27]一致。
3.2 翘嘴鲌群体遗传结构
群体遗传结构不仅可用于评价物种群体的变异水平和不同地理群体之间的关系,还可确定群体中的进化显著单元和管理单元,以及制定资源的保护和管理策略[30]。通常用遗传分化系数Fst来反映群体间遗传分化程度,若Fst值<0.05,表示各群体间不存在分化;若Fst值为0.05~0.15,表示各群体间存在中度分化;若Fst值为>0.15~0.25,表示各群体间存在高度分化[31]。总体来看,6个翘嘴鲌群体的Fst值为0.083 62(P<0.01),表明翘嘴鲌群体间具有极显著的中度遗传分化。群体间的Fst值显示,长江水系和淮河水系内各群体间没有显著遗传差异,而长江水系和淮河水系群体之间有极显著的遗传分化,6个群体的UPGMA聚类图也支持此结果。一般来说,鱼类在不同水系之间存在明显的种群分化,同一流域内的种群通常分化不明显或没有遗传分化[32-33]。翘嘴鲌的遗传分布格局可能与群体所属水系及地理位置等因素有关。从所属水系来看,太湖、滆湖及长荡湖属于长江水系,而高邮湖、白马湖和洪泽湖属于淮河水系;从地理位置来看,太湖、滆湖及长荡湖位于长江以南,高邮湖、白马湖和洪泽湖位于长江以北,水系内各群体的地理位置相对较近,而水系间各群体的地理位置相对较远。另外,修闸建坝及湖泊的生态环境差异也会导致江淮水系群体间基因交流通道受阻而产生遗传差异。从单倍型组成可看出,长江、淮河水系内各群体间拥有较多的共享单倍型,而2个水系群体间拥有的共享单倍型较少,也说明江淮水系群体间的基因交流较弱。已有研究显示,我国东部6个湖泊的翘嘴鲌群体间也有显著的遗传分化[18],本研究结果与之一致。根据翘嘴鲌群体的遗传结构,应将长江水系群体和淮河水系群体划分为不同的管理单元进行保护。
3.3 翘嘴鲌种质资源管理和保护
本研究采用线粒体Cytb基因对江淮下游6个翘嘴鲌群体的遗传多样性和遗传结构进行分析,研究结果显示,长江水系和淮河水系翘嘴鲌群体间有显著的遗传分化,因此应将长江水系和淮河水系的翘嘴鲌群体划分为不同的遗传单元进行管理和保护;翘嘴鲌群体遗传多样性具有高单倍型多样性和低核苷酸多样性模式,提示翘嘴鲌种质资源遗传多样性较低,需要采取措施恢复翘嘴鲌种群数量及提高其遗传多样性水平。根据国家禁渔政策及翘嘴鲌生物学特征,可采取以下措施:(1)强化渔业资源管理,加大执法力度,严厉打击非法捕捞行为,禁止电毒炸等作业方式;(2)控制环境污染,开展湖泊生态环境治理,恢复水生植被,构建人工鱼巢,为翘嘴鲌生存和繁殖提供良好的生态环境;(3)科学规范开展翘嘴鲌增殖放流,提高翘嘴鲌成活率,开展放流群体和野生群体的遗传评估,跟踪监测翘嘴鲌资源变化,逐步提高翘嘴鲌遗传多样性。
参考文献:
[1]乐佩琦,罗云林. 鲌亚科类系统发育初探(鲤形目:鲤科)[J]. 水生生物学报,1996,20(2):182-185.
[2]覃 亮,熊邦喜,王基松,等. 鲌属鱼类在天然水域中的生态功能及资源增殖对策[J]. 湖北农业科学,2009,48(1):233-236.
[3]徐慧东,苗畅齐,韩 英. 兴凯湖翘嘴鲌的生物学研究与养殖概况[J]. 水产学杂志,2014,27(5):59-64.
[4]黄艳飞,段国旗,彭林平. 翘嘴鲌的资源现状和生物学特征综述[J]. 安徽农业科学,2019,47(19):10-13.
[5]凌去非,谭夕东,许爱国. 澄湖翘嘴鲌的生长与资源保护[J]. 水利渔业,2006,27(4):30-32.
[6]程 鹏,樊启学,陈年林,等. 赤东湖翘嘴鲌人工繁殖和苗种培育技术研究[J]. 水利渔业,2007,28(1):22-23.
[7]呂帅帅,管卫兵,何文辉. 太湖翘嘴鲌的生物学特性和条件状况研究[J]. 江苏农业科学,2013,41(5):196-199.
[8]吕大伟,周彦锋,葛 优,等. 淀山湖翘嘴鲌的年龄结构与生长特性[J]. 水生生物学报,2018,42(4):762-769.
[9]丁德明,陈杏华,伍远安,等. 不同养殖模式翘嘴鲌肌肉营养品质比较[J]. 中国饲料,2021(1):89-95.
[10]李国庆,伍育源,秦志峰,等. 鱼类遗传多样性研究[J]. 水产科学,2004,23(8):42-44.
[11]张亚平,施立明. 动物线粒体DNA多态性的研究概况[J]. 动物学研究,1992,13(3):289-298.
[12]蓝昭军,李 强,舒 琥,等. 基于线粒体细胞色素b基因的光倒刺鲃遗传多样性与遗传结构研究[J]. 海洋与湖沼,2016,47(6):1158-1165.
[13]李大命,唐晟凯,刘燕山,等. 基于Cytb基因的江苏省湖鲚种群遗传多样性和遗传结构分析[J]. 中国农学通报,2021,37(20):144-151.
[14]李大命,唐晟凯,刘燕山,等. 江苏省4个太湖新银鱼种群遗传多样性和遗传结构分析[J]. 渔业科学进展,2020,41(5):52-60.
[15]杨子拓. 珠江流域三种鲌亚科鱼类的遗传结构及遗传多样性分析[D]. 广州:华南农业大学,2016.
[16]黄小彧. 长江水系翘嘴鲌遗传多样性研究[D]. 广州:暨南大学,2012.
[17]王 伟,陈立侨,禹 娜,等. 应用COⅡ基因部分序列分析翘嘴鲌群体的遗传多样性[J]. 大连水产学院学报,2008,23(5):403-408.
[18]伊西庆. 中国东部6个大型湖泊翘嘴鲌(Culter alburnus)遗传多样性的线粒体ND2基因序列分析[D]. 广州:暨南大学,2009.
[19]Xiao W H,Zhang Y P,Liu H Z. Molecular systematics of Xenocyprinae (Teleostei:Cyprinidae):taxonomy,biogeography,and coevolution of a special group restricted in East Asia[J]. Molecular Phylogenetics and Evolution,2001,18(2):163-173.
[20]李文静,王环珊,刘焕章,等. 赤水河半的遗传多样性和种群历史动态分析[J]. 水生生物学报,2018,42(1):106-113.
[21]王 丹,程庆武,杨镇宇,等. 三峡库区鲌属鱼类线粒体 COⅠ基因遗传多样性的初步分析[J]. 水生生物学报,2015,39(5):1054-1058.
[22]李大命,唐晟凯,刘燕山,等. 基于Cytb基因的江苏省大银鱼种群遗传多样性和遗传结构分析[J]. 上海海洋大学学报,2021,30(3):416-425.
[23]李大命,唐晟凯,刘燕山,等. 基于线粒体COⅠ基因序列的江苏省4个太湖新银鱼群体遗传多样性分析[J]. 海洋渔业,2020,42(3):277-286.
[24]Grant W,Bowen B. Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,1998,89(5):415-426.
[25]Qi P Z,Guo B Y,Xie C X,et al. Assessing the genetic diversity and population structure of Culter alburnus in China based on mitochondrial 16S rRNA and COⅠ gene sequences[J]. Biochemical Systematics and Ecology,2013,50:390-396.
[26]Sun N,Zhu D M,Li Q,et al. Genetic diversity analysis of Topmouth Culter (Culter alburnus) based on microsatellites and D-loop sequences[J]. Environmental Biology of Fishes,2021,104(3):213-228.
[27]李大命,刘 洋,唐晟凯,等. 基于Cytb基因的滆湖鲌类国家级水产种质资源保护区3种鲌鱼的遗传多样性分析[J]. 水产科技情报,2022,49(1):1-7.
[28]Fu Y X. Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection[J]. Genetics,1997,147(2):915-925.
[29]Márcia Barbosa A,Real R,Muoz A R,et al. New measures for assessing model equilibrium and prediction mismatch in species distribution models[J]. Diversity and Distributions,2013,19(10):1333-1338.
[30]OBrien S J. A role for molecular genetics in biological conservation[J]. Proceedings of the National Academy of Sciences of the United States of America,1994,91(13):5748-5755.
[31]Balloux F,Lugon-Moulin N.The estimation of population differentiation with microsatellite markers[J]. Molecular Ecology,2002,11(2):155-165.
[32]Perdices A,Sayanda D,Coelho M M.Mitochondrial diversity of Opsariichthys bidens (Teleostei,Cyprinidae) in three Chinese drainages[J]. Molecular Phylogenetics and Evolution,2005,37(3):920-927.
[33]Xia Y Z,Chen Y Y,Sheng Y. Phylogeographic structure of lenok (Brachymystax lenok Pallas) (Salmoninae,Salmonidae) populations in water systems of Eastern China,inferred from mitochondrial DNA sequences[J]. Zoological Studies,2006,45(2):190-200.
