基于GBS测序的古蔺野生茶树遗传多样性与群体遗传结构分析
2023-08-22王小萍李明红熊元元唐晓波李春华
王小萍,刘 飞,李明红,张 厅,赖 谦,刘 晓,熊元元,唐晓波,李春华,王 云
(1.四川省农业科学院茶叶研究所,成都 610066;2.泸州市经济作物站,四川 泸州 646000;3.古蔺县农业农村局,四川 古蔺 646500)
【研究意义】茶树属多年生异花授粉经济作物,起源于我国云贵高原,栽培历史悠久,主要种植于我国华南、西南、江南以及江北茶区。四川盆地位于我国西南腹地,位于26°03′~34°19′ N,97°21′~108°31′ E,属西南茶区,地势西高东低,气候、生态类型复杂多样,造就了区域内丰富的茶树种质资源,包括地方品种、有性群体种、野生茶树等。四川省野生茶树主要分布在金沙江沿岸以及龙门、邛崃山脉一带[1]。野生茶树具有品质特异、抗性强的特点,是茶树育种重要的物质基础,因此研究野生茶树的演化进程、群体遗传结构,明确野生茶树的遗传多样性和亲缘关系,对优良茶树品种的选育具有重要意义。【前人研究进展】单核苷酸多态性(SNP)是基于简化基因组测序(GBS)技术发展起来的第3代最具优势的分子标记技术,具有简单、快速、特异性强、稳定遗传、便于检测等优点[2]。张鹏等[3]利用SNP芯片技术研究107份云南玉米自交系,发现云南地区玉米种质资源遗传基础丰富,可创建新的具有良好应用潜力的杂交优势群。孙倩等[4]采用SNP和Indel标记揭示了巴西木薯的遗传多样性。周新桐等[5]利用GBS测序技术构建了甘蓝型油菜连锁图谱,并对油菜粉色花性状进行了QTL定位。Hou等[6]基于GBS测序技术对信阳蓝壳鸡面部色素性状进行了全基因组关联分析,定位到10个与面部色素性状紧密关联的SNP标记。周艳春等[7]利用SNP标记对93份无芒雀麦种质资源产量性状进行全基因组关联分析,筛选出33个核心SNP(P<0.001),有利于缩短无芒雀麦的育种进程。由此可见,GBS测序技术研究结果可靠,已广泛应用于动植物遗传多样性分析、群体遗传结构分析、QTL定位、遗传图谱构建以及全基因组关联分析等研究领域,且研究结果可靠。【本研究切入点】古蔺县位于四川省南部边缘,位于27°41′~28°20′ N,105′34′~106°20′ E,与贵州毕节县、赤水县相邻。前期调查发现,古蔺县蕴含丰富的野生茶树,多以乔木、小乔木为主,树径多大于0.3 m,最大可达2.0 m,且这些野生茶树具有较强的抗性或优异的品质性状,是茶树新品种选育的宝贵资源。【拟解决的关键问题】为分析古蔺当地野生茶树的遗传多样性与群体遗传结构,揭示其遗传分化特征,本研究以65份野生茶树为研究对象,利用GBS测序技术,基于获得的SNP分子标记研究参试茶树的遗传多样性和群体遗传结构,以期为四川省茶树种质创新、优良品种选育等提供理论依据,同时也为今后优良性状基因深度挖掘提供参考。
1 材料与方法
1.1 试验材料
供试野生茶树共65份(表1),其中古蔺野生茶树49份(以G1、G2、G3......表示)、叙永野生茶树5份(以X1、X2、X3......表示)、合江野生茶树1份(以H1表示)、崇州枇杷茶4份(以CP1、CP2、CP3......表示)、雷波野生茶4份(以LB1、LB2、LB3......表示)、云南野生茶树2份(以YD1、YD2表示)。于2019年9月取参试材料新鲜成熟叶片,-80 ℃液氮冷冻保存用于GBS测序。
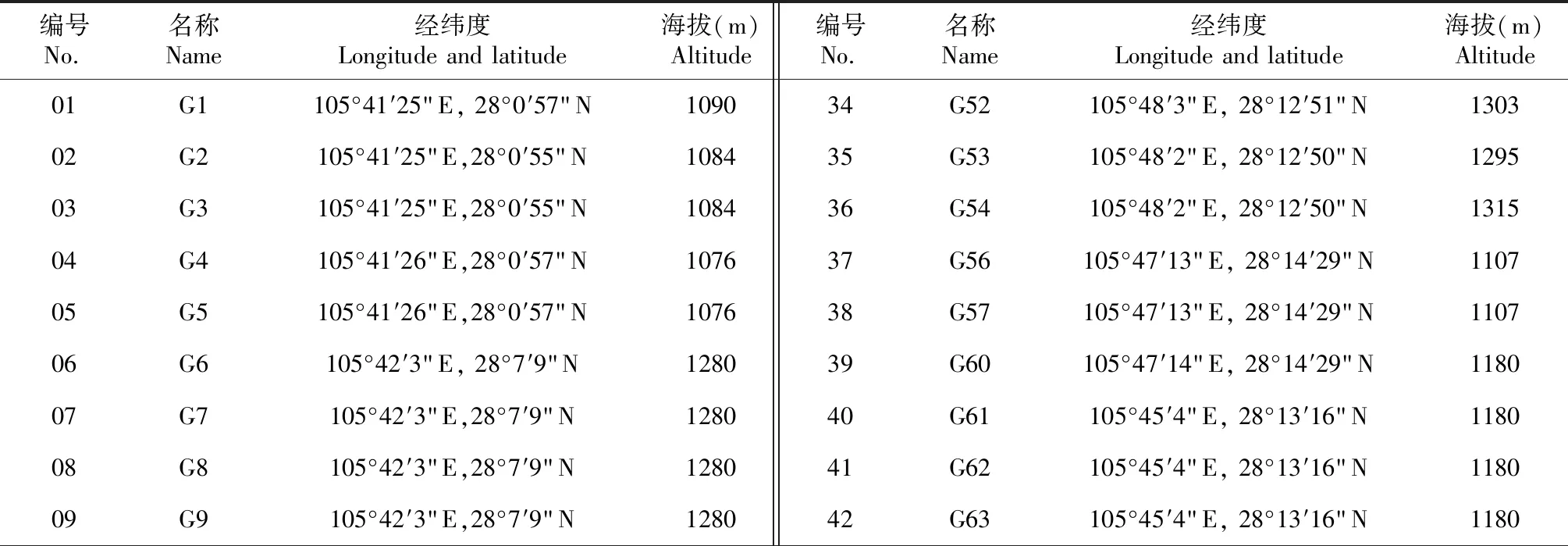
表1 65份野生茶树资源
1.2 茶树基因组DNA提取及建库测序
采用改良CTAB法提取茶树基因组DNA,使用Qubit和Nanodrop检测DNA质量。质检合格的基因组DNA由基迪奥生物科技公司进行30×的GBS测序,利用Novaseq6000测序仪平台,按照GBS实验流程,对基因组DNA进行限制性内切酶酶切,加上带barcode的测序接头,对样本进行混合,构建小片段文库(250~550 bp),采用PE125双末端测序。
1.3 SNP与InDel标记检测
用 FASTP (版本 0.18.0)[8]对 Illumina 平台的原始数据进行过滤,过滤标准如下:①去除含有未知核苷酸(N)≥10%的 reads;②去除低质量reads(质量值Q≤10 的碱基数占整条read的50%以上);③去除含接头的reads。过滤后的clean reads 用于组装分析。使用比对软件 BWA(v0.7.12)[9]采用mem算法将过滤后的reads 比对到参考基因组上,比对参数为-k32-M;比对结束后使用软件 picard(版本 1.129)对结果进行标记,并使用变异检测软件 GATK(版本 3.4-46)[101]进行群体变异检测,利用ANNOVAR(版本2)[11]对检测出的变异进行功能注释。
1.4 野生大树茶遗传多样性与遗传结构分析
采用POPGENE软件计算各地区茶树遗传多样性参数:多态性位点数(PLN)、多态位点百分比(PPL)、有效等位基因数(Ae)、观测等位基因数(Na)、最小等位基因频率(Maf)、观测杂合度(Ho)、期望杂合度(He)以及多态信息含量(PIC)和Shannon’s指数;利用VCFtools软件[12]计算各居群的群体分化指数(Fst)。采用Plink和Admixture软件[13]进行群体结构分析,估算出最佳的群体亚群数。
根据所得到的SNP信息,计算样品间距离,对样品进行聚类分析,利用treebest软件[14]通过邻接法(Neighbor-joining methods)构建SNP进化树。并基于个体间的SNP差异程度,利用R语言包(http://www.r-project.org/)进行主成分分析,根据各个样本在第一主成分(PC1)和第二主成分(PC2)两个综合指标中的数值大小,绘制二维坐标图。
2 结果与分析
2.1 野生茶树基因组测序结果与SNP注释
对65份野生茶树基因组DNA进行分析,构建GBS文库及酶切测序,共获得77.30 G数据,过滤后获得74.48 G数据,540 633 846条高质量reads,平均每份茶树资源获得1.15 G数据,8317 444条高质量reads。65份野生茶树Q20≥93.30%,Q30≥91.84%,表明测序质量较好,GC含量在40.02%~41.71%,分布正常。将高质量reads比对到茶树参考基因组上,65份野生茶树共获得799 217个变异,其中769 893个SNPs,29 324个Indels。
利用 ANNOVAR软件对769 893个高质量的SNPs进行检测(图1-a),692 570个SNPs位于基因间区(Intergenic region),占比89.96%;31 173个SNPs位于内含子区(Intronic region),占比4.05%;19 754个SNPs位于外显子区(Exonic region),占比2.57%;8994个SNPs位于转录终止位点下游区域(Downstream region),占比1.17%;7703个SNPs位于转录终止位点上游区域(Upstream region),占比1.0%;1801个SNPs位于3′端UTR变异(UTR3),占比0.23%;949个SNPs位于5′端UTR变异(UTR5),占比0.12%;812个SNPs位于转录终止位点(Downstream; Upstream),占比0.11%;137个SNPs位于可变剪切位点(Splicing region),占比0.02%。
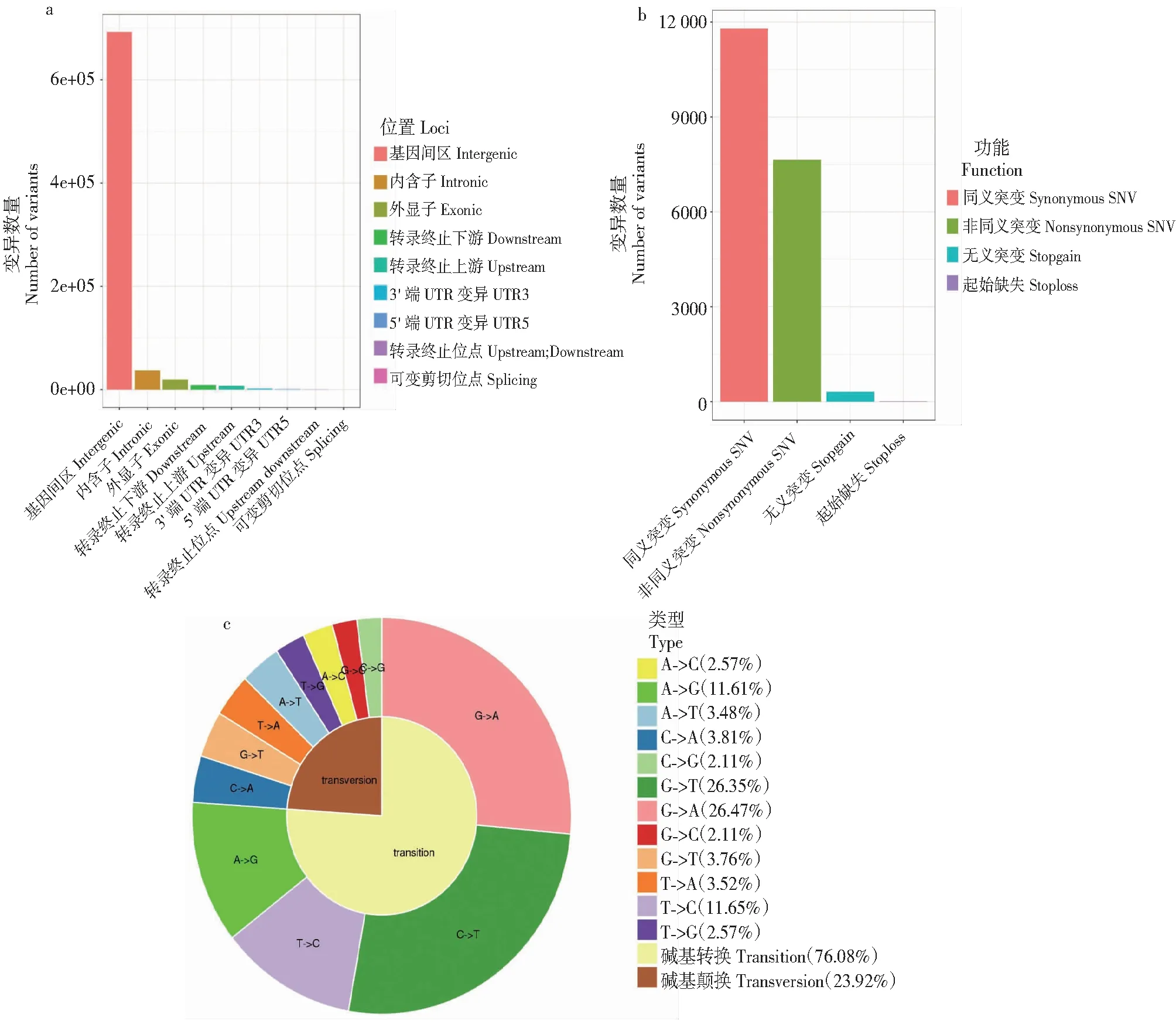
a:SNPs位置;b:SNPs功能;c:SNPs变异类型。a: Location of SNPs;b:Function of SNPs;c:Types of SNPs.图1 SNPS注释结果Fig.1 The results of SNP annotation
对编码区SNPs进行功能注释(图1-b),其中11 791个SNPs属非同义突变(Nonsynonymous),7640个SNPs属同义突变(Synonymous),311个SNPs属无义突变(Stop gain),12个SNPs属起始缺失(Stop loss)。
对SNP变异类型进行注释(图1-c),76.08%的SNPs属于碱基转换(Transition,ts),其中G->A占比26.47%,C->T占比26.53%,T->C占比11.65%,A->G占比11.61%;23.92%的SNPs属于碱基颠换(Transversion,tv),其中A->C与A->T分别占比2.57%与3.48%,G->C与G->T分别占比2.11%与3.76%,C->A与C->G分别占比3.81%与2.11%,T->A与T->G则分别占比3.52%与2.57%。表明SNP变异类型以碱基转换为主,且ts/tv值为3.18,表明SNP变异类型多以同类型核苷酸之间的变异为主。
2.2 野生茶树群体遗传多样性与遗传结构分析
从表2可知,6个群体平均有效等位基因数(Ne/Ae)为1.1284个,平均多态性位点数(PLN)为9475 456,最高为Gulin群体(2828 344),多态性位点比例(PPL)为57.33%,最低为合江野生茶树(172 911),多态性位点比例为8.53%;各群体观测等位基因数(Na)分别在0.4267~0.9147,平均为0.7677;最小等位基因频率(Maf)介于0.1514~0.5000,均值为0.3213;基因多样性指数(Nei)最高是古蔺野生茶树(0.1328),最低是云南野生茶树(0.0632),均值为0.0979;各群体观测杂合度(Ho)与期望杂合度(He)仅古蔺地区变化范围稍大,为0.0636~0.1233,其余变化范围较小;多态性信息含量(PIC)则介于0.0312~0.1025,均值为0.0596;香农指数(Shi)最高的为古蔺地区(0.199),最低的为云南地区(0.0576),均值为0.1122。

表2 参试野生茶树群体遗传多样性
从表3可知,不同地区野生茶树群体间遗传分化指数Fst变化范围为-0.7042~0.4372,云南野生茶树与崇州枇杷茶、古蔺野生茶树、雷波野生茶树、叙永野生茶树和合江野生茶树群体间遗传分化指数分别是0.3898、0.3375、0.3673、0.4372和0.3094,表明群体间存在非常强的遗传分化,其中与叙永群体间遗传分化最高,为0.4372;合江野生茶树与崇州枇杷茶、古蔺野生茶树、雷波野生茶树、叙永野生茶树种群间遗传分化均为负数,表明这些群体间存在普遍的基因交流,遗传亲缘关系较近,其中与古蔺群体遗传分化值最小,为-0.7042;古蔺野生茶树与崇州枇杷茶、雷波野生茶树、叙永野生茶树种群间遗传分化指数分别为0.04373、0.11740和-0.03481,表明古蔺群体与崇州枇杷茶群体间存在遗传分化,但分化较弱,与雷波群体间存在中等程度的遗传分化,而与叙永群体间则存在基因交流。
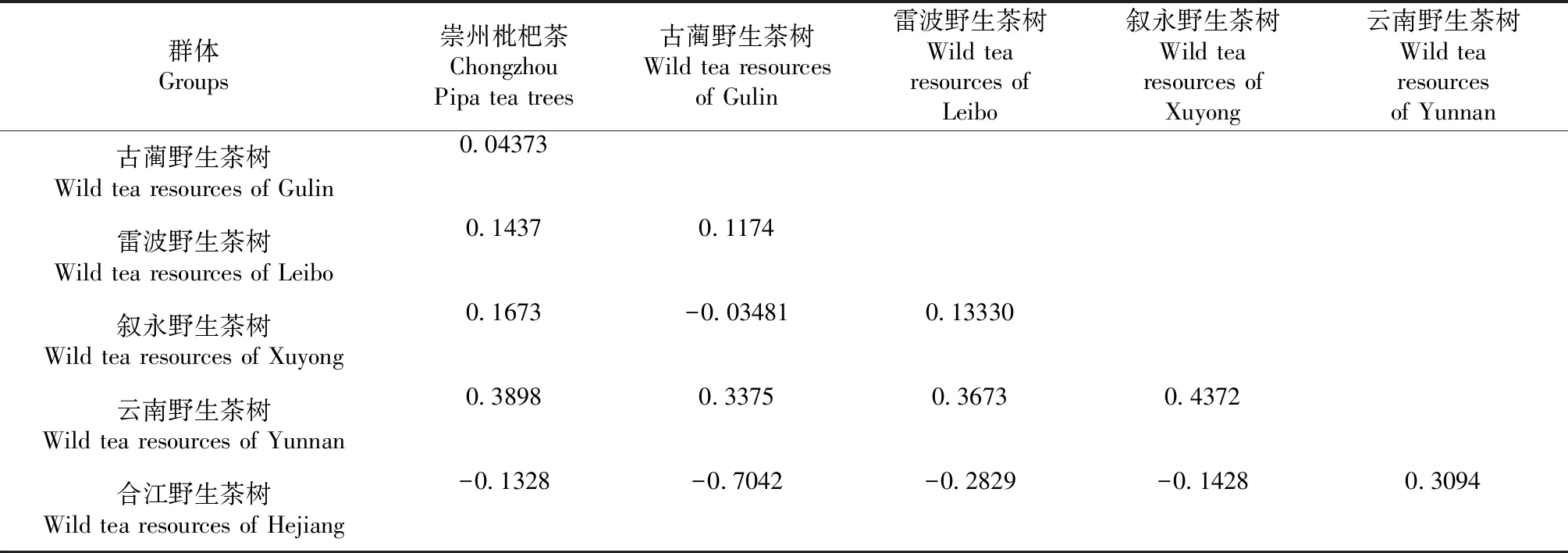
表3 野生茶树群体遗传分化指数(Fst)
利用769 893个高质量的SNPs变异,采用Plink和Admixture软件对65份野生茶树进行群体结构分析。将亚群数K值范围设定为2~9,计算不同K值下的交叉验证错误率(Cross-validation error,CV error),由图2-a可知,当K值为2时,CV值最小,因此可将参试的65份茶树种质分成2个亚群,再根据Q值的大小,将野生茶树划分到最大Q值所在的亚群(图2-b)。2个亚群中CP4、G19、LB5、YD1、YD2共5份野生茶树归为亚群Ⅰ(红色),其余60份野生茶树归为亚群Ⅱ(蓝色)。
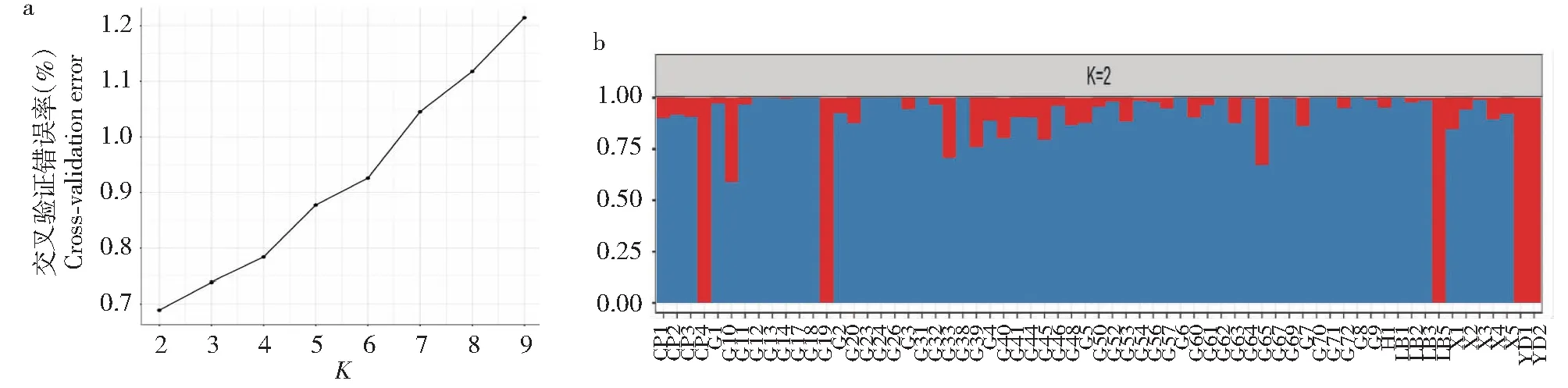
a:计算K为2~9时的CV error。b:K=2时的群体结构,在该群体结构中,红、蓝分别代表亚群1~2,根据颜色占比推断该野生茶树属于哪个亚群。a:The cross-validation error was calculated when K was 2 to 9.b:The Population structure at K=2,in this Population structure,red,blue represented subgroups 1 to 2,and the subgroup to which the individual belonged was inferred based on the proportion of the color.图2 65份野生茶树群体遗传结构Fig.2 Population genetic structure of 65 wild tea resources
2.3 野生茶树的SNP系统进化分析
利用769 893个高质量SNPs计算样品间距离,并对样品进行聚类分析,从而推断出种群间的亲缘关系远近。利用treebest软件邻接法(Neighbor-joining methods)构建进化树(图3)。65份野生茶树可以明显分为两大群,其中YD2、YD1、CP4、LB5、G19、G10为Ⅰ类群,其余59份为Ⅱ类群,这与遗传结构分析结果存在差异,系统进化分析将G10聚在Ⅰ类群,而在遗传结构分析中G10与大部分野生茶树聚为一类。同时,Ⅱ类群可以分为3个亚群(Ⅱ-a、Ⅱ-b和Ⅱ-c),其中亚群Ⅱ-a仅包含材料G65,Ⅱ-b包含G67、G69、G70、G71和G725 5份野生茶树,均源自相近地理位置,Ⅱ-c则包含其余53份野生茶树。
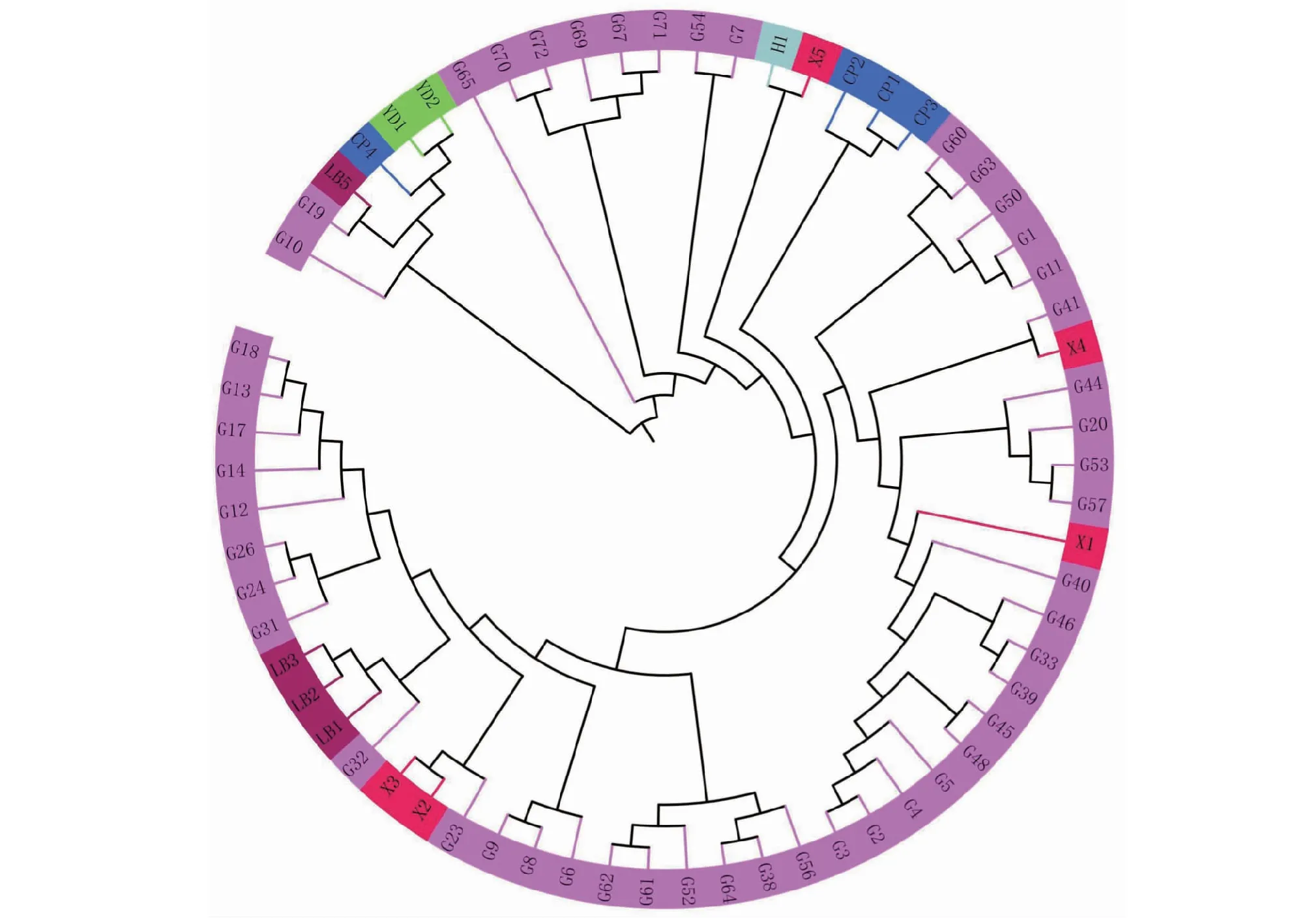
紫色为古蔺野生茶树;红色为叙永野生茶树;暗红色为雷波野生茶树;浅蓝色为合江野生茶树;蓝色为崇州枇杷茶;浅绿色为云南野生茶树。Purple was wild tea resources in Gulin;Red was wild tea resources in Xuyong;Dull red was wild tea resources in Leibo; Light blue was wild tea resources in Hejiang;Blue was Chongzhou Pipa tea; Light green was wild tea resources in Yunnan.图3 65份野生茶树系统发育进化树Fig.3 Phylogenetic tree of the 65 wild tea resources
2.4 PCA主成分分析
基于个体间SNPs差异程度,利用R软件进行主成分分析,分别利用PCA1与PCA2、PCA1与PCA3以及PCA2与PCA3绘制散点图(图4),发现PCA1与PCA2最能解释65份野生茶树的遗传变异情况,PCA1vPCA3和PC2vPC3次之。由图4-a可知,65份野生茶树能明显区分为4个亚群,亚群1包含42份古蔺野生茶树、5份叙永野生茶树、1份合江野生茶树、3份雷波野生茶树以及3份崇州枇杷茶,亚群2包含3份茶树茶树,分别来自古蔺、雷波、崇州,间于云南与大部分古蔺茶树集中点间,亚群3包含6份古蔺野生茶树,分布在Y轴上方,与亚群1大部分古蔺野生茶树集中点距离较远,亚群4则是2份云南野生茶树,和其余茶树区分明显。
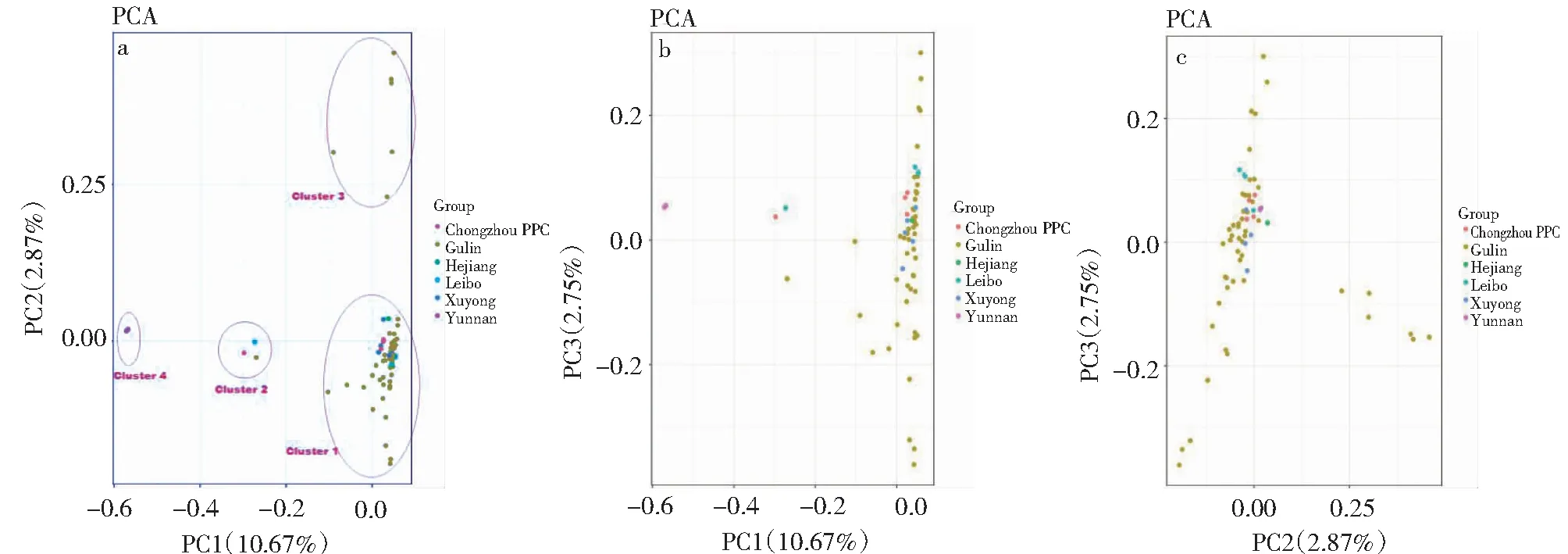
a:PC1vPC2; b:PC1vPC3;c:PC2vPC3.图4 65份野生茶树SNP基因型主成分分析Fig.4 Principal components analysis of SNP genotype of the 65 wild tea resources
3 讨 论
SNP是最常见的一种遗传变异,具有数量多、分布广的特点,通过比较分析种群内个体的SNP位点,可研究种群的遗传多样性、亲缘关系以及群体遗传结构。王丽鸳等[15]研究发现茶树SNP的分布规律,即茶树SNP分布频率高,变异类型主要是G/A与C/T的碱基转换为主。郭燕等[16]利用GBS技术对贵州久安100份古茶树资源开展全基因组简化测序分析,共获得548 597个高质量SNP,其中G/A和C/T的SNP变异类型最多,且ts/tv为2.95。本研究利用GBS技术对65份野生茶树进行简化基因组测序分析,获得769 893个高质量SNPs,变异类型以G/A与C/T为主,这与上述结果一致,但ts/tv为3.18,较上述研究高,表明参试茶树SNP变异类型较丰富。通过对外显子区域(基因编码区)19 754个SNP进行功能注释,SNP导致的非同义突变与同义突变的比例为1.54,表明参试茶树在进化过程中受到自然环境的正选择效应,环境适应能力强,同时这些变异为后续SNP标记的开发、功能基因的研究提供基础。
遗传多样性是作物群体内个体基因水平的变异与环境等多因素共同作用的结果,是物种适应环境、保持遗传潜力的基础,对种群的进化发展具有重要意义[17]。研究表明,雷波野生茶树[18]、云南野生茶树[19-20]、江华苦茶群体种[21]、贵州野生茶树[22]以及广西野生茶树[23]均具有较高的遗传多样性,而本研究中基于获取的SNP对参试茶树的遗传多样性进行分析,结果表明茶树群体的遗传多样性参数与之相比处于较低水平(基因多样性指数Nai=0.0979、期望杂合度 He=0.0745、多态信息含量PIC=0.0596、Shannon信息指数=0.1122)。推测原因可能与供试茶树的数量和种群来源有关,本研究的65份野生茶树源于四川省古蔺县(49份)、合江县(1份)、叙永县(5份)、雷波县(4份)、崇州(枇杷茶,4份)和云南省德宏芒市(2份),其中古蔺、合江和叙永地理位置相邻,气候环境较一致,在文中后续遗传分化研究中也发现这3个地方野生茶树在遗传演化的过程中,存在频繁的基因交流,遗传亲缘关系近,而源于这3个地区野生茶树达55份,占比85%,这可能是导致本研究总体遗传多样性低的原因之一。另外前人研究多选用多态性丰富的SSR标记对茶树遗传多样性进行研究,而多态性丰富的标记获得的等位基因变异数较多,检测到的有效等位基因、杂合度、香农指数等参数也较高[24-25],这可能也是研究获得较高遗传多样性参数的原因。何旭东等[26]利用SLAF-seq测序技术研究舒玛栎遗传多样性时也获得了较低的遗传多样性参数,且遗传多样性指数远低于利用SSR标记的研究结果。
群体结构分层、等位基因分布不均容易导致基因型与表型分析结果假阳性或假阴性,因此对参试群体进行群体结构研究是基因与表型关联分析的基础。本研究采用Plink和Admixture软件对65份野生茶树进行群体结构分析,当K=2时,CV值最小,由此将65份野生茶树分成2个亚群,该结果与系统进化树分析结果基本相符,而与主成分分析存在差异。主成分分析不仅将遗传距离较远的YD1、YD2划分在一起,也将其余川内野生茶树划分为3个亚群,其中亚群2分布介于亚群1与亚群4之间,且划分在亚群2的3份野生茶树在群体结构和系统进化分析中则和YD1、YD2聚在一起,亚群3则是从古蔺野生茶树中划出,因此主成分分析结果是对群体结构和系统进化分析的补充,更好地解释了野生茶树的遗传结构。
我国西南云贵高原一直被认为是茶树的原产地,四川省在地理位置上处于云贵高原的过渡地带。本研究结果显示65份野生茶树资源来自2个血统,四川崇庆、雷波、古蔺3个地区各1份材料与云南材料血统一致,表明四川与云南茶树资源存在演化进程;而17份古蔺地区和1份雷波地区的茶树组成另一血统且和前者区别明显,无任何交集,表明四川省存在原产的野生大茶树,与钟渭基[1]的四川野生大茶树系本地原产一致,也与周斌等[18]的“四川与云南的野生茶树群落相互独立”一致。本研究对四川古蔺、雷波、叙永、合江、崇州5个茶树群体结构研究中,四川纯正血统的野生茶树集中在古蔺地区,其余茶树存在2个血统杂合现象,但主要以四川血统为主,表明古蔺地区存在更原始的茶树血统,其余区域的野生茶树均以此血统为主与另一血统杂交。笔者经过近几年实地考察发现,古蔺地区野生大茶树数量较多,以原桂花乡和黄荆乡为中心广泛分布,据此推测古蔺地区及周边可能是四川野生大茶树的发源中心,这与周斌等[18]的雷波地区可能是川内野生茶树的发源中心观点不一致。因此,针对此观点,需在今后研究中进一步扩大各地区试验样本数进行验证。
4 结 论
65份古蔺野生茶树具有较低的遗传多样性,亲缘关系较近,存在频繁的基因交流;遗传结构分析可将65份野生茶树分为两个亚群。
