基因编辑技术及其在疾病治疗中的研究进展和应用前景
2021-11-06邱志超李卓霏
邱志超,李卓霏,石 宏,2
(1.昆明理工大学 灵长类转化医学研究院,云南 昆明 650500;2.国家卫计委西部孕前优生重点实验室,云南 昆明 650021)
1 基因编辑技术的发展及原理
1866年,Mendel发现遗传定律奠定了现代遗传学的发展,经过科学家150多年的不懈努力,以基因为核心的分子生物学技术已经深入治疗人类疾病的各个方向.根据欧洲罕见病组织近期的一份研究报告显示[1]:在全世界,超过3亿人患有6 000多种已确定罕见遗传病中的一种或多种,且不包括罕见病中其他非遗传性疾病的患者.1972年至2009年,基因治疗理论从首次被提出作为治疗人类遗传疾病的潜在方案[2],到入选Science杂志2009年度十大科学进展之一[3],其为包括罕见病在内的多种疾病治疗带来了新的解决方案.目前,基因治疗方案主要以病毒载体递送外源基因的方式治疗疾病,虽然取得了一定的进展,但由于某些不确定性因素会诱导机体对载体产生免疫应答反应,从而使得基因治疗方案一直存在潜在的其他致病风险.基因编辑技术的出现,为人类削弱这些潜在的致病风险提供了新的思路[4].图1展示的为不同基因编辑技术发展的主要时间线.
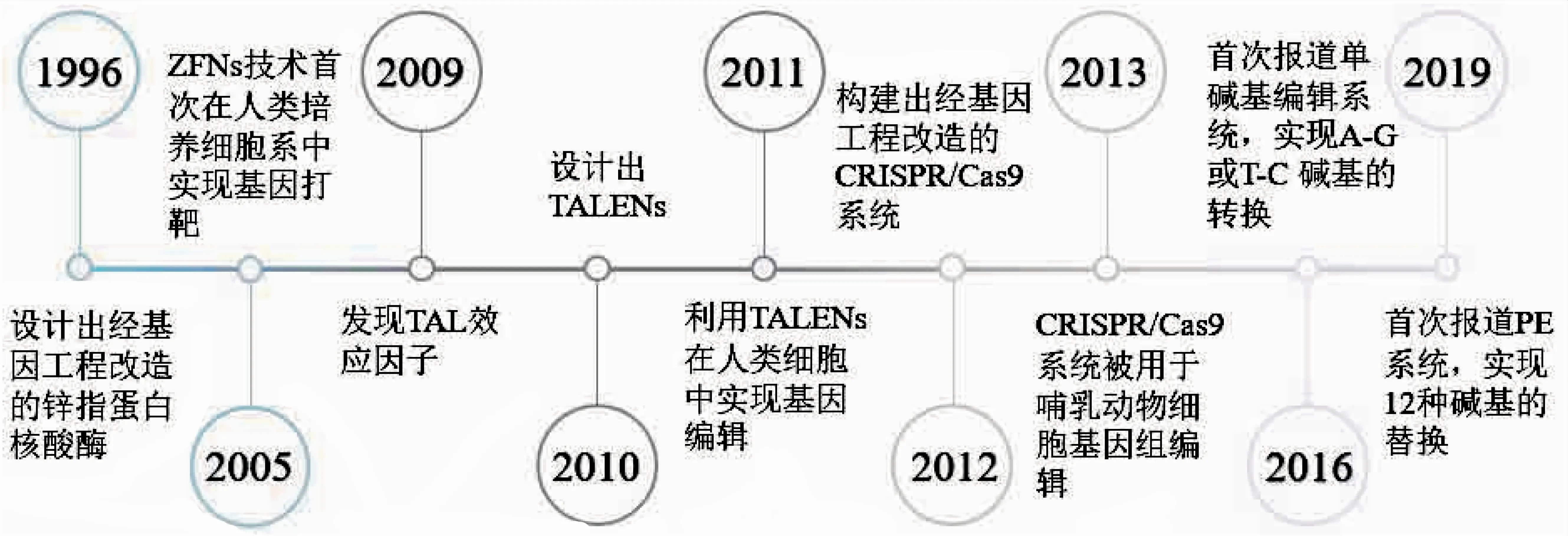
图1 基因编辑的发展时间线Fig.1 Timeline of developments in genome editing
1.1 ZFNs技术
ZFNs由决定其特异性的锌指蛋白结构域和切割DNA的Fok I核酸酶结构域共同组成.1983年,Hanas等[5]通过对非洲爪蟾TFIIIA的研究,率先报道了7S核糖核蛋白中Zn的存在,且介导TFIIIA与5S基因内部控制区域的结合.在此基础上Miller等[6]发现,TFIIIA的5S RNA体外转录过程的正常进行,需要一个40KDa含有重复锌结合域的蛋白质参与,氨基酸序列分析显示其包含9个串联相似的结构单元,每个单元大约有30个氨基酸,且含有Cys2和His2共同围绕Zn形成的ββα折叠的手指状保守结构域,即锌指结构.随后的研究[7-8]证实了α-螺旋的-1、3和6位氨基酸可以识别DNA链3′到5′方向的3个碱基,2位识别5′到3′方向的一个碱基.
基于这一发现,Kim等[9]将3个锌指结构域与1个FokI核酸酶的水解结构域进行连接,构建出了第一个锌指蛋白核酸酶.因FokI核酸酶必须二聚化后才能发挥功能,故Kim等[9]设计了2个ZFNs,当ZFNs与靶位点特异结合形成二聚体后,会导致双链断裂,最终通过非同源末端连接(Non-homologous end joining,NHEJ)或同源定向重组修复(Homology directed recombination repair,HDR)的方式进行修复,从而实现基因编辑的目的.ZFNs基因编辑技术的作用原理如图2(ZFNs)所示,其中Nuclease Dimer为二聚化后的FokI核酸酶.2019年,Sangamo Therapeutics公司[10]通过对一系列蛋白质进行改造,开发出新的ZFNs结构,有望对特定基因组位点进行更加精准编辑.目前,该技术已经在哺乳动物细胞[11-12]、果蝇[13]、斑马鱼[14]、小鼠[15]和植物[16]中得到了应用.由于构建一个特异的锌指蛋白比较费时费力,且ZFNs存在一定的脱靶现象,如何有效地解决这两个问题,也成为ZFNs技术未来主要的研究方向.
1.2 TALENs技术
Bonas等[17]首先在植物病原体黄单胞菌属中发现TALEs蛋白.2009年,两支来自德国和美国的科研团队[18-19]破译了TALE特异性结合DNA序列的机制.TALE蛋白包含一系列高度保守由33~35个氨基酸组成的重复单元,其第12和13位为两个可变氨基酸(repeat-variable diresidue,RVD),RVD可以识别DNA的四种不同碱基,TALEs蛋白即通过RVD特异性结合DNA序列.基于此发现,Christian等[20]将TALEs与二聚化后才能发挥功能的FokI核酸酶融合在一起,构成了TALENs,其具体作用原理如图2(TALENs)所示,其中每个TALEs蛋白能够识别不同碱基序列.TALENs技术已经在哺乳动物ES细胞[21]和植物[22]等生物及动物模型构建[23]的研究中得到了应用.相比于ZFNs技术,TALENs虽然具有构建过程相对简单和细胞毒性较低等优点,但也存在脱靶现象.在疾病治疗中,如何安全且高效地将其表达载体递送至病变细胞中,也是亟待解决的问题.
1.3 CRISPR/Cas9技术
CRISPR广泛存在于原核生物中,由一系列来自噬菌体或质粒DNA的间隔序列区(spacer)和高度保守的重复序列区(repeat)组成[24].1987年,Ishino等[25]首次报道了这一特征序列,此后在其他细菌和古细菌中也发现了类似重复序列[26].2002年,Jansen等[27]将该特征序列命名为CRISPR,并在含CRISPR的原核生物中,发现了4个编码核酸酶或解旋酶的Cas基因.此后,科学家发现CRISPR可以转录生成crRNA(CRISPR RNA),再与Cas蛋白进行结合,可以使宿主获得抵抗外来入侵质粒或噬菌体的能力[28].2011年,Sapranauskas等[29]发现PAM(proto-spacer adjacent motif)基序的存在,也是CRISPR/Cas系统发挥免疫功能重要因素.2012年,Jinek等[30]通过将成熟的crRNA和tracrRNA(trans-activating crRNA)结合在一起,形成的双RNA结构(即single-guide RNA,sgRNA),可以引导来源于Streptococcus pyogenes的cas9蛋白对体外DNA进行精确切割,同时还确定了Cas9蛋白的HNH核酸酶结构域切割与crRNA结合的互补DNA链,RuvC样结构域切割非互补链,其作用原理如图2(CRISPR/Cas9)所示,当sgRNA与特定目标序列结合后,Cas9蛋白会在特定位点处切割双链DNA.
2013年,Cong等[31]和Mali等[32]利用CRISPR/Cas9系统,在体外培养的哺乳动物细胞中,实现了特定基因位点的靶向修饰.此后,CRISPR/Cas9系统在小鼠[33]、斑马鱼[34]、果蝇[35]、植物[36]及动物模型构建[37]等研究中得到了应用.相比于ZFNs和TALENs技术,CRISPR/Cas系统的设计过程更加简便,可以实现多位点同时打靶.针对其存在的脱靶性问题,科学家们也设计了一系列Cas9蛋白突变体(如SaCas9-HF[38]、SpG和SpRY[39]等).近期,Broad研究所Uri Ben-David团队[40]发现Cas9蛋白会促进p53突变富集,具有潜在致癌性,所以对于CRISPR/Cas9基因编辑技术而言,还有很长的路要走.
1.4 BE及PE技术
2016年,David R.Liu团队首先报道了在不需要引入DNA双链断裂和外源供体DNA模板的条件下,就可以对单碱基进行转换的BE技术.其团队通过长度为16个碱基的XTEN接头,将胞嘧啶脱氨酶rAPOBEC1和失去催化活性的dCas9(catalytically-dead Cas9)蛋白连接在一起,组成了第一代胞嘧啶碱基编辑器BE1(rAPOBEC1-XTEN-dCas9),可以在特定窗口内实现C-T或G-A碱基的转换,经过不断的优化,最终开发出了第三代胞嘧啶碱基编辑器BE3(APOBEC-XTEN-dCas9(A840H)-UGI),经在人类细胞中测试,发现BE3比BE2编辑效率提高了2~6倍[41].2017年,David R.Liu团队又开发出了编辑效率更高,Indel出现频率更低的第四代胞嘧啶碱基编辑器BE4,同年,其团队再次报道了由腺嘌呤脱氨酶和nCas9组成的ABE系统,其可实现A-G或T-C碱基的转换[42].Base editing基因编辑技术作用原理如图2(Base editing)所示,当dCas9蛋白与胞苷脱氨酶结合时,会在特定编辑窗口内实现C-T碱基的转换.自单碱基编辑系统开发以来,已经广泛的应用于各种动物[43]和植物[44]中,并且也在人类疾病研究和育种等领域得到应用[45].
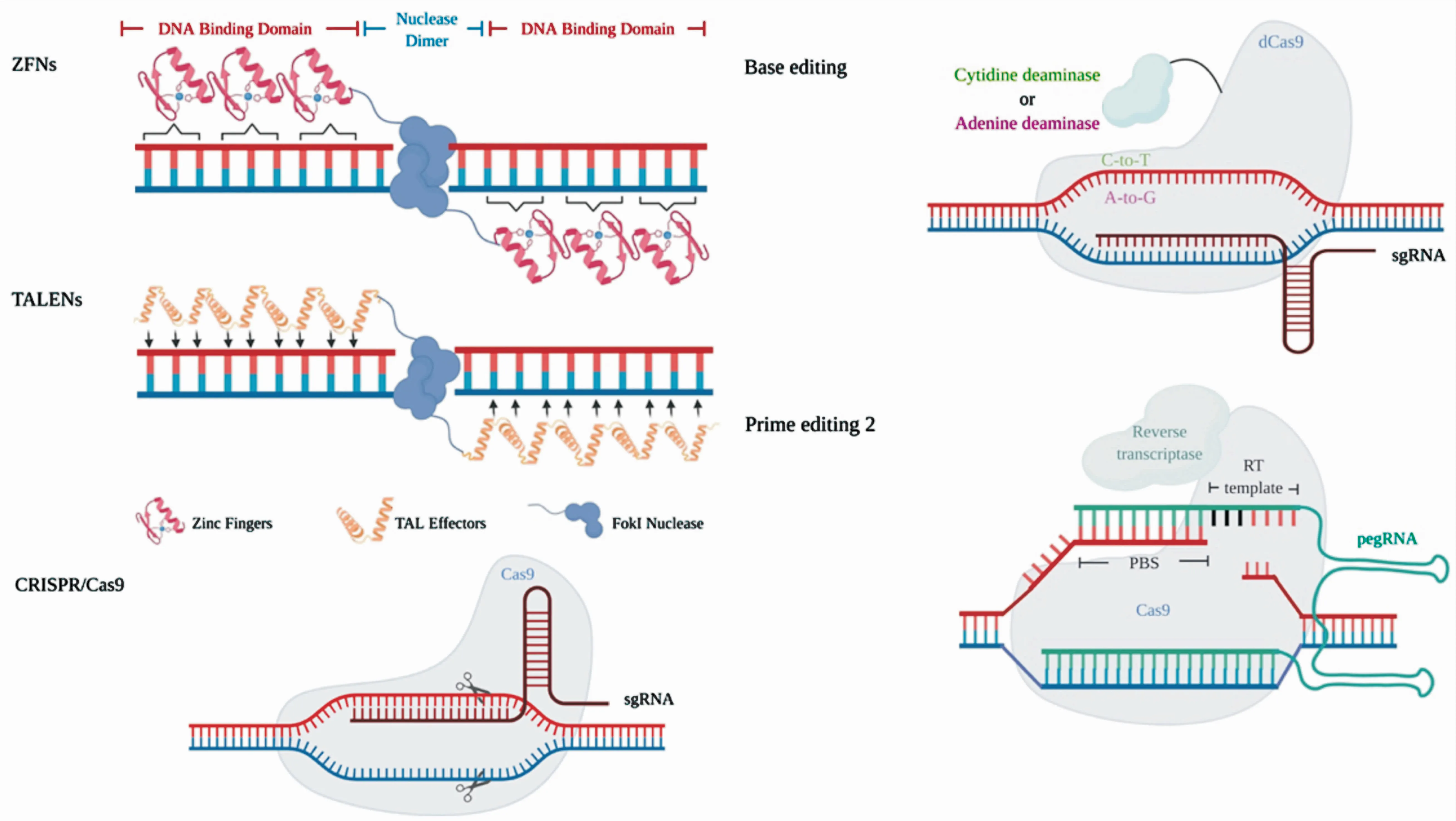
图2 不同基因编辑技术的作用原理Fig.2 Principles of different gene editing technologies
围绕着单碱基编辑系统带来的脱靶性问题,科学家们也做了许多不同的研究,2017年,Kim等[46]通过对其之前报道的Digenome-seq(digested-genome sequencing)CRISPR/Cas9脱靶检测方法加以修改,使其可以检测单碱基编辑系统的脱靶情况,结果显示BE3的脱靶率很低,且脱靶位点有别于Cas9.但是,2019年杨辉团队[47]等与高彩霞团队[48]在《Science》杂志上同期发表的两篇文章,发现BE3系统在小鼠胚胎和水稻中会出现严重的脱靶现象,而CRISPR/Cas9和ABE系统则不会出现明显的脱靶效应,同时杨辉团队也建立了一种名叫GOTI(Genomewide Off-target analysis by Two-cell embryo Injection)的脱靶检测技术.此后,借助这一脱靶检测技术,杨辉团队[49]等开发出新型单碱基编辑工具YE1-BE3-FNLS,可显著降低基因编辑脱靶效应.同年10月,David R.Liu团队报道了一种全新的基因编辑工具PE(prime editor),该技术的原理是通过将Cas9 H840A切口酶和逆转录酶偶连在一起,接着在一种含有一段 RNA 序列的 pegRNA(prime editing guide RNA)引导下,靶向结合并切开一条DNA链,逆转录生成的单链DNA会在该切口处与原始序列展开竞争,在经过优化后的PE3中,逆转录生成的单链DNA有较高概率通过细胞自我修复机制整合到基因组中,而原始序列会通过细胞的修复机制去除掉[50].其中该技术不仅可以在靠近或远离PAM位点的位置进行基因编辑,而且还可以在带来更少副产物的前提下,实现12种可能的碱基替换,该基因编辑技术的简要作用原理如图2(Prime editing 2)所示.近期,高彩霞团队与David R.Liu团队合作,成功建立并优化了适用于植物的PPE(plant prime editing)编辑系统,并在水稻和小麦基因组中实现了碱基的精准增添、替换或删除[51].从理论上来说,PE技术可以避免一些脱靶性问题,但这有待于后续的工作进行验证.表1展示的为不同基因编辑技术的优势及其局限性.

表1 不同基因编辑技术的比较Tab.1 Comparison of different gene editing technologies
2 基因编辑技术在遗传性疾病治疗中的应用
遗传性疾病是指由一个或多个致病基因所控制,或因染色体缺陷导致的一种疾病.如今,虽然可以通过药物干预或手术矫正等方案减轻患者症状,但由于并未从根本上解决问题,这些疾病仍会通过基因遗传给后代.传统的基因治疗方案理论上可以从基因水平,彻底治愈遗传病,但由于缺乏理想的基因组靶向修饰技术,一直没有取得太多突破性进展.而随着近些年基因编辑技术的不断发展,已经在遗传性疾病治疗、传染性疾病治疗和癌症治疗等领域得到了广泛的研究应用,为基因治疗领域开辟了新的途径.
单基因遗传病是指受一对等位基因控制的遗传病,其一般治疗困难,从经济及心理层面给家庭带来了巨大的负担.肥厚型心肌病(Hypertrophic cardiomyopathy,HCM)是比较常见的单基因遗传性心血管疾病,患病率约1/500[52],MYBPC3基因突变容易导致HCM的发生[53].2017年,来自中美韩的科研团队在得到伦理许可的条件下,将正常人的卵子与MYBPC3基因突变的精子通过体外受精形成受精卵,最终利用CRISPR-Cas9技术成功对致病突变基因MYBPC3进行修复,基于Digenome-seq和全基因组深度测序等检测方法,结果显示该基因编辑方法是安全有效的,并没有在脱靶区域产生新的突变[54].近几年,也有一些科研团队利用CRISPR/Cas9技术建立了HCM模型或纠正了人诱导性多能干细胞(human induced pluripotent stem cell,hiPSC)的HCM突变基因[55-57].血友病是一种单基因隐性遗传病,因患者血液中缺乏某些凝血因子,而导致患者产生严重的凝血障碍,根据不同凝血因子的缺陷可分为血友病A(hemophilia A,HA)和血友病B(hemophilia B,HB).Li等[58]利用AAV将ZFNs和正常FIX基因递送至HB小鼠模型体内,使得凝血因子恢复至正常水平的3%~7%,后续观察并未发现任何副作用.β-地中海贫血症(βthalassemia)是一种由单基因突变引起的遗传性疾病,因β珠蛋白(HBB)基因突变使得β-珠蛋白肽链合成严重不足,导致没有足够的β-链与α-链进行结合,过剩的α-链会结合在红细胞膜上,致使骨髓中的红细胞前体和血液中的红细胞被破坏,引发贫血或慢性溶血.黄军就团队选取人类三核受精卵细胞,利用CRISPR/Cas9系统切割HBB基因,同时引入正常同源DNA修复模板,达到治疗β-thalassemia的目的,但其团队同时发现该系统同源重组效率较低,且存在一定的脱靶现象,表明仍需对CRISPR/Cas9系统的特异性做进一步优化处理[59].
多基因遗传病是由环境因素和遗传因素共同导致的一类疾病,其一般受多个具有累加效应的致病基因控制,且此类疾病大多具有家族聚集现象、性别及种族差异.唇裂与腭裂(Cleft lip and palate,CLP)属于比较常见的先天性缺陷,包含唇裂(CL)、腭裂(CP)及唇腭裂(CLP)三种疾病.Beaty等[60]通过GWAS分析将GADD45G列为CLP候选基因,2019年,Lu等[61]通过CRISPR/Cas9和BE4-Gam两种不同的基因编辑方法,成功获得具有CL症状的GADD45G基因突变兔,为探索GADD45G基因与唇腭部发育机理的联系提供了一种理想的动物疾病模型.先天性心脏病(Congenital heart defect,CHD)是一种婴幼儿心血管结构发育异常导致的疾病,在约10%的死产胎儿中发现患有严重CHD,因此认为CHD是导致早期胎儿死亡的一个重要原因[62].2019年,Gifford等[63]通过对一个CHD患儿的家族进行全基因组测序,发现父亲的MRTF-B(Q670H)和MYH7(L387F)基因,及母亲的NKX2-5(A119S)基因发生突变,最终导致该儿童发病,其团队利用CRISPR/Cas9技术构建出了具有相同突变的小鼠模型,发现只有同时存在三种突变的小鼠才表现出与该CHD患儿相同的症状,说明遗传自母亲的NKX2-5基因突变加剧了MKL2和MYH7两个基因突变引起的问题.精神分裂症(Schizophrenia,SCZ)是由环境因素和遗传因素共同造成的一种精神疾病,主要临床特征表现为思维情感紊乱、妄想幻听及认知功能障碍.Lee等[64]通过使用CRISPR/Cas9技术成功抑制了FXS小鼠mGluR5基因的表达,这也是人们第一次成功编辑自闭症相关基因,为后续治疗SCZ等相关精神疾病开辟了道路.
3 基因编辑技术在非遗传性疾病治疗中的应用
传染性疾病(Infectious Diseases)是由细菌、病毒或寄生虫等病原微生物引起的疾病,这些疾病可以在人与人、动物与动物或人与动物之间相互传播,是导致人类死亡的重要原因之一.其中,常见的病毒性传染病致病原包括艾滋病病毒(HIV)、乙型肝炎病毒(HBV)、人乳头瘤病毒(HPV)及如今正在全球引起大流行的新型冠状病毒(SARS-CoV-2)等,目前应对此类病毒带来的威胁,主要解决办法为疫苗预防和药物治疗.随着人们对基因功能研究的不断深入,人们开始探索基因编辑技术在病毒性传染病治疗中的应用[65].
艾滋病也称获得性免疫缺陷综合征(acquired immune deficiency syndrome,AIDS),是一种由人类免疫缺陷病毒(human immunodeficiency virus,HIV)感染造成的疾病[66].Xu等[67]在《The New England Journal of Medicine》杂志上联合报道了世界首例基因编辑干细胞治疗HIV和白血病患者的临床研究,经基因治疗后患者的急性淋巴白血病得到完全缓解,同时经过编辑的干细胞不仅能分化为多种谱系细胞,而且能够在患者体内存活达19个月,该研究在没有引发伦理担忧的情况下,极大地推动了CRISPR基因编辑技术在临床治疗中的应用.新型冠状病毒肺炎(COVID-19)是一种由新发现的冠状病毒引起的传染性疾病,大多数患者会在接触病毒后的2~14天出现发烧、咳嗽或呼吸困难等症状,其中一些重症患者可能会导致心脏损伤,大大增加了患者的死亡风险[68].截至目前为止,全球累计新型冠状病毒感染患者已达一亿多人,并且每天感染患者的比率仍在持续增加,给全人类生命健康安全和全球经济带来了巨大的威胁和冲击,而CRISPR技术在此次新冠病毒带来的全球大流行病挑战中又起到了极其重要的作用.Gootenberg等[69]在《Science》杂志上报道了基于CRISPR/Cas13的病毒检测技术SHERLOCK(Specific High Sensitivity Enzymatic Reporter Unlocking).该检测技术可以让被切割的RNA形成比较直观的条带,针对此次新型冠状病毒,其团队设计了两个可以识别新型冠状病毒S基因和Orf1ab基因的gRNA,一旦检测样本中含有新型冠状病毒,两个gRNA就会引导Cas13切割S和Orf1ab基因,1h内就可以完成对样本的新冠病毒检测[70-71].在抑制新冠病毒研究方向,Nguyen等[72]基于一种靶向RNA的CRISPR系统CRISPR/Cas13d,利用腺相关病毒AAV将其递送至COVID-19感染患者的肺部,从而达到清除病毒的目的,目前此研究仍处于初期阶段,后续仍需动物模型及相关临床试验数据的支撑,一旦证明该治疗方法是有效的,将为全球COVID-19等RNA病毒患者提供更多的选择.
4 面临的问题与展望
4.1 脱靶问题及应对策略
ZFNs中起切割DNA作用的为二聚化FokI核酸酶,由于FokI核酸酶在二聚化过程中有可能形成同源二聚体结构,进而发生非特异性切割,并产生一定的细胞毒性.因此,人们通过对FokI核酸酶进行修饰,以提高ZFNs的特异性,并降低因脱靶效应带来的细胞毒性.例如,Szczepek等[73]通过突变一对FokI核酸酶中的两个碱基(D483R和R487D),获得R和D两个变体,这两个变体只有形成异源二聚体时才具有切割活性.TALENs作为第二代基因编辑技术,虽然其构建过程要比ZFNs更加简便,但依然没能摆脱脱靶效应带来的困扰,当TALE蛋白的DNA结合域和DNA产生非特异性结合时,有可能造成TALENs的脱靶效应.已有报道显示,当两个TALENs结合位点间隔距离为10~30 bp时,能够最大程度地减少TALE蛋白与DNA的非特异性结合[74-75].与ZFNs类似,同源二聚体的存在增加了TALENs技术的脱靶效应,对此,一些科研团队通过对FokI进行修饰改造,使得只有FokI的异源二聚体才具有剪切活性[73,76].值得注意的是,虽然现在已有其他的一些优化后的变体可以应用到基因编辑实验中,但这些都只是降低了脱靶效率,并没有彻底消除脱靶效应.
相比于以上两种基因编辑技术,CRISPR/Cas9技术具有较高的编辑效率及操作简便和成本低廉等优点,但其同样具有较高的脱靶效率.CRISPR/Cas9技术发生脱靶效应主要是由于sgRNA在基因组中存在很多脱靶位点造成的[77],此外,PAM序列及染色质结构等因素也在一定程度上影响CRISPR/Cas9系统的特异性.针对这些可能的脱靶因素,一些科研团队通过开发出了一些SpCas9类似物[78],或对sgRNA进行不同的修饰[79-80],进而达到降低脱靶效应的目的.对BE技术而言,其脱靶效应大致可以分为3个层次:可预测的脱靶、全基因组水平及转录组水平的脱靶.由于sgRNA序列的容错性及Cas9核酸酶本身的非特异性,都容易造成可预测的脱靶,针对此类位点,可以应用传统的分子生物学手段来进行检测判断.Kim等[81]通过对sgRNA进行修饰,可明显降低BE3的脱靶效应.David R.Liu团队[82]通过将BE3与工程化的Cas9进行融合,与原始BE3相比可显著降低脱靶水平.在全基因组水平上,科研人员发现相比于CBE系统,ABE系统具有较高的特异性,进一步分析可能与经过人工定向进化而来的腺嘌呤脱氨酶有关,所以开发新型胞嘧啶脱氨酶,也成了解决CBE系统在全基因组水平脱靶的潜在方案[83].对于转录组水平的脱靶,一般采取对脱氨酶进行改造的手段来减少脱靶现象的发生.2020年5月,中科院神经科学研究所联合其他科研单位,共同发布了在DNA和RNA上能够显著降低脱靶效应的单碱基编辑工具YE1-BE3-FNLS[84].与此同时,基因编辑技术在疾病治疗中所引发的伦理问题也是不容忽视的,在基因编辑领域出台相应的法律法规迫在眉睫,倘若只以道德的约束力去镇守科学界的伦理底线,伦理底线一定会被反复践踏,从而增加了基因编辑技术风险的不确定性.
4.2 发展前景
自从1996年第一个人工锌指核酸酶的问世,到此后的20多年时间里,基因编辑技术不断取得新的突破,尤其当CRISPR-Cas系统第一次在真核细胞基因组中实现基因编辑以后,更是在全球范围内掀起了基因编辑技术研发及应用的热潮.与只能通过载体释放目的基因的治疗方案相比,基因编辑技术可以实现定点敲除、插入和替换,以此达到启动或关闭某些基因的效果,同时还解决了病毒载体随机整合到染色体的问题,规避了诱导细胞癌变的发生.尤其是近几年CRISPR/Cas9和单碱基编辑技术的出现,将人们带入一个全新的精准编辑时代,推动了生命科学和临床治疗等领域的进步,在医学疾病治疗研究领域,基因编辑技术极大地推动了该领域的发展.尽管目前现有的多种基因编辑技术,在疾病治疗中仍有很多问题亟待解决,但随着研究的不断深入,人们总有一天会建立起一套安全、高效的基因编辑系统,为饱受疾病困扰的患者带来更多的希望.
