基于TCGA SpliceSeq数据库可变剪接事件构建结肠癌患者预后风险模型
2021-02-05郭萌月王若颖倪小梅
雷 鸣,郭萌月,王若颖,倪小梅,石 琼
(云南省肿瘤医院 昆明医科大学第三附属医院 云南省癌症中心,云南 昆明 650118)
结肠癌(colon adenocarcinoma,COAD)是常见的恶性肿瘤之一,发生率在消化道肿瘤中仅次于胃癌和食管癌[1]。肿瘤中存在大量异常mRNA剪接方式,二代高通量测序技术能有效获取肿瘤中mRNA的表达水平和序列信息,从而发现样本中大量的新突变、可变剪接和基因融合。可变剪接指同一个基因转录形成的mRNA前体通过不同的剪切和拼接方式产生不同的成熟mRNA的过程,所获得的mRNA被称为转录本,再进行翻译可以获得不同的蛋白质异构体[2]。可变剪接是调节基因表达和产生蛋白质多样性的重要机制,也是真核生物基因和蛋白质数量差异较大的重要原因[3],在细胞生长、分化、分裂、凋亡过程中具有重要作用,其异常与肿瘤细胞的凋亡、迁移、侵袭、耐药等密切相关[4]。癌症基因组计划(the Cancer Genome Atlas,TCGA)SpliceSeq数据库提供的可变剪接事件主要有7种类型:可变受体位点(alternate acceptor site,AA)、可变供体位点(alternate donor site,AD)、可变启动子(alternate pormoter,AP)、可变终止子(alternate terminator,AT)、外显子跳跃(exon skip,ES)、外显子互斥(mutually exclusive exons,ME)、内含子保留(retained intron,RI)[5]。本研究基于TCGA数据库中大样本COAD的RNA-Seq数据及患者临床信息,联合TCGA SpliceSeq数据库中的剪接事件数据,构建高效且可靠的COAD预后风险模型,并从SpliceAid 2数据库中下载剪接因子(splicing factor,SF)数据[6],构建其与可变剪接事件之间的调控网络,为预测患者预后奠定基础。
1 材料和方法
1.1 研究对象
从TCGA数据库(https://cancergenome.nih.gov/)中获取COAD患者肿瘤组织(398例)及癌旁组织(39例)RNA-seq Levels 3数据和对应临床信息,数据使用每百万转录本映射的reads数(Transcripts per million reads,TPM)进行标准化处理。COAD的剪接事件从TCGA SpliceSeq数据库(http//bioinformatics.mdanderson.org/TCGASpliceSeq/)下载,并获得剪接百分比(percent spliced-in index value,PSI),将PSI≥75%的可变剪接事件纳入到本研究中,SF数据从SpliceAid2数据库(http://193.206.120.249/splicing_tissue.html)中下载,共得到403个SF。
1.2 构建预后风险模型
COAD患者临床信息排除标准:(1)患者生存时间缺失或临床信息<90 d;(2)缺少病理诊断的临床信息;(3)没有临床信息。用单因素Cox回归分析进行初步变量筛选,然后将得到的相关数据纳入到Lasso回归,对数据进行特征选取和降维处理,调取R语言“glmnet”包对数据进行100次10折交叉验证,选定最小λ值为最佳λ参数值。筛选得到特征变量后,再经多因素Cox回归分析构建出预后风险模型(Risk score)为:
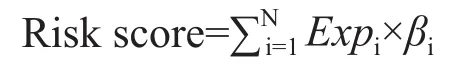
公式中Expi为各可变剪接事件在样本中的PSI值,即可能影响生存时间的有关因素;βi为多因素Cox回归系数。根据公式计算出每例患者的预后风险值,由于基因表达量呈偏态分布,故将计算得到的风险值中位数设为最佳临界值,将患者分为高风险组和低风险组,并以60个月为研究终点。
1.3 预后相关可变剪接事件(prognostic related alternative splicing events,PASE)功能富集分析
将单因素Cox回归分析筛选出的PASE导入到Cytosacpe Reactome FI插件用于蛋白互作分析。由R语言“clusterProfiler”包进行基因本体(Gene Ontology,GO)富集[包括生物过程(biological processes,BP)、分子功能(molecular function,MF)、细胞构成(cellular component,CC)]和KEGG通路分析。用R语言“UpSet”显示7种可变剪接事件组成的预后风险模型和PASE的交互作用。将R语言的“UpSet”包用于基因的可视化。
1.4 构建SF和剪接事件共表达网络
通过Pearson检验分析SF表达与PASE PSI值之间的相关性。Cytoscape 3.7软件用于构建蛋白调节网络。核心SF(Hub)由每个节点的数值和边界定,Hub的表达水平由肿瘤组织和癌旁组织来确定,并分析SF与非SF的PASE构成的互作网络。
1.5 统计学方法
采用Kaplan-Meier绘制生存曲线,用Logrank秩和检验评估高风险组、低风险组患者的总体生存率。采用受试者工作特征(receiver operating characteristic,ROC)曲线评估预后风险模型在1、3、5年生存期的预测能力,并绘制高、低风险热图。采用单因素和多因素Cox回归分析评估临床各变量及风险评分与患者预后的相关性。SF在癌旁组织和肿瘤组织中的表达差异用Wilcoxon非配对检验。
2 结果
2.1 COAD中可变剪接事件和PASE统计分析
398例COAD患者中共有9 085个基因发生了35 391次可变剪接事件,每个基因平均有4次可变剪接事件发生。其中5 635个基因发生13 087次ES,3 381个基因发生7 740次AT,2 692个基因发生6 653次AP,2 124个基因发生2 917次AA,1 833个基因发生2 524次AD,1 600个基因发生2 332次RI,137个基因发生138次ME。所有可变剪接事件中,ES(36.97%)为主要类型,ME(0.38%)是发生次数最少的类型。单个基因可能发生2个或更多可变剪接事件,为确定与COAD预后相关的PASE,利用单因素Cox回归分析对可变剪接事件进行变量筛选(P<0.05),结果显示有1 811个基因发生2 515个PASE。
2.2 构建蛋白互作网络并进行GO富集、KEGG通路分析
将821个PASE(P<0.01)所对应的649个基因导入Cytoscape Reactome FI插件中,挖掘核心调控基因,发现链接点数最多的核心调控基因分别是TP53、RELA、UBE21。随后将2 515个PASE中对应的1 810个基因进行GO富集和KEGG通路的生物信息学分析,发现GO富集共计47个BP(P<0.01),主要包括RNA剪接、细胞周期调节、mRNA加工、组蛋白修饰;14个CC(P<0.001),主要包括细胞器、线粒体基质、染色质和中心粒;17个MF(P<0.001),包括泛素类蛋白转移酶活性、转录调控、丝氨酸/苏氨酸激酶活性、细胞黏附。KEGG通路有10个与生存相关的具有显著性差异(P<0.01)的通路,其中5个主要涉及泛素介导的蛋白水解通路、剪接体通路、HIF-1信号通路、脂肪酸代谢、结直肠癌相关信号通路。见图1。
2.3 利用PASE构建COAD预后风险模型
数据中基因数量多而样本量较少,因此用Lasso回归分析进行变量筛选。为了更为精确和方便地描述可变剪接事件,本研究对COAD中每个可变剪接事件独立编号,例如,PCNP-65964-AA,PCNP是基因名,65964是在COAD中剪接事件的唯一编号,AA是剪接事件类型。在7种可变剪接事件混合模型中,当最佳λ参数值为0.056时,共筛选出8个PASE。基于这8个PASE,用多因素Cox回归分析进行变量筛选,最后构建出由8个PASE组成的预后风险模型(Risk score)。Risk score=(-17.504×HMGXB3-74054-RI)+(-104.165×PPP3CA-70095-ES)+(-1.947×KIAA1522-1632-AP)+(-6.171×SPINK1-73963-AT)+(-2.783×ZNF765-51718-AT)+(-13.186×PDCD2-78502-AA)+(-10.491×PTPRD-85850-ES)+(2.272×RAB3IP-23343-AP)。以风险值中位数(0.919)为最佳临界值,将COAD患者划分为高风险组(风险值>0.919,173例)和低风险组(风险值≤0.919,174例)。热图结果显示,随着风险值的升高,PASE在2个组中的表达水平也明显升高或降低。Kaplan-Meier曲线分析结果显示,高风险组患者总体生存率较低风险组低,且2个组差异有统计学意义[风险比(hazard ratio,HR)=2.921,95%可信区间(confidence interal,CI)=1.890±4.512,P<0.001)。用ROC曲线对预后风险模型进行预测性能的评价(COAD患者1、3、5年的生存率),结果显示COAD患者1年生存率的曲线下面积[(area under curve,AUC)=0.860]好于3年(AUC=0.803)和5年(AUC=0.704)的。见图2。
2.4 临床相关参数和预后风险模型的单因素和多因素Cox回归分析
为评估预后风险模型是否独立于其他临床变量,我们对其分别进行了单因素和多因素的Cox回归分析。纳入的临床变量包括年龄、性别、息肉史、肿瘤浸润、淋巴结转移、远处转移、临床分期。在单因素Cox回归分析中,肿瘤浸润、淋巴结转移、远处转移、临床分期、预后风险模型都与患者总体生存时间呈显著负相关(P<0.001)。经过多因素Cox回归分析调整后,预后风险模型依然与患者总体生存时间呈显著负相关(P<0.001)。见表1和表2。
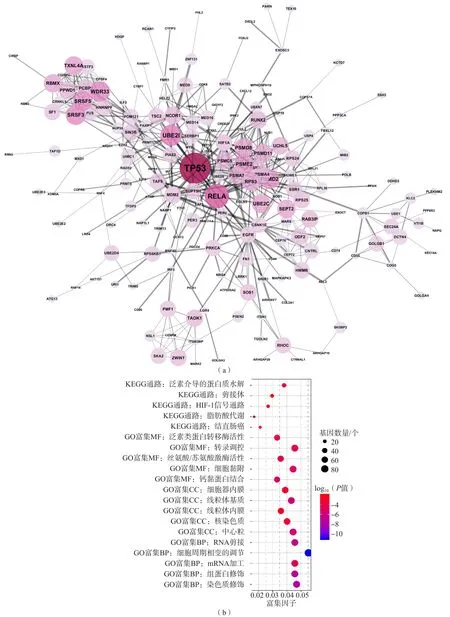
图1 PASEs的生物信息学分析

图2 PASE构建的COAD预后风险模型价值评价
2.5 PASE与其对应mRNA表达量的相关性分析
对预后风险模型的8个PASE对应的基因mRNA表达量在肿瘤组织和癌旁组织间进行比较,发现ZNF765、KIAA1522、HMGXB3、RAB3IP、PDCD2有差异(P<0.01)。8个PASE对应的基因的生存曲线分析结果显示,高风险组、低风险组之间没有显著性差异(P>0.05)。见图3和图4。
2.6 生存相关SF的风险因素
从TCGA数据库中提取到COAD相关的387个SF表达量,用单因素Cox回归分析得到22个与生存相关的SF,其中只有RBM3、GRSF1、RBM47的HR<1,属于保护性因素;其他19个SF的HR>1,属于危险性因素。见表3。
2.7 PASE和SF共表达网络
对2 515个PASE与22个生存相关的SF表达量进行相关性检验,并将结果导入到Cytoscape 3.71软件中构建互作网络,发现有420个PASE的PSI值与15个SF有相关性(P<0.01),其中296个PASE与15个SF呈负相关;381个PASE与14个SF呈正相关。见图5。
2.8 与生存相关的SF的mRNA表达量比较
我们将15个生存相关的SF在COAD癌旁组织和肿瘤组织中的表达量进行分析,发现有14个SF有显著性差异(P<0.05),其中C9orf78、CCDC130、CLK2、DHX38、GRSF1、ISY1、KHSRP、RBM17、RBM3、SART1和ZC3H18属于危险因素;NRIP2、NOVA2和RBM47属于保护性因素;NUMA1无差异(P>0.05)。见图6。

表1 预后风险模型单因素Cox回归分析结果

表2 预后风险模型多因素Cox回归分析结果
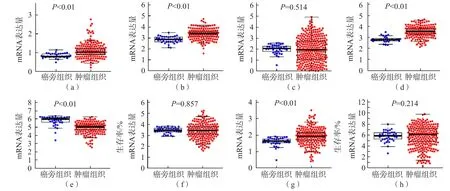
图3 预后风险模型中8个PASE对应基因的mRNA表达量

图4 预后风险模型中8个PASE对应基因的生存曲线分析

表3 22个生存相关SF的单因素Cox回归分析结果

图5 SF与生存相关剪接事件在COAD中的相关性分析

图6 COAD癌旁组织和肿瘤组织中生存相关SF的mRNA表达量
3 讨论
目前,COAD的可变剪接事件研究主要是以小样本的测序方式进行,COAD的可变剪接事件分析还鲜有报道。本研究从TCGA SpliceSeq数据库得到的COAD可变剪接事件和PSI值显示,共有9 085个基因发生了35 391次可变剪接事件,ES(36.9%)为主要类型,ME(0.38%)是发生次数最少的类型。KAHLES等[7]的研究结果显示,可变剪接事件在不同的肿瘤类型中出现的频率各不相同,但ES都是最高的,与本研究结果相符。本研究将可变剪接事件与生存资料合并进行单因素Cox回归分析,发现共有2 515个PASE,其所对应的基因有1 811个。“UpSet”结果显示,部分基因至少有2种以上PASE发生,说明可变剪接事件在肿瘤发生、发展过程中起重要作用。将821个PASE(P<0.01)所对应的649个基因导入到Cytoscape Reactome FI插件中,筛选出核心基因TP53、RELA、UBE21。TP53、RELA、UBE21是共表达网络的核心节点,有望成为COAD治疗的分子靶点。WU等[8]利用TCGA SpliceSeq数据库下载和分析了肝癌的PASE,构建了8个风险评估模型,由ES事件组成的风险模型预测效果最好,AUC为0.898。为评估PASE在COAD中的预后诊断价值,本研究构建了预后风险模型,混合剪接事件构成的预后风险诊断模型高、低风险有显著性差异(P<0.001),ROC曲线的AUC为0.860(1年生存率),在COAD患者的风险分层中表现准确。为了评估预后风险模型是否独立于其他临床变量,本研究分别采用单因素和多因素Cox回归分析评估临床病理参数和预后风险模型对COAD患者预后的影响,发现预后风险模型与患者总体生存时间呈显著负相关(P<0.001)。对构成预后风险模型的8个PASE对应的基因的癌旁组织与肿瘤组织mRNA表达量进行分析,发现并无统计学差异(P>0.05)。基因和其可变剪接事件可能执行不同的生物学功能,其具体的机制尚未被完全阐明,需要进行进一步探讨。
在肿瘤中,SF表达异常可能会形成特定促癌剪切异构体,从而导致癌症发生。为了阐明SF和PASE之间复杂的调控关系,本研究构建了互作网络,有420个PASE的PSI值与15个SF有相关性(P<0.01),其中296个PASE与15个SF存在负相关关系;381个PASE与14个SF存在正相关关系。这说明有的SF具有双重调控功能,有的PASE同时受到不同的SF调控。在互作网络中,SF细胞分裂周期样激酶2与SF富含丝氨酸/精氨酸剪接因子3 2个同类型的PASE存在相反的调控关系,说明同一基因的同类型可变剪接事件可能执行不同的生物学功能。SRSF3和CLK2的mRNA表达量几乎无相关性,而CLK2的4个可变剪接事件CLK2-8053-AP、CLK2-8054-AP、CLK2-8055-AA和CLK2-8056-AA则不属于PASE,因此基因的mRNA表达量并不能很好地描述基因的功能,基因的生物学功能可能要依靠占优势的可变剪接事件进行描述。SF在调控可变剪接事件中起到了重要的作用,其核酸序列的突变或者表达水平的改变都可能影响可变剪接事件[9],但SF如何调节可变剪接事件的类型目前尚不清楚。通过上调或下调SF的表达水平都可能促进或者抑制肿瘤的进展[10]。分析COAD患者生存资料得到的22个与生存相关的SF中,大部分SF高表达时患者生存期缩短,这些SF的表达水平可能是被类似的可变剪接事件所调控。相关性分析结果显示,15个与生存相关的SF中有14个在癌旁组织和肿瘤组织中有差异(P<0.05),多数在肿瘤组织中表达量上调,说明在COAD发生、发展过程中与PASE相关的SF过表达,对患者预后不利。各组间核心调控因子CLK2在COAD的临床分期、T分期、组织分级中表达有差异(P<0.05),而在N分期和M分期中无差异(P>0.05),说明SF的高表达多与患者预后不良有关。
可变剪接事件的发生更像是细胞为了应对外来的刺激信号而快速产生的特殊表型,用于执行特定的功能。相对于庞大的基因组,目前检测到的剪接产物很少,剪接机制的研究还处于初级阶段。本研究尝试用多个可变剪接事件类型构建预后风险模型,得到了满意的效果。对于数据而言,可变剪接事件可单独成为肿瘤诊断和预后的标志物。然而,以单一类型可变剪接事件作为标志物是不够充分的,联合多个可变剪接事件来提高诊断和预后的敏感性,也符合临床研究的发展趋势。本研究还构建了SF与PASE之间的互作网络,这为COAD的预后风险模型的构建提供了帮助。
