乳头状肾细胞癌差异表达基因和通路生物信息学分析
2019-09-10崔嘉晗赵明牛海涛王新生
崔嘉晗 赵明 牛海涛 王新生

[摘要]目的探讨生物信息学分析对乳头状肾细胞癌(PRCC)差异表达基因和通路的意义。方法分析癌症基因组图谱(TCGA)数据库中PRCC的RNA测序数据,应用生物信息学分析筛选差异表达基因,并对候选基因依据功能和信号通路进行富集分析。结果 分析TCGA中PRCC和癌旁正常组织的RNA测序数据,筛选4 639个差异表达的基因。对筛选差异表达基因进行基因本体及代谢通路富集分析后得到818个基因本体术语(GO terms)和110条信号通路术语(pathway terms)。结论 运用edgeR包以及基因本体及信号通路分析,筛选了PRCC与癌旁正常组织差异表达基因及富集的信号通路。
[关键词]癌,肾细胞;基因本体;信号通路;遗传关联研究;计算生物学
[中图分类号]R730.26[文献标志码]A[文章编号] 2096-5532(2019)04-0402-05
[ABSTRACT]ObjectiveTo explore the significance of bioinformatics in the analysis of differentially expressed genes and pathways in papillary renal cell carcinoma (PRCC).MethodsThe RNA sequencing data of PRCC in the Cancer Genome Atlas database (TCGA) were analyzed. Differentially expressed genes were identified by bioinformatics analysis, and candidate genes were then enriched according to their functions and the signaling pathways in which they were involved.ResultsRNA sequencing data of PRCC and adjacent normal tissues in TCGA were analyzed and 4639 differentially expressed genes were screened out. After enrichment analyses of gene ontology and metabolic pathways for these differentially expressed genes,818 gene ontology terms and 110 pathway terms were obtained.ConclusionUsing the edgeR package and gene ontology and pathway analyses, the differentially expressed genes and enriched signaling pathways between PRCC and adjacent normal tissues were screened out.
[KEY WORDS]carcinoma, renal cell; gene ontology; signaling pathway; genetic association studies; computational biology
腎细胞癌(RCC)是最常见的肾脏癌症,占所有成人恶性肿瘤的2%~3%[1-2]。有20%~30%的病人被诊断为RCC时已经发现远处转移。此外,另有20%的病人接受肾切除术后会复发并发展成为转移性RCC [3-4]。转移性RCC病人生存率低,预后极差[5-6]。乳头状肾细胞癌(PRCC)是第二常见的RCC类型,占所有RCC的10%~20%[1]。PRCC可根据组织病理学特征进一步分为两种主要亚型[7-9]。癌症基因组图谱(TCGA)是临床信息和基因测序数据的大型综合集合。既往RCC研究主要以透明细胞RCC为主,缺乏PRCC相关研究。为了探究PRCC涉及的信号传导途径和驱动基因并且进行系统分析,本文利用TCGA进行数据挖掘,筛选PRCC中差异表达基因及富集的信号通路。现将结果报告如下。
1材料与方法
1.1实验样本信息收集
于 TCGA 数据库(https://cancergenome.nih.gov/)中收集与PRCC相关的RNA的测序数据(Level3),包括289例PRCC组织和32例癌旁非癌肾组织。
1.2筛选PRCC中异常表达的mRNAs
利用genecode数据库筛选mRNA测序数据,共涵盖20 271个mRNA数据。随后以R语言包edgeR计算异常表达的mRNAs(差异倍数>2并且错误发现率(FDR)校正后P值<0.01)。排除超过75%的样品中表达<2的mRNA。
1.3基因本体分析和富集途径分析
使用在线数据库DAVID(https://david.ncifcrf.gov/)进行差异基因的基因本体分析和富集途径分析。基因本体分析和富集途径分析均以P值<0.05作为临界标准。
1.4基因集富集分析
基因组富集分析使用Java GSEA软件。基因组富集分析软件及注释基因集均由Broad研究所网站(http://www.broadinstitute.org/gsea/index.jsp)下载。基因集P<0.05并且FDR<0.25被认为具有统计学差异。
1.5统计学处理
使用R语言(版本号3.5.1)中的edgeR包筛选PRCC和癌旁正常组织中不同表达的基因,筛选标准为FDR校正后P值<0.01和差异倍数>2的基因。对于GO和KEGG途径分析,其中P<0.05的GO术语或途径是有统计学差异的。
2结果
2.1癌与癌旁配对样本差异表达的mRNA
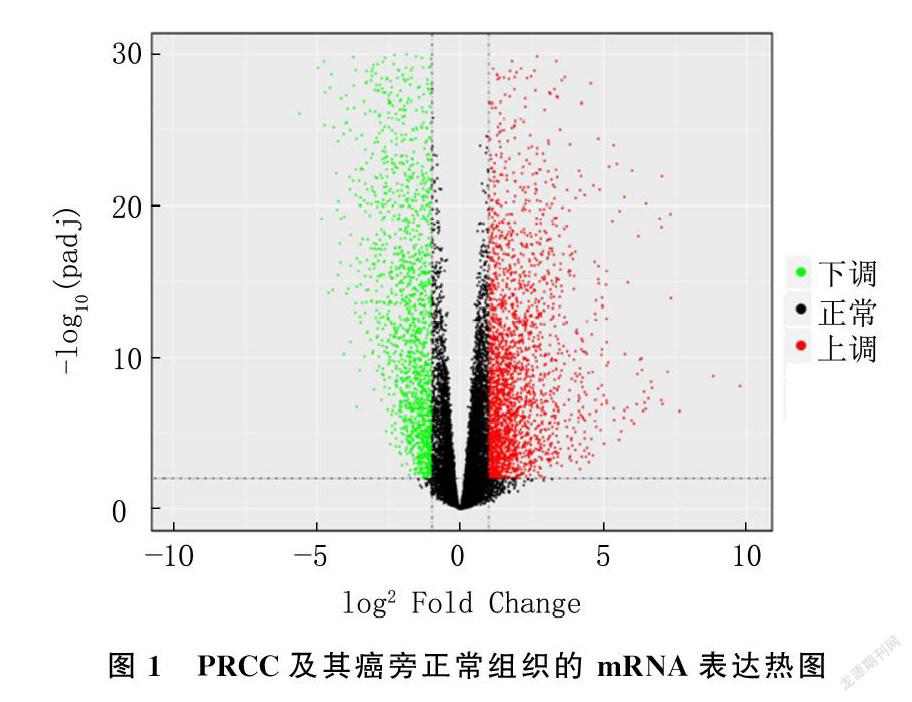
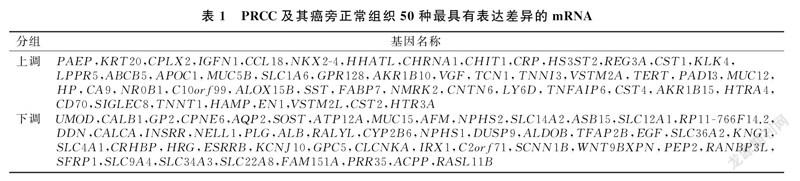
对PRCC标本及癌旁正常组织的mRNA测序数据进行差异基因分析(差异倍数>2并且FDR校正后P值<0.01),筛选4 639个差异表达的基因,在样品中2 638个mRNA转录物被上调并且2 001个mRNA转录物被下调。火山图见图1。其中,PRCC及其癌旁正常组织中50种最具有表达差异的mRNA(从倍数变化最大到最小列出)见表1。
利用在线基因功能分类工具(https://david.ncifcrf.gov/gene2gene.jsp),基于功能相似性对筛选出的50种最具有表达差异的基因进行分组。上调组筛选出1个功能相关基因组,基因为SIGLEC8、CNTN6、VSTM2A、VSTM2L。下调组筛选出2个功能相关基因组,其中一个基因组为SLC34A3、SCNN1B、SLC22A8、SLC9A4、SLC12A1,另外一个基因组为AQP2、MUC15、SLC22A8、SLC14A2。检索相关文献,我们发现SIGLEC8表达的增强预测了PRCC病人的不良预后[10]。另外,MUC15在PRCC中的表达降低[11],但是其余大部分基因在PRCC中的作用仍不明确。
2.2差异表达基因的基因本体分析
利用在线数据库DAVID分析差异表达基因,以P<0.05作为临界标准。基因本体分析分为3个功能组:分子功能组、生物过程组和细胞成分组。在生物过程组中,上调的基因主要富集于信号转导、免疫反应、炎症反应等,下调基因主要富集于信号转导、细胞黏附等。在细胞成分组中,上调基因主要富集在膜的组成部分、质膜、细胞外区域等,下调基因主要富集于膜的组成部分、质膜等。在分子功能组中,上调基因主要富集于钙离子结合、受体结合等,下调基因主要富集于钙离子结合、蛋白质同源二聚化活性等。其中,PRCC标本转录组上调基因最显著富集的5个基因本体术语见图2A~C,下调基因最显著富集的5个基因本体术语见图2D~F。
2.3信号通路富集分析
使用KEGG PATHWAY在線网站进行差异基因的功能和信号传导途径富集。上调的基因主要富集于细胞因子-细胞因子受体相互作用、神经活性配体-受体相互作用等,下调的基因主要富集于代谢途径、神经活性配体-受体相互作用、癌症的途径等。见图2G、H。
2.4基因集富集分析
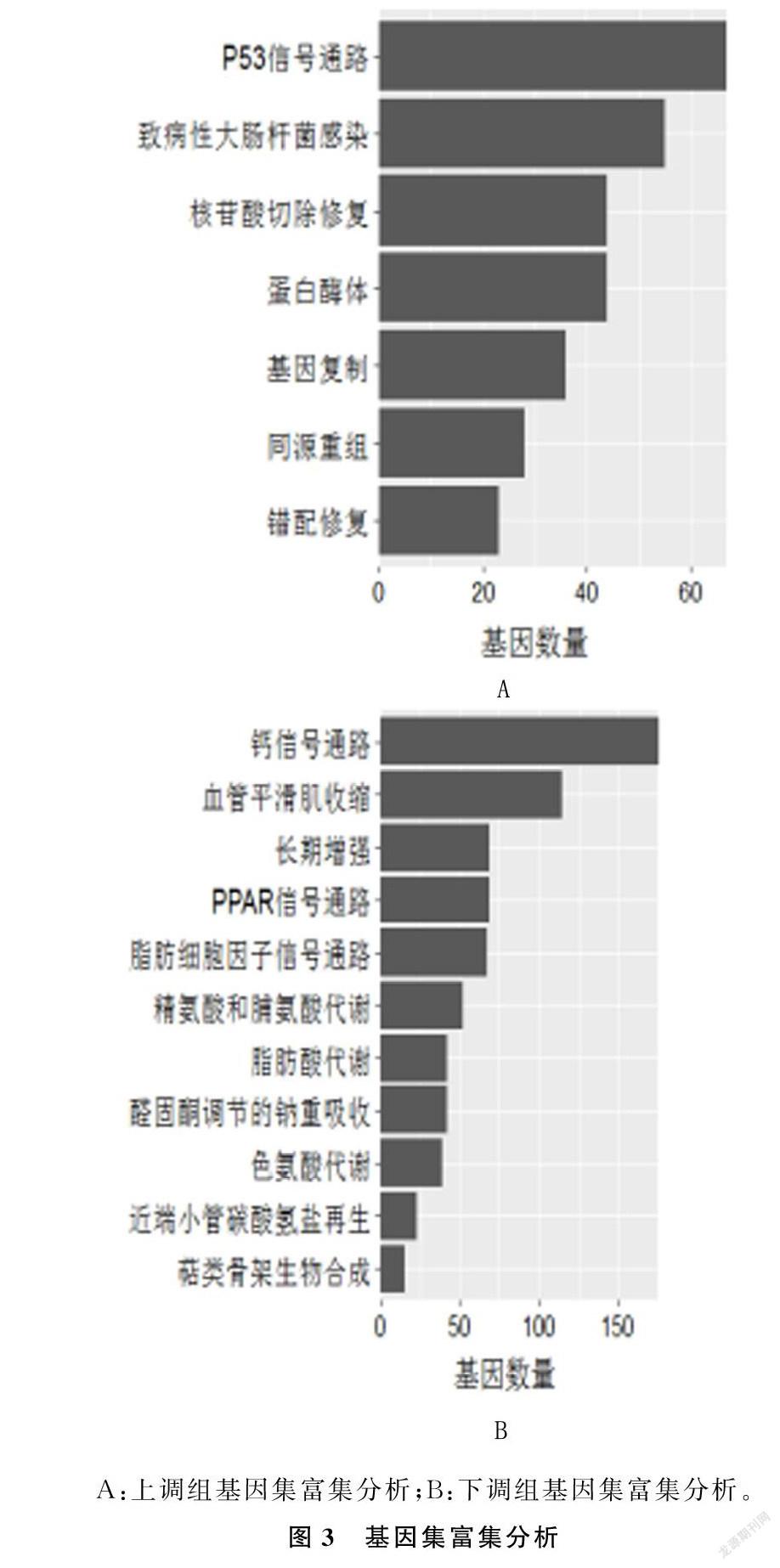
基因集富集分析通过关注具有共同生物功能、染色体定位或调节的基因组,从而筛选出可能与疾病表型相关的过量表达的基因或蛋白质。我们使用KEGG基因组作为基因集对PRCC及其癌旁正常组织的转录组数据进行基因集富集分析。确定了7个上调途径以及12个下调途径。见图3A、B。
3讨论
在过去的几十年中,已经进行了大量的基础和临床研究,揭示PRCC形成和发展的原因和潜在机制[12-15]。已报道PRCC具有代表性遗传基因突变和失调的途径包括:MET基因突变、HIPPO途径、NRF2途径、染色质修饰基因突变等[9]。本研究应用生物信息分析技术深入分析TCGA数据集,并在第一步中确定了4 639个差异表达的基因(2 638个上调和2 001个下调)。在第二步中,对筛选差异表达基因进行基因本体分析及代谢通路分析,本研究中筛选的上调基因主要富集在信号转导、免疫反应、炎症反应、膜的组成部分和钙离子结合等途径中;下调基因主要富集在信号转导、细胞黏附、膜的组成部分和钙离子结合等途径中。改变的基因和信号转导途径为理解PRCC的分子基础提供相关数据,从细胞通路及代谢途径更好揭示PRCC内在分子机制,探究预后的生物标志物及治疗靶点。既往研究已提示上皮细胞黏附及其活化分子在高级别RCC中的过度表达可能是预后的有用标志[16]。在PRCC发生骨转移病人的组织样本和原代细胞中,检测到钙离子受体表达明显增强[17-18]。本研究中,同样观察到钙离子结合途径发生明显改变,与有关文献报道相符合。以前研究表明,肿瘤的发生与炎症状态有密切关系[19],这与本研究显示免疫反应、炎症反应相关基因集在PRCC上调具有一致性。此外,既往研究发现肿瘤可建立肿瘤内免疫抑制环境,以支持其生长并促进免疫逃避[20-21],本研究结果同样显示肿瘤内免疫反应的改变。肿瘤内的代谢物水平的增加可以促进癌症的发生和发展,可导致细胞信号、酶活性或代谢通量的改变[22]。散发性和家族性2型PRCC检测到增强的有氧糖酵解,并且具有抗氧化反应的细胞表型[23-24]。
本研究第三步中,利用基因集富集分析方法分析筛选的差异表达基因的代谢途径,结果表明P53信号通路和DNA修复途径在PRCC中上调。DNA损伤的修复在维持基因组完整性方面起着关键作用[25]。多种常规DNA修复途径的多功能DNA修复中心经常在癌症中被改变[26]。有研究表明,DNA损伤修复途径中不利等位基因数量的增加,会增加RCC风险[27]。不过也有证据表明,DNA修复基因多态性可能不会影响RCC的易感性,但可能会导致DNA修复能力的改变,从而影响肿瘤的进展[28]。肿瘤抑制基因p53是人类癌症中最常见的突变基因,在细胞凋亡、基因组稳定性和抗血管生成中发挥作用,可以介导RCC细胞周期停滞和细胞凋亡[29]。研究表明,P53阳性表达与RCC病人预后不良和晚期临床病理特征相关,这表明P53是一个潜在有效的治疗靶点[30]。
综上所述,利用生物信息学分析,我们共筛选了4 639个候选基因,其富集于信号转导、钙离子结合、细胞因子-细胞因子受体相互作用、代谢途径、P53信号通路和DNA修复等途径。这些发现可提高我们对PRCC病因和潜在分子事件的理解,这些候选基因和途径有可能成为PRCC的治疗靶点。
[参考文献]
[1]COURTHOD G, TUCCI M, MAIO M D, et al. Papillary renal cell carcinoma: a review of the current therapeutic landscape [J]. Crit Rev Oncol Hematol, 2015,96(1):100-112.
[2]SIEGEL R L, MILLER K D. Cancer statistics, 2019[J]. CA-Cancer J Clin, 2019,96(1):7-34.
[3]LJUNGBERG B, CAMPBELL S C,CHO H Y, et al. The epidemiology of renal cell carcinoma[J]. European Urology, 2011,60(4):615-621.
[4]KING S C, POLLACK L A, LI J, et al. Continued increase in incidence of renal cell carcinoma, especially in young patients and high grade disease: United States 2001 to 2010[J]. J Urol, 2014,191(6):1665-1670.
[5]SHUCH B, HAHN A W, AGARWAL N. Current treatment landscape of advanced papillary renal cancer[J]. J Clin Oncol, 2017,35(26):2981-2983.
[6]CONNOR W J, DONSkOV F, FRACCON A P, et al. Characterizing the outcomes of metastatic papillary renal cell carcinoma[J]. Cancer Medicine, 2017,6(5):902-909.
[7]ALGABA F, AKAZA H, LOPEZ-BELTRAN A, et al. Current pathology keys of renal cell carcinoma[J]. Eur Urol, 2011,60(4) :634-643.
[8]KLATTE T, PANTUCK A J, SAID J W, et al. Cytogenetic and molecular tumor profiling for type 1 and type 2 papillary renal cell carcinoma[J]. Clin Cancer Res, 2009,15(4):1162-1169.
[9]LINEHAN W M, SPELLMAN P T, RICKETTS C J, et al. Comprehensive molecular characterization of papillary renal-cell carcinoma[J]. N Engl J Med, 2016,374(2) :135-145.
[10]OU C, LIU L, WANG J, et al. Enhancement of Siglec-8 expression predicts adverse prognosis in patients with clear cell renal cell carcinoma [J]. Urol Oncol, 2017,35(10):607-607.
[11]KING R J, YU F, SINGH P K. Genomic alterations in mucins across cancers[J]. Oncotarget, 2017,8(40):67152-67168.
[12]ALBIGES L, GUEGAN J, FORMAL A L, et al. MET is a potential target across all papillary renal cell carcinomas: result from a large molecular study of pRCC with CGH array and matching gene expression array[J]. Clin Cancer Res, 2014,20(13):3411-3421.
[13]LIU K, REN Y, PANG L, et al. Papillary renal cell carcinoma: a clinicopathological and whole-genome exon sequencing study[J]. Int J Clin Exp Pathol, 2015,8(7):8311-8335.
[14]KOVAC M, NAVAS C, HORSWELL S, et al. Recurrent chromosomal gains and heterogeneous driver mutations characterise papillary renal cancer evolution[J]. Nat Commun, 2015,6(1):6336.
[15]DURINCK S, STAWISKI E W, PAVIA-JIMENEZ A, et al. Spectrum of diverse genomic alterations define non-clear cell renal carcinoma subtypes[J]. Nat Genet, 2015,47(1):13-21.
[16]ZIMPFER A, MARUSCHKE M, REHNS, et al. Prognostic and diagnostic implications of epithelial cell adhesion/activating molecule(EpCAM)expression in renal tumours:a retrospective clinicopathological study of 948 cases using tissue microarrays[J]. BJU Int, 2014,114(2):296-302.
[17]BRENNER W, HABER T, JUNKER K, et al. Bone metastasis by renal cell carcinoma. Importance of Calcium and calcium-sensing receptor[J]. Urologe A, 2015,54(6):839-843.
[18]FREES S, BREUKSCH I, HABER T, et al. Calcium-sensing receptor(CaSR)promotes development of bone metastasis in renal cell carcinoma[J]. Oncotarget, 2018,9(21):15766-15779.
[19]KORNILUK A, KOPER O, KEMONA H, et al. From inflammation to cancer[J]. Ir J Med Sci, 2017,186(1):57-62.
[20]BUSSE A, ASEMISSEN A, NONNENMACHER A, et al. Systemic immune tuning in renal cell carcinoma:favorable prognostic impact of TGF-beta1 mRNA expression in perip-
heral blood mononuclear cells[J]. J Immunother, 2011,34(1):113-119.
[21]BINNEWIES M, ROBERTS E W, KERSTEN K, et al. Understanding the tumor immune microenvironment (TIME) for effective therapy[J]. Nature Medicine, 2018,24(5):541-550.
[22]SULLIVAN L B, GUI D Y, VANDERHEIDEN M G. Altered metabolite levels in cancer: implications for tumour biology and cancer therapy[J]. Nat Rev Cancer, 2016,16(11):680-693.
[23]OOI A, WONG J C, PETILLO D, et al. An antioxidant response phenotype shared between hereditary and sporadic type 2 papillary renal cell carcinoma[J]. Cancer Cell, Oct 18, 2011,20(4):511-523.
[24]SRINIVASAN R, RICKETTS C J, SOURBIER C, et al. New strategies in renal cell carcinoma:targeting the genetic and metabolic basis of disease[J]. Clin Cancer Res, 2015,21(1):10-17.
[25]MARGULIS V, LIN J, YANG H, et al. Genetic susceptibility to renal cell carcinoma:the role of DNA double-strand break repair pathway[J]. Cancer Epidemiol Biomarkers Prev, 2008,17(9):2366-2373.
[26]DIETLEIN F, THELEN L, REINHARDT H C. Cancer-specific defects in DNA repair pathways as targets for personalized therapeutic approaches [J]. Trends Genet, 2014,30(8),326-339.
[27]LIN J, PU X, WANG W, et al. Case-control analysis of nucleotide excision repair pathway and the risk of renal cell carcinoma[J]. Carcinogenesis, 2008,29(11):2112-2119.
[28]SAKANO S, HINODA Y, OKAYAMA N, et al.The association of DNA repair gene polymorphisms with the development and progression of renal cell carcinoma[J]. Ann Oncol, 2007,18(11):1817-1827.
[29]LIU Q J, SHEN H L, LIN J, et al. Synergistic roles of p53 and HIF1alpha in human renal cell carcinoma-cell apoptosis responding to the inhibition of mTOR and MDM2 signaling pathways[J]. Drug Des Devel Ther, 2016,10(1):745-755.
[30]WANG Z, PENG S, JIANG N, et al. Prognostic and clinicopathological value of p53 expression in renal cell carcinoma:a meta-analysis[J]. Oncotarget, 2017,8(60):102361-102370.
(本文編辑 于国艺)
