黄花大苞姜花药发育qRT-PCR内参基因筛选
2018-05-30张俊平王英强
张俊平 王英强


摘 要: qRT-PCR技術具有定量准确、灵敏度高、重复性好等特点,被广泛用于基因表达分析。内参基因的稳定性对于准确分析实验结果非常重要。该研究以黄花大苞姜(Caulokaempferia coenobialis)花粉母细胞时期(PMC)、四分体时期(TET)、成熟花粉时期(MP)的花药组织为材料,基于3个阶段花药转录组表达谱数据以及常用传统内参基因,筛选出Glyceraldehyde 3-phosphate dehydrogenase (GAPDH)、Malate dehydrogenase (MDH)、α-tubulin3 (TUA3)、β-tubulin7 (TUB7)和Actin6 (ACT6)作为候选内参基因,进行qRT-PCR分析;并运用BestKeeper、geNorm和Normfinder软件综合分析5个候选内参基因在黄花大苞姜花药发育过程中的表达稳定性。结果表明:MDH和TUB7的表达最稳定,ACT6的稳定性最差;分别以MDH和TUB7作为内参,分析GBE1在黄花大苞姜花药发育中的表达模式,并与该基因在花药转录组中的表达模式做相关系数分析,3种表达模式结果一致,进一步验证了MDH和TUB7的表达稳定性。这说明MDH和TUB7适合作为qRT-PCR分析黄花大苞姜花药发育过程中相关基因表达模式的内参基因。该研究结果为黄花大苞姜花药发育分子机制相关研究奠定了基础,也为姜科花药发育相关内参基因的选择提供了参考。
关键词: 黄花大苞姜, 花药, 内参基因, qRT-PCR, BestKeeper, geNorm, Normfinder
中图分类号: Q943.2
文献标识码: A
文章编号: 1000-3142(2018)01-0076-08
Selection of reference genes for quantitative real-time PCR during anther development of Caulokaempferia coenobialis (Zingiberaceae)
ZHANG Junping1, WANG Yingqiang1, 2*
( 1. Guangdong Provincial Key Laboratory of Biotechnology for Plant Development, College of Life Sciences, South China Normal University, Guangzhou 510631, China; 2. Guangzhou Key Laboratory of Subtropical Biodiversity and Biomonitoring College of Life Sciences, South China Normal University, Guangzhou 510631, China )
Abstract: Quantitative real-time PCR (qRT-PCR) has been widely used in gene expression analysis with quantitative accuracy, high sensitivity and good repeat ability. The stability of the reference genes is very important to accurate analysis of the target genes expression. Anther tissues from pollen mother cells stage (PMC), tetrad stage (TET) and mature pollen stage (MP) of Caulokaempferia coenobialis (Zingiberaceae) were used to select reference genes. According to the developing anther transcriptome expression profile data and traditional reference genes reported, Glyceraldehyde 3-phosphate dehydrogenase (GAPDH), Malate dehydrogenase (MDH), alpha tubulin3 (TUA3), beta tubulin7 (TUB7) and Actin6 (ACT6) were selected as candidate reference genes for qRT-PCR analysis. The expression stability of five candidate reference genes during the anther development was comprehensive analysis by BestKeeper, geNorm and Normfinder software. The results showed that the expression of MDH and TUB7 were the most stable, and the ACT6 was the worst. The expression patterns of GBE1 during anther development was obtained based on MDH and TUB7 as reference gene respectively. And the expression stability of MDH and TUB7 was further verified by correlation coefficient analysis (Pearson) with the anther transcriptome expression profile data. The results showed that MDH and TUB7 could serve as qRT-PCR reference gene to analyse the gene expression pattern related to anther developing in C. coenobialis. The study would provide research fundamental data for molecular mechanism research of anther development in C. coenobialis, and reference for the selection of reference genes in other Zingiberaceae species during anther development.
Key words: Caulokaempferia coenobialis, anther, reference gene, quantitative realtime PCR(qRT-PCR), BestKeeper, geNorm, Normfinder
黄花大苞姜(Caulokaempferia coenobialis)是中国特有的姜科大苞姜属植物,具有独特的花粉滑动自花授粉机制(Wang et al, 2004, 2005),花粉呈粘液状,与拟南芥、水稻的干性花粉显著不同。因此,其花药发育相关的研究,对于了解和补充精密的花药发育分子机制具有重要意义。但目前关于黄花大苞姜花药发育的相关研究却尚还未见有报道。 qRT-PCR技术具有定量准确、特异性强、灵敏度高及高通量等优点,被广泛用于基因表达分析(Nolan et al, 2006; Huggett et al, 2005)。但由于RNA提取质量、逆转录效率等因素无法完全统一,目的基因表达准确性会受到一定影响,因此不能真实反映基因的表达情况,需要内参基因来矫正和标准化,而一个好的内参可以消除这些影响(Vanguilder et al, 2008; Derveaux et al, 2010)。因此,筛选特定条件下最适内参基因对于准确分析目的基因的表达情况至关重要。
本研究基于黄花大苞姜花粉母细胞时期(Pollen mother cell stage,PMC)、四分体时期(Tetrad stage, TET)和成熟花粉时期(Mature pollen stage, MP)的花药转录组数据,结合已经报道的植物qRT-PCR内参基因(胡瑞波等,2009;牙库甫江等,2011)。筛选出了表达相对稳定的5个基因Glyceraldehyde 3-phosphate dehydrogenase(GAPDH)、Malate dehydrogenase(MDH)、α-tubulin3(TUA3)、β-tubulin7(TUB7)和Actin6(ACT6)作为候选内参基因,进行qRT-PCR分析。并运用BestKeeper(Pfaffl et al, 2004)、Normfinder(Andersen et al, 2004)和geNorm(Vandesompele et al, 2002)软件综合分析5个候选内参基因在黄花大苞姜花药发育过程中的表达稳定性,以筛选出表达最稳定的内参基因,为开展黄花大苞姜花药发育相关基因的研究奠定基础。
1 材料与方法
1.1 材料
本研究以黃花大苞姜不同发育阶段的花药组织为材料,于5—7月采自广东惠州南昆山自然保护区。收集花粉母细胞时期、四分体时期和成熟花粉时期的花药组织,液氮速冻后,于-80 ℃保存,直至提取RNA。每个发育阶段取3个生物学重复,共9个样品。花药发育阶段经醋酸洋红压片,显微观察确定。
1.2 总RNA提取和反转录cDNA合成
用Omega BioTek公司的Plant RNA Kit(America)试剂盒,参照说明书方案二(用于次生代谢物多的植物组织)提取花药组织的总RNA。RNase-free DNase 1(Takara Bio, Japan)去除RNA中的DNA。1.2%的琼脂糖凝胶电泳和Nano-100超微量分光光度计用于检测RNA的纯度和完整性。cDNA的合成采用PrimeScriptTM Ⅱ1st Strand cDNA Synthesis Kit(TaKaKa, Japan)试剂盒,取1.6 μg RNA,参照说明书反转录生成cDNA的第一条链,产物-20 ℃保存。
1.3 内参基因的选择及特异性引物设计
结合已经报道的常用内参基因,以及其在本实验室3个阶段发育花药的转录组数据中的表达情况,以基因在3个花药发育阶段的表达量(FPKM)差异倍数均小于2为标准。最终确定表达量差异较小的GAPDH、MDH、TUA3、TUB7、ACT6基因作为候选内参基因。特异性引物的设计采用NCBI内嵌的Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计,根据荧光定量引物设计原则,以及SYBR Premix Ex TaqTM II (Tli RNaseH Plus)荧光定量试剂对引物的要求选择最优引物,并将引物对转录组数据库比对,确定引物的专一性。引物信息见表1。
1.4 候选内参基因的qRT-PCR反应
采用BIO-RAD公司的CFX96TM Real-Time PCR Detection System进行荧光定量PCR反应,定量试剂采用SYBR Premix Ex TaqTM II (Tli RNaseH Plus)试剂盒(Takara Bio, Japan)。反应体系:cDNA 0.25 μL; SYBR Premix Ex Taq II(Tli RNaseH Plus)(2×)10 μL; Primer F(10 μmol·L-1)0.8 μL; Primer R(10 μmol·L-1)0.8 μL;加ddH2O补至20 μL体系。每个样品设3次技术重复。反应程序:预变性95 ℃ 30 s,40个循环;95 ℃ 5 s,60 ℃ 30 s。生成溶解曲线,用于判断扩增特异性。
1.5 引物扩增效率计算
等量取所有样品的cDNA,混合均匀后稀释5个梯度,每个梯度5倍,即模板浓度分别为初始浓度的1、1/5、1/52、1/53、1/54倍。每个反应设3个重复。用Bio Rad CFX Manager软件分析进行数据分析。以模板浓度的对数值为横坐标,以 Ct值为纵坐标绘制标准曲线,得到斜率K和相关系数R2。通过公式E=(5-1/k_1)×100%,计算引物扩增效率(E)。
1.6 数据处理与分析
采用BestKeeper、geNorm和Normfinder 3个软件对5个候选内参基因在黄花大苞姜花药发育过程中的表达稳定性进行分析。BestKeeper软件可直接采用基因表达Ct值进行分析,而geNorm和Normfinder软件需将Ct值经ΔCt方法转换后才能用于数据分析。
2 结果与分析
2.1 RNA质量检测
3个发育阶段的黄花大苞姜花药组织提取总RNA后,用1.2%琼脂糖凝胶电泳检测RNA完整性和纯度,结果显示RNA电泳条带清晰,无可见污染(图1)。Nano-100检测RNA的浓度和纯度,结果表明A260/280均在2.0~2.1之间,表明RNA完整性好。A260/230在1.9~2.1之间,表明纯度较高,可用于后续实验。
2.2 扩增效率与扩增特异性分析
以5倍为浓度梯度稀释5个梯度,qRT-PCR获得的GAPDH、MDH、TUA3、TUB7、ACT6的标准曲线斜率、扩增效率和相关系数(R2)见表1。5个候选内参基因中除ACT6扩增效率有点低只有77.8%,相关系数(R2)仅有0.889之外,GAPDH、MDH、TUA3、TUB7的引物扩增效率E均大于100%,相关系数(R2)均大于0.990,符合qRT-PCR对引物扩增效率的要求。溶解曲线分析结果表明,5个候选内参基因的溶解曲线均呈现明显单一的主峰,相同样品间的曲线重复性良好(图2),说明引物的特异性良好,可用于后续实验。
2.3 候选内参基因的表达水平分析
Ct值与基因的表达量呈反比,Ct值越大,基因的表达量越低;反之,Ct值越小,代表基因的表达量越高。5个候选内参基因在黄花大苞姜花药发育3个阶段的Ct值见图3,5个候选内参基因在黄花大苞姜不同花药发育阶段中表达的Ct值在18~27之间,其中ACT6和TUB7的表达量较低,GAPDH的表达量最高。
2.4 候选内参基因表达稳定性分析
BesterKeeper软件是基于内参基因Ct值的标准差(SD)和调节系数标准差SD(±x - fold)来评判基因的表达稳定性,直接对基因表达Ct值进行分析。SD值越小,表达越稳定,程序默认临界值为1,当SD值大于1时,认为该基因表达不稳定。分析结果显示,ACT6的SD值大于1,其余均小于1(表2),表明除ACT6之外,其余4个候选内参均符合作为内参基因的标准,表达最稳定的是TUB7。5个候选内参基因的稳定性排序依次为TUB7>MDH>GAPDH>TUA3>ACT6。
geNorm V3.5软件是基于平均表达稳定值M来确定候选内参基因的稳定性。qRT-PCR实验获得的Ct值,需经ΔCt方法转换后方可用于分析。 M值越大,基因的表达稳定性越低,M值越小,基因的表达越稳定。一般认为,M值等于1.5為基因表达稳定的界限,M值大于1.5的基因, 不宜作为内参基因。本研究5个候选内参基因的M值均小于不稳定的是ACT6。5个候选内参基因的表达稳定性依次为MDH/TUB7>GAPDH>TUA3>ACT6(图4:A)。此外,geNorm软件还可以通过计算候选内参基因的配对变异值Vn/n+1来分析最适内参基因数。
软件默认Vn/n+1临界值为0.15,即当Vn/n+1<0.15时,n个内参基因即可满足内参基因的要求。图4:B结果显示V3/4<0.15,即需要3个内参基因MDH、TUB7和GAPDH组合来作为内参基因。
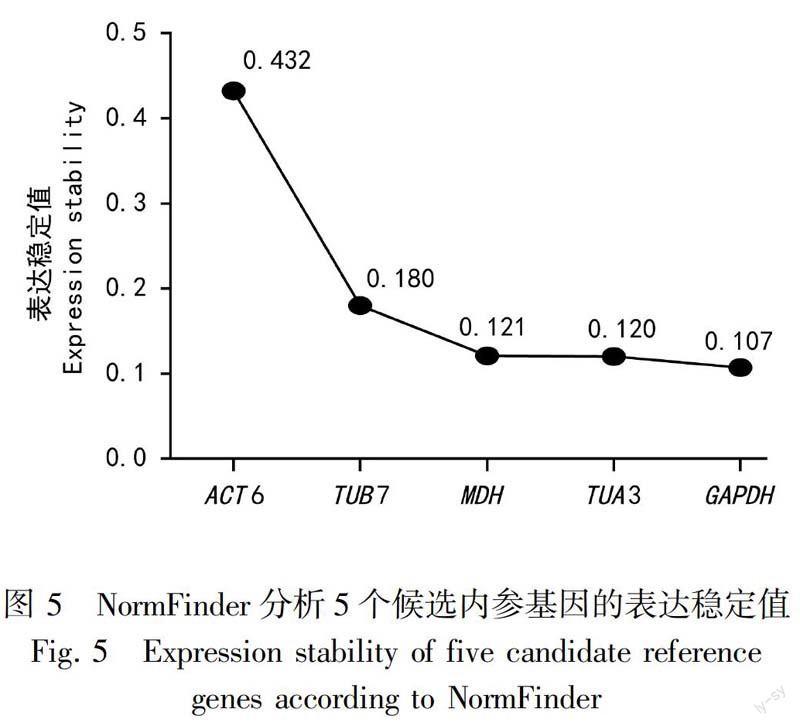
NormFinder是基于组内方差和组间方差来计算基因表达稳定性的,基因表达的Ct值也需要经ΔCt方法转换后方可用于分析。稳定值越大,代表基因的表达稳定性越差,反之,稳定性值越小代表,基因表达越稳定。图5结果表明ACT6的表达稳定性最差,GAPDH的表达稳定性最高。5个候选内参基因的表达稳定性依次为GAPDH>TUA3>MDH>TUB7>ACT6。
2.5 内参基因稳定性的验证
综合考虑BestKeeper、geNorm和Normfinder 3个软件的分析结果,认为MDH和TUB7在黄花大苞姜花药发育中表达相对最稳定。分别以MDH和TUB7作为内参基因,以PMC时期作为对照,采用ΔΔCT法,对黄花大苞姜花药发育过程中与淀粉和蔗糖代谢相关的1,4-α-葡聚糖支链酶基因(1,4-alpha-glucan branching enzyme,GBE1)在花药发育过程中的相对表达模式进行分析。并与花药转录组中该基因的表达模式(FPKM)两两做相关系数分析(Pearson),来验证筛选出的表达相对较稳定的内参基因的可靠性。结果表明,以MDH或TUB7作为内参基因获得的GBE1在花药发育中的表达模式,与该基因在转录组中的表达模式相关系数r分别为0.966和0.946,它们两个之间的相关系数r为0.998,表明3种途径获得的GBE1在黄花大苞姜花药发育中的表达模式一致,因此认为MDH和TUB7可作为分析黄花大苞姜花药发育过程中相关基因表达模式的内参基因。
3 讨论与结论
qRT-PCR技术已被广泛用于研究基因表达的研究,但是定量结果的准确性与内参基因的稳定性表达密切相关,而并不存在所谓恒定表达的内参基因(牙库甫江等,2011;Huis et al, 2010; Chen et al, 2011)。Mallona et al(2010)筛选矮牵牛叶和花发育中表达稳定的内参基因,NormFinder、BestKeeper、geNorm和qBasePlus 4种软件综合分析结果显示,EF1a在Mitchell品系中最稳定,CYP在V30品系中最稳定,GAPDH在两个品系中都最不稳定。Xu et al(2014)对中国白菜花蕾发育中qRT-PCR内参基因进行筛选,geNorm分析结果显示可育系与不育系花蕾发育过程中表达最稳定的是TUB和GAPDH。Jin et al(2013)分析了不同色系瓜叶菊花发育过程中的8个候选内参基因的表达稳定性,结果表明在所有样本中SAND和ACT最稳定,但是在不同色系中的结果有所不同,蓝色和粉红色品系中SAND和ACT最稳定,TIP41和ACT在白色系中最稳定,PP2A和ACT在洋红色系中最稳定。而本研究发现传统常用内参基因GAPDH、MDH、TUA3、TUB7、ACT6,在黄花大苞姜花药发育过程中表达最稳定的基因是MDH和TUB7,最不稳定的基因是ACT6。因此筛选特定条件下,稳定表达的内参基因对于准确分析目的基因的表达非常重要(Bustin et al, 2010)。
NormFinder、BestKeeper和geNorm软件是目前最常用于评估内参稳定的软件,但是由于采用不同的算法(Pfaffl et al, 2004;Andersen et al, 2004;Vandesompele et al, 2002),对内参基因稳定性的分析结果会有一些差异,目前已经有基于这些软件的研究与分析,并得到了稳定表达的内参基因(李晗等,2016;刘文哲等,2016;苏晓娟等,2013;刘艷霞等,2016)。本研究也采用了这3个软件对GAPDH、MDH、TUA3、TUB7、ACT6在黄花大苞姜花药发育中的表达稳定性进行了分析。BestKeeper分析结果显示,除了ACT6以外,其余4个基因均符合作为内参的要求,MDH和TUB7的表达最稳定,ACT6的表达稳定性最差。geNorm分析认为5个候选内参基因都符合作为内参的标准,MDH和TUB7的表达最稳定,ACT6的表达稳定性最差。而NormFinder则分析认为表达最稳定的基因是GAPDH和TUA3,ACT6表达稳定性最差,但是MDH与TUB7的稳定性值与最稳定的GAPDH的差异不大。本研究选择了表达较为稳定的MDH和TUB7分别作为内参基因分析GBE1在黄花大苞姜花药发育中的表达模式,并与该基因在花药发育转录组中的表达模式做相关系数分析,该基因的3种表达模式结果一致。因此,认为MDH和TUB7可作为qRT-PCR分析黄花大苞姜花药发育相关基因表达情况的内参基因。
参考文献:
ANDERSEN CL, JENSEN JL,NTOFT TF, 2004. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon canc data sets [J]. Cancer Res, 64: 5245-5250.
BUSTIN SA, BEAULIEU JF, HUGGETT J, et al, 2010. MIQE précis: Practical implementation of minimum standard guidelines for fluorescence-based quantitative real-time PCR experiments [J]. BMC Mol Biol, 11(1):1-5.
CHEN K, FESSEHAIE A, ARORA R, 2011. Selection of reference genes for normalizing gene expression during seed priming and germination using qPCR in Zea mays and Spinacia oleracea [J]. Plant Mol Biol Rep, 30(2): 478-487.
DERVEAUX S, VANDESOMPELE J, HELLEMANS J, 2010. How to do successful gene expression analysis using Real-time PCR [J]. Methods, 50(4): 227-230.
HU RB, FAN CM, FU YF, 2009. Reference gene selection in plant real-time quantitative reverse transcription PCR (qRT-PCR) [J]. J Agric Sci Technol, 11(6): 30-36. [胡瑞波, 范成明, 傅永福, 2009. 植物实时荧光定量PCR内参基因的选择 [J]. 中国农业科技导报, 11(6): 30-36.]
HUGGETT J, DHEDA K, BUSTIN S, et al, 2005. Real-time RT-PCR normalization; strategies and considerations [J]. Genes Imm, 6(4): 279-284.
HUIS R, HAWKINS S, NEUTELINGS G, 2010. Selection of reference genes for quantitative gene expression normalization in flax (Linum usitatissimum L.) [J]. BMC Plant Biol, 10(1): 1-14.
JIN X, FU J, DAI S, et al, 2013. Reference gene selection for qPCR analysis in cineraria developing flowers [J]. Sci Hortic, 153(153): 64-70.
LI H, LI ZL, LI XY, et al, 2016. Selection of reference genes for real-time fluorescence quantitative PCR in different tissues and stigma development from ornamental kale [J]. Bull Bot Res, 36(4): 565-572. [李晗, 李治龙, 李晓屿, 等, 2016. 羽衣甘蓝不同组织及柱头发育实时荧光定量PCR内参基因的筛选 [J]. 植物研究, 36(4): 565-572.]
LIU YX, LAN XX,CAO J, et al, 2016. Screening of qRT-PCR rerference genes for Chenopodium album and C. glaucum of Chenopodiaceae [J]. Guihaia, 36(12): 1511-1518. [刘艳霞, 兰欣欣, 曹婧, 等, 2016. 黎科植物藜和灰绿藜实时荧光定量PCR内参基因的选择 [J]. 广西植物, 36(12): 1511-1518.]
LIU WZ, NIU MY, LI XY, et al, 2016. The selection of reference genes for quantitative PCR in Betula luminifera [J]. Sci Silv Sin, 52(8):29-37. [刘文哲, 牛明月, 李秀云, 等, 2016. 光皮桦实时荧光定量PCR内参基因的筛选 [J]. 林业科学, 52(8): 29-37.]
MALLONA I, LISCHEWSKI S, WEISS J, et al, 2010. Validation of reference genes for quantitative real-time PCR during leaf and flower development in Petunia hybrida [J]. BMC Plant Biol, 10(1): 4.
NOLAN T, HANDS RE, BUSTIN SA, 2006. Quantification of mRNA using real-time RT-PCR [J]. Nat Protocols, 1(3): 1559-1582.
PFAFFL MW, TICHOPAD A, PRGOMET C, et al, 2004. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper—Excel-based tool using pair-wise correlations [J]. Biotechnol Lett, 26: 509-515.
SU XJ, FAN BG,YUAN LC,et al, 2013. Selection and validation of reference genes for quantitative RT-PCR analysis of gene expression in Populus trichocarpa [J]. Chin Bull Bot, 48(5): 507-518. [苏晓娟, 樊保国, 袁丽钗, 等, 2013. 实时荧光定量PCR分析中毛果杨内参基因的筛选和验证 [J]. 植物学报, 48(5): 507-518.]
VANDESOMPELE J, PRETER KD, PATTYN F, et al, 2002. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes [J]. Genome Biol, 3(7):1-11.
VANGUILDER HD, VRANA KE, FREEMAN WM, 2008. Twenty-five years of quantitative PCR for gene expression analysis [J]. Biotechniques, 44(5): 619-626.
WANG YQ, ZHANG DX, RENNER SS, et al, 2004. A new self-pollination mechanism [J]. Nature, 431(7004): 39-40.
WANG YQ, ZHANG DX, RENNER SS, et al, 2005. Self-pollination by sliding pollen in Caulokaempferia coenobialis (Zingiberaceae) [J]. Int J Plant Sci, 166(5): 753-759.
XU X, YANG Z, SUN X, et al, 2014. Selection of reference genes for quantitative real-time PCR during flower bud development in CMS7311 of heading Chinese cabbage (Brassica rapa L. ssp. pekinensis ) [J]. Acta Physiol Plant, 36(3): 809-814.
YAKUPJAN, HAXM, GUAN B, et al, 2011. Research progress in plant reference genes [J]. Biotechnol Bull, (7): 7-11. [牙庫甫江, 阿西木, 关波, 等, 2011. 植物基因表达转录分析中内参基因的选择与应用 [J]. 生物技术通报, (7): 7-11.]
