缺血性脑卒中患者的循环免疫circRNA-miRNA-mRNA轴预测*
2024-02-26薛如卉张广炜
薛如卉,刘 丹,张广炜
(1.内蒙古科技大学包头医学院,内蒙古包头 014040;2. 内蒙古科技大学包头医学院第一附属医院神经内三科)
急性缺血性卒中(Acute Ischemic Stroke,AIS)是一种由大脑血液流量减少,导致脑细胞损伤引起的医疗紧急情况[1]。最近的研究表明,对急性缺血性卒中的早期识别、紧急介入治疗以及在专门的卒中中心进行治疗可以显著降低卒中相关的发病率和死亡率[2]。MRI检测急性缺血性卒中的敏感度优于CT,是急性缺血性卒中诊断的重要手段[3]。由于计算机断层扫描(CT)经济、便捷,CT仍然作为卒中诊断的首选影像学方法[4]。然而,由于院前快速识别卒中的方法有限,对AIS患者的及时干预往往受到阻碍。因此,目前迫切需要一种高效且便捷的方法用于急性缺血性脑卒中的快速诊断。随着测序技术和生物信息学的发展,越来越多研究提出AIS患者在外周血中差异表达某些circRNAs[5-6]、miRNAs[7]可能是诊断AIS的生物标志物或潜在的治疗靶点。据此,本研究采用生物信息学方法,预测可能的免疫相关circRNA-miRNA-mRNA调控网络,分析circRNAs、miRNAs在缺血性卒中的分子诊断和个体化治疗中的作用。
1 材料与方法
1.1微阵列数据 从基因表达综合数据库(GEO)中获得了IS的3个微阵列数据集GSE16561、GSE195442和GSE117064。GSE16561基于GPL6883平台,包括63个外周血样本,病例组(共39例)入组标准为:年龄>18岁,MRI诊断为缺血性卒中,对照组(共24例)为非卒中神经健康者。GSE195442是基于GPL31275平台,采用芯片技术对10例急性缺血性卒中患者和10例对照组患者的血浆外泌体进行微阵列和生物信息学分析,研究了血浆外泌体中circRNA的图谱和推定功能。GSE117064基于GPL21263平台,总共1 785份样本的血清microRNA谱,其中包括173名脑血管疾病患者,1 612名非脑血管疾病对照。
1.2方法
1.2.1差异表达基因(Differentially Expressed Genes,DEGs)的鉴定 使用R软件的limma包对下载的数据集GSE16561进行差异表达基因(DEGs)的筛选。两组间的DEGs采用学生t检验和log2fold change(FC)值进行进一步的筛选。在阈值P-Value<0.05和|log2foldchange(FC)|>0.5的情况下,通过R软件ggplot2包构建火山图,识别具有统计学意义的DEGs。
1.2.2PPI网络建设及模块分析 DEGs的PPI网络是通过STRING v11.5在线工具[相互作用基因检索搜索工具,STRING(https://string-db.org/)]构建的。利用Cytoscape 3.9.1插件CytoNCA中的介数中心度(Betweenness Centrality)计算了PPI网络的拓扑结构。根据中心性评分,确定PPI网络中的关键节点,然后推导出评分较高的前15个基因。
1.2.3功能富集分析 基于DAVID数据库(https://david.ncifcrf.gov/),我们对15个关键基因进行了GO和KEGG通路富集分析,GO分析包括以下领域:生物过程(Biological Process,BP)、细胞组分(Cellular Component,CC)和分子功能(Molecular Function,MF),P-Value<0.05的具有统计意义。
1.2.4与靶基因相关的miRNAs 使用mirTargets 2.0,一个发现基因调控网络中miRNA-mRNA相互作用的平台,将前15个中心基因定位到相应的miRNAs上,构建miRNAs-mRNAs调控网络。
1.2.5差异表达circRNAs的筛选及靶标miRNAs的预测 采用GEO2R对数据集GSE195442中的IS组和对照组的血清样本进行差异circRNAs的分析。具有P-Value<0.05和|log2foldchange(FC)|>1的circRNAs被认为是差异表达的circRNAs。结合Yang等人[8]的文献挑选出显著差异[P-Value<0.05,|log2fold change(FC)|>1]并且已经经过PCR验证过的差异circRNAs。使用Circular RNA Interactome 预测circRNAs的靶标miRNAs。通过Cytoscape 3.9.1绘制miRNAs和circRNAs调控网络。
1.2.6circRNA-miRNA-mRNA调控网络预测 分别对根据基因预测的miRNAs和根据circRNAs预测的miRNAs取交集,预测可能的circRNA-miRNA-mRNA调控网络。使用在线可视化软件Draw Venn Diagram生成miRNAs的维恩图。预测可能的circRNA-miRNA-mRNA调控网络。
1.2.7免疫相关circRNA-miRNA-mRNA调控网络的筛选 在ImmPort数据库中获得了免疫相关基因(Immune-related genes,IRGs),并将IRGs与我们预测可能的circRNA-miRNA-mRNA调控网络中的基因取交集,筛选出免疫相关的circRNA-miRNA-mRNA调控网络。
1.2.8免疫相关circRNA-miRNA-mRNA调控网络中的miRNAs验证及构建 使用GSE117064数据集对circRNA-miRNA-mRNA调控网络中的miRNAs进行验证,并且进一步构建可能的免疫相关circRNA-miRNA-mRNA调控网络。
2 结果
2.1IS的差异基因(DEGs)的鉴定 根据限制条件,在GEO数据库中有GSE16561,包括对照和IS样本。测序信息来自于人外周血单个核细胞。GSE16561芯片属于GPL6882,其中包括24个对照样本和39个IS样本。根据P-Value<0.05和|log2fold change(FC)|>0.5的标准,在GSE16561数据集中,获得了651个DEGs,其中502个表达上调,149个表达下调。
2.2PPI网络的构建与枢纽基因的鉴定 用v11.5 STRING构建DEGs的PPI网络,用Cytoscape 3.9.1进行可视化。利用Cytoscope 3.9.1的CytoNCA确定PPI网络的关键节点,获得STAT3、PTEN、FOXO3、CREBBP、GLTSCR2等15个关键基因。根据Cytoscope 3.9.1产生的节点度评分,确定潜在的前15个枢纽基因。结果显示,STAT3(评分12 023.7)和PTEN分子(评分11 404.3)是最显著的基因。其余为FOXO3(评分7 853.2)、CREBBP(评分7 664.1)、GLTSCR2(评分5 516)、RPL3(评分5 148)、STAT1(评分4 869.4)、MYH9(评分4 324.8)、SYNJ1(评分4 311.4)、ITGAM(评分3 988.3)、IL2RB(评分3 768.2)、AGO2(评分3 201.6)、MMP9(评分3 143.9)、TLR2(评分2 963.1)、CD247(评分2 923.0)。见图1。

图1 前15个关键基因
2.3GO功能富集分析和KEGG富集途径 采用David平台对15个关键基因进行GO功能富集分析,获取48个显著差异的GO项目,包括6个细胞组分(Cell Component,CC)、33个生物过程(Biological Process,BP)(取前10个)、9个分子功能(Molecular Function,MF)。DEGs介导的BP主要集中在RNA聚合酶Ⅱ启动子转录的正向调控、信号转导、基因表达的正向调控等方面。CC的发生结果主要集中在胞液、细胞质等部位。MF的结果表明,蛋白结合、相同的蛋白质结合是主要富集项目。KEGG富集途径的结果显示,DEGs主要参与癌症通路、PD-L1在癌症中的表达和PD-1检查点通路、乙型肝炎、Th17细胞分化。见图2~图5。

图2 GO功能富集分析(BP)

图3 GO功能富集分析(CC)

图4 GO功能富集分析(MF)
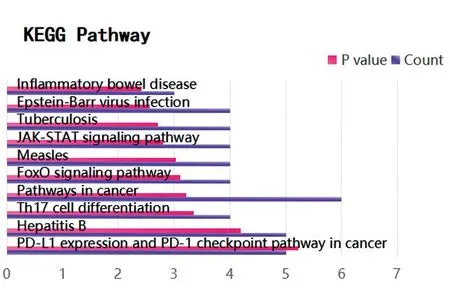
图5 KEGG富集分析
2.4整合的miRNA-mRNA调控网络 根据mirTargets 2.0网络分析数据库预测前15个关键基因的靶标miRNAs,构建预测的miRNA-mRNA调控网络。
2.5差异表达circRNAs的筛选及靶标miRNAs的预测 采用GEO2R对IS组和对照组的血清样本进行差异circRNAs的分析,具有P-Value<0.05和|log2fold change(FC)|>1的circRNAs被认为是差异表达的circRNAs。结合文献挑选出显著差异[P-Value<0.05,|log2fold change(FC)|>1]并且已经经过PCR验证过的差异circRNAs。使用Circular RNA Interactome 预测目标miRNAs。见图6。
2.6circRNA-miRNA-mRNA调控网络预测 使用在线可视化软件Draw Venn Diagram生成维恩图。分别对根据基因预测的miRNAs和根据circRNAs预测的miRNA取交集,预测可能的circRNA-miRNA-mRNAs调控网络。
2.7免疫相关circRNA-miRNA-mRNA调控网络的筛选 在ImmPort数据库中获得了免疫相关基因(IRGs),并将IRGs与我们预测可能的circRNA-miRNA-mRNA调控网络中的基因取交集。结果表明,重叠基因CD247、IL2RB、STAT1、STAT3被鉴定为免疫相关枢纽基因,之后我们在预测的circRNA-miRNA-mRNA调控网络中筛选出了免疫相关circRNA-miRNA-mRNA调控网络。
2.8免疫相关circRNA-miRNA-mRNA调控网络中的miRNAs验证 使用GSE117064数据集对circRNA-miRNA-mRNA调控网络中的miRNAs进行验证[P-Value<0.05,|log2fold change(FC)|>0.5],提示hsa-miR-892a、hsa-miR-1276、hsa-miR-938、hsa-miR-1208、hsa-miR-338-3p在缺血性卒中患者体内表达上调,hsa-miR-1248在缺血性卒中患者体内表达下调。
2.9免疫相关circRNA-miRNA-mRNA调控网络的构建 研究发现hsa_circ_0112036在缺血性卒中患者体内表达下调,hsa-miR-892a、hsa-miR-1276、hsa-miR-938、hsa-miR-1208、hsa-miR-338-3p在缺血性卒中患者体内表达上调,CD247、IL2RB在缺血性卒中患者体内表达下调。相反,hsa_circ_0093708在缺血性卒中患者体内表达上调,hsa-miR-1248在缺血性卒中患者体内表达下调,STAT1在缺血性卒中患者体内表达上调。发现hsa_circ_0112036可能作为hsa-miR-892a、hsa-miR-938、hsa-miR-1276、hsa-miR-1208、hsa-miR-338-3p的分子海绵调控缺血性卒中的发生发展。hsa_circ_0093708可能作为hsa-miR-1248的分子海绵调控缺血性卒中的进展。并且构建了免疫circRNA-miRNA-mRNA调控网络。然而,这些因子还需要在未来的研究中得到进一步的验证。见表1。

表1 预测的循环免疫circRNA-miRNA-mRNA网络关键因子
3 讨论
卒中仍然是世界范围内的主要死亡原因之一[9],目前急性缺血性卒中(Acute ischemic stroke,AIS)患者的数量仍然普遍,因此发现一种新的机制来解决AIS至关重要。近年来,针对IS发生发展的分子机制研究已经取得显著性的成就,许多研究者报道了microRNA调控IS。例如,Tiedt等人[7]发现循环的microRNA(miR-125a-5p、miR-125b-5p和miR-143-3p)与AIS相关,可能作为AIS早期诊断标志物。Burlacu等人[10]荟萃分析提示血清miR-9和中性粒细胞miR-29b是深入随访研究IS最有希望的生物标志物,相比microRNAs,circRNAs具有高稳定性[11],并且circRNAs具有作为miRNA海绵的潜力[12],机制上,越来越多的研究结果表明circRNAs通过作为miRNAs的分子海绵参与IS的发生发展。有研究表明,circ_0101874过表达通过靶向miR-335-5p来增强PDE4D表达,以促进缺血性脑卒中的神经元损伤[13]。circPRDX3通过海绵miR-641抑制IS的发展,从而增加NPR3表达并使MAPK途径失活[14]。这些结果可能有助于寻找IS的潜在治疗靶点。
目前,缺血性脑卒中中越来越多的circRNA-miRNA-mRNA调控网络被发现。Wang等人[15]成功构建了circRNA介导的免疫相关内源竞争RNA(competing endogenous RNA,ceRNA)网络。Cao等人[16]构建了免疫相关ceRNA网络,从分子机制上为调控IS提供了新的看法。研究表明,circ_0006459的消耗通过miR-940/FOXJ2通路保护人脑微血管内皮细胞(Human brain microvascular endothelial cells,HBMEC)免受氧-葡萄糖剥夺(Oxygen-glucose deprivation,OGD)诱导的损伤,为缺血性卒中提供了可能的治疗靶点[17]。circFUNDC1敲低通过miR-375抑制PTEN,减轻氧葡萄糖剥夺引起的人脑微血管内皮细胞损伤[18]。circUSP36 通过 miR-139-3p/SMAD3/Bcl2 信号轴减轻缺血性卒中损伤[19]。circVRK1敲低通过调节miR-150-5p/MLLT1通路削弱了HBMEC中OGD诱发的损伤,可能为IS治疗提供了靶点[20]。此外,药物也可以通过circ_0008146/miR-709/Cx3cr1轴发挥神经保护作用,抑制脑缺血/再灌注损伤后的细胞凋亡和炎症[21]。
目前关于非编码RNA以及circRNA-miRNA-mRNA网络与免疫反应的研究也越来越多。研究表明,circRNA以及circRNA-miRNA-mRNA网络被发现可以在免疫浸润、免疫逃逸、免疫治疗等多方面调节免疫反应。例如,Chen等人[22]将circPRDM4鉴定为肝细胞癌中缺氧相关的circRNA,circPRDM4被发现在缺氧状态下通过促进HIF-1α募集到CD274启动子上,从而调节肝细胞癌PD-L1,促进肝细胞癌中的细胞免疫逃逸。circCRIM1的沉默驱动IGF2BP1介导的非小细胞肺癌免疫逃逸[23]。在免疫浸润方面,Ying等人[24]发现FAS介导的circRNA-miRNA-mRNA网络可以调节脑梗死中的免疫细胞浸润。在免疫治疗方面,有研究表明,HNRNPL诱导的circFAM13B通过新型circFAM13B/IGF2BP1/PKM2级联反应,最终抑制免疫逃逸,增强免疫治疗敏感性,因此,circFAM13B可作为指导膀胱癌患者免疫治疗的生物标志物[25]。有趣的是,microRNA也可以参与调节免疫反应。通过影响免疫浸润,被microRNA-92a家族调节的SYT1,NEFM,GAP43和GRIA1被确定为帕金森病的诊断生物标志物[26]。miR-155被发现和炎症、免疫密切相关,miR-155的免疫和炎症作用表现为调节巨噬细胞极化、NK细胞杀伤、Th17细胞和Th1/Th2细胞分化[27]。综上所述,非编码RNA以及circRNA-miRNA-mRNA网络在免疫中发挥了重要的调节作用,免疫反应又作为缺血性卒中病理生理过程中重要的环节,所以进一步研究与免疫相关的非编码RNA以及circRNA-miRNA-mRNA网络,为解决缺血性卒中提供了新的思路,既与缺血性脑卒中相关又与免疫相关的非编码RNA,可能成为缺血性脑卒中治疗的新兴生物标志物。
本研究采用生物信息学方法,预测可能的免疫相关circRNA-miRNA-mRNA调控网络。研究发现hsa_circ_0112036可能作为hsa-miR-892a、hsa-miR-938、hsa-miR-1276、hsa-miR-1208、hsa-miR-338-3p的分子海绵调控缺血性卒中的发生发展。hsa_circ_0093708可能作为hsa-miR-1248的分子海绵调控缺血性卒中的进展。尽管如此,所预测的免疫相关circRNA-miRNA-mRNA调控网络还需要在未来的研究中得到进一步的验证。基因调控网络在IS的病理生理过程中扮演着重要的角色。这一发现将有助于更好地了解脑卒中的发病机制,并为IS提供不同的治疗角度。本研究也存在一些局限性,一方面,目前的研究涉及基因数量仍然有限;另一方面,在构建的circRNA-miRNA-mRNA网络中,还需进一步的实验验证。
综上所述,本研究在IS中发现了潜在的circRNA-miRNA-mRNA免疫相关调控网络,且在文献中尚未见报道。免疫相关circRNA-miRNA-mRNA调控网络的可能性及其生物学意义仍有待进一步研究,为今后从机制上寻找circRNA、miRNA与IS的关系提供更多可能。
