基于WGCNA的谷子苗期冷胁迫应答基因网络构建与核心因子发掘
2023-11-02张会王越越赵波张丽玲郄倩茹韩渊怀李旭凯
张会, 王越越, 赵波, 张丽玲, 郄倩茹, 韩渊怀, 李旭凯
(1.山西农业大学生命科学学院,山西 太谷 030801; 2.山西农业大学山西省后稷实验室,太原 030031;3.山西农业大学农学院,山西 太谷 030801)
谷子(Setaria italica)古称稷、粟、粱,是起源于我国黄河流域的古老作物,一万多年前由青狗尾草(Setaria viridis)驯化而来[1]。谷子是二倍体(2n=2X=18),基因组较小(约440 Mb)[2-4],具有耐旱、耐贫瘠、适应性强等特点,被誉为“五谷之首”,与C3 模式作物水稻(Oryza sativa)“优势互补”“水旱对照”“南北呼应”,成为旱生C4 禾谷类作物分子育种研究的模式植物[5]。本实验室通过对‘晋谷21’进行EMS(ethylmethane sulfonate)诱变获得矮秆超早熟突变体材料xiaomi,xiaomi生育期短,约60 d,其全基因组测序的完成为本研究奠定了基础[4]。
植物生存缺乏自主移动性,易遭受低温、干旱、盐碱等非生物胁迫,严重影响植物的生长发育[6]。低温胁迫对作物整个生育过程都会造成不同程度的影响,如种子萌发、植株生长发育、产量和品质形成等[7]。近年来,国内外多个研究团队在水稻耐冷相关的数量性状位点(quantitative trait locus,QTL)克隆与调控机制解析等方面取得了一定进展[8]。水稻低温感受器基因COLD1编码膜定位蛋白,可与G 蛋白α 亚基RGA1 互作以感知低温,激活Ca2+通道,并增强G 蛋白GTP 酶活性;COLD1能够正向调控下游转录因子OsDREB1B和OsDREB1C的表达,并偶联叶绿体中的维生素E-维生素K1 代谢调控网络,进而调控水稻的耐冷性[9-10]。Ca2+作为第二信使,在冷信号的激活和转导中发挥重要作用。研究发现,过表达膜定位的钙通道蛋白OsCNGC9 可以增强水稻幼苗的耐冷性:在转录水平,OsCNGC9受转录因子OsDREB1A的直接转录激活调控;在蛋白水平,激酶OsSAPK8 可与OsCNGC9 互作并使其磷酸化,进而激活Ca2+的内流[11];AP2 转录因子家族的CBF/DREBs 转录因子在单、双子叶植物中高度保守,可以促进多个冷相关基因(COR)的表达,增强植株的耐冷性[12]。此外,OsWKRY71/94/45/76、OsMYB3R-2/30、OsTCP-5/6/8/21、OsbZIP38/52/71/73、OsSPL3/14/17、OsMADS57和OsSLR1等转录因子基因均直接参与水稻的低温胁迫调控[13]。玉米中也有较多耐冷相关QTL 定位和候选基因预测的报道,但克隆的基因较少[14]。谷子中耐冷基因定位及克隆的研究较为匮乏。因此,挖掘谷子中的耐冷基因有助于进一步探索谷子的抗逆机理,阐明其分子遗传机制,有助于推进谷子耐冷遗传改良进程。
加权基因共表达网络分析(weighted gene coexpression network analysis, WGCNA)是在鉴定出表达模式相似的基因集合(module)的基础上,解析不同基因集合与不同分组样品表型之间的关联性,绘制基因集合之间的调控网络并鉴定关键调控基因的方法[15]。该方法多被用于研究共表达网络与植物性状之间的生物学关系,能够辅助挖掘与目标性状高度关联的核心基因。
本研究利用谷子的根、叶、穗、茎等不同组织、不同冷处理条件下的33 份转录组数据,利用WGCNA 方法构建谷子共表达网络模块,并进行差异表达分析,将冷胁迫处理组与对照组进行比较,设置错误发现率(false discovery rate,FDR)阀值为0.01 筛选差异,与WGCNA 模块相结合,在模块中找出谷子抗冷基因,预测与谷子耐冷相关的候选基因,为进一步研究谷子的抗逆机理奠定基础。
1 材料与方法
1.1 谷子RNA-Seq数据的获取与分析
谷子品种‘豫谷1 号’(Yugu 1)冷胁迫处理的部分转录组数据中来源于NCBI(national center for biotechnology information)的SRA(sequence read archive)数据库(https://www.ncbi.nlm.nih.gov/sra/),从‘豫谷1 号’12 d 的幼苗期开始,分别在6 ℃冷胁迫处理0.0、0.5、1.0、3.0、6.0、16.0、24.0 h,在每个时间点分别从对照组和冷胁迫处理组同时取样,进行RNA 测序[16]。xiaomi突变体的11 个不同组织、时期的转录组数据来自于本实验室[4],数据已上传NGDC(national genomics data center)的GSA(genome sequence archive)数 据 库(https://bigd.big.ac.cn/);其他部分为本实验室RNA-seq 测序所得。
将‘晋谷21’(JG21)、‘牛毛白’(NMB)和‘平定大谷’(PD)种子播种于山西省太谷县(37°25′13″ N,112°35′26″ E)大田。取‘晋谷21’出苗2周整株幼苗的混合叶、种植30 d的倒2叶及灌浆期S2、S4 时期未成熟的种子;对于‘牛毛白’及‘平定大谷’均只取灌浆期S2、S4 时期未成熟的种子。将所有样品取样后立即置于液氮中,随后采用RNAprep 纯植物试剂盒(天根生化科技有限公司)分离总RNA,并送样测序,每个样本3 个生物学重复。
对于NCBI 下载的数据首先将SRA 格式文件利用SRAtoolkit 软件的fast-dump 命令转换为fastq序列文件[17]。所有的测序原始数据利用FastQC对其进行质控[18],然后利用Trimmomatic 软件(软件中参数threads 设为10,phred 设为33)去除接头,过滤掉低质量的reads,得到clean data[19]。利用Hisat2(参数phred 设为12)将clean data 比对到本团队构建的谷子超早熟突变体xiaomi高质量基因组[20]。利用featurecounts[21]对数据的reads计数,之后根据基因表达的TPM(transcripts per million)值,编写计算TPM 值的R 语言代码,构建基因表达矩阵[22]
式中,xi和xj分别表示比对到基因i上的reads数和比对到任一基因j上的reads 数;Li表示基因i的外显子长度的总和;Lj表示任一基因j的外显子长度总和。
1.2 共表达网络构建
基因的表达量矩阵来自于谷子不同组织不同处理下的基因表达量。参照Langfelder[23]的方法,使用R 软件(R version 3.4.4)中的WGCNA 包(R version 1.6.6)构建加权基因共表达网络。利用WGCNA 包中的pickSoftThreshold 函数计算权重值,根据谷子转录组数据不同软阈值下的网络拓扑结构分析结果,选择软阈值power=12,使用默认参数利用函数blockwiseModules 构建无尺度网络。
1.3 谷子耐冷基因检索及差异表达基因分析
通过国家水稻数据中心(http://www.ricedata.cn/ontology/)检索已报道的水稻耐冷相关基因,通过blastp 将其蛋白序列与谷子xiaomi的蛋白库进行比对[24],获取与水稻耐冷相关基因的蛋白序列相似性最高的谷子基因,统计这些基因分布最多的3个模块。
将count 数据文件在百迈克云平台进行基因差异表达分析,采用FDR 来筛选差异,设置FDR阀值为0.01 且|log2FC|>2 进行差异表达基因分析,用在线软件(http://jvenn.toulouse.inra.fr/app/example.html)制作韦恩图[25],并统计差异表达基因在上述3个模块中的分布数目。
1.4 模块的GO富集分析
提取出含冷胁迫基因最多的3 个模块,利用TBtools 软件对3 个模块中的所有基因进行GO 富集分析[26];之后选出模块内与冷GO注释相关的基因,利用Cytoscape(version 3.6)对模块中的这些基因进行可视化[27]。
1.5 冷胁迫候选基因功能注释
将核心基因在水稻(https://rapdb.dna.affrc.go.jp)及玉米数据库(https://www.maizegdb.org)中进行同源比对,设置阈值E-value<10-5且Identity>85%进行筛选,参照水稻和玉米中的同源基因功能进行注释。
1.6 冷胁迫应答候选基因的实时荧光定量PCR(RT-qPCR)分析
为了验证冷胁迫应答候选基因的表达模式,对其中6个候选基因进行了RT-qPCR分析。将‘预谷1 号’播种12 d 后对其进行6 ℃冷胁迫处理,分别在处理0、3、6、24 h时,从对照组和冷胁迫处理组取样(地上部),每组样本取3个生物学重复,使用博迈德Green qPCR Master Mix 荧光定量试剂盒进行RT-qPCR 验证,检测候选基因对冷胁迫的响应情况。用NCBI 的Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)进行引物设计,如表1 所示。内参基因为谷子Actin基因,并采用2-ΔΔCt方法[28]计算基因的相对表达量。

表1 RT-qPCR引物序列Table 1 RT-qPCR primer sequence
2 结果与分析
2.1 谷子加权基因共表达网络构建
利用WGCNA 包中的goodSamplesGenes 函数检测并过滤掉表达量偏低的基因,最终获得32 017 个高表达基因。利用WGCNA 包内的pickSoftThreshold 函数计算并选择权重值β=12(R2=0.8),以实现所有基因构成的关系网络符合无尺度网络分布。按权重值β=12,计算并获得每2 个基因间的拓扑重叠矩阵(topological overlap matrix,TOM);之后采用动态剪切算法进行共表达网络模块划分,最终得到44个共表达模块,如图1所示,图中每种颜色代表聚成一簇的基因所形成的模块。其中darkturquoise 模块包含的基因最多,有7 415 个;navajowhite 模块包含的基因最少,仅37个。
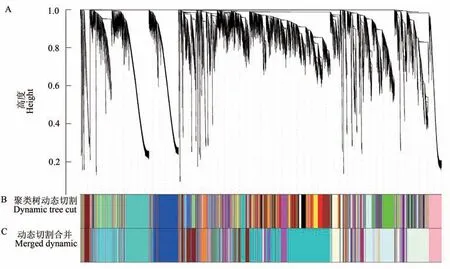
图1 基因聚类树和样品分割Fig. 1 Gene cluster dendrogram and module detecting
2.2 冷胁迫相关模块提取
利用水稻已报道的与冷胁迫直接相关的基因在谷子数据库中进行检索比对,共鉴定到68 个冷胁迫候选基因。通过映射查找,这些基因分布于18 个共表达网络模块,其中,darkturquoise、turquoise、blue 模块中所含的耐冷基因数量较多,分别为13、10、8个。
为了评估冷胁迫相关基因映射选取模块的准确性,对谷子冷胁迫处理0.5、1.0、3.0、6.0、16.0和24.0 h 的转录组数据进行差异表达基因分析。结果显示,turquoise 模块中不同处理下的差异基因数量最多,占所有差异基因数的12.69%;darkturquoise 模块中的差异基因数次之,占12.06%;blue模块中差异基因数占7.68%。
对6 种冷胁迫处理的差异表达基因进行分析(图2),发现有9 个基因在6 种冷胁迫时间下均差异表达。通过同源比对,这9 个基因在水稻和拟南芥(Arabidopsis thaliana)中的同源基因主要涉及TPR、AP2、WRKY、bHLH、TIFY、PAO2 和MYB家族以及赤霉素、冷调控等相关功能(表2),这些家族均与植物非生物胁迫应答相关[29-31]。

图2 谷子6个冷胁迫时间差异表达基因分析Fig. 2 Analysis of differentially expressed genes in foxtail millet under cold stress

表2 谷子6个冷胁迫处理条件下同时差异表达基因Table 2 Differentially expressed genes under 6 cold stress conditions in foxtail millet
进一步比较这9 个基因在6 种冷胁迫处理条件下的表达模式,结果(图3)显示,与各处理的对照组相比,除Si6g03220在各处理下均呈上调表达外,其他8个基因在0.5和1.0 h冷处理下呈下调表达,而在3.0、6.0、16.0 和24.0 h 冷处理下呈持续上调表达。其中Si3g01950与Si3g02940以及Si2g38970与Si9g50340随着处理时间延长,上调表达的强度呈递增趋势(图3A)。同时,对这9 个基因在xiaomi全生育期的表达模式进行分析,发现,Si6g03220、Si3g01950、Si3g02940和Si9g50340在全生育时期的表达量均较高(图3B)。

图3 差异表达基因的表达模式Fig. 3 Expression patterns of differentially expressed genes
2.3 模块的GO富集分析
选取冷响应差异表达基因数量最多的模块,并结合谷子中与水稻已报道耐冷基因同源最多的模块,进行GO 富集分析。对冷胁迫基因数最多的3个模块中的所有基因进行GO 功能富集分析,富集结果涉及到生物学过程(biological process,BP)、分子功能(molecular function,MF)和细胞组 分(cellular component,CC)。由 图4 可 知,darkturquoise 模块富集到了对寒冷的响应(GO:0009409)、低温驯化(GO:0009631)等相关的调控通路;turquoise 模块富集到了半胱氨酸类型的肽酶活性(GO:0008234)、作用于蛋白质的催化活性(GO:0140096)及脱落酸激活的信号通路(GO:0009738)等相关的调控通路;blue模块富集到了多种激酶活性的调控通路,如氧化还原酶活性(GO:0016491)、碳水化合物激酶活性(GO:0019200)。

图4 模块特有的基因GO富集结果Fig. 4 GO enrichment results of module specific gene
从上述GO 富集结果中挑选与冷胁迫相关的基因,进行权重共表达网络分析,并利用Cytoscape 软件对网络进行可视化,如图5 所示。同时对模块darkturquoise、turquoise 及blue 内权重较高的核心基因分别在水稻和拟南芥数据库中进行同源基因注释(部分见表3)。结果表明,这些基因涉及ERF、bHLH 和WRKY 等家族,而这些家族基因均与植物非生物胁迫应答相关[29-32],可作为谷子耐冷胁迫研究的候选基因。

图5 3个模块中与冷胁迫相关基因的共表达网络Fig. 5 Co-expression network of genes relative to cold stress in 3 modules

表3 3个模块中与冷胁迫相关的核心基因的功能注释Table 3 Functional annotation of hub genes relative to cold stress in 3 modules
2.4 目标模块中冷胁迫候选基因的筛选及功能分析
筛选与核心基因高连通性的基因作为候选基因,为了进一步了解这些候选基因的功能,通过功能注释及文献报道发现候选基因都与冷胁迫相关。Si8g19810和Si7g12250在水稻和玉米中的同源基因均属于bHLH 家族;Si4g26860和Si7g26280在水稻和玉米中的同源基因属于bZIP家族;Si1g19560和Si9g44570属于ERF 家族;这几个家族的转录因子都与植物非生物胁迫应答相关[33-39]。因此,通过WGCNA 构建冷胁迫基因共表达网络,并进一步筛选具有冷胁迫相关生物学意义的候选基因是可行的。从目标模块中的筛选获得的候选基因可作为谷子冷胁迫相关基因挖掘的可用资源,可作为后续研究中重点关注的对象。
2.5 候选基因冷胁迫应答的RT-qPCR分析
为进一步验证候选基因对冷胁迫的响应模式,对其中6 个核心胁迫应答基因进行RT-qPCR验证,结果(图6)显示,与对照相比, 6个基因在不同冷胁迫处理时间后的表达量均呈现出上调趋势,其表达模式与转录组分析结果(图3)基本一致,证明利用转录组数据分析得到的冷胁迫响应差异表达基因是可靠的。
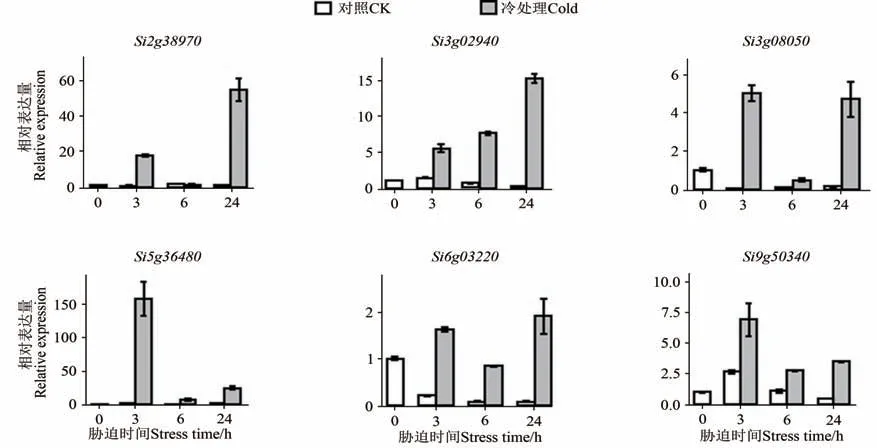
图6 6个核心基因在冷胁迫不同时期的表达Fig. 6 Expression of 6 hub genes in different times under cold stress
3 讨论
3.1 共表达网络分析富集到多个冷胁迫相关的基因
本研究通过对谷子冷胁迫处理组及对照组多个组织、不同时间的33 份RNA-seq 数据进行分析,构建了共表达网络,根据聚类情况将其划分为44 个模块。利用水稻数据库中已报道与冷胁迫相关的基因,在谷子蛋白数据库中比对寻找谷子中的同源基因,并与划分的模块进行比较,发现在18 个模块中都能找出与冷胁迫相关的基因。对冷胁迫相关基因数量最多的3 个模块进行了GO功能富集分析,各个模块都得到了与冷胁迫应答相关的功能富集结果,并且发现不同模块内的响应存在差异。其中,darkturquoise 模块富集到了对寒冷的响应(GO:0009409)、低温驯化(GO:0009631)等相关的调控通路;turquoise模块富集到了半胱氨酸类型的肽酶活性(GO:0008234)、作用于蛋白质的催化活性(GO:0140096)及脱落酸激活的信号通路(GO:0009738)等相关的调控通路;blue模块富集到了多种激酶活性的调控通路,如氧化还原酶活性(GO:0016491)、碳水化合物激酶活性(GO:0019200)。
对turquoise模块共表达网络中的核心基因进行分析发现,核心基因Si4g02480注释到了对寒冷的响应(GO:0009409),且该基因在水稻蛋白数据库中的同源基因为脱水应答元件结合蛋白,属于DREB转录因子的ERF亚族基因。此亚族基因通过与启动子区域的顺式作用元件特异性结合来调控与干旱、高盐和低温等非生物胁迫相关的应答基因表达[33],因此在植物抵抗非生物胁迫过程中起重要作用[34];此外,ERF 基因家族可以促进其下游抗逆相关基因的表达,提高植物的抗逆境胁迫能力[35]。本研究对各模块的候选基因进行注释发现,Si9g05520在水稻和玉米中的同源基因LOC_Os03g58890和GRMZM2G054224都 属 于 氧化还原酶,其酶活性受温度影响;另有2 个候选基因属于bHLH 转录因子;有4 个基因属于ERF 转录因子。研究表明,bHLH 转录因子家族参与植物的生长发育,其表达受盐、冷等非生物胁迫诱导[36],还可以响应低温,进而调控植物生长发育[37];AP2/ERF 家族受低温诱导,同时激活或抑制下游基因的表达[38]。本研究除检测到ERF转录因子外,还有MYB、bZIP、WRKY、bHLH、TCP 转录因子。据报道,MYB 转录因子对高温具有耐受性[39];bZIP在植物应对盐害、干旱、冷害、机械损伤以及渗透胁迫的非生物胁迫中发挥重要作用[40];WRKY 转录因子可结合目标基因启动子上的作用元件W-BOX(TGACCA/T),从而调控下游目的基因的表达,调节植物适应低温胁迫,其编码蛋白质通常表现为抑制作用,在低温胁迫下,下游靶基因的表达量下调[41]。由此表明,多个转录因子家族与冷胁迫相关,因此,本研究中发现的候选基因为后续谷子耐冷育种提供了基因资源。
3.2 Si6g03220 可能是谷子冷胁迫响应的关键候选基因
利用‘豫谷1 号’在6 个不同冷处理时间下的差异表达基因集,并结合全生育期的表达模式进行分析,发现Si6g03220在全生育期都有较高表达,且在不同冷胁迫处理下均上调表达,该基因被注释为乙烯反应相关转录因子11,属AP2 基因家族。研究表明,当植物受到冷害和冻害刺激时,体内的乙烯含量显著增加[42-43];而乙烯在植物冷胁迫应答中具有一定的调控作用[44]。因此,该基因可作为研究谷子耐冷机制的重要候选基因。
本研究通过对谷子多时期、多组织的冷胁迫数据进行共表达网络分析,得到了44 个模块,其中有3 个模块的冷胁迫响应基因数较多,进一步对他们进行功能富集分析,筛选出了与冷胁迫相关的候选基因;通过对‘豫谷1号’在6个不同冷胁迫处理中的差异表达基因进行分析,获得了9 个高置信度的候选基因;转录组数据和RT-qPCR 验证表明,这些基因与冷胁迫应答密切相关。以上研究结果为谷子冷胁迫响应机理研究奠定了基础,得到的候选基因为谷子抗逆育种研究提供重要资源。
