基于遗传算法的计算机辅助策略优化孕酮适配体*
2023-10-10唐春花江寒冰卢晓玲陈美仑刘一杰
唐春花 江寒冰 杨 洁 卢晓玲 陈美仑 魏 铮 刘一杰 余 鹏
(中南大学湘雅药学院,长沙 410013)
孕酮(progesterone,P4)是一种天然的雌激素,与女性的月经周期、怀孕及胎儿发育有着密切的关系[1],然而,激素滥用导致的水源、食品中的P4含量过高却会给人们带来极大的身体危害[2]。因此,不论是在临床治疗用药,还是在环境保护方面,对P4浓度的监测都十分重要。常见的P4检测方法有免疫法和色谱法,这两种方法灵敏度高,特异性强,但存在着价格昂贵、操作繁琐、专业性强、不适于现场检测等缺点[3]。核酸适配体是一段单链寡聚核苷酸,它可以与靶标分子特异性结合,因此可作为新型识别分子应用于分析检测领域。适配体可以通过体外选择过程从随机核苷酸文库中获得,这种方式称为指数富集的配体系统进化技术(systematic evolution of ligands by exponential enrichment,SELEX)[4]。考虑到初始文库的多样性有限,且在PCR过程中存在偏好性,可能导致潜在的高亲和力适配体丢失,有研究者根据其筛选经验预估SELEX的成功率约为50%[5]。
计算机辅助优化策略(in silicomaturation,ISM)可作为一种针对适配体筛选后优化、改善适配体功能的方法。有研究者指出,应用ISM对SELEX筛选后的序列进行重筛选可在一定程度上弥补SELEX技术的局限性[6-7]。如模拟生物进化算法的遗传算法(genetic algorithm,GA),将亲代适配体通过选择、交叉和变异产生子代,从有希望的亲本序列中进化出改良的适配体,通过反复选择、进化,在经ISM优化后进行体外评价。2005年,Ikebukuro等[8]首次将基于GA的ISM应用于核酸适配体的优化,用于抑制凝血酶的DNA适配体的重筛选。虽然ISM已经被应用于筛选多个靶标的高亲和力适配体且不需要重复SELEX过程[7-13],但迄今为止,还没有研究者尝试使用ISM来提高适配体对P4的亲和力。
P4属于小分子化合物,已有研究指出针对小分子靶标选择的适配体比其他靶标选择的适配体亲和力低[14]。筛选小分子靶标适配体的主要挑战之一是靶标-核酸复合物和游离核酸的分离,由于靶标的分子质量较小,并不会导致游离核酸和靶标-核酸复合物之间存在较大的质量差异,分离过程更加复杂,这也是传统SELEX技术在筛选小分子靶标适配体时面临的主要挑战[15]。针对小分子适配体实验筛选的困难,ISM则可作为一种有效的补充选择,来改善小分子靶标适配体的功能。
虽然ISM的核酸文库容量比传统SELEX的容量小,倘若每一个子代中的所有序列(10~50个序列)都进行合成和评估,对于高通量筛选来说,ISM过程仍然是昂贵、繁琐和耗时的。同时,研究人员也不可能通过确定每次传代过程中适配体-靶标复合物的结构特征,来选择最好的亲本适配体进行传代。在ISM中,毫无疑问以有效的、节约时间和成本的手段来识别适配体-靶标复合物之间的相互作用对筛选高质量核酸适配体起着非常重要的作用。而分子对接则可以作为研究适配体与靶标结合时产生何种亲和力以及亲和力大小的一个理想的工具[16-17]。目前学界面临的主要问题之一是在分子对接时ssDNA适配体及其靶点的研究缺乏准确预测ssDNA三维结构的工具。最近也有研究者提出了通过先预测与DNA相对应的RNA三维结构,再将其转换为DNA的方法来预测ssDNA的三维结构[18]。
此外,由于结合试验的灵敏度较低,目前还没有标准化的分析方法来表征小分子适配体对靶标的亲和力大小[19-20]。近来有研究人员提出,对于适配体-靶标复合物的解离常数(equilibrium dissociation constant,KD)的测定,大致方法可分为基于分离策略(如透析法、超滤法、凝胶电泳法、毛细管电泳法等)或均相策略(荧光强度法、荧光各向异性法、圆二色谱法、紫外可见分光光度法及表面等离子体共振法等)[21-22]。纳米金(AuNPs)比色法是一种现象明显、易制备、操作简单、耗时短的表征方法,目前已有广泛的应用,只是AuNPs比色法一般并不用于KD值的准确测定,而是多用于亲和性、特异性检测与传感器的制备[22-23]。荧光检测法在测定适配体-靶标复合物时具有诸多优势,它可以在适配体与靶标处于平衡时进行测定,避开了结合速率和解离速率的问题。因此,本研究在结合已有研究的基础上,首先使用AuNPs比色法对经ISM优化后的适配体亲和力进行初步验证,随后根据核酸适配体结构转换信号检测靶标的原理,将标记有羧基荧光素(carboxy fluorescein,FAM)的核酸适配体与标记猝灭基团(black hole quencher 1,BHQ1)的适配体互补链配对杂交,通过P4竞争适配体的结合位点使荧光恢复,建立了一种基于核酸适配体结构转换开关的荧光共振能量转移(fluorescence resonance energy transfer,FRET)方法对适配体的KD值进行测定[24]。
2017年,P4适配体经过截短后,这里称为P4S-0,在适配体传感器的构建中得到了应用[25]。本研究描述了一种基于GA的ISM与分子对接相结合的方法,来筛选和优化出高亲和力和选择性的P4适配体(图1);此外,还提出使用Mfold和RNAComposer分别预测ssDNA的二级结构和三级结构,使用Discovery Studio将RNA修改为DNA,最后对结构进行能量最小化处理,作为分子对接中的ssDNA适配体的三维模型的输入结构;在亲和力验证阶段,首先使用AuNPs比色法对适配体进行初步表征,随后采用基于FRET的荧光法对适配体的KD值进行测定,优化后的候选适配体与其互补序列如表1所示。

Table 1 Candidate aptamers and their complementary sequences

Fig.1 The genetic algorithm-based in silico maturation strategy combined with molecular docking to obtain progesterone-binding aptamer with improved affinity
1 材料与方法
1.1 P4适配体的计算机辅助优化方法
1.1.1 初始文库的生成
轮盘赌算法原理与操作过程:GA中的轮盘赌是一种简单的选择方法,即每个个体的选择概率和其适应度值成比例,适应度越大,选中概率也越大,类似于商场的抽奖轮盘[26]。此处以每条适配体与P4进行分子对接打分的分值作为算法中的适应度值,打分越高,那么适应度越高,该序列成为亲本的可能性越大。后续每次参与变异的亲本序列、选择变异的类型与选择发生变异的位置等都离不开轮盘赌算法。
首先利用GA构造DNA序列库,使用给定的P4适配体序列P4S-0产生初始亲代DNA序列库。初始序列库由3种类型的适配体组成,包括分别由1个点突变、2个点突变和不同交叉(单点、双点和多点交叉)产生的3组适配体,共60条序列。所有GA过程均在Microsoft Visual Studio(2022)[27]中使用C++语言编写的程序实现,设定恒定的交叉百分比(0.8)和突变率(0.2)[28]。执行此程序后,得到了初始文库的60条序列。
1.1.2 基于GA的P4适配体迭代优化步骤
产生第一代序列(G1):初始文库包括60条序列,根据初始文库中序列的分子对接结果,选择打分排名前5的序列,根据对接结果的打分值对初始序列以不同的适应度值(即出现率)进行复制,并随机配对选择亲本,通过轮盘赌选择方法对序列进行单点、双点和多点交叉(程序随机产生1个0到2的随机整数,0对应发生单点交叉,1对应发生两点交叉,2对应发生多点交叉),产生20条不重复的子代序列作为G1。将得到的适配体再次进行分子对接。
产生第二代序列(G2):将G1和初始文库中对接打分排名前5的序列作为亲代,将单碱基突变随机引入序列以产生1组20条新序列作为G2。具体操作为:同样以不同的出现率按照轮盘赌方法选择亲代,然后再产生1~25内(序列长度为25)的随机数决定亲代的哪个位置发生单碱基突变,突变后的碱基种类也是由随机数的产生决定,随机产生0~3之间的整数,每个整数对应一种碱基(0对应A、1对应T、2对应C、3对应G),如果产生的碱基与原碱基相同,则重新产生随机数决定。此时产生了20条新序列作为G2,继续进行分子对接。
产生第三代序列(G3):选择初始文库、G1、G2代中打分排名前5的序列,引入双碱基突变以产生20条新序列作为G3。具体操作为:根据不同的出现率和轮盘赌方法,来选择每次突变的亲代,选择亲代后产生两个不同的随机整数来选择发生碱基突变的序列位置(随机数范围就是序列长度范围1~25),再产生0~3的随机整数来表示突变后的碱基(0对应碱基A、1对应碱基T、2对应碱基C、3对应碱基G),若突变后的碱基与亲代序列的原位置碱基相同,就重新产生随机数来重新选择,直到选择到不同的碱基为止,突变后将序列与亲代、已经产生过的突变序列进行比较,如果重复,那么将重新在该亲代上选择突变位置和突变后的碱基。此时产生了20条新序列作为G3代,同样进行分子对接。
所有GA优化步骤和适配体对接打分结果见图S1和表S1,在初始文库、G1、G2和G3代中选择5条打分最高的序列作为候选适配体。
1.1.3 适配体二级结构的预测
使用Mfold Web服务器(http://www.unafold.org/hybrid2-same.php)对初始文库以及G1、G2、G3代序列的二级结构进行预测[29]。预测在温度为25℃,离子浓度与后续荧光实验缓冲液(pH 7.6)的离子组分相同时,即在120 mmol/L Na+、20 mmol/L Mg2+下热稳定最好的结构。
1.1.4 适配体三级结构的建模和优化
对ssDNA适配体三级结构的预测主要包括3个步骤。首先,ssDNA的二级结构已经通过Mfold Web服务器预测出,将预测的二级结构通过RNA Composer服务器建立相对应的3D RNA模型[30]。随后,使用Discovery Studio 4.5[31]将ssRNA结构中的尿嘧啶转换为胸腺嘧啶,将核糖核酸转换为脱氧核糖核酸,即转换为ssDNA的三级结构。随后,使 用 Molecular Operating Environment(MOE,2014)[32]将ssDNA的三级结构进行能量最小化处理,在Amber力场下使用最陡下降法使斜率小于0.000 01 kcal/mol²。
通过对蛋白质数据库(https://www.rcsb.org/)中的24个ssDNA序列进行核磁共振解析,评价该方法的准确性。用Visual Molecular Dynamics(VMD)[33]软件比较了核磁共振求解的糖-磷酸盐骨架结构和用上述方法预测的糖-磷酸盐骨架结构之间的均方根偏差(RMSD)来确定结构的相似度。
1.1.5 分子对接
使用分子对接软件AutoDockTools(ADT,1.5.6)[34]对GA产生的ssDNA适配体与P4的亲和力大小进行评估。将能量最小化后的ssDNA适配体结构视为刚性结构,随后对P4小分子进行检测配体中心与扭转键的操作。为了覆盖适配体结构中的所有活性位点,创建大小在126 Å×126 Å×126 Å(x,y,z),间距为0.375 Å以内的对接盒子。使用拉马克遗传算法构象搜索,建立了100次遗传算法运行的所有对接模拟,种群规模为1 502 500 000次能量评估的最大数量,每次运行27 000代。根据结合能、相互作用类型(主要是氢键、疏水作用)和结合位点等分子对接结果对最佳配合物进行评分和筛选。
1.1.6 对接结果的图像表征
使用Pymol[35]将适配体-P4复合物的结构进行图像表征。
1.2 实验方法
为了验证经ISM优化后的适配体亲和力与特异性,将适配体进行了如下的实验评估。首先,使用AuNPs比色法初步验证,比较候选适配体的亲和力大小[22],原理如图2所示。随后构建如图3的适配体结构开关准确测定候选适配体的KD值。根据适配体结构开关检测靶标的原理,在候选适配体的两条互补链上从3'和5'端分别标记荧光基团FAM和猝灭基团BHQ1,当候选适配体与两条互补链碱基配对杂交时,标记了荧光基团的FDNA与标记了猝灭基团的BDNA相互靠近,荧光猝灭;通过P4竞争结合候选适配体FDNA的结合位点,使两条互补链从适配体上解离下来,荧光恢复。以此核酸适配体结构开关法评估候选适配体的亲和力和特异性。

Fig.2 The schematic for the evaluation of candidate aptamer based on colorimetric method

Fig.3 The schematic for the evaluation of candidate aptamer based on FRET and the aptamer structure switch
1.2.1 实验试剂与仪器
孕酮(源叶生物科技有限公司,上海),双酚A、雌二醇、睾酮和皮质醇(阿拉丁生化科技股份有限公司,上海)、适配体及其互补序列(生工生物工程公司,上海)、Tris-HCl(福州奥研实验器材有限责任公司,福州),氯化钠、氯化镁和吐温-20(Tween-20)(阿拉丁生化科技股份有限公司,上海)、纳米金胶体(先丰纳米,南京)。
F-7100荧光分光光度计(HITACHI,日本),设置激发波长为480 nm,激发和发射狭缝宽度设定为5 nm和5 nm;Synerg Lx多功能酶标仪(博腾仪器有限公司,美国);ZQPW-70A全温振荡培养箱(莱玻特瑞仪器设备有限公司);AUY220-0.1 mg分析天平(岛津,日本);TGL-20M台式高速冷冻离心机(迈克尔实验仪器有限公司,湖南);Q-LAB20-DV超纯水仪(启沁环保科技公司,湖南)。
1.2.2 AuNPs比色法初步验证候选适配体的亲和力
AuNPs比色法的实验原理如图2所示,先对实验中NaCl和适配体的浓度进行优化,具体见文档S1和图S2,S3。条件优化后进行适配体亲和力的定性分析,分别量取25 μl 3 μmol/L适配体溶液与25 μl系列浓度梯度 P4 溶液(0、0.5、1.0、2.0、4.0、6.0、8.0、10.0 mg/L),震荡混匀,在室温下孵育30 min后,再加入150 μl AuNPs溶液和10 μl 2.5 μmol/L NaCl溶液,室温孵育5 min后观察混合溶液的颜色变化,记录溶液吸光度变化情况,计算吸光度比值A620/A520,以P4浓度为横坐标,吸光度比值A620/A520为纵坐标,绘制工作曲线。
1.2.3 基于适配体结构开关的FRET法测量适配体的KD值
a.适配体KD值的测量
构建适配体结构开关(图3),并优化适配体与互补链的浓度、结构开关形成时间、P4孵育时间以及探索溶液中Mg2+浓度和Tween 20含量对结构开关的影响,上述条件优化见文档S1和图S4~S8。依据上述条件优化后的结果,测定在不同浓度P4的情况下结构开关体系的荧光信号变化强度。分别配制 0、10、20、30、50、100、500、1 000 nmol/L的P4溶液。将缓冲液稀释好的0.1 μmol/L FDNA、0.2 μmol/L适配体、0.3 μmol/L BDNA各50 μl置于95℃水浴锅中加热10 min,取出后迅速转移到碎冰中冷却降温。然后取适配体、FDNA、BDNA一起孵育15 min,后加入50 μl P4溶液在37℃恒温下孵育30 min,使二者得到充分结合,在测定前加入300 μl纯净水将反应体系补足到500 μl,利用荧光分光光度计测定每组荧光信号值。以P4浓度为横坐标x,以荧光信号恢复百分比(F1-F2)/F2为纵坐标y,其中F1是加入P4之后的荧光值,F2是阴性对照组(P4为0)的荧光值。通过GraphPad Prism 8.0 拟合不同浓度P4与适配体结合的非线性饱和曲线,根据曲线y=Bmax/(KD+x)计算KD值,Bmax指的是适配体与P4的最大结合率,是常量。根据此方法测定以上对接打分最高的5条候选适配体与靶标复合物的KD值。
b.通过实验评估适配体的选择性
经ISM优化后,适配体对P4的特异性同样需要鉴定。因此,本实验选取了双酚A、雌二醇、睾酮、皮质醇作为干扰物,将这些物质配制成浓度为0.1 μmol/L的溶液。0.1 μmol/L FDNA、0.2 μmol/L P4适配体和 0.3 μmol/L BDNA 各 50 μl先孵育15 min,然后再加入0.1 μmol/L各物质的溶液50 μl,在测定前加入300 μl ddH2O将反应体系补足到500 μl,利用荧光分光光度计测定出不同物质的荧光信号变化强度,以不同物质种类为横坐标x,以荧光信号恢复值(F1-F2)为纵坐标y作图。
2 结果与讨论
2.1 候选适配体的热力学性质

Fig.4 Frequency of secondary structures in groups from the initial library and following generations (G1,G2,G3)
初始文库包含60条ssDNA,包含3种不同类型的二级结构,包括简单发夹环(hairpin loop)、内环(internal loop)和多分支环(multibranch loop)。子代G1、G2、G3中的适配体二级结构更加多样化(图4)。初始文库中二级结构形成的最小自由能在-0.01~-4.42 kcal/mol,G1代中为-0.03~-10.21 kcal/mol,G2代为-0.19~-4.98 kcal/mol,G3代为-0.27~-10.8 kcal/mol。初始适配体P4S-0具有内环结构(图5),其自由能约为-0.35 kcal/mol。初始库和子代文库中选择的几条候选适配体二级结构如图5所示,结果表明,不同的GA操作可以成功地创建一个虚拟文库,不同子代的核酸序列在二级结构和热力学性质上具有多样性。
2.2 适配体的三维建模
通常,虚拟筛选需要得到受体和配体分子的三维结构。由于目前缺乏对ssDNA进行准确三维建模的计算工具,且难以通过实验确定文库中所有核酸序列的三维结构,许多研究人员不得不转而寻找较为可靠的预测ssDNA三维结构的方法。Jeddi和Saiz[18]在2017年首次报道了一种预测ssDNA适配体三维结构的方法,这里本文学习这种建模思路并进行了一定修改。主要预测步骤包括:a.构建二级结构;b.构建等效的三维ssRNA结构;c.根据报道的步骤,将三维ssRNA结构转换为ssDNA结构。本文在步骤b中使用了RNAComposer而不是文献中的Assemble2/Chimera;此外,并非使用VMD,而是使用Discovery Studio 4.5将ssRNA模型转换为等效的ssDNA 3D结构,并使用MOE软件包进行能量优化。通过对24个具有已知结构的ssDNA进行3D建模,每个预测的ssDNA结构都与从PDB数据库下载的相应ssDNA结构对齐,并计算了每对结构之间的相应均方根偏差(RMSD)以测量相似程度。图6显示了24个预测的3D结构(ssDNA-黄色)和从PDB数据库下载的24个NMR结构(ssDNA-绿色)[36-56]的叠加,以及RMSD的计算值。结果表明,本方法能够准确预测各种ssDNA的结构,序列范围是7~27个核苷酸,得到的RMSD值范围在2.51~9.02 Å的,通常在4 Å附近,平均值为5.6 Å。然后,利用本方法预测所有新的适配体的三维结构,并用于分子对接研究。
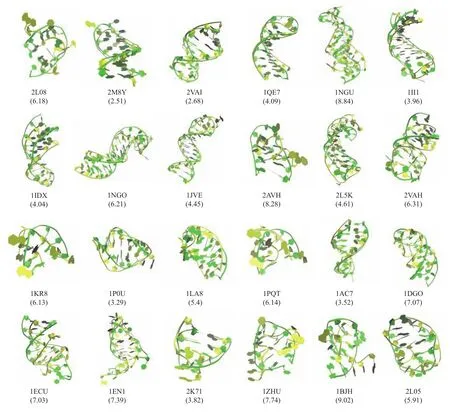
Fig.6 Alignment of the 3D structures predicted by our pipeline (yellow) and the corresponding experimental ones obtained from PDB (green) for the 24 ssDNA
2.3 使用计算机辅助策略优化P4适配体
利用分子对接技术研究P4与120条候选适配体的结合能、结合位点和相互作用模式,其中包括来自初始文库和G1、G2、G3代的序列。在初始文库和不同子代中,ssDNA适配体的预测结合能分别在-4.26~-6.7 kcal/mol和-4.46~-7.48 kcal/mol(表S1)。
初始适配体P4S-0与P4的相互作用主要是疏水作用和氢键作用,对接能量为-4.63 kcal/mol,主要是P4S-0的这几个残基——DA2、DT3和DT4与P4进行相互作用。P4S-0的DA2残基与P4的20位酮羰基形成氢键(图S9)。
在初始文库和后几代的打分排名靠前的序列中,大多数具有简单的简单发夹环结构(图5),其自由能等于或低于P4S-0。对接结果指出,发夹环适配体的结合位点主要位于同一环区,而多分支环适体的结合位点主要位于同一环区的循环区域外。通常,未配对残基可以作为结合位点参与靶标识别,因为这些残基更灵活,有更多可用的供体或受体[57]。此外,对接结果也表明,候选适配体的平均结合能从初始库(-5.08 kcal/mol)到G1(-5.98 kcal/mol)、G2(-5.93 kcal/mol)、G3(-5.85 kcal/mol)大幅降低,G3代的结合能又有一些升高。在经过这几轮GA后的局部搜索后,似乎对适配体序列进行了适当优化,而进一步的修改可能导致了序列的分化,导致功能的丧失。
在G1打分最高的序列中,适配体P4G1-14、P4G1-6和P4G1-7对P4的亲和度最高。适配体P4G1-14、P4G1-6和P4G1-7的结合能分别为-7.48、-6.9和-6.86 kcal/mol。P4G1-14与P4的结合模式为沟槽结合,主要作用力有氢键和疏水作用。P4G1-6与P4主要通过疏水作用和氢键产生相互作用,其中疏水作用占主导,结合模式为沟槽结合。P4G1-7与P4的结合位点在非环区,作用模式也是沟槽结合。
在G2代中,P4G2-14和P4G2-20的对接打分最高,对接能量分别为-6.76和-6.95 kcal/mol。P4G2-14与P4的相互作用模式为插入结合,P4G2-20与P4的相互作用模式为沟面结合,相互作用力中疏水作用占主导。以上适配体与P4具体相互作用的具体碱基和2D拓扑图(图S9,S10),可以更直观地体现适配体与目标物的具体作用位点、作用力的方向等。
基于热力学性质和对接分析,筛选出P4G1-14、P4G2-14、P4G1-6、P4G1-7和P4G2-20这5条适配体最佳候选适配体,随后通过实验进行评价,实验部分以P4S-0为对照。
2.4 初步验证候选适配体的亲和力
在优化后的NaCl浓度、适配体浓度的条件下,基于AuNPs比色法对原适配体P4S-0与候选适配体(P4G1-14、P4G2-20、P4G1-6、P4G1-7和P4G2-14)进行了亲和力比较。在与系列浓度梯度的P4溶液(0、0.5、1.0、2.0、4.0、6.0、8.0、10.0 mg/L)充分混合均匀后,观察溶液颜色变化,A620/A520比值越高,说明适配体与靶标的亲和力越强。随着P4浓度的升高,在浓度为0~4 mg/L时,各核酸适配体吸光度比值A620/A520逐渐上升,说明此时P4浓度不够,AuNPs上仍吸附有过量的核酸适配体,还未完全与P4结合,受核酸适配体的保护,AuNPs产生聚集不明显。随着P4浓度的升高,越来越多AuNPs上吸附的核酸适配体脱落与P4结合,AuNPs产生的聚集效应越来越明显,直到反应完全,A620/A520基本不变,此时AuNPs、核酸适配体与P4三者的结合达到了平衡。另外,从图7可以看出,在同等条件下,候选适配体P4G1-14的吸光度比值A620/A520较高,明显高于其他适配体,说明P4G1-14与P4的亲和力最强,原始适配体P4S-0的吸光度比值最低,其余4条候选适配体的亲和力均高于P4S-0。从上述分析可初步得出,优化后的候选适配体亲和力均较P4S-0有了较大提升。只是AuNPs比色法并不能对适配体的KD值进行准确测定。

Fig.7 Variation of A620/A520 of AuNPs solution of candidate aptamers with increasing P4 concentration
2.5 适配体的KD值
2.5.1 适配体KD值的测定
使用基于适配体结构开关的FRET法测定适配体靶标复合物的KD值,KD值越小,说明适配体与靶标的亲和力越高。以上候选适配体与原始适配体的KD测定结果如图8所示,并与AutoDOCK对接打分结果进行比较(表2)。结果表明,经以上GA优化后的候选适配体相比于原始适配体P4S-0,对P4的亲和性有了较大提高,其中,P4G1-14的亲和力提升最明显,其KD值为4.457,相比于P4S-0亲和力提升了约3倍,其次是P4G2-20、P4G1-6、P4G1-7 和 P4G1-14,KD值分别为 6.432、8.582、10.45、11.34,此方法得出的KD值亲和大小顺序与对接打分的结果基本一致。这种实验的优势在于,检测荧光信号可以在适配体和P4处于平衡状态时进行KD测定,这不仅避开了结合速率或解离速率的问题,同时允许在各种缓冲溶液中进行测定,这可能是其他分离模式的分析技术难于实现的[58]。但是,由于小分子适配体的亲和力鉴定目前缺乏标准化的分析方法,此方法仅可作为判断的部分依据,如果有其他亲和力鉴定方式作为辅助将会使结果更加可靠。

Table 2 The KD value of aptamer was measured by fluorescence detection method based on the structure switch of aptamer and compared with the docking score
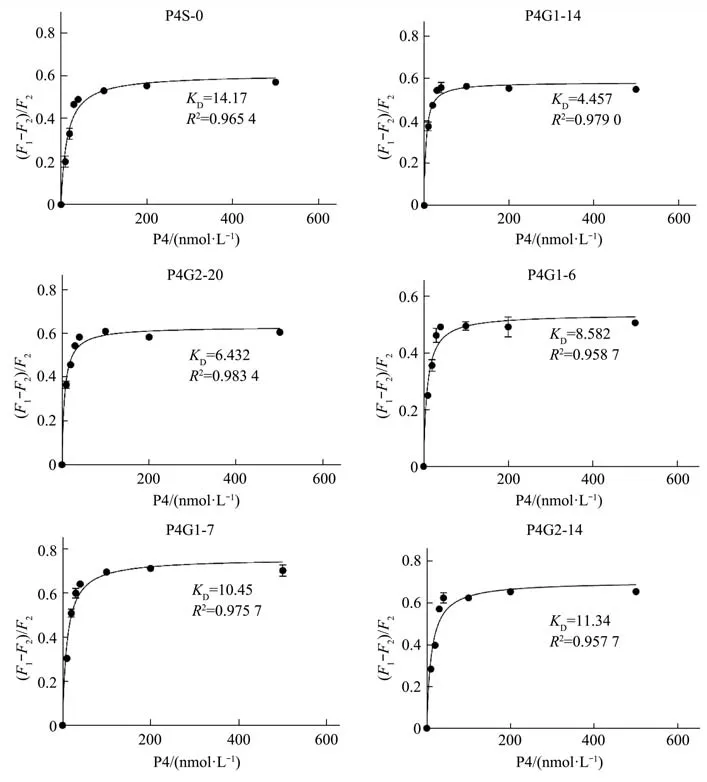
Fig.8 Saturation curve of candidate aptamers binding to P4
此外,方法中的测试结果可能会受到FDNA与BDNA的解链温度的影响,当这两条链GC含量高时,解链温度会随之升高[59],使得链解离更加困难,导致测得的解离常数偏小,这一点会对实验结果造成影响。例如P4S-0的BDNA链中GC含量约为80%,而P4G1-14的BDNA中GC含量为58%,解链温度差距的影响可能会对实验结果造成一定偏差。本实验没有设置第二种有效的方法测定KD值,无法与FRET法的结果相佐证,是本实验的局限性所在。
2.5.2 通过实验评估适配体的特异性
如图9所示,几个候选适配体对P4的响应值都是最高的,几种干扰物都只能引起部分响应。其中,双酚A的干扰性最小,而雌二醇、睾酮和皮质醇由于拥有与P4一样的甾体结构,所以候选适配体保存着对这几种甾体物质的识别能力,对雌二醇、睾酮和皮质醇的响应值较双酚A高。从图9中可以看出,在P4与其他干扰物共存的情况下,候选适配体仍对P4具有较高的特异性,候选适配体可用于进一步的传感器设计开发。

Fig.9 Fluorescence response of aptamer structural switch to different targets
3 结论
本研究成功地使用基于GA的ISM策略,与分子对接结合,提高了之前报道的P4S-0适配体对P4的亲和力。计算机打分结果与实验的一致性证实了此策略是设计或优化适配体功能的有效工具。分子对接技术可在分子水平上阐明受体与配体之间的相互作用,有助于设计或优化所需的适配体。然而,缺乏对生物大分子3D结构预测软件是计算机辅助技术发展的重要限制。本文通过先分别预测序列的二级结构以及相对应的RNA三级结构,后将RNA转换为DNA并经过能量优化得到ssDNA的三级结构,能够较准确预测DNA适配体的3D结构,首次预测了原始适配体P4S-0与P4的结合位点和相互作用类型。此外,对不同适配体-P4复合物的分析表明,较低的结合能、较简单的二级结构、较低的DG和相互作用类型是得到高亲和力、高选择性适配体的重要因素。
得到经ISM优化的适配体后,对适配体的亲和力和特异性验证是本研究的关键步骤。本研究首先采取了AuNPs比色法对候选适配体的亲和力进行初步验证,由于AuNPs比色法并不能准确测定适配体的KD值,于是构建了基于结构开关的FRET法准确测量适配体的KD值。基于适配体结构开关检测适配体对P4亲和力的方法,可以避开结合速率或解离速率的问题,同时允许在各种缓冲溶液中进行测定,在表征小分子结合适配体方面所表现出的优势可以用于开发高效的荧光适配体传感器。但该方法的缺陷在于,对于小分子配体的亲和力鉴定没有一种标准化的分析方法,应当多引入几种适配体亲和力测定方法对计算机优化适配体的结果加以佐证,且此方法中互补链的不同解链温度会对结果造成偏差,是FRET法的局限性所在,同时,也应该补充灵敏度实验以更直观地体现优化后的适配体拥有更强的功能。
其他改善适配体功能的策略,如结合序列的共轭或者构建多价适配体、稳定适配体的结构或将疏水基团引入适配体,可通过此方法流程进行研究。
附件见本文网络版(http://www.pibb.ac.cn或http://www.cnki.net):
PIBB_20230021_Doc S1.pdf
PIBB_20230021_Figure S1.pdf
PIBB_20230021_Figure S2.pdf
PIBB_20230021_Figure S3.pdf
PIBB_20230021_Figure S4.pdf
PIBB_20230021_Figure S5.pdf
PIBB_20230021_Figure S6.pdf
PIBB_20230021_Figure S7.pdf
PIBB_20230021_Figure S8.pdf
PIBB_20230021_Figure S9.pdf
PIBB_20230021_Figure S10.pdf
PIBB_20230021_Table S1.pdf
