基于结构化深度聚类网络的miRNA-疾病关联预测
2023-10-08张正涛
胡 华,张正涛
(1.枣庄学院 信息科学与工程学院,山东 枣庄 277160;2.中国矿业大学 计算机科学与技术学院,江苏 徐州 221116)
0 引言
microRNA(缩写为miRNA)是一种内源性小RNA,长度约为21~25个核苷酸。miRNA对体细胞的调控具有重要意义[1]。传统生物试验方法对miRNA疾病进行关联预测研究存在诸多瓶颈,如试验周期长、成本高等问题。近年来,越来越多的研究者通过机器学习和深度学习的方法研究miRNA和疾病间的关联,但目前大多数miRNA疾病关联预测方法无法将miRNA和疾病数据自身的特性及其结构很好的结合。本文提出一种新的深度聚类网络模型预测miRNA和疾病的关联,称为结构化深度聚类网络(a structural deep clustering network model for predicting miRNA-disease associations,SDCNMDA),将图卷积神经网络视为深度聚类网络中的子模块,使miRNA和疾病的结构信息通过图卷积神经网络整合到深度聚类中。本模型首先通过传递算子将自编码器所学到的表征传递到相应的GCN(Graph Convolutional Network)层,并引入双重监督机制,以整合两种不同的深度神经网络结构(GCN和自编码器),并指导整个模型的更新。通过上述方法,miRNA和疾病数据的多重结构与自编码器所学到的表征自然地结合在一起,并且能充分结合中心节点的低阶和高阶邻居。
1 方法
1.1 人类miRNA-疾病关联
本试验采用HMDD v2.0[2]和HMDD v3.0[3]数据集对模型进行验证。HMDD v2.0数据集包括495种miRNA和383种疾病,5 430条经试验验证的miRNA-疾病关联。HMDD v3.0数据集中包含 850种疾病与1 057种miRNA,32 226 条经试验证实的 miRNA-疾病关联。采用二元矩阵A(nd×nm)表示miRNA与疾病的关联,nd表示疾病的数目,nm表示miRNA数目。元素A(di,mj)等于1时,表示疾病di与miRNAmj有关联;否则,表示两者之间没有关联。
1.2 miRNA功能相似性
基于表型相似的疾病与功能相似性的miRNA发生关联基本生物学假设,WANG等[4]提出了一种计算miRNA功能相似性的模型。miRNA功能相似性试验数据下载地址为“https://www.cuilab.cn/files/images/cuilab/misim.zip”,以此构建出二维矩阵MFS,其包含495行和495列,存储了miRNA的功能相似性。
1.3 疾病语义相似性
常见的疾病相似性有两种计算方法。XUAN等[5]基于MeSH(Medical Subject Headings)数据库,将每种疾病抽象出的数据结构用有向无环图(Directed Acyclic Graph,DAG)表示。通过MeSH数据库和有向无环图计算疾病语义相似性。用DAG(d)=(d,T(di),E(di)来描述疾病di,其中T(di)表示疾病di的祖先节点及其自身,E(di)表示从祖先节点到疾病di的边集合。然后,计算疾病dm对疾病di所作的贡献
(1)
式中:△为贡献衰减因子,在试验中设置为0.5。疾病di的语义值可以描述为:
(2)
然后,利用式(1)(2)将疾病的语义相似性公式改写为:
(3)
如果不同DAG同一层中的疾病数量不同,则不应默认指定疾病的相同语义贡献值,这会导致计算的语义贡献值不准确。因此,第二种常见的计算疾病dm对疾病di的语义贡献值的计算方法修正为:
(4)
疾病di的语义贡献值的计算为:
(5)
利用式(4)(5),疾病di和疾病dj之间的疾病语义相似性公式为:
(6)
为了获得疾病语义的增强信息,整合式(3)和(6),疾病di和dj间的语义相似性DSS(di,dj)的计算公式为:
(7)
1.4 miRNA与疾病的高斯相互作用谱核相似性
考虑到MFS邻接矩阵和DSS邻接矩阵的稀疏性,使用CHEN等[6]提出的高斯相互作用谱核相似性矩阵。miRNA高斯相互作用谱核相似性矩阵计算公式为:
GM(mi,mj)=exp(-φm‖MGIP(mi)-MGIP(mj)‖2)
式中:GM是miRNA的高斯相互作用谱核相似性矩阵,二元向量MGIP(Mi)用于表示miRNA的相互作用谱,φm用来控制核函数带宽,计算如下:
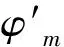
GD(di,dj)=exp(-φm‖DGIP(di)-DGIP(dj)‖2)
式中:GD是疾病的高斯相互作用谱核相似性矩阵,参数nd表示疾病数量。
1.5 miRNA和疾病的集成相似性
考虑到miRNA功能相似性矩阵的稀疏性,将miRNA功能相似性矩阵与疾病的高斯相互作用谱核相似性矩阵融合,得到miRNA的集成相似性矩阵IM,计算公式如下:
类似地,疾病的集成相似性矩阵ID的计算公式如下:
2 算法描述
为获得miRNA与疾病之间的潜在关联,基于深度聚类网络思想,提出了一种新的图卷积模型SDCNMDA,模型可描述为以下6个步骤:
(1)基于HMDD v2.0构建miRNA-疾病异构二分图;
(2)将miRNA节点与疾病特征投影到同一维向量空间;
(3)利用K最邻近(K-Nearest Neighbor,KNN)算法分别构建miRNA和疾病节点的新相似性,并且利用miRNA与疾病的原始集成相似性进行特征拼接,从而增强miRNA和疾病的特征表示;
(4)利用基于自编码器的深度神经网络,学习miRNA和疾病节点的特征表示;
(5)利用传递算子,将自编码器学到的miRNA和疾病的新特征表示作为参数输入到图卷积网络中,进而获得miRNA和疾病节点数据自身表示和结构表示;
(6)通过双监督机制进行监督,对模型进行监督训练。
SDCNMDA模型架构如图1所示。
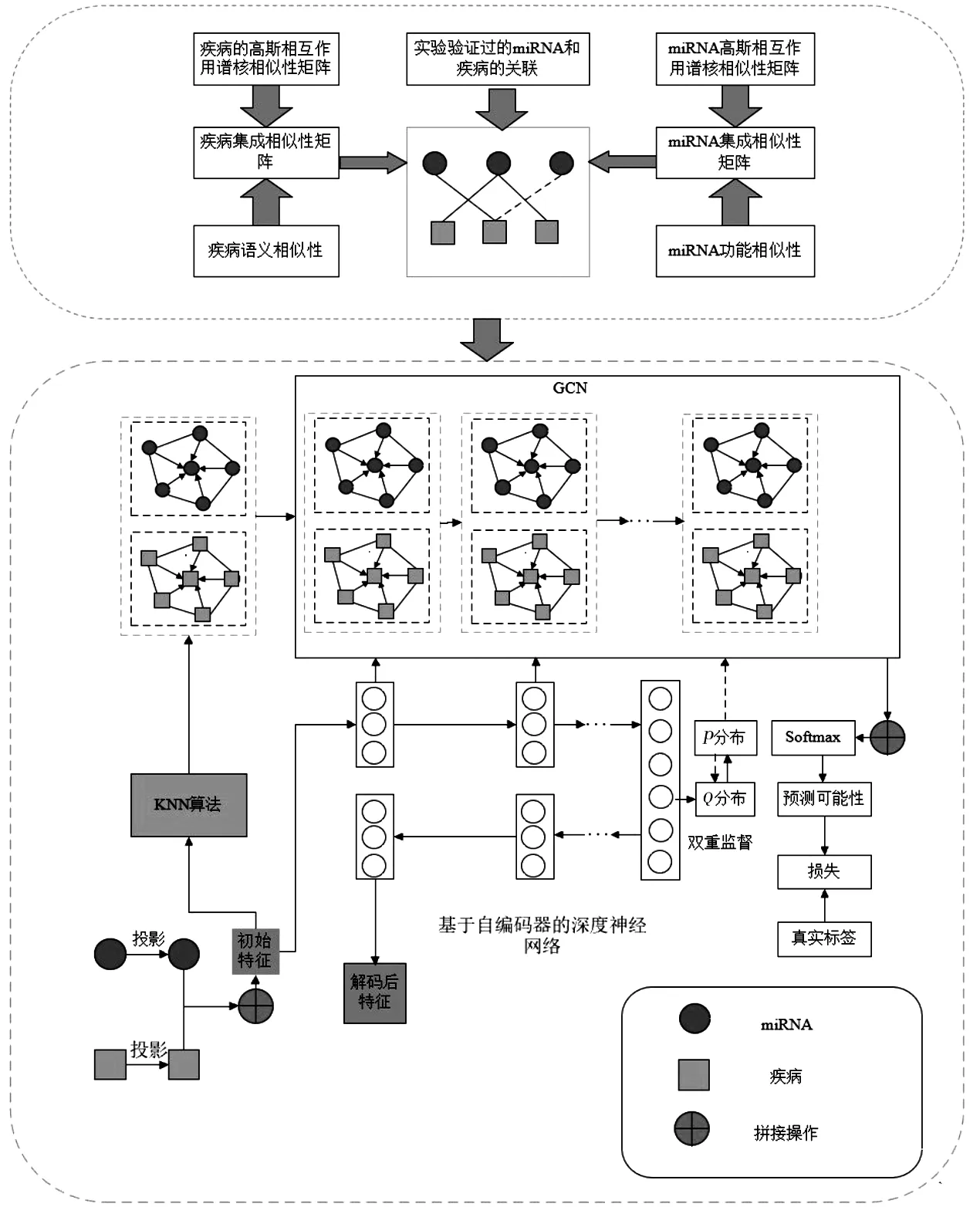
图1 SDCNMDA模型架构图
2.1 构建miRNA和疾病的异构图
模型首先读取疾病语义相似性矩阵DSS和miRNA功能相似性矩阵MFS。由于矩阵DSS和MFS的稀疏性,模型选择用疾病高斯相互作用谱核相似性矩阵对疾病语义相似性矩阵中的0元素进行填充。同理,采用miRNA高斯相互作用谱核相似性矩阵对miRNA功能相似性矩阵的0元素进行填充。其次,本模型从HMDD v2.0中获得的189 585条miRNA和疾病的关联关系中,选取5 430条已经证实存在的关联作为正样本。为了保证正负样本平衡,模型选择从剩余的尚未证实存在关联的数据中随机挑选出5 430条miRNA和疾病的未知关联作为负样本数据。选择878个异质节点构建图,其中疾病节点标签为1,miRNA节点标签为0,因为miRNA和疾病的关联是无向图,总共生成21 720条边。miRNA的特征可以描述为一个495维向量Fmi:
Fmi=(υmim1,υmim2,υmim3,…,υmim494,υmim495),
式中:Fmi表示miRNA集成相似性矩阵IM的第i行,υmimj是mi和mj之间的集成相似度值。
此外,疾病的特征可以用相同的方式描述为383维向量Fdi,计算过程如下:
Fdi=(υdid1,υdid2,υdid3,…,υdid382,υdid384),
式中:Fdi表示疾病集成相似性矩阵ID的第i行,υdidj是di和di之间的集成相似度值。
考虑到图的异质性,miRNA节点和疾病节点位于不同的特征空间中。采用495×64维度的权重矩阵Wm,将miRNA和疾病节点投影到相同维度的特征空间中。miRNA的转化特征可以表示为:
Hm=Wm·Fm
式中:Hm是miRNA经矩阵Wm转化后的特征,Fm是miRNA的原始特征IM。
类似地,疾病特征的转换可以表示为:
Hd=Wd·Fd
式中:Fd是疾病的原始特征ID,Hd是原始特征Fd经过383×64矩阵Wd变换后的疾病特征。
2.2 基于K最邻近法的特征增强
模型构造一个基于原始数据的KNN图,对于miRNA和疾病节点,是以离散而非连续形式存在的,采用一种离散型相似度计算方法Dot-product来计算第i个miRNA节点mi和第j个miRNA节点的相似度,计算过程如下:


2.3 基于自编码器的深度神经网络
为了增强miRNA和疾病节点的表征能力,本模型还构建了一种DNN模块,由编码器和解码器组成。miRNA和疾病的初始节点特征分别输入到l层自动编码器中,得到新的节点特征输出,第i层自编码器的计算过程如下:
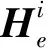
接着计算miRNA和疾病节点特征在经过l层编码器后,在将输出矩阵输入到解码器中,第i层解码器的输出:
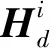
为了防止过拟合,在损失函数中加入l2范数,此子模块损失函数

2.4 GCN模块
自编码器能够从miRNA和疾病特征数据中学习到数据本身的特性,但忽略了数据间结构的关系。通过传递算子在GCN中传播深度神经网络(Deep Neural Network,DNN)中的新表征,集成自编码器后的GCN可以学习到miRNA和疾病数据本身和数据之间的结构关系。对于普通图卷积层的第i层定义为:
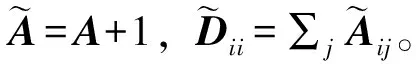

考虑到自编码器能够学习到数据本身并且包含不同miRNA和疾病节点中有效的特征信息,而GCN能够从数据结构中学习到新的特征表示,本模型新设计一种传递算子将自编码器的输出与GCN的输出进行结合,计算过程如下所示:

GCN在融合了自编码器的l层的输入和输出关系后的特征表示为:

在经过该拼接操作后,在逻辑结构上将miRNA和疾病节点的边看作整体,在对其使用softmax函数进行边分类,计算过程如下所示:
2.5 双重监督机制
模型加入双重监督模块[7]将半监督模型GCN和无监督模型自编码器结合成新的聚类模型并对GCN和自编码器进行端到端训练,模型使用Student-t函数进行计算拼接后的miRNA和疾病节点和聚类中心向量之间的相似度,计算过程如下:
式中:hi是H(L)的第i行,μj是通过在预训练自编码器k-means初始化的表征,υ是Student' st-分布的自由度,qij是样本i被分配给类别j的概率,Q=[qij]为样本分配的分布。
为了miRNA与疾病组成的边的数据表征更接近聚类中心,对Q中的每个元素进行平方和归一化,从而提高聚类凝聚性,计算过程如下所示:
(8)
P=[pij]通过对于Q中的每个分配进行平方和归一化,从而使得分配具有更高的置信度。
该模块的损失函数定义如式(9)所示,通过最小化P和Q分布之间的KL散度损失,DNN模块可以学习到miRNA和疾病边特征的有效表示,使得边数据表示更靠近聚类中心。由于P分布和Q分布之间有着紧密联系,在式(8)中,P分布由Q分布计算,在式(9)中,P分布又监督Q分布的更新,所以被视为一种双监督机制。
(9)
如果将聚类分配视为真值标签,可能会带来噪声等各种问题,从而影响miRNA和疾病边预测的准确性,所以本模型需要新的聚类分布Z。因此,可以使用分布P来监督分布Z,过程如下:
最后SDCNMDA模型的总损失定义为:
L=LDNN+αLgcn+βLclu,
式中:L为样本中miRNA和疾病组成的边设置标签,选择分布Z中的软分配作为最终聚类结果。因为GCN学习的表示包含两种不同的信息。最后给第i条miRNA和疾病的边的标签,计算过程如下:
3 试验结果与分析
3.1 试验设置
本模型基于深度图库(Deep Graph Library,DGL),使用5折交叉验证法分别评估模型在HMDD v2.0和HMDD v3.0上的性能。将Adam优化器用于优化模型,二进制交叉熵函数用于计算模型的损失,以获得预测值与实际值之间的接近度。为了获得最佳性能,对模型进行多次调整超参数。在试验开始时,隐藏层尺寸设置为64,输出层尺寸设置为64,编码器和卷积层数设置为3,丢失率为0.3,学习率设置为0.001,KNN算法中的K设置为3,传递算子设置为0.5,权重衰减设置为0.005。
3.2 性能评估
使用5折交叉验证来评估模型的性能。首先,将10 860条样本,包括5 430条正样本和5 430条负样本,随机打乱后均匀地分为5个子集,每个子集有2 172条样本;然后每次选择其中的1个子集作为测试集,其余4个子集作为训练集。采用准确率用准确率(Accuracy)、精确率(Precision)、召回率(Recall)以及 F1 值(F1-score)作为模型的评价指标。模型在HMDD v2.0数据集上的测试结果如表1所示。

表1 SDCNMDA模型在HMDD v2.0的5折交叉验证结果
从表1可以看出,模型的平均准确率为85.34%,精确率为82.74%,召回率为89.29%,F1值为85.89%,标准差分别为0.60%、1.19%、0.45%、0.92%。
在HMDD v3.0数据集上同样进行试验。测试结果如表2所示。

表2 SDCNMDA模型在HMDD v3.0的5折交叉验证结果
从表2可以看出,SDCNMDA模型取得了87.13%的平均准确率,平均精确率为84.54%,平均召回率为90.88%,平均F1值为87.59%,而标准差分别为0.53%、0.82%、0.83%、0.57%。
此外,模型还绘制了受试者工作特征(Receiver Operating Characteristic,ROC)曲线直观显示模型的预测能力,并计算了 ROC 曲线下面积AUC评 估 模 型 的 预 测 能力。在不同的数据集上,5折交叉验证的ROC曲线如图2所示。
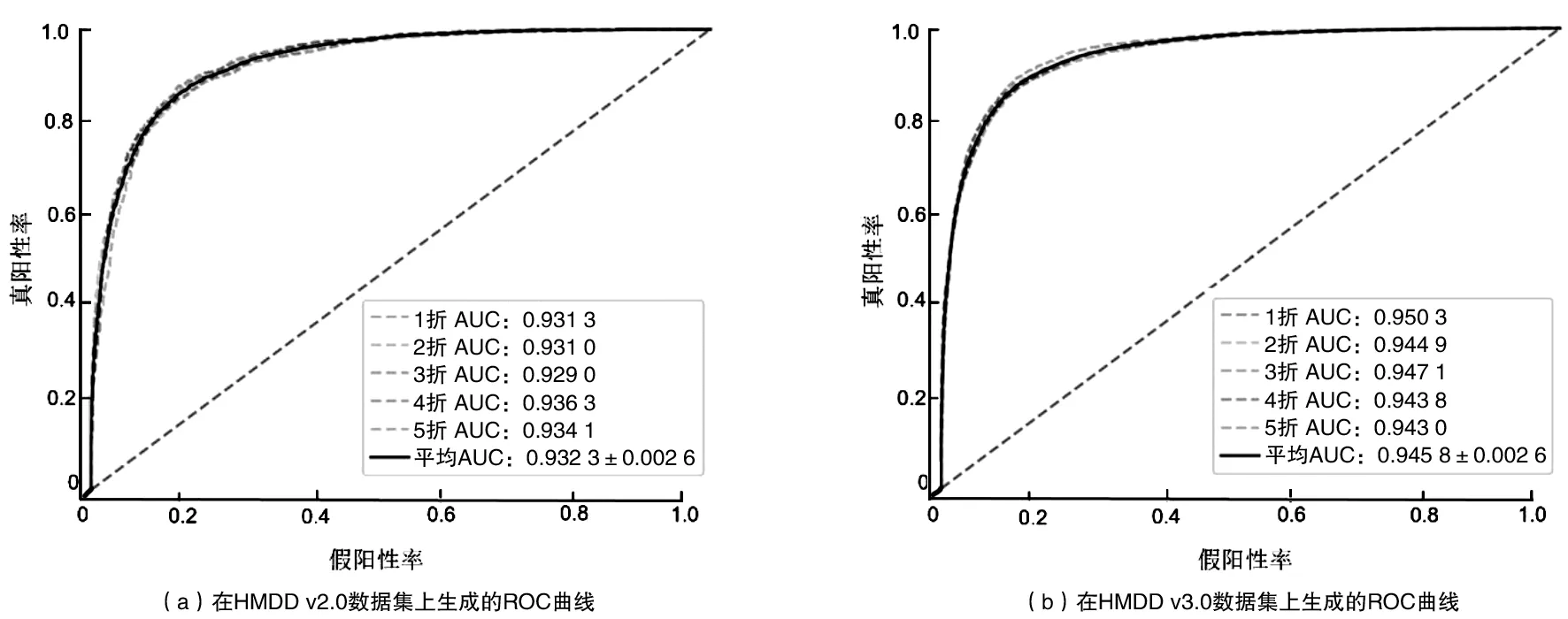
图2 SDCNMDA模型基于5折交叉验证的试验结果
由图2可以看出,本文提出的模型在HMDD v2.0取得的AUC值为93.23%,每折的AUC分别为93.13%、93.10%、92.90%、93.63%和93.41%,在HMDD v3.0取得的AUC值为94.58%,每折的AUC值分别为95.03%、94.49%、94.71%、94.38%和94.30%。
3.3 与其他最新方法的比较
为了评估SDCNMDA模型的性能,将本模型与近年来提出的7个常见模型(SACMDA[8]、KBMMDA[9]、PBMDA[10]、M2GMDA[11]、EDTMDA[12]、MSCHLMDA[13]和MCLPMDA[14])进行了比较,比较结果如表3所示。

表3 SDCNMDA模型在HMDD v2.0和五折交叉验证下与其他模型的性能比较
从表3可以发现,SDCNMD模型的AUC值高于上述7个模型。由于AUC值越高,模型性能越好,故SDCNMDA模型具有良好的预测效果。
4 结论
目前大多数现有miRNA疾病关联预测方法侧重于miRNA和疾病数据自身的特性,往往忽视 miRNA和疾病数据的自身结构。本文提出了采用结构化深度聚类网络来预测miRNA和疾病的关联,将miRNA和疾病的结构信息有效整合到深度神经网络中,使得模型能够学习到miRNA和疾病的数据本身的特性和结构特性。首先,SDCNMDA模型将miRNA和疾病的集成相似性作为特征与K最邻近法得到的新特征拼接后,投入自编码器,再将自编码器中获得的新特征通过传递算子传递到图卷积层,同时利用双监督机制对模型进行监督训练。在HMDD v2.0和HMDD v3.0数据集上,模型SDCNMDA在5折交叉验证法下的平均AUC分别为93.23%和94.58%。通过与领域最新方法的比较,本方法显示出良好的预测性能。
