粗山羊草种质遗传多样性及群体结构的ISSR分析
2023-08-20魏飒张海惠王玉泉吴晓军胡喜贵茹振钢
魏飒 张海惠 王玉泉 吴晓军 胡喜贵 茹振钢
摘 要: 粗山羊草分布范圍广,遗传变异丰富,被认为是改良普通小麦的重要基因源。为深入了解不同来源粗山羊草种质的遗传多样性和群体结构,该研究利用ISSR分子标记对56份粗山羊草种质进行了遗传多样性和群体结构分析。结果表明:(1)16个ISSR引物共检测170条多态性位点,每个ISSR引物多态性位点为3~18条,平均为10.63条;多态性信息(PIC)变异范围为0.17~0.85,平均为0.67。(2)粗山羊草4个群体的遗传多样性比较显示,中亚粗山羊草的群体遗传多样性水平最高(He=0.225 4,I=0.355 7),群体间的基因流较低(Nm=1.638 6)。(3)聚类结果在遗传相似系数约0.67处,来源于塔吉克斯坦6份和土库曼斯坦2份粗山羊草种质材料聚成一类(Group 2);其他48份种质材料形成一大类(Group 1),其中Group 1可进一步分成3个Sub亚类,呈现出来源相同的粗山羊草种质材料倾向聚在一起。(4)群体结构分析将56份粗山羊草种质分为5个群体,其中,来源于西亚伊朗V群体种质材料遗传背景比较一致,混杂程度相对较低;进一步分析各群体Q值,发现IV群体种质材料亲缘关系的来源相对复杂,遗传多样最为丰富。该研究结果可为粗山羊草种质亲缘关系解析、种质多样性保护提供重要参考依据,为其科学利用以及进化研究奠定基础。
关键词: 粗山羊草, 遗传多样性, 聚类分析, 群体结构, ISSR分析
中图分类号: Q948
文献标识码: A
文章编号: 1000-3142(2023)07-1317-09
收稿日期: 2022-08-26
基金项目: 河南省重大公益专项(201300110800); 河南省高校重点科研项目(21A210007)。
第一作者: 魏飒(1998-),硕士研究生,研究方向为种质资源创新与利用,(E-mail)1478441271@qq.com。
通信作者: 胡喜贵,博士,副教授,研究方向为作物种质资源挖掘与利用,(E-mail)xiguihu@126.com。
Genetic diversity and population structure of Aegilops
tauschii accessions based on ISSR method
WEI Sa1, ZHANG Haihui2, WANG Yuquan1, WU Xiaojun1, HU Xigui1*, RU Zhengang1
( 1. Henan Institute of Science and Technology, Xinxiang 453003, Henan, China; 2. Xinxiang
Institute of Engineering, Xinxiang 453700, Henan, China )
Abstract: Aegilops tauschii is considered as an important gene source for improving common wheat, which has wide distribution and rich genetic variation. In order to understand genetic diverstity and population structure of A. tauschii from different origins, ISSR markers were used to evaluate genetic diversity and population structure of 56 A. tauschii accessions. The results were as follows:(1) The 170 polymorphic bands were detected by 16 ISSR primers, and polymorphic bands of each ISSR primer ranged from 3 to 18, with an average of 10.63. The variation of polymorphism information content(PIC)ranged from 0.17 to 0.85, with an averaged of 0.67. (2) The comparison among four populations indicated that the population genetic diversity of Central Asia was the highest (He=0.225 4, I=0.355 7) and gene flow between populations was relatively low (Nm=1.638 6). (3)The clustering results showed that 56 A. tauschii accessions were divided into two groups at the genetic similarity coefficient 0.67, of which eight accessions from Tajikistan and Turkmenistan were clustered in Group 2. And, the Group 1 including 48 accessions could be further divided into three sub-groups, which indicated that A. tauschii accessions with clustering together have the same origin. (4) Based on population structure analysis, 56 A. tauschii accessions were divided into five populations, of which the V population from Iran in West Asia had relatively consistent genetic background and relatively low degree of hybridization. Furthermore, the Q value analysis of populations showed that the genetic relationship of IV population were relatively complex, producing the most abundant genetic diversity. The results of this study can provide an important reference for analysis of genetic relationship, protection of biodiversity, and lay a foundation for the scientific utilization and evolution research of A. tauschii.
Key words: Aegilops tauschii, genetic diversity, clustering analysis, population structure, ISSR analysis
粗山羊草(Aegilops tauschii, DD, 2n=2x=14)别称为节节麦,属于禾本科小麦族(Triticeae)山羊草属(Aegilops),被认为是普通小麦(Triticum aestivum, AABBDD, 2n=6x=42)D基因组祖先的供体种(Lagudah et al., 1991; Dvorak et al., 2012)。粗山羊草主要分布在欧亚大陆中部,其起源中心位于里海南部海岸和阿塞拜疆,而后分别向东越过土库曼斯坦的科彼特山脉扩散到中国的新疆和黄河中部地区,向西穿越土耳其东南的山谷传播至叙利亚中部地区(Lubbers et al., 1991)。已有研究表明,粗山羊草D基因组具有丰富遗传变异性,如抗逆性(Abbas et al., 2021)、抗病性(Olson et al., 2013)、优异品质基因(Hsam et al., 2001)等,已被作为改良普通小麦的重要种质资源之一(Lagudah et al., 1991; Dvorak et al., 2012; 赵昕鹏等, 2019; 郜晓峰等, 2021)。
核心种质的收集和评价是种质的应用、创新及作物新品种选育的基础,尤其对供体物种遗传多样性和群体结构的研究不仅能了解物种亲缘关系,还可以为其合理高效的保护和利用提供理论基础(Mourad et al., 2020)。粗山羊草的遗传多样性已从形态学、生理学、种子储藏蛋白、同工酶、分子标记等进行广泛研究(William et al., 1993; Ghasemzade et al., 2008; Mahjoob et al., 2021)。其中,因为分子标记不受表型条件局限,可早期完成,所以被公认为是研究遗传多样性和群体结构高效、灵活的方法(Abouzied et al., 2013)。Lubbers等(1991)对不同来源102份粗山羊草25个位点的RFLP分析,发现来自里海的粗山羊草变异最大,阿富汗次之,而土耳其和巴基斯坦的变异最小,并进一步支持粗山羊草起源于里海南部海岸。孔令让等(1998)利用RAPD标记分析粗山羊草两个亚种的基因组DNA多态性,表明Aegilops tauschii ssp. tauschii 多态性明显高于A. tauschii ssp. strangulata。Pestsova等(2000)利用SSR标记分析113份粗山羊草遗传多样性,发现来自高加索地区的粗山羊草多样性比中亚地区的丰富,同时,将其划分为两大类且与地理分布一致。
基于内部简单重复序列(inter-simple sequence repeats,ISSR)是一种显性分子标记,具有较高的多态性、重复性、稳定性且操作简便等诸多优点,已广泛应用在埃及小麦(Egyptian wheat)(Abdel-Lateif & Hewedy, 2018)、硬粒小麦(Triticum durum)(Aslan-Parviz et al., 2020)、钩刺山羊(Aegilops triuncialis)(Khodaee et al., 2021)、大麦(Hordeum vulgare)(张超等,2020)等小麦及野生近缘属种的遗传多样性研究。目前,利用ISSR标记研究粗山羊草遗传多样性的研究报道较少,仅李玉阁等(2017)利用9个ISSR标记研究了75份中国粗山羊草的遗传多样性,结果将中国粗山羊草划分为黄河流域和新疆两大群体,并筛选到5份具有独特变异的黄河流域类型,进一步研究认为黄河流域种群是从伊朗或土库曼斯坦南部地区直接传播而来(Wei et al., 2008; Su et al., 2020)。普通小麦的D基因组来源于有限粗山羊草类群,但多倍化和进化“瓶颈”导致其遗传基础日益狭窄,与A、B基因组相比,小麦D基因组的遗传多样性尤其匮乏。为了利用不同来源粗山羊草类群拓展小麦D基因组,首先要解析粗山羊草种质遗传多样性。因此,本研究利用16个ISSR标记对不同来源的56份粗山羊草种质开展遗传多样性和群体结构研究,旨在了解它们的遗传多样性和群体结构,解析它们的亲缘关系,以期为种质多样性保护和科学利用提供理论基础。
1 材料与方法
1.1 试验材料
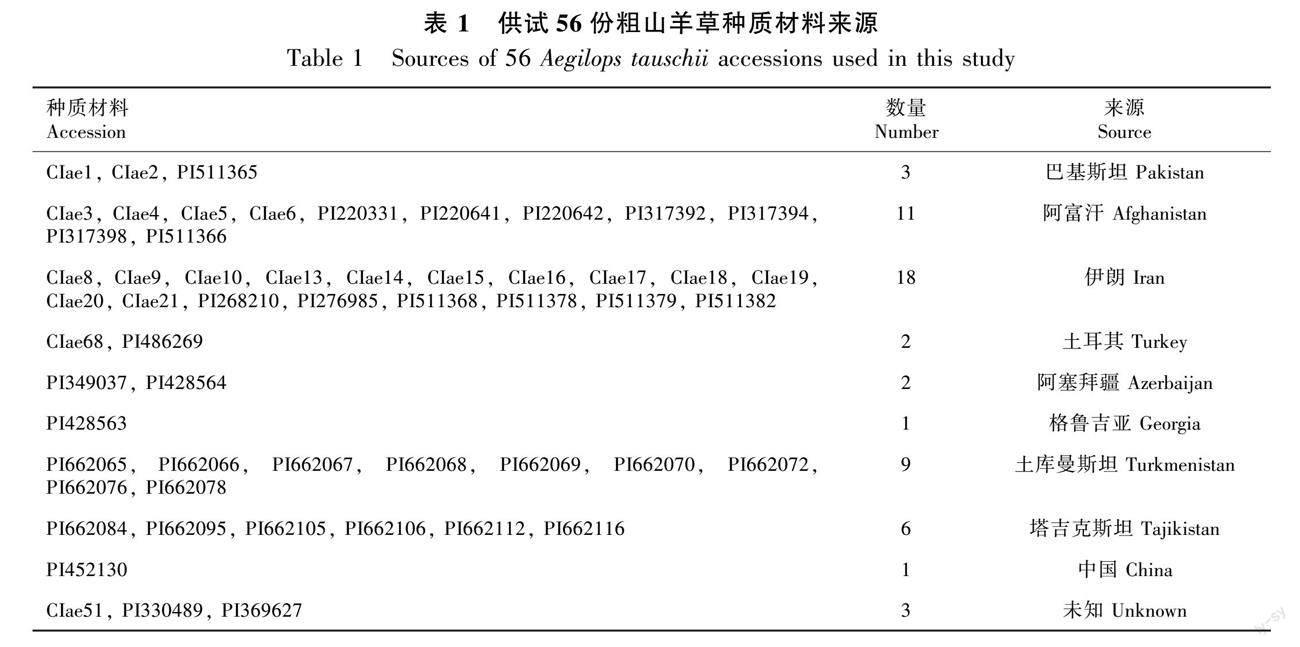
56份粗山羊草(Aegilops tauschii)种质材料(表1),其中来源于西亚23份(伊朗18份、土耳其和阿塞拜疆各2份、格鲁吉亚1份),中亚26份(阿富汗11份、土库曼斯坦9份、塔吉克斯坦6份),南亚3份(巴基斯坦3份),东亚1份(中国1份),未知3份,均由美国种质资源库(National Genetic Resource Program, NPGS)馈赠,在河南科技学院小麦中心繁殖并保存。
1.2 DNA提取
按照Yan等(2002)2×CTAB的方法,从56份粗山羊草种质材料的幼嫩叶片中提取基因组DNA,并经Nanodrop 2000(美国赛默飞世尔)检测DNA浓度和纯度。
1.3 ISSR-PCR扩增分析
参照Khodaee等(2021)研究,选用扩增清晰且重复性好的16个ISSR引物(上海生物工程技术服务有限公司合成)对56份粗山羊草种质材料进行遗传多样性分析(表2)。
ISSR-PCR扩增体系20 μL:10 μL 2×Taq PCR StarMix(北京康润诚业生物科技有限公司)、6 μL ddH2O、2 μL ISSR引物(10 pmol·μL-1)、2 μL DNA(50~60 ng·μL-1)。其PCR扩增程序如下:94 ℃预变性5 min;94 ℃变性30 s,53.7~60.5 ℃(视不同引物而定)退化45 s,72 ℃延伸2 min,共35個循环;72 ℃再延伸7 min,4 ℃继续保存。PCR扩增产物经1.2%变性琼脂糖凝胶电泳分离,D2000 bpDNA分子量标准(天根生化科技有限公司)作为对照,观察结果,照相并保存。
1.4 数据分析
依据ISSR扩增电泳图,分别读取数据,相同迁移率处有带为1,无带为0,模糊不清楚为999,并建立二元数据矩阵。利用PowerMarker 3.2.5(Liu & Muse, 2005)软件分析多态性信息含量(polymorphism information content,PIC);应用Popgene 1.32(Yeh et al., 1999)软件计算群体观察到的等位基因(Na)和有效等位基因数(Ne)、Neis基因多样性指数(He)和Shannons信息指数(I)、不同群体间的总基因多样性(Ht)、群体内的基因多样性(Hs)、群体间的基因多样性(Dst)、遗传分化系数(Gst)和基因流(Nm);采用Ntsys 2.1(Rohlf, 2000)软件,基于简单匹配系数(simple matching coefficient,SM)进行非加权平均法(UPGMA)聚类分析。
将数据转换成Structure 2.3.4(Pritchard et al., 2000)软件的格式,进行群体遗传结构分析。将K值设为2~20,基于模型每个K值进行10次分析,再通过Structure Harvester(Earl & Vonholdt, 2012)确定最合适的K值,构建遗传结构图。同时,计算群体中种质材料变异源于该群体中的概率Q值,用于分析其遗传成分。
2 结果与分析
2.1 56份粗山羊草种质ISSR引物多态性
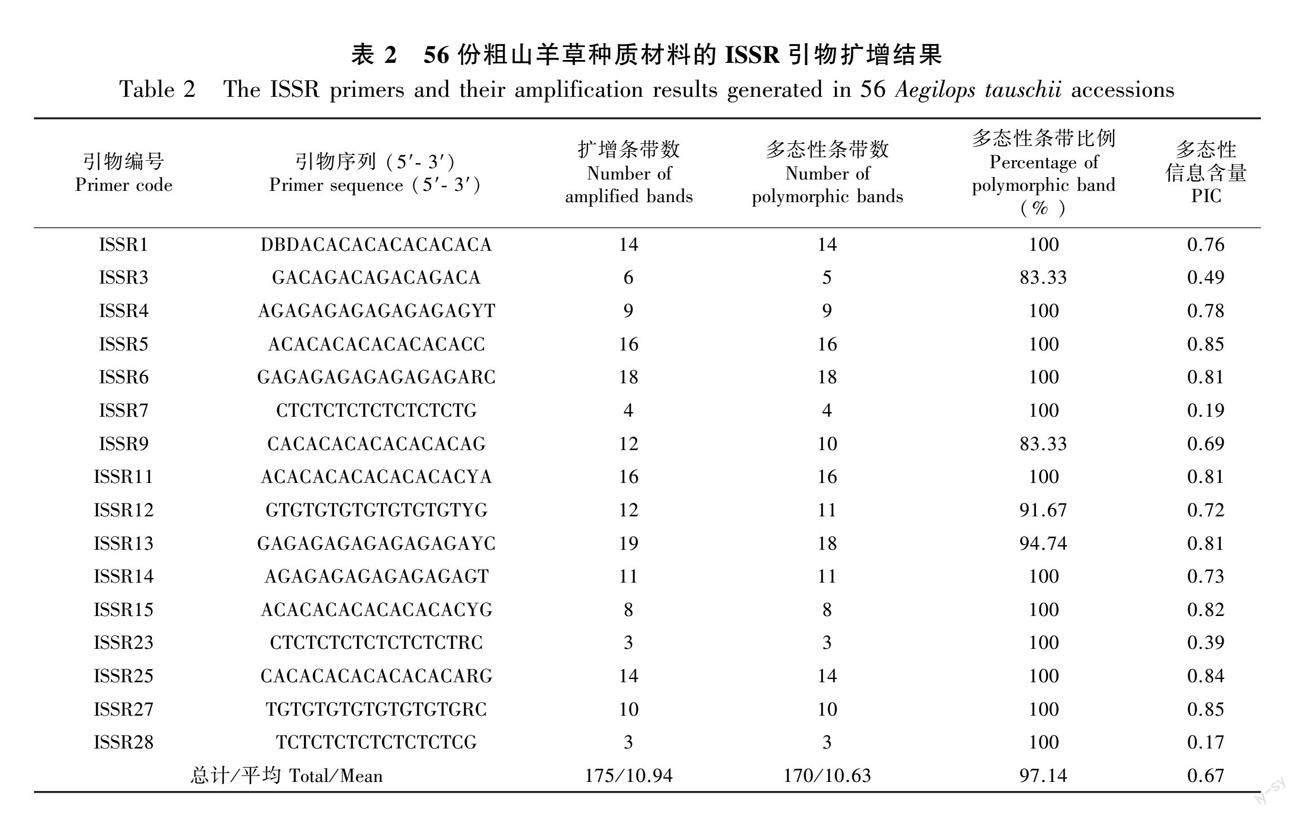
16个ISSR引物在56份粗山羊草种质材料中扩增出175条清晰稳定的条带,其中,ISSR13引物扩增条带最多(19条),ISSR23和ISSR28引物扩增带最少(3条),平均为10.94条。各ISSR引物的多态性条带数为3~18条,平均为10.63条,多态性位点百分率为97.14%。ISSR引物PIC在0.17(ISSR28)~0.85(ISSR5和ISSR27)之间,平均为0.67。其中12个ISSR引物的PIC值高于平均值,占75%(表2)。Botstein等(1980)提出了衡量基因变异程度高低的PIC,并将PIC分成3个程度:高度(PIC>0.50)、中度(0.25≤PIC≤0.50)和低度(PIC<0.25)。本研究中16个ISSR引物PIC中,除引物ISSR3(PIC=0.49)、ISSR7(PIC=0.19)、ISSR23(PIC=0.39)和ISSR28(PIC=0.17)外,其他均表现为高度多态性信息。
2.2 56份粗山羊草种质群体遗传多样性和分化程度分析
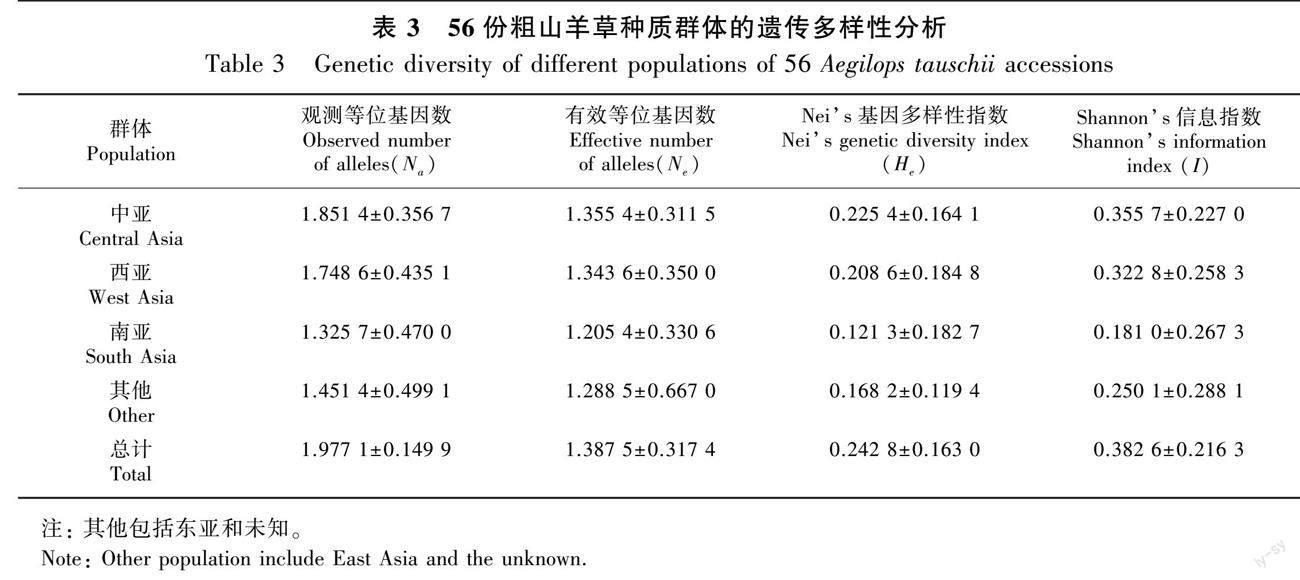
56份粗山羊草种质材料按中亚、西亚、南亚和其他4个群体进行遗传多样性分析,由表3可知,4个群体的观测等位基因数(Na)、有效等位基因数(Ne)、Neis基因多样性指数(He)与Shannons信息指数(I)由高到低排序为中亚>西亚>其他>南亚。这说明中亚粗山羊草的群体遗传多样性水平最高,西亚次之,其他和南亚的相对较低。进一步分析群体基因的分化程度,由表4可知,56份粗山羊草种质材料的总基因多样性(Ht)为0.236 0,4个群体间发生一定的遗传分化(Gst=0.233 8),而群体内基因多样性比值为0.766 2(Hs/Ht), 并存在中等程度的基因交流(Nm=1.638 6)。这说明粗山羊草种质群体间的遗传分化水平较低,大部分变异以群体内为主。
2.3 56份粗山羊草种质的聚类分析
利用Ntsys 2.1软件对56份粗山羊草种质材料的ISSR遗传多样性结果进行聚类分析,可将52份粗山羊草种质材料分开,占92.86%,其种质材料间的配对遗传相似系数(genetic similarity coefficient,GS)变化范围为0.64~0.94(图1)。由聚类结果(图1)可知,在遗传相似系数约0.67处,来源于塔吉克斯坦6份(PI662084、PI662095、PI662105、PI662106、PI662112和PI662116) 和土库曼斯坦2份(PI662076和PI662078)种质材料聚成一类(Group 2),其他48份粗山羊草种质材料形成一大类(Group 1)。而在遗传相似系数约0.70处,可进一步将Group 1分成3个亚类:来源于8个国家的26份种质材料组成Sub 1亚类,其中具有相同来源仍聚在一起,如土库曼斯坦(PI662065、PI662066、PI662067、PI662068、PI662069、PI662070、PI662072)、阿富汗(CIae3、CIae4、CIae5、CIae6、PI220331、PI220641、PI220642、PI317392、PI317394、PI317398、PI511366)等;除3份种质材料(土耳其PI486269和未知PI330489、PI369627)外,Sub 2亚类中种质材料主要来源于伊朗(CIae8、CIae9、CIae10、CIae13、CIae15、CIae16、CIae17、CIae18、CIae19、CIae20、CIae21、PI268210、PI276985、PI511368、PI511378、PI511379、PI511382);Sub 3亞类种质材料来源于阿塞拜疆(PI349037和PI428564)。同时,发现未知CIae51与阿富汗PI220642,未知PI330489和PI369627与伊朗PI276985聚在一起,可推测未知CIae51来源于阿富汗,而未知PI330489和PI369627来源于伊朗。本研究聚类分析结果将大部分粗山羊草种质材料可聚类分开,说明供试粗山羊草种质之间具有一定的遗传差异,也证明了ISSR标记在粗山羊草种质分析方面具有较好的效果。
2.4 56份粗山羊草种质的遗传结构分析
基于ISSR数据,利用Structure 2.3.4软件分析56份粗山羊草种质群体的遗传结构,绘制K与ΔK的关系图(图2),K=5时,ΔK最大。因此,可将56份粗山羊草种质材料分成5个群体。由图3可知,V群体的种质材料主要来源于西亚的伊朗,遗传背景比较一致,其混杂程度相对较低,而其他群体间混合程度相对偏高,尤其IV群体。从遗传结构整体分析也发现,相同来源的种质材料倾向聚在一起,这与聚类分析(图1)的结果相似。
由56份粗山羊草种质材料在各群体中Q值分布(表5)可知,15份种质材料Q值小于0.6,占26.8%,Q值大于0.8和0.9的种质材料分别占50.0%和42.9%,这说明群体中大部分种质材料的亲缘关系比较单一。对各群体Q值大于0.8的进行对比,发现V群体的Q值最大,为83.3%,而IV最小,仅为26.7%。这揭示了IV群体种质材料亲缘关系的来源相对复杂,遗传多样最为丰富。
3 讨论与结论
3.1 ISSR标记分析粗山羊草种质遗传多样性
近年来,分子标记在生物多样性和进化关系方面研究中发挥着重要的作用,ISSR分子标记是基于PCR技术发展起来的一种DNA多态性检测技术。本研究利用16个ISSR标记对56份粗山羊草种质的遗传多样性进行分析,结果表明多态性位点百分率为97.14%,观测等位基因数为1.977 1,有效等位基因数为1.387 5,Neis基因多样性指数为0.242 8,Shannons信息指数为0.382 6, 群体内基因遗传多样性为0.180 9; 李玉阁等(2017)用9个ISSR标记研究75份中国粗山羊草(新疆、河南和陕西)的遗传多样性,多态性位点百分率、观测等位基因数、有效等位基因数、Neis基因多样性指数、Shannons信息指数、群体内基因遗传多样性分别为89.31%、1.993 4、1.471 5、0.273 4、0.415 2、0.236 7。经比较同物种的两个群体,本研究粗山羊草群体(主要来源西亚和中亚)除多态性位点百分率外,各项指标均略低于中国粗山羊草群体,表明粗山羊草种质从西亚和中亚向东传入中国的新疆和黄河中部地区后,中国区域内群体种质发生部分变异,进而提升了遗传多样性水平。另外,利用ISSR研究其他物种遗传多样性, 如钩刺山羊引物多态性条带平均为9.50条、PIC为0.30(Khodaee et al., 2021);硬粒小麦引物多态性条带平均为10.00条、PIC为0.42(Aslan-Parviz et al., 2020);大麦引物多态性条带平均为8.313条(张超等,2020)。与以上不同物种比较,粗山羊草ISSR引物多态性及PIC较高,这有可能与本研究粗山羊草种质材料的地理来源广有关。
3.2 粗山羊草群体遗传结构
Mizuno等(2010)和Sohail等(2012)对不同地理分布的粗山羊草种质遗传结构进行分析,将其分成两个亲缘谱系:L1(隶属于Aegilops tauschii ssp. tauschii)和L2(隶属于A. tauschii ssp. strangulata),并表明少数L2谱系的粗山羊草参与了普通小麦D基因组的起源;而未参与起源的L1谱系的粗山羊草具有丰富遗传变异。Wang等(2013)进一步按地理分布,将两个亲缘谱系细划分成西(W)亚系和东(E)亚系,其中L1W亚系位于土耳其东部、 亚美尼亚、 阿塞拜疆和伊朗西部,L1E亚系位于伊朗中部到中国西部;L2W和L2E亚系分别位于亚美尼亚到阿塞拜疆区域和阿塞拜疆南部到伊朗北部的里海沿岸区域。Pestsova等(2000)利用SSR标记分析不同地理来源的113份粗山羊草遗传多样性,发现高加索地区(格鲁吉亚、亚美尼亚和俄罗斯的达吉斯坦地区)的粗山羊草多样性最为丰富,而中亚(乌兹别克斯坦和土库曼斯坦)粗山羊草多样性最低。本研究56份粗山羊草种质材料按4个群体的遗传多样性对比,表明来自中亚(阿富汗、土库曼斯坦和塔吉克斯坦)的粗山羊草群体遗传多样性水平最高(He=0.225 4,I=0.355 7),这与Pestsova等(2000)研究结果相反,这可能归因于来自中亚阿富汗和塔吉克斯坦的粗山羊草种质变异较大。王亚娟等(2010)基于SSR标记对78份粗山羊草进行聚类分析,在遗传相似系数0.77处划分成6个主要类群;而Sohail等(2012)基于DarT标记对81份粗山羊草进行聚类分析,将其分成3大类(Group A/B/C),Group C类主要来源伊朗。本研究聚类结果在遗传相似系数约0.67处,56份粗山羊草种质材料主要聚成2大类(Group 1和Group 2),其中Group 1进一步分成3个Sub亚类;群体结构将其分成5个群体。两种分析方法在分类数量上略有差异,但均呈现出来源相同的粗山羊草种质材料倾向聚在一起,本研究结果进一步支持种质材料间的亲缘关系与其地理来源有一定的相关性(王亚娟等,2010; Sohail et al., 2012)。同时,本研究56份粗山羊草种质材料中Q值小于0.6的15份,仅占26.8%,也进一步说明大部分种质材料的亲缘关系比较单一,以群体内或地理区域变异为主。因此,粗山羊草种质在传播过程中,群体内种质的基因交流或重组是造成不同群体粗山羊草差异的主要原因。
3.3 粗山羊草保护生物学意义
自然遗传变异是植物生物学最重要的基础资源之一(Koornneef et al., 2004)。普通小麦是四倍体小麦和粗山羊草天然雜交后经过染色体加倍而形成的,其起源、驯化及现代育种过程,排斥了供体物种的大量遗传变异(Dubcovsky & Dvorak, 2007; Dvorak et al., 2012)。模拟普通小麦起源过程而创制的人工合成小麦,是重新引入供体物种遗传变异的重要桥梁,在小麦育种中的应用潜力很大(Hao et al., 2019)。但Waines(1998)研究发现,许多粗山羊草物种的栖息地已经遭受破坏甚至面临危机,尤其是在地中海东部沿岸地区。因此,从遗传多样性保护来看,了解粗山羊草种群结构十分重要。本研究对不同来源的56份粗山羊草种质进行遗传多样性和群体结构分析,了解它们的亲缘关系,对每个种群或地区选取代表性的种质保护,可为今后粗山羊草自然遗传变异研究与利用提供基础。
参考文献:
ABBAS A, YU HY, CUI HL, et al., 2021. Genetic diversity and synergistic modulation of salinity tolerance genes in Aegilops tauschii Coss [J]. Plants, 10(7): 1393.
ABDEL-LATEIF KS, HEWEDY OA, 2018. Genetic diversity among Egyptian wheat cultivars using SCoT and ISSR markers [J]. SABRAO J Breed Genet, 50(1): 36-45.
ABOUZIED HM, ELDEMERY SMM, ABDELLATIF KF, 2013. SSR-based genetic diversity assessment in tetraploid and hexaploid wheat populations [J]. British Biotechnol J, 3(3): 390-404.
ASLAN-PARVIZ M, OMIDI M, RASHIDI V, et al., 2020. Evaluation of genetic diversity of durum wheat (Triticum durum Desf.) genotypes using inter-simple sequence repeat (ISSR) and caat box-derived polymorphism (CBDP) markers [J]. Genetika, 52(3): 895-909.
BOTSTEIN D, WHITE RL, SKOLNICK M, et al., 1980. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. Am J Hum Genet, 32(3): 314-331.
DUBCOVSKY J, DVORAK J, 2007. Genome plasticity a key factor in the success of polyploid wheat under domestication [J]. Science, 316: 1862-1866.
DVORAK J, DEAL KR, LUO MC, et al., 2012. The origin of spelt and free-threshing hexaploid wheat [J]. J Hered, 103(3): 426-441.
EARL DA, VONHOLDT BM, 2012. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method [J]. Conser Genet Resour, 4(2): 359-361.
GAO XF, ZHOU Y, SONG DY, et al., 2021. Construction of core collection of Chinese Aegilops tauschii Coss. germplasm resource based on spike morphological traits and molecular markers [J]. J Plant Genet Resour, 22(2): 361-370. [郜曉峰, 周仪, 宋丹阳, 等, 2021. 基于穗形特征与分子标记进行中国节节麦核心种质的创建 [J]. 植物遗传资源学报, 22(2): 361-370.]
GHASEMZADE R, BEHAMTA MR, AGHAII MJ, et al., 2008. Intra- and inter-population diversity of Iranian Aegilops tauschii based on seed storage protein electrophoresis [J]. Int J Agric Biol, 10(4): 463-465.
HAO M, ZHANG LQ, ZHAO LB, et al., 2019. A breeding strategy targeting the secondary gene pool of bread wheat: introgression from a synthetic hexaploid wheat [J]. Theor Appl Genet, 132(8): 2285-2294.
HSAM SLK, KIEFFER R, ZELLER FJ, 2001. Significance of Aegilops tauschii glutenin genes on breadmaking properties of wheat [J]. Cereal Chem, 78(5): 521-525.
KHODAEE L, AZIZINEZHAD R, ETMINAN AR, et al., 2021. Assessment of genetic diversity among Iranian Aegilops triuncialis accessions using ISSR, SCoT, and CBDP markers [J]. J Genet Eng Biotechnol, 19: 5.
KONG LR, DONG YC, JIA JZ, 1998. Random amplified polymorphism of DNA analysis in Aegilops tauschii [J]. Acta Bot Sin, 40(3): 223-227. [孔令让, 董玉琛, 贾继增, 1998. 粗山羊草随机扩增多态性DNA研究 [J]. 植物学报, 40(3): 223-227.]
KOORNNEEF M, ALONSO-BLANCO C, VREUGDENHIL D, 2004. Naturally occurring genetic variation in Arabidopsis thaliana [J]. Ann Rev Plant Biol, 55(1): 141-172.
LAGUDAH ES, APPELS R, BROWN AHD, 1991. The molecular-genetic analysis of Triticum tauschii, the D-genome donor to hexaploid wheat [J]. Genome, 34(3): 375-386.
LI YG, SU YZ, ZHANG DL, et al., 2017. Genetic diversity of Aegilops tauschii accessions native to China revealed by ISSR markers [J]. J Triticeae Crops, 37(1): 30-39. [李玉閣, 苏亚中, 张大乐, 等, 2017. 中国节节麦基于ISSR标记的遗传多样性分析 [J]. 麦类作物学报, 37(1): 30-39.]
LIU K, MUSE SV, 2005. PowerMarker: an integrated analysis environment for genetic marker analysis [J]. Bioinformatics, 21(9): 2128-2129.
LUBBERS EL, GILL KS, COX TS, et al., 1991. Variation of molecular markers among geographically diverse accessions of Triticum tauschii [J]. Genome, 34(3): 354-361.
MAHJOOB MMM, GORAFI YSA, KAMAL NM, et al., 2021. Genome-wide association study of morpho-physiological traits in Aegilops tauschii to broaden wheat genetic diversity [J]. Plants, 10(2): 211.
MIZUNO N, YAMASAKI M, MATSUOKA Y, et al., 2010. Population structure of wild wheat D-genome progenitor Aegilops tauschii Coss.: implications for intraspecific lineage diversification and evolution of common wheat [J]. Mol Ecol, 19(5): 999-1013.
MOURAD AMI, BELAMKAR V, BAENZIGER PS, 2020. Molecular genetic analysis of spring wheat core collection using genetic diversity, population structure, and linkage disequilibrium [J]. BMC Genom, 21(1): 1-12.
OLSON EL, ROUSE MN, PUMPHREY MO, et al., 2013. Introgression of stem rust resistance genes SrTA10187 and SrTA10171 from Aegilops tauschii to wheat [J]. Theor Appl Genet, 126(10): 2477-2484.
PESTSOVA E, KORZUN V, GONCHAROV NP, et al., 2000. Microsatellite analysis of Aegilops tauschii germplasm [J]. Theor Appl Genet, 101: 100-106.
PRITCHARD JK, STEPHENS M, DONNELLY P, 2000. Inference of population structure using multilocus genotype data [J]. Genetics, 155(2): 945-959.
ROHLF FJ, 2000. NTSYS-pc: numerical taxonomy and multivariate analysis system Version 2.1 [Z]. New York: Exeter Software.
SOHAIL Q, SHEHZAD T, KILIAN A, et al., 2012. Development of diversity array technology (DArT) markers for assessment of population structure and diversity in Aegilops tauschii [J]. Breed Sci, 62(1): 38-45.
SU YZ, ZOU MW, ZHU YM, et al., 2020.Analysis of population structure and origin in Aegilops tauschii Coss. from China through SNP markers [J]. Genet Resour Crop Evol, 67(4): 923-934.
WAINES JG, 1998. In situ conservation of wild relatives of crop plants in relation to their history [M]//DAMANIA AB, VALKOUN J, WILCOX G, et al. The Origins of Agriculture and Crop Domesticatio. ICARDA, Apeppo, Syria: 300-306.
WANG JR, LUO MC, CHEN ZX, et al., 2013. Aegilops tauschii single nucleotide polymorphisms shed light on the origins of wheat D-genome genetic diversity and pinpoint the geographic origin of hexaploid wheat [J]. New Phytol, 198(3): 925-937.
WANG YJ, WANG CY, LIU XL, et al., 2010. Genetic diversity of Aegliops tauschii based on SSR marker [J]. J Agric Biotechnol, 18(3): 493-500. [王亚娟, 王长有, 刘新伦, 等, 2010. 基于SSR标记的粗山羊草遗传多样性分析 [J]. 农业生物技术学报, 18(3): 493-500.]
WEI HT, LI J, PENG ZS, et al., 2008. Relationships of Aegilops tauschii revealed by DNA fingerprints: the evidence for agriculture exchange between China and the West [J]. Prog Nat Sci, 18(12): 1525-1531.
WILLIAM MDHM, PEA RJ, MUJEEB-KAZI A, 1993. Seed protein and isozyme variations in Triticum tauschii (Aegilops squarrosa) [J]. Theor Appl Genet, 87: 257-263.
YAN ZH, WAN YF, LIU KF, et al., 2002. Identification of a novel HMW glutenin subunit and comparison of its amino acid sequence with those of homologous subunits [J]. Chin Sci Bull, 47(3): 222-226.
YEH FC, YANG RC, BOYLE T, 1999. POPGENE version 1.32 Microsoft windows-based freeware for populations genetic analysis [Z]. Edmonton: University of Alberta.
ZHANG C, Cairangzhuoma, KA MX, et al., 2020. Genetic diversity of naked barley analyzed by ISSR molecular markers [J]. Mol Plant Breed, 18(4): 1193-1201. [張超, 才让卓玛, 卡毛先, 等, 2020. 应用ISSR分子标记分析裸大麦的遗传多样性 [J]. 分子植物育种, 18(4): 1193-1201.]
ZHAO XP, ZHOU Y, L LL, et al., 2019. Genetic diversity of Aegilops tauschii Coss. and its utilization in improving common wheat [J]. Biotechnol Bull, 35(7): 181-189. [赵昕鹏, 周云, 吕琳琳, 等, 2019. 节节麦遗传多样性及在改良普通小麦中的应用 [J]. 生物技术通报, 35(7): 181-189.]
(责任编辑 周翠鸣)
