水稻OsCDF1基因突变效应及其基因组变异分析
2023-07-20胡艳娟王天穹王晓雪
胡艳娟 薛 丹 耿 嫡 朱 末 王天穹 王晓雪
水稻基因突变效应及其基因组变异分析
胡艳娟 薛 丹 耿 嫡 朱 末 王天穹 王晓雪*
沈阳农业大学水稻研究所, 辽宁沈阳 110866
开花期(又叫抽穗期)影响水稻的产量、品质和地区适应性。在拟南芥中, Cycling DOF Factor 1 (CDF1)蛋白质是()的转录抑制因子, 负向调节拟南芥的开花期。但是, 水稻中的同源蛋白OsCDF1的功能尚不完全清楚。为了揭示OsCDF1在水稻中的生物学功能及其对开花期的影响, 本研究利用CRISPR/Cas9基因编辑技术设计定向敲除水稻基因的2个靶位点, 构建基因的敲除载体; 通过农杆菌介导的方法分别转化北方粳稻品种沈农9816, 创制基因的突变体; 分析沈农9816和突变体在田间种植下的开花期和产量性状的差异。主要研究结果为: 创制了在第1个外显子的第16 bp处缺失5个碱基和在第2个外显子的338 bp处单碱基A插入的纯合突变。序列比对分析表明, 这2种类型的突变均造成移码和蛋白翻译提前终止。在自然长日照条件下,突变体的开花期比野生型沈农9816晚4 d以上, 其产量高于野生型。对单倍型和单倍型网络分析发现,在不同品种中进化出高度多样性。本研究成功利用CRISPR/Cas9基因编辑技术敲除了水稻基因, 为进一步研究基因功能提供了理论参考, 也为水稻遗传改良提供了潜在的基因和种质资源。
水稻; CRISPR/Cas9; OsCDF1; 开花期; 产量相关性状; 单倍型
作为我国第一口粮作物, 水稻()在我国总产已经连续11年稳定在2亿吨以上。尽管当前稻谷产需形势较好, 但人口的不断增加和可耕地面积的减少, 提高水稻产量仍然是农业生产面临的挑战[1]。开花是水稻从营养生长向生殖生长的重要转换点, 开花期是水稻分子育种的生物学基础之一, 是决定品种地区与季节适应性的重要农艺性状。水稻在开花之前分蘖旺盛, 生长迅速, 为开花后的生殖生长提供强有力的基础[2]。水稻品种如果过早开花, 营养生长量小, 不能充分利用光照和温度资源, 形成产量的物质基础不足, 从而导致产量降低; 相反, 如果水稻品种过晚开花, 在适宜的生长季节内不能完成开花和籽粒的发育, 也会导致产量降低。因此, 水稻开花期与其产量密切相关。随着现代分子生物学技术的发展, 利用正向和反向遗传学相结合的方法, 一些与水稻开花期相关的基因被定位和克隆[3-4]。
开花期由许多环境因素共同决定的, 光周期是其中最主要的一个因素。光周期调控植物开花期途径包括光受体、生物钟和成花基因(floral integrator genes)[5-8]。拟南芥()是双子叶植物的模式系统, 是典型的低温长日照植物。拟南芥中成花素基因()促进开花[9-10]。在长日照条件下, 转录因子CONSTANS (CO)诱导的表达, 从而促进开花[11]。CO含有2种锌指结构域, 一个位于N端介导蛋白互作, 另一个是位于C端的CCT (constans, constsans-like, and timing of cab expression 1)结构域[12]。生物钟相关蛋白质GIGANTEA (GI)能够调控的表达, 影响拟南芥开花[13]。()和在相同组织器官中表达, CDF1能够与FLAVIN- BINDING, KELCH REPEAT, F-BOX 1 (FKF1)形成复合体结合到的启动子区, 抑制转录[14-16]。此外, 转录因子CDF1是和表达时间的关键调节因子。在拟南芥开花期的上午, TOPLESS (TPL)蛋白能够与CDF1蛋白含有的non-EAR基序保守结构域结合形成CDF-TPL转录复合体, 抑制和的表达[17]。FKF1属于F-box蛋白家族, 可以直接与靶蛋白CDF1相互作用, 通过多泛素化介导的26S蛋白酶体降解CDF1, 提高和的表达, 是开花期的正向调节因子。因此,突变延迟开花[18]。
水稻是典型的短日照作物, 与拟南芥相比, 水稻调控开花期通路尚不完善。在水稻中,()是第1个被克隆的与开花期相关的基因,是拟南芥的同源基因[19-20]。水稻Heading date 3a (Hd3a)和RICE FLOWERING LOCUS T 1 (RFT1)都是拟南芥FT的同源蛋白, 与拟南芥中通路类似, OsGI通过影响来调控成花素的表达, 最终影响开花[8,19]。与拟南芥不同的是, Early heading date 1 (Ehd1)促进和的表达是水稻特有的开花期相关的一条途径[21]。OsCDF1是拟南芥CDF1在水稻中的同源蛋白。在长日照条件下,的超表达植株提前开花, 并且改变了水稻的株型, 例如株高变矮、叶夹角变小、一次枝梗数和二次枝梗数降低[22-23]。但是,突变对水稻开花期和产量的影响尚不完全清楚。
CRISPR/Cas9基因组编辑系统作为目前有效的基因编辑系统, 在作物性状改良及培育新品种上有广阔的应用前景[24]。本研究利用沈农9816 (Shen nong 9816, SN9816)为背景材料, 通过CRISPR/Cas9基因组编辑技术定点敲除在水稻中的同源基因, 获得了突变体。以T3代分离出的非转基因纯合突变体和为试验材料, 对SN9816和突变体的开花期及产量相关性状进行了调查和分析。并对基因组的遗传变异和单倍型进行分析, 为进一步研究基因的功能, 利用创制新的种质资源提供了理论依据。
1 材料与方法
1.1 试验材料
于2018—2021年在沈阳农业大学农学院进行, 选用沈阳农业大学水稻研究所培育的SN9816粳稻品种为试验材料。沈农9816是沈阳农业大学水稻所1998年以江西丝苗为母本, 以辽粳454为父本人工去雄杂交, 再以辽粳454为轮回亲本回交系选育而成。大肠杆菌() TOP10和农杆菌() EHA105菌株感受态细胞由沈阳农业大学农学院水稻研究所实验室自制。pRGEB32载体由华中农业大学谢卡斌老师提供[25]。限制性内切酶I和T4DNA连接酶购自NEB (北京)有限公司; 琼脂糖凝胶回收试剂盒等从天根生化科技(北京)有限公司订购; 引物合成及测序由华大基因科技(北京)有限公司完成。
1.2 基因编辑载体的构建
通过日本RAP-DB网站(http://rapdb.dna.affrc.go. jp/)获得(Os03g0169600, LOC_Os03g07360) 基因的基因组DNA序列, 利用德国癌症研究中心的E-CRISP Design (www.e-crisp.org/E-CRISP/designcrispr)设计guide RNA (gRNA)引物。利用日本RAP-DB网站BLAST检验引物特异性。在正向引物5′端加上黏性末端GGCA, 在反向引物5′端加上黏性末端AAAC。引物双链化后与经I酶切后的pRGEB32载体连接。用pRGEB32载体上I酶切位点的侧翼序列设计的正/反向引物gRF/R扩增片段, 测序无误后将连接好的载体通过液氮冻融法转入农杆菌。提取农杆菌质粒, 反转到大肠杆菌感受态中, 再次测序无误后, 载体构建完成。所用引物见表1。

表1 引物及序列信息
1.3 编辑株系的突变位点检测
利用农杆菌介导的水稻成熟胚遗传转化法将构建好的载体转入SN9816愈伤组织中[26-27]。提取T1代植株幼苗叶片DNA作为模板, 通过转基因植株的潮霉素筛选标记(hygromycin, Hyg)引物, PCR扩增Hyg元件, 可以扩增出Hyg元件的植株, 即为T1代阳性植株。然后用gRNA的侧翼序列设计突变位点检测引物。用突变位点检测引物扩增, 测序分析PCR产物, 检测编辑位点是否发生突变。将发生突变的植株种植到T2代, 提取植株幼苗叶片DNA, 通过PCR扩增Hyg元件, 不能扩增出Hyg元件的植株, 即为Transfer-DNA (T-DNA)分离的基因编辑植株。所用引物见表1。
1.4 生物信息学分析
利用MEGA-X软件对SN9816和突变体进行OsCDF1蛋白序列比对。通过Jalview软件展示比对结果。通过ROBETTA网站(https://robetta.bakerlab. org/)预测SN9816和突变体的蛋白三级结构。
1.5 光周期响应分析
将T-DNA分离的基因编辑植株播种于试验田中, 按当地常规水肥管理制度进行管理。播种之日起, 至稻穗随着茎秆的伸长而露出1 cm之日, 之间所经历的天数即为开花期。
1.6 水稻产量相关性状分析
将T-DNA分离的纯合基因编辑植株种植于沈阳农业大学试验田中。4月上旬播种, 5月中旬插秧, 行株距30.0 cm × 13.3 cm, 施标准氮肥975 kg hm–2, 分3段5次施入; 施二铵112.5 kg hm–2, 做基肥一次性施入; 施钾肥112.5 kg hm–2, 作基肥一次性施入。水层管理采用浅湿干间歇灌溉; 注意防治稻曲病、纹枯病和稻瘟病。在收获时, 每份试材每个小区选取有代表性的9个单株, 将穗子从穗茎节下面剪下来, 分单株放入信封中, 风干2周后进行考种。测量穗长、穗重、一次枝梗数、二次枝梗数、穗总粒数、穗实粒数、穗瘪粒数、粒长、粒宽、粒厚、结实率、千粒重和单株产量。以上试验2019—2021年进行, 共计3次生物学重复。利用GraphPad Prism 9软件计算种间差异, 以≤0.05为显著性差异。
1.7 表达模式分析
以SN9816为材料, 取7 d地上幼苗和地下根部, 三叶期至四叶期取生长点, 水稻孕穗期取剑叶、倒二叶、倒三叶、茎节、叶鞘和幼穗, 以及播种后50~110 d, 每间隔10d取一次完全展开的新叶, 提取上述全部样品RNA, 反转录成cDNA。以(Os03g0718100, LOC_Os03g50885)作为内参, 通过qRT-PCR分析在水稻中的不同组织器官和不同发育时期的表达量, 以2–ΔCT法计算基因的相对表达量。所用引物见表1。
1.8 单倍型以及单倍型网络分析
通过RiceVarMap2 (http://ricevarmap.ncpgr.cn/)和RFGB (https://www.rmbreeding.cn/)网站分析的基因组DNA变异、单倍型和单倍型网络。网站中包括栽培水稻的类型有籼稻、粳稻、Aus稻、中间型。籼稻又可分为具有华南种质的籼稻I、含有东南亚种质的籼稻II和籼稻中间型。粳稻又可分为温带粳稻、热带粳稻和粳稻中间型。
2 结果与分析
2.1 gRNA引物的设计以及植物转基因载体的构建
根据基因的基因组DNA序列, 选取CDS编码区(+4 ~ +23) bp作为第1个靶点序列, (+24 ~ +26) bp作为protospacer adjacent motif (PAM)序列。选取CDS编码区(+321 ~ +340) bp作为第2个靶点序列, (+341 ~ +343) bp作为PAM序列(图1-A)。利用RAP-DB网站分别BLAST两个gRNA引物, 均具有特异性。
将gRNA引物双链化, 并与pRGEB32载体连接。用阳性克隆的检测引物gRF和gRR扩增连接后的载体, 条带长度350 bp (图1-B), 测序结果显示gRNA引物连入载体中(图1-C)。将重组载体转入农杆菌感受态细胞EHA105中, 再次测序, 结果显示无误。2个重组构建分别命名为和。
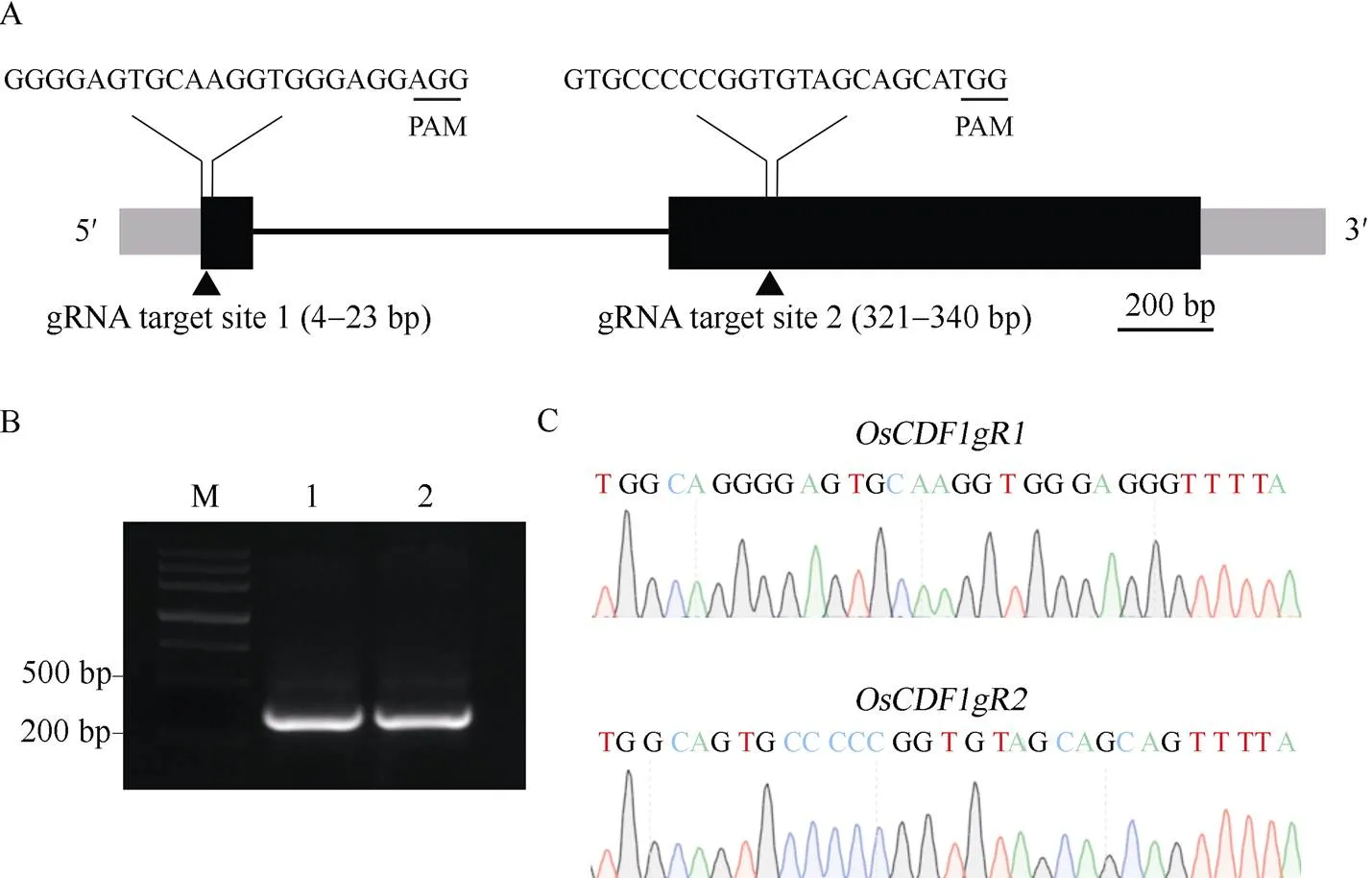
图1 OsCDF1gRNA靶位点示意图及含有OsCDF1 gRNA的阳性克隆筛选
A:靶位点示意图。灰色矩形为5'和3' untranslated region (UTR); 黑色矩形为外显子; 黑线为内含子。比例尺为200 bp。PAM: 前间隔序列邻近基序; 黑色下画线表示PAM序列。B: PCR扩增连接到pRGEB32载体的gRNA。M: DNA marker III; 1~2: PCR产物。C: 含有OsCDF1的gRNA的阳性克隆测序结果。
A:target site in schematic diagram. The grey, bluerectangles, and black lines represent 5' or 3' UTRs, exons and introns, respectively. Bar: 200 bp. PAM: protospacer adjacent motif. The sequences underlined indicate PAM sequences. B: the amplification of the fragments containing gRNAs in pRGEB32 vector by PCR. M: DNA marker III; 1–2: PCR products. C: the sequencing results of the positive clones containing gRNAs of.
2.2 非转基因纯合oscdf1s突变体的筛选
将2个重组构建和分别转入SN9816愈伤组织中, 通过农杆菌介导的遗传转化法各获得15株和17株T1代阳性植株(图2-A)。利用突变位点检测引物扩增T1代阳性植株靶点区域并测序。另外, 测序对比了沈农9816的该区间DNA序列, 证明沈农9816的测序结果与日本晴参考基因组的序列相同。结果显示, 重组构建再生转化植株中, 有11株发生突变, 有2种突变类型(图2-B)。重组构建再生转化植株中, 有7株发生突变, 有1种突变类型(图2-B)。
为最终获得非转基因纯合突变体, 提取T2代植株幼苗叶片DNA, 通过PCR扩增元件。选取扩增后无条带的植株DNA, 利用突变位点检测引物扩增靶点区域并测序。综合2个构建结果, 最终获得在第1个外显子的第16 bp处缺失5 bp碱基和在第2个外显子第221 bp处插入单碱基A的2种纯合突变类型(图2-C, D), 将其分别命名为和。
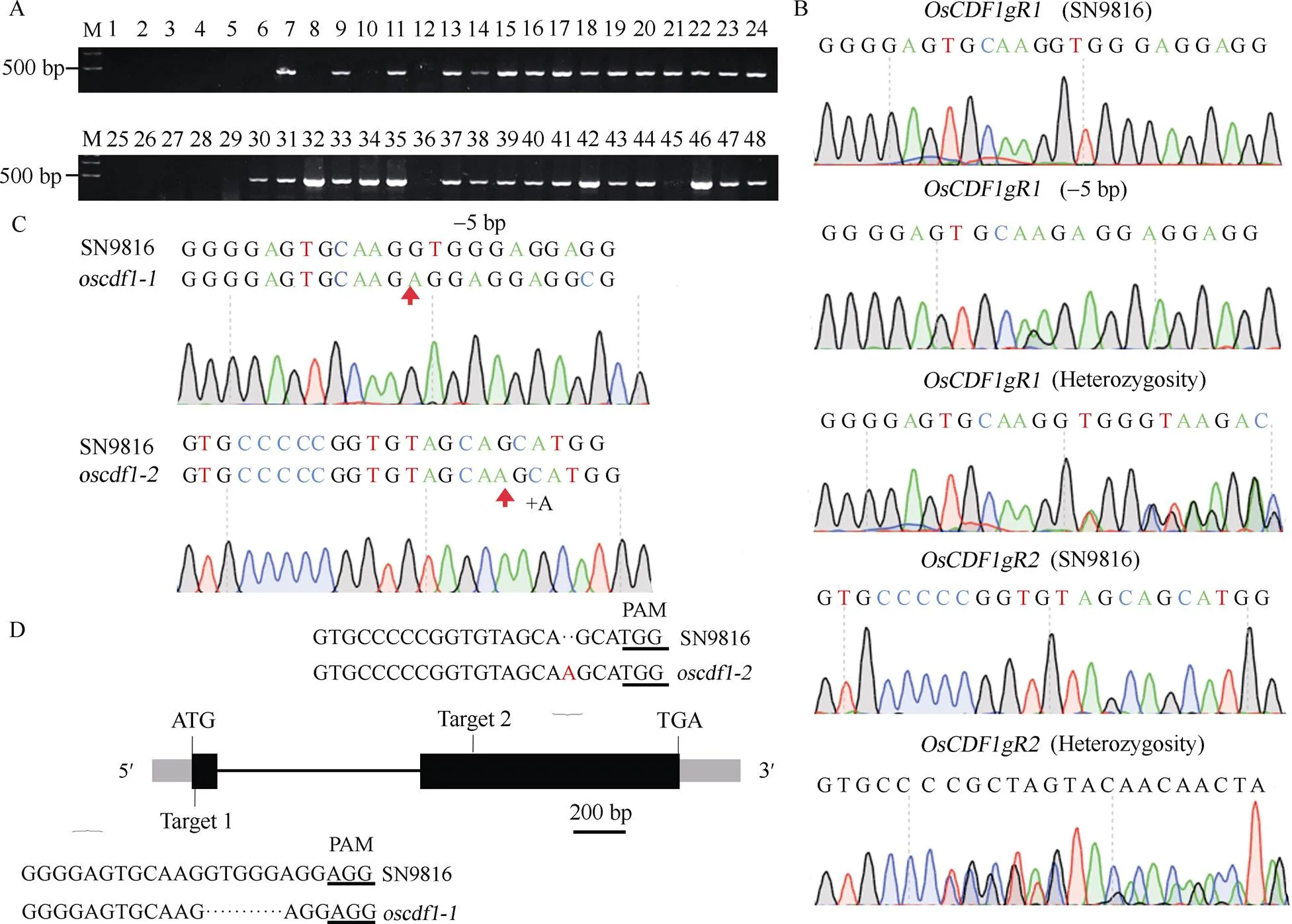
图2 纯合不含T-DNA的oscdf1突变体筛选及分析
A: PCR扩增基因片段结果。M: DNA marker III; 1~48: PCR产物, 1: 模板是ddH2O。B: T1代突变类型分析。C: T2代突变类型分析。红色箭头表示突变位点。D: 靶位点核苷酸变化。灰色矩形为5'和3' UTR; 黑色矩形为外显子; 黑线为内含子。PAM: 前间隔序列邻近基序; 黑色下画线表示PAM序列。标尺200 bp。
A: PCR amplification offragments. M: DNA marker III; 1–48: PCR products; 1: template is ddH2O. B: mutation type analysis in T1generation. C: the mutation type analysis in T2generation. Red arrows show the mutation sites. D: the change of the target sites. The grey, blue rectangles, and black lines represent 5' or 3' UTRs, exons, and introns, respectively. PAM: protospacer adjacent motif. The sequences underlined represent PAM. Bar: 200 bp.
2.3 OsCDF1突变蛋白的生物信息学分析
为确认蛋白水平是否发生了有效突变, 分析比对了突变蛋白序列。结果表明, 2种突变分别在第6个氨基酸处由缬氨酸变为精氨酸、第112个氨基酸处丝氨酸变为赖氨酸, 并产生移码突变, 最终导致翻译提前终止(图3-A, B)。编码氨基酸数由野生型的441个缩短为36个和178个(图3-B)。
蛋白质三级结构预测表明,和突变蛋白的三级结构与野生型相比更加简单, 螺旋结构明显减少, 破坏了OsCDF1的三级结构(图3-C)。综上所述, 一系列分析表明在核酸及蛋白水平都发生了突变, 表明基因功能可能受到影响。
2.4 oscdf1突变体开花期调查
在自然长日照条件下, 种植SN9816和突变体和, 并调查了开花期, 结果显示, 突变体(106 d ± 4 d)比野生型(100.5 d ± 1.5 d)晚5d以上抽穗, 突变体(105 d ± 1 d)比野生型 (100.5 d ± 1.5 d)晚4d以上抽穗(图4)。
2.5 oscdf1突变体产量相关性状的调查
纯合和突变植株经田间种植试验, 对其穗部相关农艺性状调查发现,突变体与野生型SN9816相比穗长极显著变短(图5-A, D),但单株穗数极显著升高(图5-C)。突变体的一次枝梗数与野生型SN9816无显著差异(图5-B, E), 而二次枝梗数与野生型SN9816相比显著下降(图5-F)。
对野生型SN9816和突变体粒部相关农艺性状调查发现, 除了突变体的粒长极显著长于野生型SN9816外,突变体的粒宽和粒厚均与野生型SN9816无显著差异(图5-G~K)。突变体的每穗饱粒数极显著下降(图5-L)。突变体的结实率极显著低于野生型SN9816, 但千粒重显著高于野生型SN9816 (图5-M, N)。表明基因突变影响正常田间种植模式下的植株农艺性状和产量构成因素。
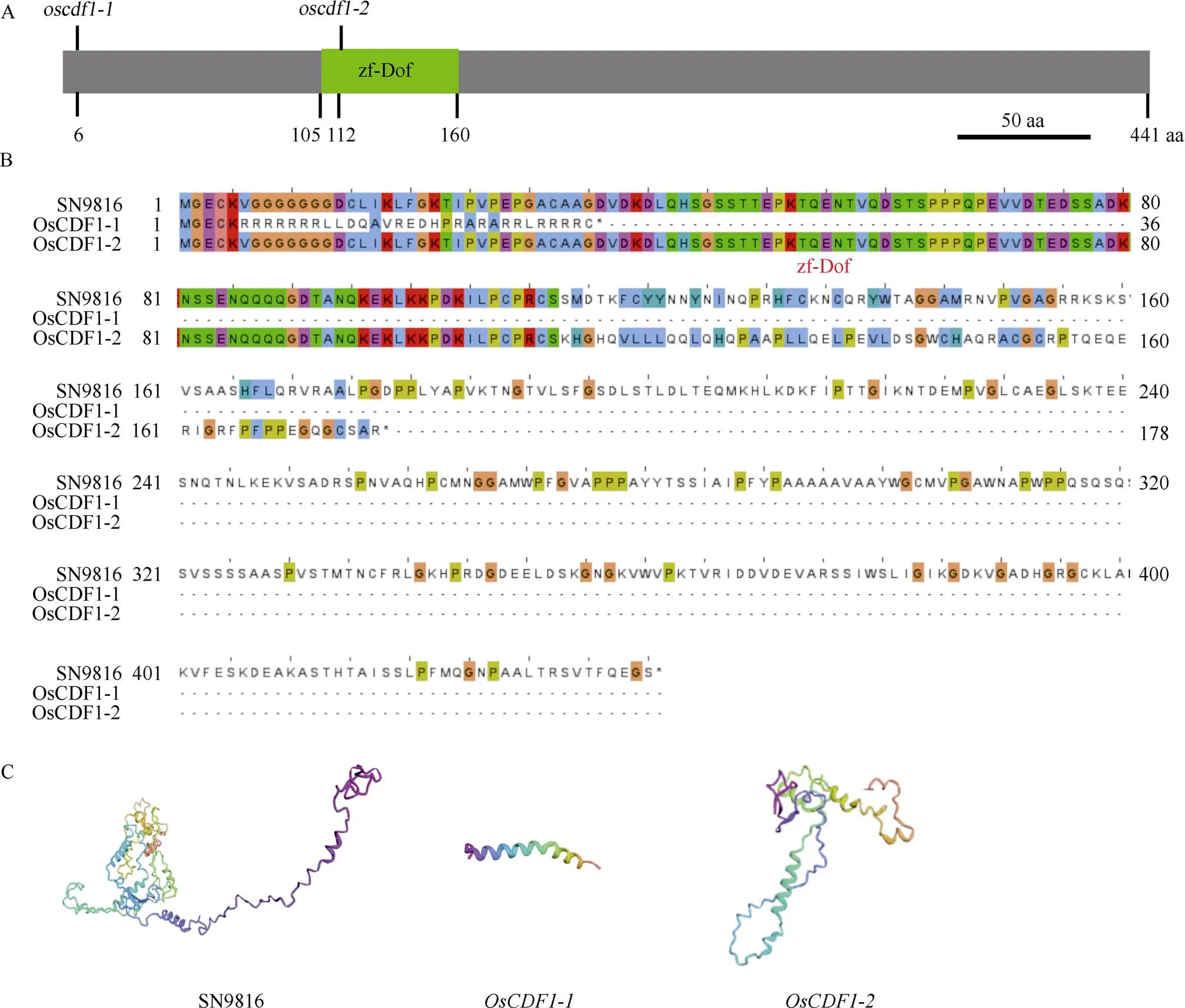
图3 OsCDF1突变蛋白的氨基酸变化及三级结构分析
A: 靶位点氨基酸变化。标尺为50 aa。B: 突变蛋白比对分析。C: 三级结构分析。
A: the amino acid change of the target sites. Bar: 50 aa. B: the blast analysis of the mutant proteins. C: the tertiary structure.
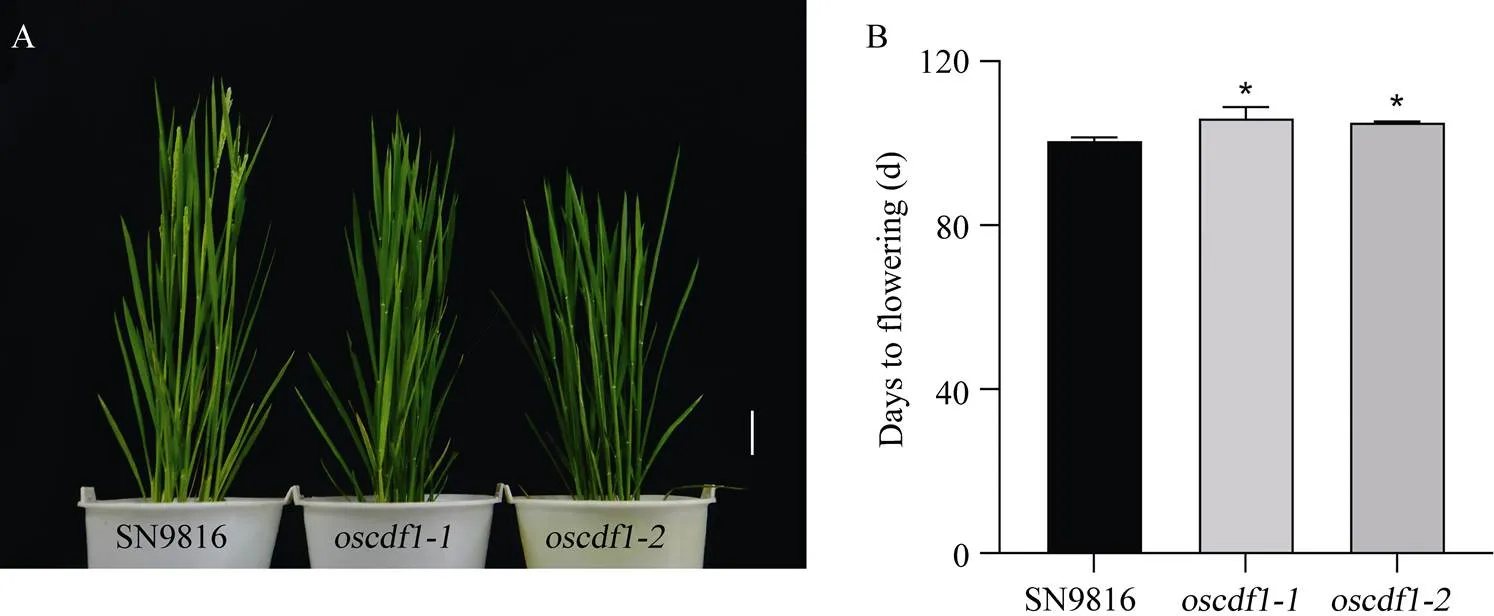
图4 oscdf1突变体开花期表型调查
A:突变体表型。白线为标尺, 10 cm。B:突变体开花期表型分析(≥ 15)。*表示< 0.05水平差异显著。
A: phenotype ofmutants. Bar: 10 cm in white line. B: flowering time ofmutants (≥ 15). *:< 0.05.
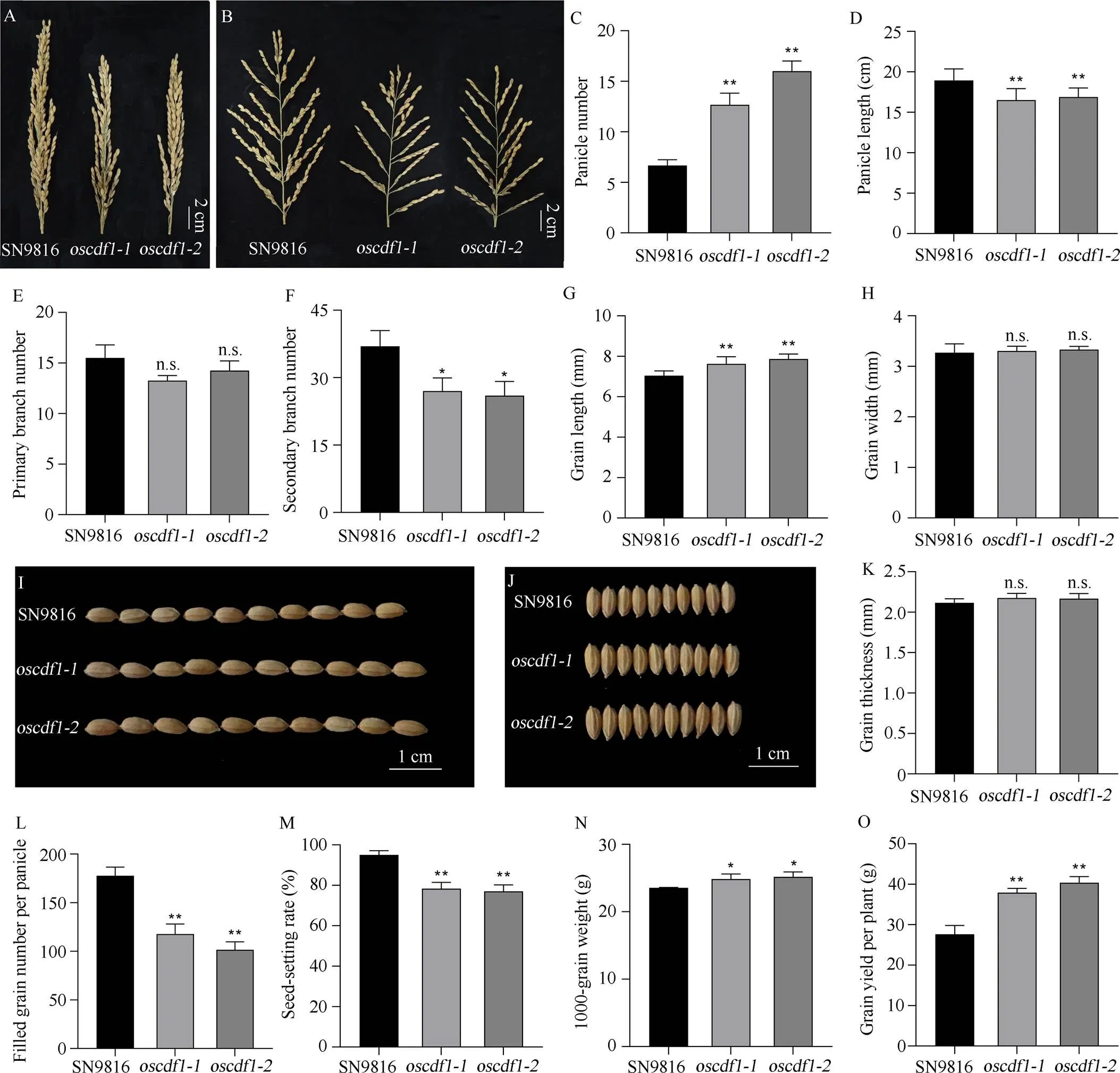
图5 水稻oscdf1突变体和野生型的农艺性状比较
A: 野生型(WT)和突变体穗部表型; B:突变体抽穗期表型分析; C: 单株穗数; D: 穗长; E: 一次枝梗数; F: 二次枝梗数; G: 粒长; H: 粒宽; I: 粒长表型; J: 粒宽表型; K: 粒厚; L: 每穗粒数; M: 结实率; N: 千粒重; O: 单株产量。C~F和I~N的数据为平均值±标准差(≥ 15)。*表示< 0.05水平差异显著; **表示< 0.01水平差异显著; n.s.表示> 0.05水平差异显著。
A: the morphology of panicle between WT andmutant; B: the morphology of branch between WT andmutant; C: panicle number; D: panicle length; E: primary branch number; F: secondary branch number; G: grain length. H: grain width; I: grain length; J: grain width; K: grain thickness; L: the filled grain number per panicle; M: seed-setting rate; N: 1000-grain weight; O: yield weight per plant. Data in C–F and I–J are means ± SDs (≥ 15). *:< 0.05; **:< 0.01; n.s.: no significant difference.
分析野生型SN9816和突变体的单株产量发现,突变体的单株产量极显著高于野生型SN9816 (图5-O)。综上所述,突变体的单株穗数、粒长和千粒重的升高是导致水稻产量升高的主要原因。
2.6 OsCDF1表达模式分析
对在7d SN9816幼苗和根、孕穗期SN9816的剑叶、倒二叶、倒三叶、幼穗、茎、叶鞘以及三叶期的生长点中的表达量检测发现,在SN9816水稻不同组织中的表达量存在较大差异, 其中在叶片、茎、叶鞘和幼苗中的表达量相对较高, 其次为幼穗, 在根系和生长点中的表达水平最低(图6-A)。
检测SN9816自播种之日起第50、60、70、80、90、100和110天完全展开的新叶中的基因表达量, 发现在大部分生育时期内都有较高的表达量(图6-B)。表明可能影响水稻生育期内的生长发育。
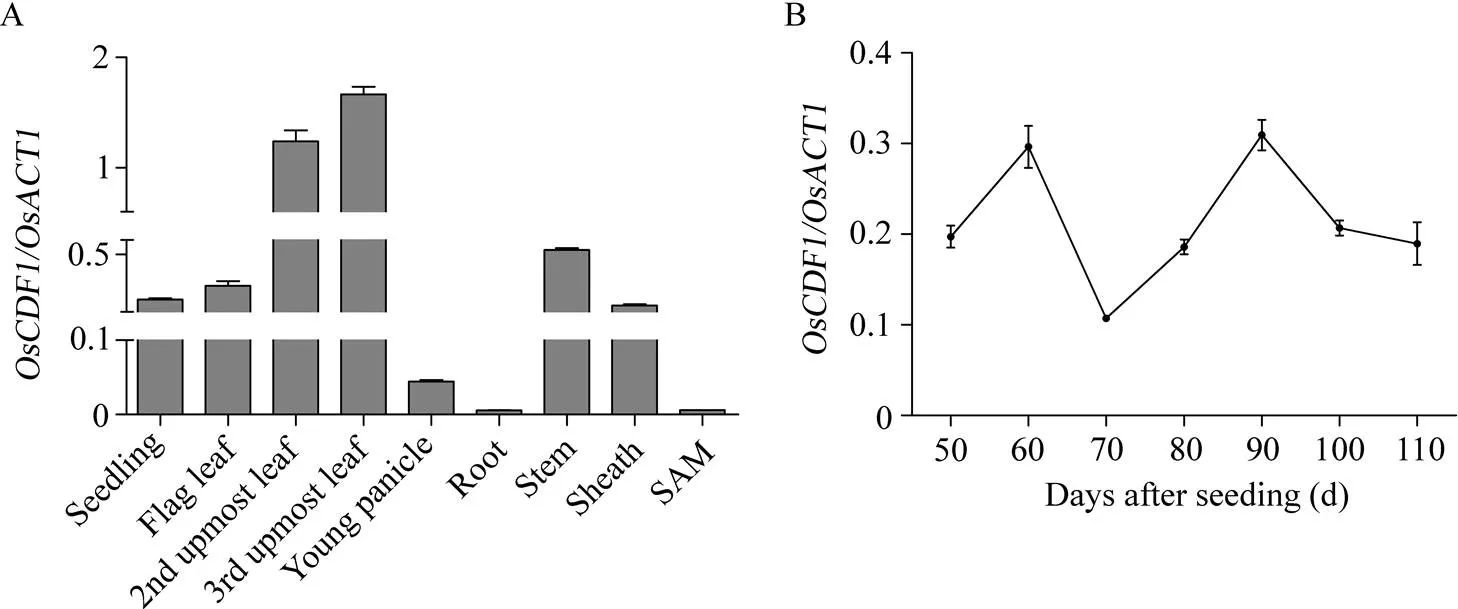
图6 OsCDF1基因表达模式分析
A:组织特异性表达分析。B:在不同发育时期的表达量; A和B的数据为平均值±标准差。
A: tissue-specific analysis of. B: the relative expression level ofdifferent developmental stages. Data in A and B are means ± SDs.
2.7 OsCDF1的单倍型分析
通过RiceVarMap2 (http://ricevarmap.ncpgr.cn/)网站分析的单倍型。在位点中, 一共有22个基因变异, 包括单核苷酸多态性(single nucleotide polymorphisms, SNPs)和插入/缺失 (Insertions/Deletions, InDels)多态性。其中有6个变异分布在第2个外显子上, 其中有4个是同义变异(图7- A, B)。另外, 2个功能变异可能会破坏的C端的功能(图7-B)。
在4726份水稻材料中分析的单倍型, 其中包括籼稻I、籼稻II、籼稻III、籼稻中间型、热带粳稻、温带粳稻、粳稻中间型、Aus稻和中间型。结果显示, 共有7组单倍型。第I组和第II组占总水稻材料份数的比重最多(图7-C), 且所有类型的水稻材料都含有单倍型I。说明第I组和第II组是的主要单倍型。此外, 在粳稻和Aus稻中各含有5种单倍型, 在籼稻和中间型中各有7种单倍型, 表明在不同品种中进化出高度多样性。通过RFGB网站分析海南地区7组单倍型平均抽穗期数据。不同单倍型开花期不同, 第V组单倍型平均开花期最长, 第VI组单倍型平均开花期最短(图7-C)。
3 讨论
水稻是世界三大粮食作物之一, 更是我国的主要粮食作物。因此, 水稻产量对保障我国粮食安全和社会经济稳定起到关键作用。2021年, 我国大部分水稻种植区气象条件良好, 水稻产量有所提高[1]。然而, 由于近年来气候变化、部分地区的自然灾害频发、水稻种植面积减少等原因, 影响水稻总产的稳定性[28]。因此, 除了保障水稻种植面积外, 提高单产才能稳定总产, 这也是近年来育种家持续关注的问题。过去, 育种家利用传统自然变异和物理或化学诱变手段获得突变体, 但过程随机性强、成本高、周期长、效率低, 在一定程度上限制了作物品种改良。近些年, 育种家将新兴的CRISPR/Cas9基因组编辑技术应用到作物品种改良中, 该技术简单、快速、高效。
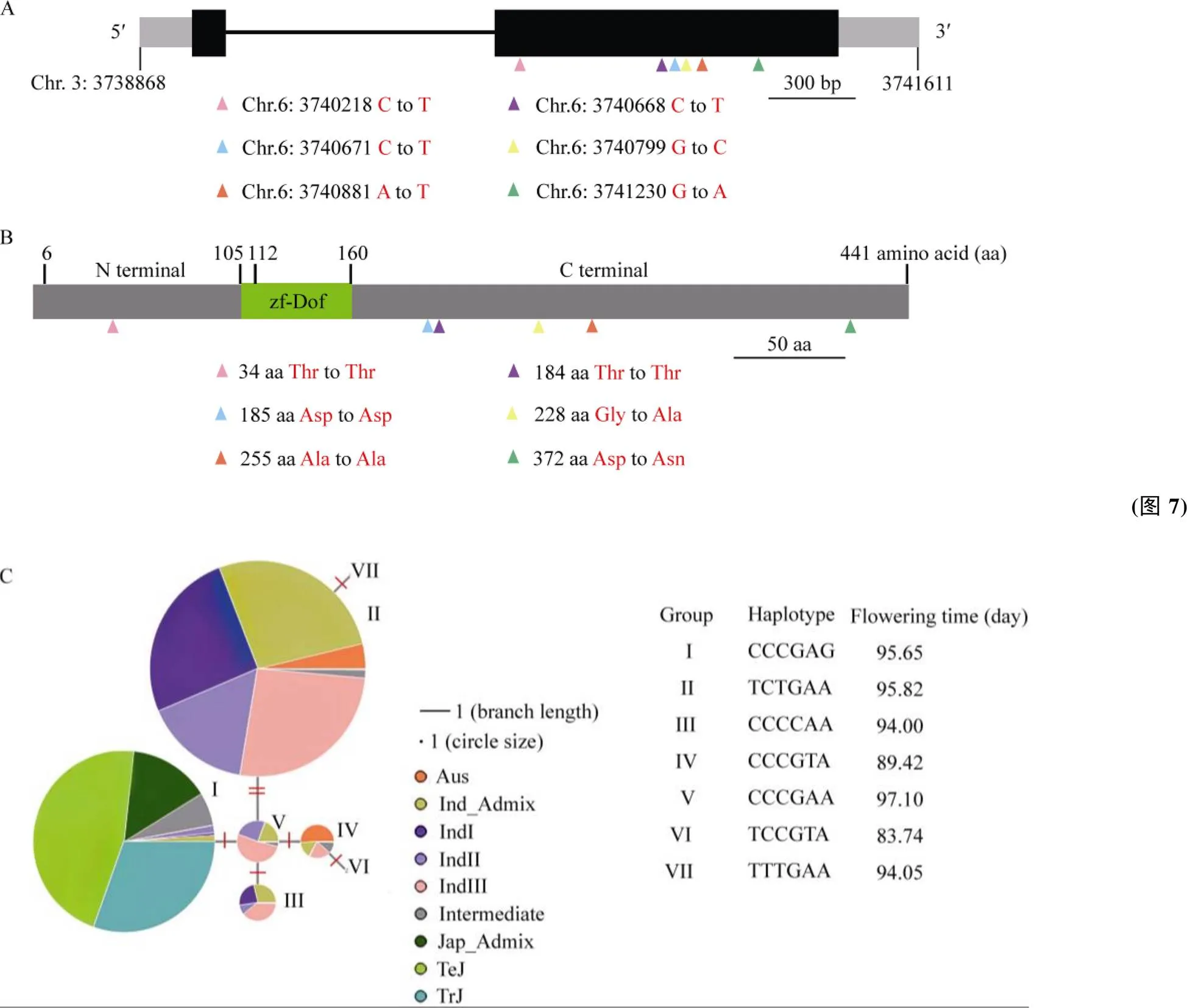
图7 OsCDF1基因变异与单倍型网络分析
A:基因结构及6种变异位置。黑色矩形代表外显子; 灰色矩形代表5'非编码区和3'非编码区; 黑线代表内含子。B:OsCDF1蛋白结构及6种变异位置。不同染色三角形的位置代表6种变异的位置。C: 6种基因变异的单倍型网络, 红线代表2种单倍型之间突变数量。
A:gene structure and location of the six genomic variations. Black rectangles represent the two exons; Grey rectangles represent the five' untranslated region (5' UTR) and 3' UTR; Black lines represent the intron. B: RFT1 protein structure and location of the six genomic variations. Triangles in different colors indicate the locations of the genetic variations. C: the haplotype network of the six genomic variations. The red lines represent the number of mutations between two haplotypes.
CRISPR/Cas9基因组编辑技术是作物遗传品种改良的一种强大的工具。该技术可以将负调控作物某些性状的基因敲除或者弱化其表达, 以达到目标性状。比如, 敲除大麦()中3个的同源基因, 能够提高大麦对白粉病的抗性[29]。在水稻中敲除转录因子可以增强水稻对稻瘟病的抗性[30]。此外, CRISPR/Cas9基因组编辑技术还在玉米()[31]、大豆(L.)[32]、花生()[33]以及小麦()[34]中有所应用。本研究利用CRISPR/Cas9基因组编辑技术成功创制出突变体。利用2种不同编辑位点获得2种不同编辑类型: 一种是在距起始密码子ATG第16 bp处缺失5 bp碱基, 在106 bp出现终止密码子TGA; 另一种是在距起始密码子ATG第338 bp处插入单碱基A, 在532 bp处出现终止密码子TGA (图4-A)。2种突变类型编码氨基酸数均有所变短(图4-B), 2种突变蛋白的三级结构也变得更加简单, 螺旋结构明显减少(图4-C)。以上表明在核酸及蛋白水平都发生了突变, 表明基因功能可能受到影响。
真核生物基因组上有很多重复的同源序列, 靶位点序列有较高的GC含量, 这些都会导致CRISPR/Cas9基因组编辑技术的脱靶效应[35]。因此, 本研究在设计靶位点时利用CRISPR/Cas9专用网站, 并在水稻基因组中BLAST检验靶位点的特异性。虽然CRISPR/Cas9系统不断被优化, 但其脱靶效应还是会存在。正如本研究中2个靶位点共获得32株转基因当代阳性植株, 结果确定被编辑的植株只有18株。对于基因组编辑技术在水稻基因功能研究中的应用, 如果发生脱靶突变, 可能需要查明脱靶突变是否会导致表型变异, 干扰目标突变对表型的分析。可以通过与亲本杂交后分离的后代来确定目标突变与非目标突变。如果脱靶突变导致性状变异, 这些突变实质上等同于自然和诱导突变, 并可以在新的品系中保留。因此, 在育种计划中, 没有必要排除所有脱靶突变。
植物基因表达与转录因子密不可分, Dof蛋白是植物特有的一类转录因子[36]。虽然Dof结构域蛋白在植物生长发育过程中发挥着重要的转录激活或抑制的作用, 但其理论机制仍有待进一步研究[37]。Shigyo提出, Dof结构域蛋白可能与光调控机制有关,CDF1正是含有这种结构域的蛋白之一[38]。在拟南芥中,-都被报道与光周期开花途径有关[14]。CDF1是根据日长特异性调控光周期开花的一个关键机制。之前的报道中证明, 在拟南芥开花时期的上午CDF1的non-EAR结构域能够与TPL结合形成CDF-TPL转录复合体, TPL的N端结合域很大程度上削弱了CDF1蛋白对其靶基因的抑制能力, 即抑制拟南芥开花期相关基因和的表达, 导致拟南芥延迟开花, 这种机制在傍晚则不存在[17]。在之前的报道中以水稻的超表达为材料证明能够促进开花, 并对光周期有响应[22]。本研究则利用基因编辑突变体为研究对象, 为在光周期通路中调控水稻开花期进一步提供理论证据。
尽管一些开花期相关基因被发掘, 但研究用的遗传背景材料各不相同, 很难用同一标准对开花期机制进行合理评价。很难将达到最佳产量的开花期基因集于一体, 这对育种家们来说也是一项挑战。因此, 研究不同种质水稻开花期和相关基因的功能具有重要意义。本研究分析了的基因变异和单倍型, 为水稻开花期研究提供了基因资源, 有利于后续鉴定优异的高产材料。基因编辑与基因的优异单倍型相结合, 这已然成为水稻高产品种分子选育的重要策略。
本研究利用CRISPR/Cas9基因编辑技术成功创制出2种突变体, 筛选出去除转基因标记的水稻SN9816突变体和, 对水稻基因的基因功能进行了初步探索。的突变导致SN9816的开花期延迟和单株产量提高。纯合突变体的获得, 不仅为深入研究基因功能提供了试验材料, 也为进一步获得开花期相关基因的双重或多重突变体奠定了基础, 同时, 为北方粳稻品种的生育期、产量和地区适应性的改良奠定了基础。
4 结论
本研究利用CRISPR/Cas9基因编辑技术, 定点敲除基因, 成功获得2种去除转基因标记的水稻SN9816突变体。在自然长日照条件下, 突变体和表现晚花, 单株产量提高,表明能够促进水稻开花。在水稻不同组织器官以及不同生育时期均有表达, 并影响水稻产量。在不同品种中进化出高度多样性。本研究结果对深入研究的功能及育种应用具有理论和实践意义。
[1] 徐春春, 纪龙, 陈中督, 方福平. 2021年我国水稻产业形势分析及2022年展望. 中国稻米, 2022, 28(2): 16–19. Xu C C, Ji L, Chen Z D, Fang F P. Situation analysis of China’s rice industry in 2021 and its outlook in 2022., 2022, 28(2):16–19 (in Chinese).
[2] 万建民. 中国水稻分子育种现状与展望. 中国农业科技导报, 2007, (2): 1–9. Wan J M. Current situation and prospect of rice molecular breeding in China., 2007, (2): 1–9 (in Chinese with English abstract).
[3] 张海淼, 李洋, 刘海峰, 孔令广, 丁新华. 水稻重要农艺性状调控基因及其育种利用研究进展. 生物技术通报, 2020, 36(12): 155–169. Zhang H M, Li Y, Liu H F, Kong L G, Ding X H. Research progress on regulatory genes of important agronomic traits and breeding utilization in rice., 2020, 36(12): 155–169 (in Chinese with English abstract).
[4] 郭韬, 余泓, 邱杰, 李家洋, 韩斌, 林鸿宣. 中国水稻遗传学研究进展与分子设计育种. 中国科学: 生命科学, 2019, 49: 1185–1212. Guo T, Yu H, Qiu J, Li J Y, Han B, Lin H X. Advances in rice genetics and breeding by molecular design in China., 2019, 49: 1185–1212 (in Chinese with English abstract).
[5] Izawa T, Oikawa T, Sugiyama N, Tanisaka T, Yano M, Shimamoto K. Phytochrome mediates the external light signal to repress FT orthologs in photoperiodic flowering of rice., 2002, 16: 2006–2020.
[6] Mouradov A, Cremer F, Coupland G. Control of flowering time: interacting pathways as a basis for diversity., 2002, 14: 111–130.
[7] Yanovsky M J, Kay S A. Molecular basis of seasonal time measurement in., 2002, 419: 308–312.
[8] Ryosuke H, Shuji Y, Shojiro T, Masahiro Y, Ko S. Adaptation of photoperiodic control pathways produces short-day flowering in rice., 2003, 422: 719–722
[9] Kardailsky I, Shukla V K, Ahn J H, Dagenais N, Christensen S K, Nguyen J T, Chory J, Harrison M J, Weigel D. Activation tagging of the floral inducer FT., 1999, 286: 1962–1965.
[10] Kobayashi Y, Kaya H, Goto K, Iwabuchi M, Araki T. A pair of related genes with antagonistic roles in mediating flowering signals., 1999, 286: 1960–1962.
[11] Samach A, Onouchi H, Gold S E, Ditta G S, Schwarz-Sommer Z, Yanofsky M F, Coupland G. Distinct roles of CONSTANS target genes in reproductive development of., 2000, 288: 1613–1616.
[12] Robson F, Costa M M, Hepworth S R, Vizir I, Piñeiro M, Reeves P H, Putterill J, Coupland G. Functional importance of conserved domains in the flowering-time gene CONSTANS demonstrated by analysis of mutant alleles and transgenic plants., 2001, 28: 619–631.
[13] Yasushi K, Detlef W. Move on up, it’s time for change: mobile signals controlling photoperiod-dependent flowering., 2007, 21: 2371–2384.
[14] Imaizumi T, Schultz T F, Harmon F G, Ho L A, Kay S A. FKF1 F-Box protein mediates cyclic degradation of a repressor of CONSTANS in, 2005, 309: 293–297.
[15] Sawa M, Nusinow D A, Kay S A, Imaizumi T. FKF1 and GIGANTEA complex formation is required for day-length measurement in., 2007, 318: 261–265.
[16] Fornara F, Panigrahi K C S, Gissot L, Sauerbrunn N, Rühl M, Jarillo J A, Coupland G.DOF transcription factors act redundantly to reduce CONSTANS expression and are essential for a photoperiodic flowering response., 2009, 17: 75–86.
[17] Goralogia G S, Liu T K, Zhao L, Panipinto P M, Groover E D, Bains Y S, Imaizumi T. CYCLING DOF FACTOR 1 represses transcription through the TOPLESS co-repressor to control photoperiodic flowering in., 2017, 92: 244–262.
[18] Imaizumi T, Schultz T F, Harmon F G, Ho L A, Kay S A. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in., 2005, 309: 293–297.
[19] Yano M, Katayose Y, Ashikari M, Yamanouchi U, Monna L, Fuse T, Baba T, Yamamoto K, Umehara Y, Nagamura Y, Sasaki T., a major photoperiod sensitivity quantitative trait locus in rice, is closely related to theflowering time gene CONSTANS., 2000, 12: 2473–2484.
[20] Izawa T, Takahashi Y, Yano M. Comparative biology comes into bloom: genomic and genetic comparison of flowering pathways in rice and., 2003, 6: 113–120.
[21] Kazuyuki D, Takeshi I, Takuichi F, Utako Y, Takahiko K, Zenpei S, Masahiro Y, Atsushi Y. Ehd1, a B-type response regulator in rice, confers short-day promotion of flowering and controls FT-like gene expression independently of Hd1., 2004, 18: 926–936.
[22] Li D J, Yang C H, Li X B, Gan Q, Zhao X F, Zhu L H. Functional characterization of rice., 2009, 229: 1159–1169.
[23] Wu Q, Li D Y, Li D J, Liu X, Zhao X F, Li X B, Li S G, Zhu L H. Overexpression ofaffects plant architecture in rice (L.)., 2015, 6: 833.
[24] 单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展. 遗传, 2015, 37: 953–973. Dan Q W, Gao C X. Research progress of genome editing and derivative technologies in plants., 2015, 37: 953–973 (in Chinese with English abstract).
[25] Xie K B, Minkenberg B, Yang Y N: Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system., 2015, 112: 3570–3575.
[26] Li D, Xu H, Sun X X, Cui Z B, Zhang Y, Bai Y G, Wang X X, Chen W F. Differential transformation efficiency ofrice varieties developed in northern China., 2015, 15: 162–168.
[27] Zhu M, Hu Y J, Tong A Z, Yan B W, Lyu Y P, Wang S Y, Ma W H, Cui Z B, Wang X X. LAZY1 controls tiller angle and shoot gravitropism by regulating the expression of auxin transporters and signaling factors in rice., 2021, 61: 2111–2125.
[28] 孔冬艳, 陈会广. 近40年来中国农作物与耕地受灾时空特征及影响因素分析. 长江流域资源与环境, 2020, 29: 1236–1246. Kong D Y, Chen H G. Spatial-temporal characteristics and influencing factors of agricultural crop and cultivated land disaster in China in recent 40 years., 2020, 29: 1236–1246 (in Chinese).
[29] Wang Y P, Cheng X, Shan Q W, Zhang Y, Liu J X, Gao C X, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew., 2014, 32: 947–951.
[30] Wang F J, Wang C L, Liu P Q, Lei C L, Hao W, Gao Y, Liu Y G, Zhao K J. Enhanced rice blast resistance by CRISPR/Cas9- targeted mutagenesis of the ERF transcription factor gene., 2016, 11: e0154027.
[31] Wang Y X, Liu X Q, Zheng X X, Wang W X, Yin X Q, Liu H F, Ma C L, Niu X M, Zhu J K, Wang F. Creation of aromatic maize by CRISPR/Cas., 2021, 63: 1664–1670.
[32] 侯智红, 吴艳, 程群, 董利东, 芦思佳, 南海洋, 甘卓然, 刘宝辉. 利用CRISPR/Cas9技术创制大豆高油酸突变系, 作物学报, 2019, 45: 839–847. Hou Z H, Wu Y, Cheng Q, Dong L D, Lu S J, Nan H Y, Gan Z R, Liu B H. Creation of high oleic acid soybean mutation plants by CRISPR/Cas9., 2019, 45: 839–847 (in Chinese with English abstract).
[33] 张旺, 冼俊霖, 孙超, 王春明, 石丽, 于为常. CRISPR/Cas9编辑花生基因研究. 作物学报, 2021, 47: 1481–1490. Zhang W, Xian J L, Sun C, Wang C M, Shi L, Yu W C. Preliminary study of genome editing of peanutgenes by CRISPR/ Cas9., 2021, 47: 1481–1490 (in Chinese with English abstract).
[34] Zhang J H, Zhang H T, Li S Y, Li J Y, Yan L, Xia L Q. Increasing yield potential through manipulating of an ARE1 ortholog related to nitrogen use efficiency in wheat by CRISPR/Cas9., 2021, 63: 1649–1663.
[35] Fu Y F, Foden J A, Khayter C, Maeder M L, Reyon D, Joung J K, Sander J D. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells., 2013, 31: 822–826.
[36] Riechmann J L, Heard J, Martin G, Reuber L, Jiang C, Keddie J, Adam L, Pineda O, Ratcliffe O J, Samaha R R, Creelman R, Pilgrim M, Broun P, Zhang J Z, Ghandehari D, Sherman B K, Yu G.transcription factors: genome-wide comparative analysis among., 2000, 290: 2105–2110.
[37] Shuichi Y. Dof domain proteins: plant-specific transcription factors associated with diverse phenomena unique to plants., 2004, 45: 386–391.
[38] Shigyo M, Tabei N, Yoneyama T, Yanagisawa S. Evolutionary processes during the formation of the plant-specific Dof transcription factor family., 2007, 48: 179–185.
Mutation effects ofgene and its genomic variations in rice
HU Yan-Juan, XUE Dan, GENG Di, ZHU Mo, WANG Tian-Qiong, and WANG Xiao-Xue*
Rice Research Institute, Shenyang Agricultural University, Shenyang 110866, Liaoning, China
Flowering time (heading date) affects yield, quality, and regional adaptability of rice. The Cycling DOF Factor 1 (CDF1) protein is a transcriptional repressor of() and negatively regulates flowering time in. However, the biological functions of OsCDF1 in rice is not quite clear. To explore the biological functions of OsCDF1 and its effects on flowering time control in rice, we constructed two binary vectors carrying guide RNAs targetinggene via CRISPR/Cas9 system. The resultant plasmids were transferred into SN9816 which was the variety widely cultivated in northern China by using an-mediated transformation, and the mutations ofwas firstly generated in SN9816. The flowering time and yield related traits of SN9816 andmutants were investigated in the paddy field. The main results were as follows: Two homozygouslines were identified, including a five bp deletion at 16th bp of the first exon and a single base pair A insertion at 338th bp of the second exon. Sequence alignment analysis revealed that the two types of mutations resulted in frame-shift and premature translation termination. Mutations ofdelayed flowering time, but increased yield under natural long day conditions in rice. Analysis ofgenetic variations and haplotype networks revealed that the rice accessions had evolved high genomic diversity inlocus. The knockout mutants ofcreated by CRISPR/Cas9 provided the theoretical basis to further study the role ofgene in rice and the potential gene and germplasm resources for genetic improvement in rice.
rice; CRISPR/Cas9; OsCDF1; flowering time; yield related traits; haplotype
2022-10-28;
2023-02-21;
2023-03-16.
10.3724/SP.J.1006.2023.22062
通信作者(Corresponding author):王晓雪, E-mail: wangxx@syau.edu.cn
E-mail: 2020200056@stu.syau.edu.cn
本研究由国家重点研发计划项目(2017YFD0300107)和国家自然科学基金项目(32070642)资助。
This study was supported by the National Key Research and Development Program of China (2017YFD0300107) and the National Natural Science Foundation of China (32070642).
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20230315.1424.002.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).
