比较转录组分析花生种子休眠调控网络
2023-07-20王菲菲张胜忠胡晓辉CHUYe崔凤高赵立波张天雨郭进涛于豪谅苗华荣
王菲菲 张胜忠 胡晓辉 CHU Ye 崔凤高 钟 文 赵立波 张天雨 郭进涛 于豪谅 苗华荣,* 陈 静,*
比较转录组分析花生种子休眠调控网络
王菲菲1张胜忠1胡晓辉1CHU Ye2崔凤高1钟 文3赵立波4张天雨3郭进涛5于豪谅6苗华荣1,*陈 静1,*
1山东省花生研究所, 中国山东青岛 266100;2佐治亚大学蒂芙顿校区园艺系, 美国佐治亚州蒂芙顿 31793;3山东省种子管理总站, 中国山东济南 250100;4青岛市农业技术推广中心, 中国山东青岛 266071;5新郑市和庄农业服务中心, 中国河南新郑 451150;6烟台枫林食品股份有限公司, 中国山东烟台 264108
种子休眠性是花生重要且复杂的农艺性状, 对花生(L.)的产量和品质影响巨大。为深入揭示花生种子休眠维持和解除的分子调控网络, 本研究以强休眠品种花育52号(HY52)和弱休眠突变株系M23、M67为试材, 种子吸胀处理(0 h、12 h、24 h)后测定其激素ABA和GA含量并进行转录组测序。吸胀12 h时M23和M67中GA含量显著高于HY52, ABA含量和ABA/GA比值则低于HY52。测序共得到31,373个差异表达基因(DEGs), 其中ABA和GA生物合成和信号转导相关的基因在种子休眠维持和解除过程中发生显著变化, 挖掘到50个ABA相关基因、8个GA相关基因、49个乙烯相关基因和13个生长素相关基因。此外, 还鉴定到许多参与碳水化合物和脂质代谢、氨基酸代谢途径相关的DEG, 挖掘到糖代谢相关基因5个、脂质代谢相关基因4个; 昼夜节律调控也可能参与花生种子休眠解除。这表明, 花生种子休眠维持和解除的调控是一个复杂网络, 植物激素平衡调控可能只是其中一个重要部分。
花生; 休眠维持; 休眠解除; 转录组; 植物激素; 氨基酸代谢
种子休眠是指在特定时间范围内、适宜的生长环境下, 具备萌发能力的种子不发芽的现象, 是植物为抵抗和适应不良环境的一种适应性性状[1-2]。它是植物长期进化过程中形成的自我保护机制, 是植物界中普遍存在的特性[3]。种子休眠是极其重要和复杂的农艺性状, 对农业安全生产有重要意义。农作物如小麦、大麦、水稻和花生等在长期驯化、培育过程中, 注重追求高产高效而忽视了保留适度的种子休眠性, 导致种子休眠性减弱甚至丧失, 使得产量和食用品质下降、种用安全隐患(易受黄曲霉侵染, 产生黄曲霉毒素)等一系列问题[4-5]。因此, 揭示种子休眠调控机制并选育具有适度休眠性的农作物品种/系是作物育种家重要的研究方向之一。
目前, 关于种子休眠的生物学机制仍未得到全面解析, 种子会随着储藏时间的增加而逐渐失去休眠性[6-7]。早在50多年前就已报道, 植物激素脱落酸(abscisic acid, ABA)和赤霉素(gibberellin, GA)及其两者间的平衡在调控种子休眠和萌发中发挥主要作用[8-10]。ABA诱导和维持种子休眠而GA则促进休眠解除和种子萌发[7], 在种子休眠和萌发过程中, 一系列ABA和GA合成、代谢及信号转导相关的基因表达发生变化[7]。除此之外, 其他植物激素如乙烯(ethylene, ETH)、生长素(auxin)、细胞分裂素(cytokinins, CTK)和油菜素内酯(brassinosteroids, BR)也在调控种子休眠和萌发中发挥重要作用[7,11]。种子萌发和休眠受多个因子调控, 包括植物激素和休眠蛋白等, 这些调控信息将在种子吸胀过程中被整合处理, 最终决定是否萌发[10]。
花生是食用油和植物蛋白的重要来源, 是我国重要的油料和经济作物。由于花生地上开花地下结果, 其种子休眠调控更加复杂, 为花生种子休眠研究带来更多挑战。前人研究主要集中在花生休眠品种筛选鉴定、相关QTL定位[4,12-14], 但是花生休眠关键基因的挖掘及克隆相对滞后。转录组测序是高通量筛选差异表达基因的有效方法, 利用转录组分析种子休眠和萌发的研究已有报道[15-18], Xu等[19]对休眠不深的鲁花14号的新收获种子(freshly harvested seed, FS)、后熟种子(after-ripening seed, DS)和新萌发种子(newly germinatedseed, GS)进行比较转录组测序分析, 在GS vs DS的比较组中, 许多上调基因与植物激素合成和信号转导相关, 包括生长素信号、油菜素内酯合成和信号转导及GA、ABA信号转导的大部分元件[19]。通过测定下胚轴和胚根、胚芽、子叶中的激素含量, 发现在种子吸胀过程中, ABA含量逐步下降, ABA/GA3比值在吸胀4 h达到最大值, 随后下降。其他物种也有相关报道, 低温处理能打破豆梨(Dence)休眠, 对低温处理的种子进行转录组测序分析发现, 多个差异表达基因(differentially expressed genes, DEGs)与植物激素(ABA, GA, CTK, ETH, BR, JA)的代谢和信号转导相关。在种子休眠解除过程中, 与淀粉和蔗糖代谢、脂质代谢相关的基因以及编码转录因子的基因也有差异表达[20]。
尽管种子休眠维持和解除的基因调控网络及在拟南芥和谷类作物中取得了巨大突破[20-21], 但有关双子叶植物花生种子休眠和萌发的机制尚不清楚。为揭示花生种子休眠维持和解除机制, 本研究选用强休眠品种花育52号(Huayu 52, HY52)和由HY52诱变而来的2个弱休眠材料M23和M67为试材, 吸胀处理(0 h、12 h、24 h)后进行转录组测序。同时对HY52进行全长转录组测序, 以获得更好的基因功能注释。同时测定了种子中ABA和GA含量, 分析种子吸胀过程中植物激素变化, 旨在揭示花生休眠维持和解除过程中关键基因的完整表达谱, 为花生适度休眠育种提供新的思路。
1 材料与方法
1.1 植物材料及生长条件
试验材料系花育52号(Huayu 52, HY52)及经EMS诱变而来的M4突变体M23和M67[5], 其中花育52号为山东省花生研究所育成的花生品种, 具有强休眠性, M23和M67则为2个具有弱休眠性的突变材料。试验材料种植在山东省莱西市实验农场(36.86°N, 120.53°E), 种植方式为起垄双行单粒播种,行距40.0 cm, 株距16.7 cm, 覆膜。5月上旬播种, 种子成熟后收获晾晒10 d后进行种子休眠性检测, 剩余种子置于纸袋中室温保存。参照胡晓辉等[5]的方法, 以种子萌发率来评价种子休眠性强弱。
1.2 cDNA文库构建、测序和数据分析
本研究选择对种子进行吸胀处理, 分析该时期种子激素含量及转录组信息变化。基于胡晓辉等[5]前期研究, 选择吸胀0 h、12 h和24 h 3个处理进行深入分析。每个供试材料选取饱满无损的种子10粒, 置于培养皿中, 吸胀0 h、12 h、24 h时分别取样用于RNA提取并构建转录组文库, 转录组测序设3个生物学重复。以TRIzol法进行RNA提取并进行DNase I处理, 质检合格后送杭州联川生物技术股份有限公司建库测序。二代转录组测序平台为Illumina HiSeq4000, HY52的全长转录组测序平台为PacBio Sequel。
1.3 差异表达基因和富集分析
测序产生的原始数据(rawdata)经cutadapt过滤得到有效数据(cleandata)。所有有效数据经Hisat v2.0比对到栽培花生参考基因组上(https://peanutbase. org/peanut_genome)[22]。计算每个转录本的FPKM(每千碱基转录序列的预期片段数/百万碱基对测序)值来表示归一化的基因表达量。根据公式(表达倍数=FPKMA/FPKMB)计算基因表达量的倍数变化, 将表达差异倍数log2|Fold Change|>1且FDR<0.05的基因定义为差异表达基因(DEGs)。差异表达基因基于GO (Gene Ontology)数据库和KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库进行功能注释并按照生物过程(biological process, BP)、细胞组分(cellular component, CC)和分子功能(molecular function, MF)对其富集分析, 找出差异基因显著富集的GO term和KEGG term。基于短时间序列表达式挖掘(short time-series expression minor, STME)聚类方法将差异基因聚类到不同的表达谱中[23]。
1.4 数据处理及作图
用Graphpad Prism 8软件对测定数据进行显著性检验和绘图。使用联川生物云平台在线工具(https://www.omicstudio.cn/tool)进行差异基因GO功能分析和KEGG分析。
2 结果与分析
2.1 HY52与M23、M67的休眠表型差异
HY52萌发率为35%, 而M23和M67萌发率分别为78.33%和83.33%, 三者萌发率间有显著差异, HY52为强休眠品种, M23和M67为弱休眠材料(图1-A)。3种材料中GA含量均随吸胀时间增加而升高(图1-B), 而ABA含量则呈下降趋势(图1-C)。吸胀0 h时, GA和ABA含量在HY52、M23、M67之间并无显著差异, 而吸胀12 h和24 h时M23和M67中GA含量显著高于HY52 (图1-B), M67中ABA含量和ABA/GA比值则显著低于HY52 (图1-C, D), 吸胀12 h时M23中ABA含量和ABA/GA比值也低于HY52 (图1-C, D)。这与HY52、M23、M67的休眠表型一致。种子吸胀过程中, HY52、M23、M67中ABA/GA比值分别从1335、986、676降至676、651、343 (图1-D)。

图1 HY52、M23和M67的休眠性差异
A: HY52、M23和M67的萌发率, B~D: 吸胀12 h和24 h的GA含量、ABA含量和ABA/GA。GA: 赤霉素; ABA: 脱落酸; HY52: 花育52号。星号表示通过单因素方差分析与HY52相比有显著性(*<0.05, **<0.01, ***<0.001)。
A: germination rate of dormant peanut variety HY52, weak dormant accessions M23 and M67, B–D: GA content, ABA content and ABA/GA of HY52, M23, and M67 after imbibition at 0, 12, and 24 h, respectively. GA: gibberellin; ABA: abscisic acid; HY52: Huayu 52. Asterisks indicate significant differences compared to HY52 by one-way ANOVA (*<0.05, **<0.01, and ***<0.001).
2.2 转录组测序整体分析
本研究供试的27个样本, 共获得原始序列数目大约为39,391,916~63,130,436。经过滤, 有效序列数目为39,016,270~62,578,018, 有效碱基为5.98~9.39 Gb。平均GC含量为46.38%, 有效序列的Q30超过94.81%。有效序列的比率为88.92%~99.20%, 比对到基因组上的平均比率超过92.51% (表1)。另外, 为了对差异表达基因进行更好的注释, 对休眠品种HY52进行了全长转录组测序, 在KOG (Kyoto Encyclopedia of Genes and Genomes)、KEGG (Kyoto Encyclopedia of Genes and Genomes)、NR (Non- redundant Protein Database)、Swiss Prot (Non- redundant Protein Database)和GO (Gene Ontology)数据库中都进行了功能注释(表2)。
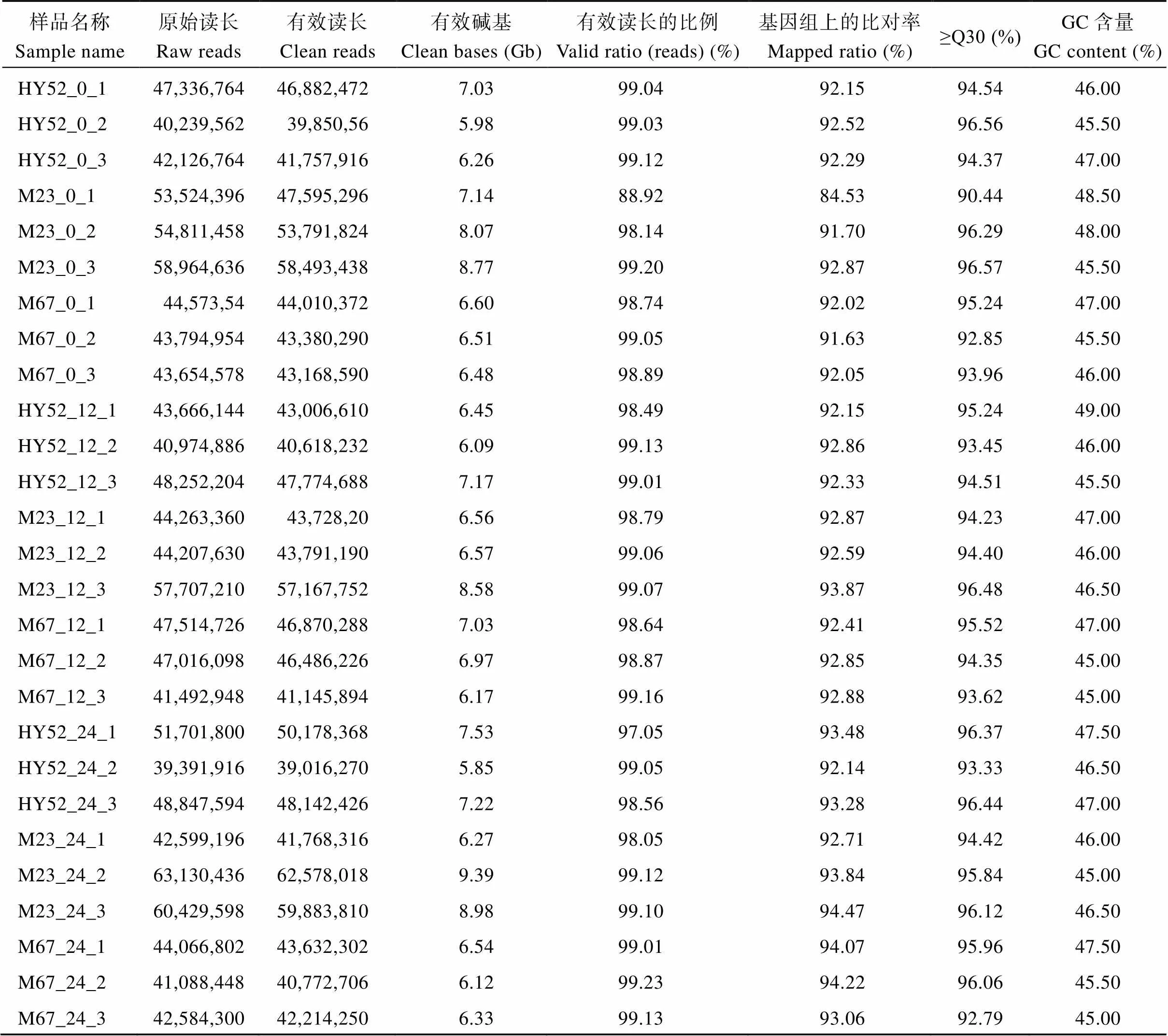
表1 参试样品的转录组数据质量

表2 花育52号全长转录组的功能注释
2.3 转录组差异表达基因及表达谱分析
根据<0.05和|log2FC|≥1的标准, 所有样本中共鉴定到15,746个差异表达基因。HY52吸胀12 h后有1392个上调基因和1702个下调基因, 吸胀24 h后有2001 (上调)/2075 (下调)个差异基因。M23吸胀12 h和24 h的差异表达基因数分别为1766/1522和1384/2498, M67吸胀12h和24 h的差异表达基因数分别为1868/2796和2061/2368 (图2-A)。韦恩图展示了吸胀和对照组中相同和特异的差异表达基因数, HY52和M23的4个比较组中共有的上调基因有40个(图2-C), 下调基因有15个(图2-D); HY52和M67的4个比较组中共有的上调基因有61个(图2-E), 下调基因有51个(图2-F)。3个材料的比较组中, M23 (M23_12 vs M23_0, M23_24 vs M23_0)和M67 (M67_12 vs M67_0, M67_24 vs M67_0)中的特有差异表达基因数量比HY52 (HY52_12 vs HY52_0, HY52_24 vs HY52_0)中多(图2-C~F), 说明M23和M67在吸胀过程中有更多的基因参与休眠解除。另外, 在吸胀12 h时HY52和M23中上调的差异表达基因数是156, 下调的差异表达基因数是116, 吸胀24 h时分别为287 (上调)和532 (下调); HY52和M67在吸胀12 h和24 h的差异表达基因数分别为272 (上调)/335 (下调)和542 (上调)/459 (下调)。
为深入探究在种子休眠解除过程中的基因表达谱变化, 利用STEM算法[23]将HY52中52,747个差异表达基因、M23中的54,233个差异表达基因和M67中的53,060个差异表达基因聚类到16个表达谱中。根据显著性分析将HY52中26,816个差异表达基因聚类到6个表达谱中: 5个上调的表达谱(profiles 11, 15, 13, 12, 8)和一个双相表达谱(profile 14) (图3-A)。与之类似, M23中25,999个差异表达基因聚类到7个表达谱中, M67中24,681个差异表达基因聚类到6个表达谱中(图3-B, C)。结果表明, 3个材料中有5个相同的上调表达谱, 包括11、15、13、12和14, 而2个下调表达谱4和0只出现在M23中, 表达谱8则在HY52和M67中同时存在。M23中上调基因数目(profiles 11和15)大于HY52中上调基因数量, M67中上调基因数目(profiles 15, 13, 12和8)大于HY52和M23在这几个表达谱中的基因数量。M23和M67都是弱休眠材料却有不同表达谱, 可能是由突变位点的差异导致。

图2 不同处理差异表达基因比较及韦恩图
HY52: 花育52号; DEGs: 差异表达基因。HY52: Huayu 52; DEGs: differentially expressed genes.
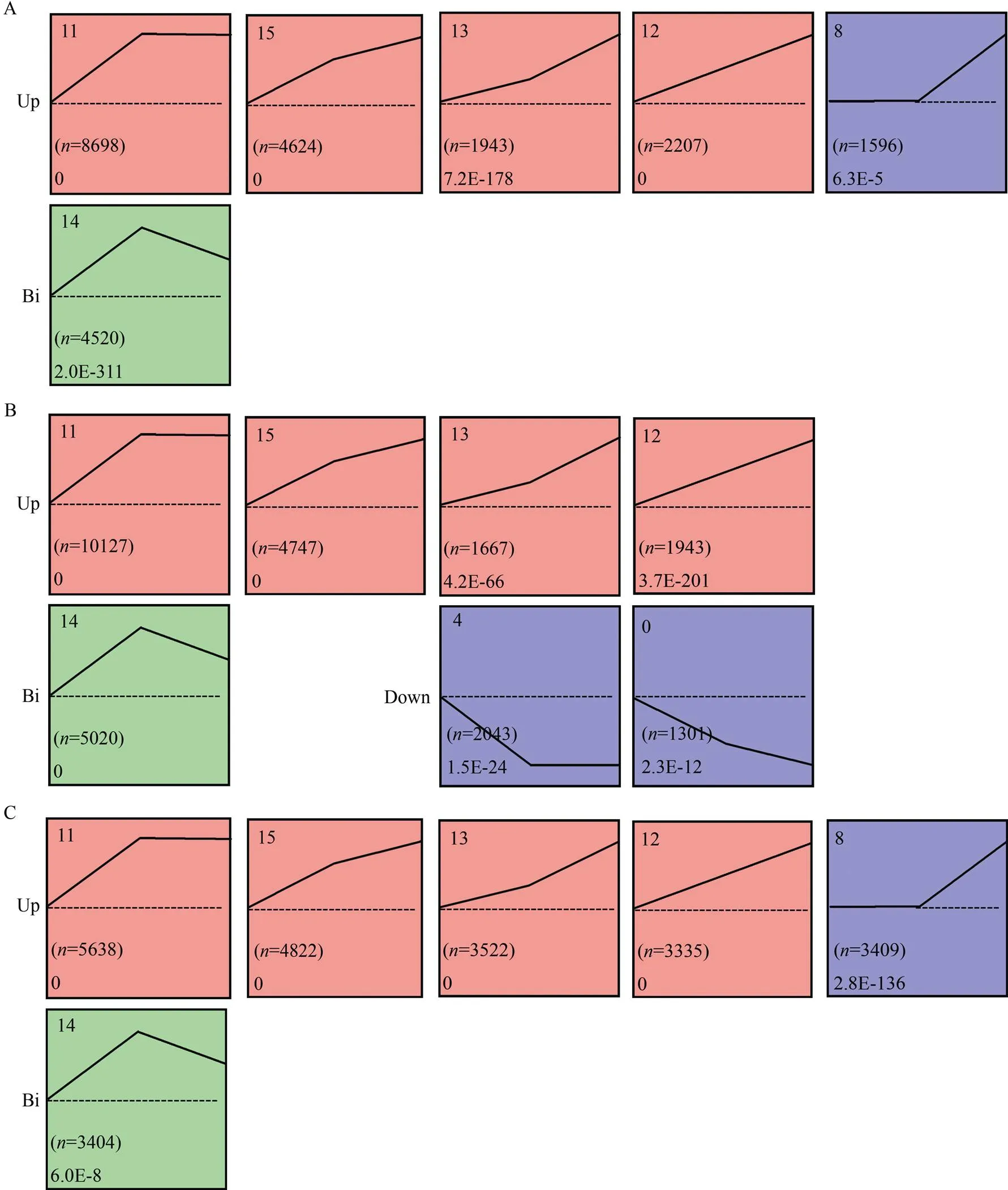
图3 HY52、M23和M67的差异基因表达谱
HY52 (A)和M67 (C)的表达谱聚成2个组, 即上调和双向表达谱, M23 (B)则聚成3个组, 即上调、下调和双向表达谱。表达谱编号标记在左上角, 每个表达谱对应的值标记在左下角, 括号中为每个表达谱中差异基因的数量。HY52: 花育52号。
Profiles of HY52 (A) and M67 (C) were clustered into two groups, namely Up (upregulated) and Bi (biphasic expression pattern), however, M23 (B) were clustered into three groups, namely Up (upregulated), Down (downregulated), and Bi (biphasic expression pattern). Profile numbers are indicated in the top left-hand corner, and the corresponding-values for each profile are shown in the bottom left-hand corner. The number of DEGs with each profile is shown in the brackets. HY52: Huayu 52.
2.4 花生休眠维持相关差异表达基因的富集分析
为揭示种子休眠维持的机制, 对6个比较组(HY52_0 vs M23_0, HY52_0 vs M67_0, HY52_12 vsM23_12, HY52_24 vs M23_24, HY52_12 vs M67_12, HY52_24 vs M67_24)进行了GO富集分析。与预期一致, 结果表明激素和胁迫相关的GO条目在HY52 vs M23和HY52 vs M67的比较组中显著富集, 如脱落酸刺激的细胞反应(cellular response to abscisic acid stimulus)、赤霉素反应(response to gibberellin)、生长素反应(response to auxin)、胁迫反应(response to stress)、盐胁迫反应(response to salt stress)、脱落酸激活的信号通路(abscisic acid- activated signaling pathway)、茉莉酸介导的信号通路(jasmonic acid mediated signaling pathway)、乙烯结合(ethylene binding)、乙烯激活的信号通路(ethylene- activated signaling pathway) (图4)。除此之外, 还有很多防御相关的条目, 如防御反应(defense response)、活性氧代谢的正调控(positive regulation of reactive oxygen species metabolic process)、种子耐干燥性的获得(acquisition of desiccation tolerance in seed)。氨基酸代谢相关的条目包括硫胺素生物合成(thiamine biosynthetic process)、谷氨酸代谢过程(glutamate metabolic process)和天冬氨酸代谢过程(aspartate metabolic process)。碳水化合物代谢相关的条目也参与这个过程, 如半乳糖代谢过程(galactose metabolic process)、阿拉伯糖合成过程(arabinose biosynthetic process)、半乳糖氨基转移酶活性(galactinol-sucrose galacosyl-transfererase activity)。另外还富集到昼夜节律的负调控(negative regulation of circadian rhythm)、脂肪酸合成过程(fatty acid biosynthetic process)和三羧酸循环酶复合体(tricarboxylicacid cycle enzyme complex)等条目。
图4 花生种子休眠保持过程中差异基因GO富集分析
Fig. 4 GO enrichment of the DEGs during peanut dormancy maintenance period
HY52: 花育52号。HY52: Huayu 52.
为分析差异表达基因所影响的代谢通路, 对前20个KEGG途径进行了富集分析。KEGG分析表明, α-亚麻酸代谢(alpha-linolenic acid metabolism)、不饱和脂肪酸生物合成(biosynthesis of unsaturated fatty acids)、氨基糖和核糖代谢(amino sugar and nucleotide sugar metabolism)、柠檬酸循环(citratecycle (TCA cycle))、甘油酯代谢(glycerolipid metabolism)、半乳糖代谢(galactose metabolism)显著富集。前人研究表明长链脂肪酸可以作为一种萌发抑制剂, 有可能是通过抑制胚胎生长发挥作用[24]。另外, 许多氨基酸合成和代谢相关的条目, 如缬氨酸、亮氨酸和异亮氨酸合成(valine, leucine and isoleucine biosynthe- sis)、β-丙氨酸代谢(beta-alanine metabolism)、酪氨酸代谢(tyrosine metabolism)、苯丙氨酸、酪氨酸和色氨酸合成(phenylalanine, tyrosine and tryptophan biosynthesis)、苯丙氨酸代谢(phenylalanine metabolism)、精氨酸和脯氨酸代谢(arginine and proline metabolism)、酪氨酸代谢(tyrosine metabolism)、色氨酸代谢(tryptophan metabolism)也在HY52 vs M23 和HY52 vs M67的比较组中同时富集到(图5)。除此之外, 蛋白外运(protein export)和昼夜节律(circadian rhythm-plant)也在HY52 vs M67中显著富集(图5-F)。
2.5 花生休眠解除相关差异表达基因的富集分析
为揭示休眠解除机制, 将M23_12 vs M23_0, M23_24 vsM23_0, M67_12 vs M67_0, M67_24 vs M67_0比较组进行了GO和KEGG富集分析(图6和图7)。KEGG富集分析结果表明, 差异表达基因富集在碳水化合物代谢相关的条目, 如甘油磷脂代谢(glycerophospholipid metabolism)、糖胺聚糖降解(glycosaminoglycan degradation)和氨基酸代谢相关的条目, 如苯丙氨酸、酪氨酸和色氨酸合成(phenylalanine, tyrosine and tryptophan biosynthesis)、甘氨酸、丝氨酸和苏氨酸代谢(glycine, serine and threonine metabolism)、精氨酸和脯氨酸代谢(arginine and proline metabolism)。另外, 还富集到了柠檬酸循环(citrate cycle (TCA cycle))、蛋白外运(protein export)、昼夜节律(circadian rhythm-plant)等条目(图7)。而且在M23_24 vs M23_0比较组中, 富集到了质子转运ATP酶活性(proton-transporting ATPase activity)和质子转运ATP合酶活性(proton- transporting ATP synthase activity) 2个条目。

HY52: 花育52号。HY52: Huayu 52.
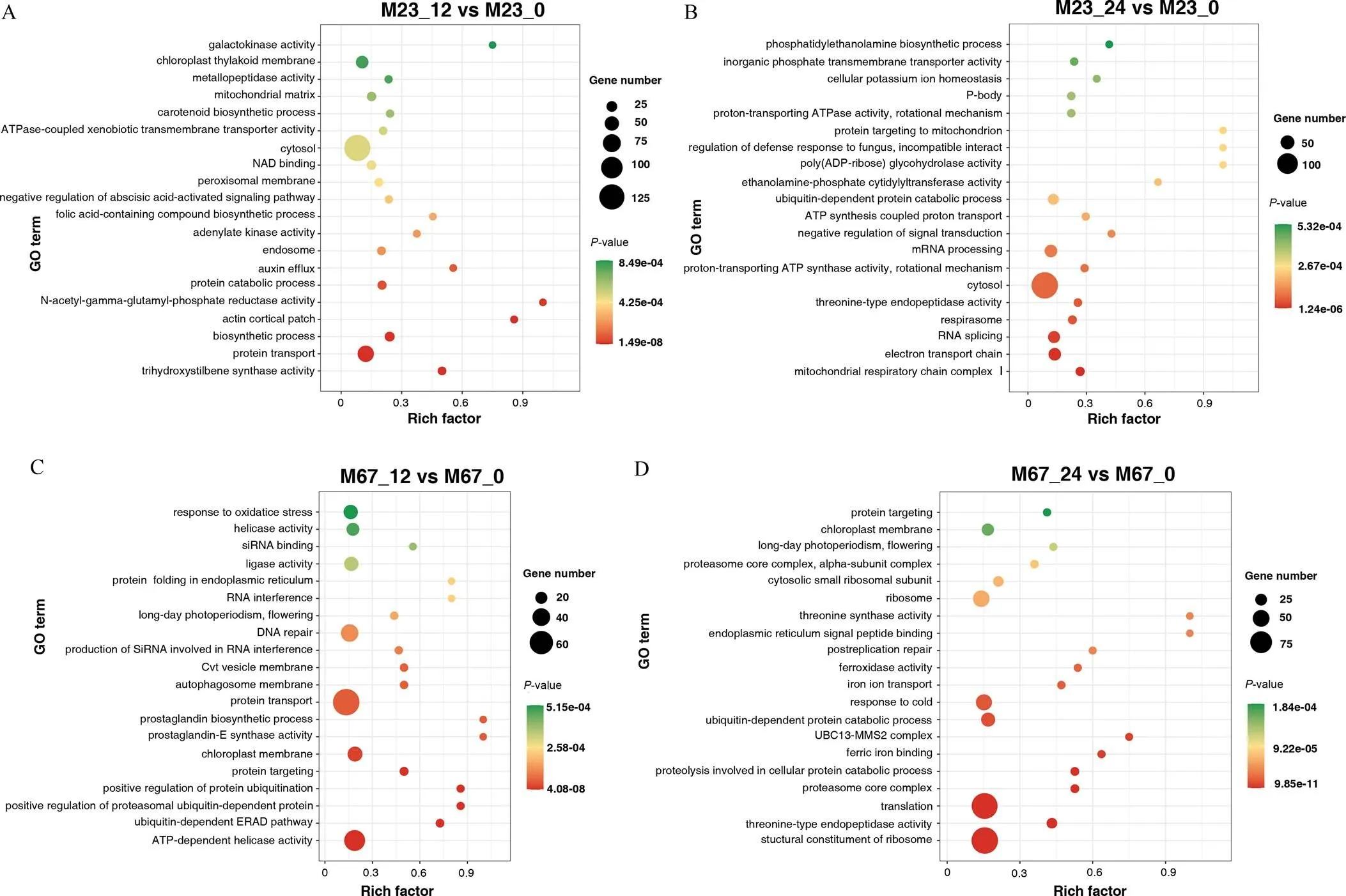
图6 花生种子休眠解除过程中差异基因GO富集分析
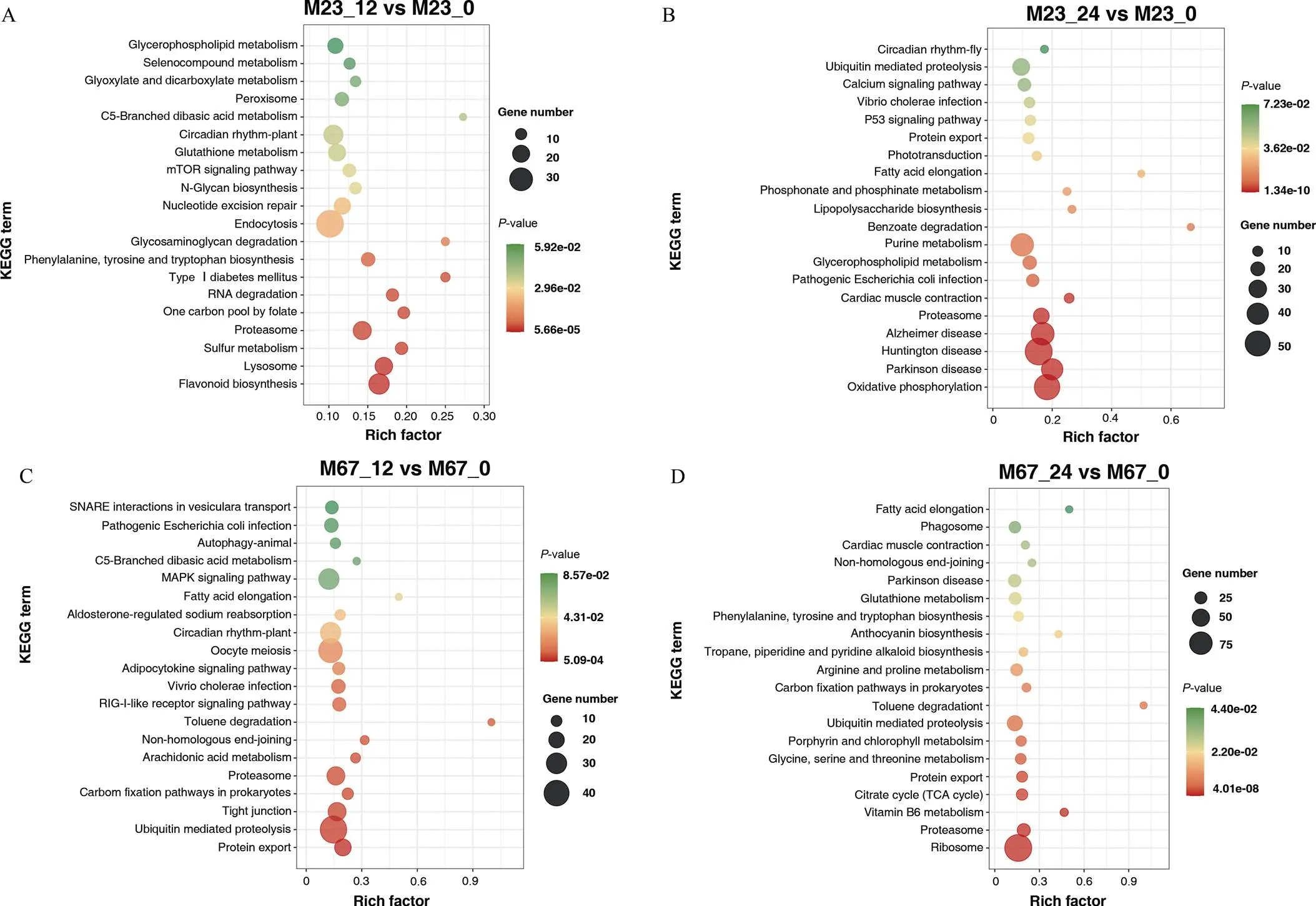
图7 花生种子休眠解除过程中差异基因KEGG富集分析
2.6 花生休眠维持和解除过程中与植物激素合成、代谢和信号转导相关的差异表达基因
种子休眠和萌发是通过植物激素网络调控, 包括ABA、GA、ETH和Auxin等, 植物激素合成、代谢和信号转导相关的基因在种子休眠维持和解除过程中显著表达。因为M67中ABA含量和ABA/GA比值均低于M23 (图1-E, F), 而且HY52 vs M67比HY52 vs M23有更多的差异表达基因(图2-B), 因此以下分析以HY52 vs M67展示。
ABA 8’-羟化酶(CYP707A)是参与ABA代谢的关键酶, 有3个编码CYP707A的基因在HY52和M67中都显著上调(图8)。9个ABA受体()基因随着吸胀时间增加而上调, ABA信号转导相关的34个(编码蛋白磷酸酶2C)基因和4个基因也在吸胀过程中差异表达(图8)。
GA合成相关基因, 如1个(编码ent-贝壳杉酸氧化酶)和2个(编码赤霉素20氧化酶)基因都在休眠解除过程中上调, 但是参与GA失活的2个(编码赤霉素2氧化酶)基因也上调表达(图8)。此外, 1个赤霉素受体基因和2个信号转导元件也参与这个过程。
乙烯是调控种子休眠解除的另一个关键植物激素, 外源施加乙烯能打破种子休眠[7]。乙烯合成基因(2个)、(7个)和5个乙烯受体基因包括, 都在HY52和M67吸胀过程中上调表达(图8)。另外, 还有35个转录因子也在吸胀过程中有差异表达。生长素合成和信号转导相关的基因, 如1个(编码吲哚-3-丙酮酸单加氧酶)、4个(编码生长素上调小RNA)和8个(编码生长素反应因子)在HY52和M67的种子休眠解除过程中差异表达(图8)。
2.7 花生休眠维持和解除过程中与碳水化合物和脂质合成和代谢相关的差异表达基因
KEGG富集分析结果表明, 在HY52_12 vs M23_12中显著富集了氨基糖和核糖代谢(amino sugar and nucleotides sugar metabolism)、α-亚麻酸代谢(alpha-linolenic acid metabolism) (图5-C)条目, 在HY52_24 vs M23_24中显著富集了不饱和脂肪酸合成(biosynthesis of unsaturated fattyacids) (图5-D)条目。在HY52_24 vs M67_24中显著富集了半乳糖代谢(galactose metabolism)、甘油酯代谢(glycerolipid metabolism)、不饱和脂肪酸合成(biosynthesis of unsaturated fatty) (图5-F)条目。在M23_12 vs M23_0和M23_24 vs M23_0中分别富集了糖胺聚糖降解(glycosaminoglycan degradation)和甘油磷脂代谢(glycerophospholipid metabolism) (图7-A, B)条目。基于以上富集分析结果, 对糖和脂质代谢相关的基因进行筛选, 挖掘到9个相关的基因在种子休眠维持和解除过程中发生表达变化(图9)。葡萄糖-6-磷酸/磷酸转运蛋白(GPT)参与淀粉和葡萄糖转化, 其编码基因在种子休眠解除过程中上调(图9-B)。参与脂质合成的3-磷酸甘油酰基转移酶(GPAT)基因在种子休眠解除过程中上调, 参与脂质代谢的3个长链酰基辅酶A合酶(LACS)基因的表达量也在此过程中发生变化(图9-A)。以上所有差异表达基因在M67中的表达量均高于HY52, 表明休眠种子更难以调动能量储备。

图8 花生种子吸胀过程中与激素合成、代谢和信号转导相关的差异基因表达热图
A: ABA、GA、乙烯和生长素的生物合成、代谢和信号转导示意图; B: 与植物激素相关的差异表达基因热图。HY52: 花育52号。
A: the diagram of biosynthesis, metabolism, and signal transduction in ABA, GA, ethylene, and auxin; B: the heatmaps of the relative expression patterns of DEGs related to plant hormones. HY52: Huayu 52.

图9 花生种子吸胀过程中与碳水化合物、脂质合成、代谢相关的差异基因表达热图
A: 脂质代谢相关差异表达基因热图; B: 碳水化合物合成和代谢相关差异表达基因热图。HY52: 花育52号。
A: the relative expression patterns of DEGs related to lipid metabolism displayed by heatmap; B: the relative expression patterns of DEGs related to carbohydrate synthesis and metabolism displayed by heat map. HY52: Huayu 52; DEGs: differentially expressed genes.
3 讨论
3.1 花生休眠维持和解除过程中植物激素网络的调控
脱落酸和赤霉素是调节种子休眠状态的2种主要激素, 它们通过调节激素含量和信号转导的平衡发挥拮抗作用[7,25]。ABA维持种子休眠, 而GA促进种子萌发。前人研究表明种子休眠是由较高的ABA/GA比值调控的, 休眠解除或萌发则是通过促进GA合成和ABA降解导致ABA/GA比值下降来实现的[26-29]。综上, GA负调控种子休眠而ABA正调控种子休眠。值得注意的是, 吸胀过程中, 虽然M23和M67中ABA/GA比值都显著低于HY52, 但M67中ABA/GA比值更低, 暗示了M67的休眠性更弱。本研究中, GA含量在强休眠品种HY52和弱休眠种质M23和M67中都随着吸胀时间而增加(图1-B)。但ABA含量在M23和M67中随吸胀进程而降低, 而且含量低于HY52 (图1-C)。ABA/GA比值随吸胀进程降低, M23和M67中ABA/GA比值显著低于HY52 (图1-D)。尽管HY52中ABA/GA比值有大幅度下降, 但是其休眠状态并未被打破, 暗示了HY52具有很强的休眠性, 解除休眠可能需要更低的ABA/GA比值。拟南芥和烟草中, ABA含量在吸胀过程中积累[26,30]。同时, 在休眠解除过程中ABA代谢也会加快, 因此能测到的ABA含量是生物合成代谢后的净值[27]。Lu14是一个休眠不深的花生品种, 在子叶吸胀4 h、16 h、28 h和52 h的过程中ABA/GA3比值从38.26极速降低到3.82[19]。本研究中, HY52、M23、M67中的ABA/GA比值分别从1335到676、986到651、676到343 (图1-F), ABA/GA比值较高的原因可能是本研究中GA含量和ABA含量的单位不同。
GA生物合成基因的表达模式与GA含量变化一致(图8-B), 除了植物激素合成相关的基因外, 信号转导相关的蛋白也参与种子休眠解除[31-32]。ABA受体基因/在HY52和M67中上调, 但是却下调了(图8-B)。在拟南芥中, PYL/PYR家族成员通过与ABA信号转导途径中的负调控因子PP2C互作发挥冗余功能[25]。据报道, 包括ABI1、ABI2在内的多种PP2C参与了种子休眠调控[33]。GA信号转导元件的突变也能影响种子萌发, DELLA蛋白基因的功能缺失挽救了其不萌发的表型[34]。GA受体GID和下游蛋白DELLA也是种子休眠维持和解除的重要调控因子。与预期一致, 本研究中在弱休眠花生M67中上调, 但是负调控因子则下调(图10)。表明ABA和GA相关的差异表达基因在花生种子休眠维持和解除中发挥作用。
生长素也是调控种子休眠维持和解除的重要植物激素, 已有报道表明, IAA参与ABA的互作从而促进种子休眠[35-37]。在种子休眠解除过程中, 拟南芥、豌豆和小麦中IAA含量增加[38-40]。与前人研究结果一致, IAA信号相关基因, 如TIR1、IAA14和TCP均上调(图10), 表明生长素可能在花生种子休眠维持和解除中发挥作用。
3.2 花生休眠维持和解除过程中碳水化合物和氨基酸代谢与调控
种子萌发是一个耗能的过程, 三酰基甘油(triacyl-glycerol, TAG)是花生主要的脂质储备物质。在萌发过程中, TAG被脂肪酶水解为脂肪酸, 生成的脂肪酸被转运进入过氧化物酶体经β氧化和乙醛酸循环为萌发后的生长过程提供能量和C骨架。
淀粉、油脂和蛋白质等贮藏物质是种子从休眠到发芽转变的重要能源[19,41-42], 在种子休眠解除过程中, 淀粉、油脂和蛋白质分别降解为可溶性糖、脂肪酸和氨基酸。虽然淀粉不是花生中的主要碳储备, 但在种子休眠解除过程中, 参与淀粉和蔗糖转化的5种GPT均上调(图9-B)。油脂(三酰基甘油或TAG)和蛋白质占花生种子含量的80%[43], 因此可能是主要的能量储备。本研究结果表明, 参与脂质生物合成和代谢的3个长链酰基辅酶A合酶(LACS)基因可能参与种子休眠解除过程(图9-A)。
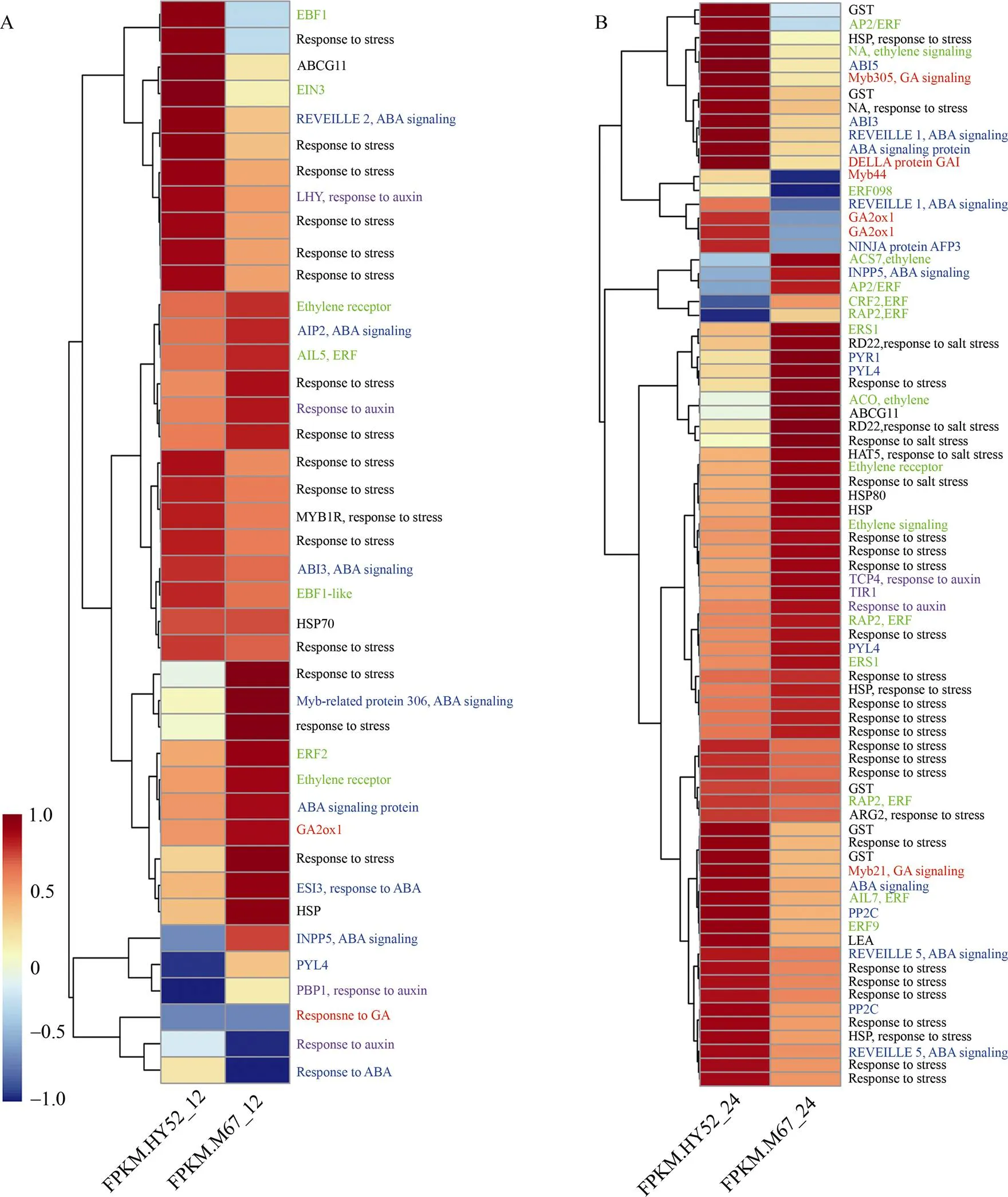
图10 HY52_12 vs M67_12 (A) and HY52_24 vs M67_24 (B)比较组前20个GO条目中与植物激素和胁迫相关的差异表达基因热图分析
HY52: 花育52号。HY52: Huayu 52.
与差异表达基因的结果一致, 与碳水化合物代谢相关的GO条目“阿拉伯糖生物合成过程”在HY52_12 vs M23_12 (图4-A)和HY52_12 vs M67_12显著富集(图5-A); “半乳糖-蔗糖半乳糖基转移酶活性”在HY52_24 vs M23_24 (图4-B)和HY52_24 vs M67_24 (图5-B)中显著富集; “营养库活性”和“半乳糖代谢过程”分别在HY52_12 vs M23_12 (图4-A)和HY52_12 vs M67_12 (图5-A)显著富集。与三酰基甘油代谢相关的GO条目“三羧酸循环酶复合物”、“脂肪酸生物合成”在HY52_24 vs M67_24中显著富集(图5-B)。表明, 淀粉和油脂代谢在花生种子休眠解除中也可能发挥作用, 但是需要更多的实验数据支持。
本研究结果表明, 氨基酸代谢可能参与了花生种子的休眠, 花生种子富含蛋白质, 可降解为氨基酸, 表明蛋白质可能是种子休眠解除和萌发的贮藏储备, 与氨基酸生物合成和代谢相关的KEGG条目在HY52 vs M23和HY52 vs M67中显著富集(图7和图8)。氨基酸不仅在大麦萌发后生物活性物质(如细胞蛋白和分泌的水解酶)的生物合成中发挥重要作用[44-45], 而且是许多具有多功能的代谢物的前体[46]。Sato等[46]的研究表明, 主效休眠基因编码丙氨酸转氨酶(AlaAT)调控大麦种子休眠。
3.3 花生种子休眠维持和解除过程中的其他调控因子
值得注意的是, 在HY52 vs M23和HY52 vs M67比较组中还富集到了一些GO条目, 如“胁迫反应” “活性氧代谢调控” “种子耐旱性获得” (图4和图5)。在种子萌发过程中, 种子由相对静止状态转入活跃状态, 重新生长的过程类似于胁迫响应。大多数休眠种子在种子发育后期具有抗干燥性, 因此可以在干燥状态下存活很长时间。然而, 由于活性氧(ROS)的产生和氧化损伤的发生, 非休眠的种子并不耐干燥[7]。
此外, GO条目“昼夜节律的负调控”在HY52_12 vs M67_12中显著富集(图4-E), KEGG条目“昼夜节律”在HY52_24 vs M67_24中富集(图5-F), 这表明昼夜节律也可能参与种子休眠调控。植物昼夜节律是内源性产生和自我维持的特性, 因此它在恒定的环境条件下持续存在, 通常是恒定的光照(或黑暗)和恒定的温度[47]。在拟南芥中, 生物钟基因,()和()在整合环境信号控制休眠解除中发挥重要作用[48]。本研究结果表明, 休眠维持和解除的过程可能是昼夜节律的重新调整。
4 结论
本研究综合分析了强休眠品种HY52和弱休眠种质M23和M67在种子休眠维持和解除过程中的调控网络。结果表明, 植物激素(脱落酸、赤霉素、乙烯和生长素)的生物合成和信号转导在调控种子休眠维持和解除中起关键作用。此外, 能量储备(碳水化合物和脂质)、氨基酸代谢、昼夜节律也可能参与了种子休眠解除过程, 本研究拓展了对花生种子休眠维持和解除调控的认知, 也为花生休眠育种奠定了理论基础。
[1] Hilhorst H W. Standardizing seed dormancy research., 2011, 773: 43–52.
[2] Baskin J M, Baskin C C. A classification system for seed dormancy., 2007, 14: 1–16.
[3] Fenner M, Thompson K, The Ecology of Seeds. New York: Cambridge University Press, 2005. pp 99–104.
[4] Wang M L, Wang H, Zhao C, Tonnis B, Tallury S, Wang X, Clevenger J, Guo B. Identification of QTLs for seed dormancy in cultivated peanut using a recombinant inbred line mapping population., 2021, 40: 208–217.
[5] 胡晓辉, 崔凤高, 张胜忠, 苗华荣, 张智猛, 陈静. 花生种子休眠特异突变材料的创制及理化因素研究花生学报, 2018, 47(1): 33–37. Hu X H, Cui F G, Zhang S Z, Miao H R, Zhang Z M, Chen J. Study on the creation and physicochemical factors of peanut seed dormancy mutant., 2018, 47(1): 33–37 (in Chinese with English abstract).
[6] Shi J, Shi J, Liang W, Zhang D. Integrating GWAS and transcriptomics to identify genes involved in seed dormancy in rice, 2021, 134: 3553–3562.
[7] Finkelstein R, Reeves W, Ariizumi T, Steber C. Molecular aspects of seed dormancy, 2008, 59: 387–415.
[8] Sondheimer E, Tzou D S, GalsonE C. Abscisic acid levels and seed dormancy, 1968, 43: 1443–1447.
[9] Kallio P, Piiroinen P. Effect of gibberellin on the termination of dormancy in some seeds, 1959, 183: 1830–1831.
[10] Nee G, Xiang Y, Soppe W J. The release of dormancy, a wake-up call for seeds to germinate, 2017, 35: 8–14.
[11] Kucera B, Cohn M A, Leubner-Metzger G. Plant hormone interactions during seed dormancy release and germination, 2007, 15: 281–307.
[12] Zhang M, Zeng Q, Liu H, Qi F, Sun Z, Miao L, Li X, Li C, Liu D, Guo J, Zhang M, Xu J, Shi L, Tian M, Dong W, Huang B, Zhang X. Identification of a stable major QTL for fresh-seed germination on chromosome Arahy. 04 in cultivated peanut (L.), 2022, 10: 1767–1773.
[13] 郝西, 张俊, 刘娟, 臧秀旺, 董文召, 汤丰收. 不同花生品种种子休眠性鉴定种子, 2018, 37(8): 1–3. Hao X, Zhang J, Liu J, Zang X W, Dong W Z, Tang F S. Identification of seed dormancy of diffident varieties., 2018, 37(8): 1–3 (in Chinese with English abstract).
[14] 任明刚, 何大智, 冯明友, 李婵, 杨如英, 张超, 穆航. 贵州78份地方花生品种的休眠性及相关分析种子, 2020, 39(11): 55–58. Ren M G, He D Z, Feng M Y, Li C, Yang R Y, Zhang C, Mu H. Dormancy and correlation analysis of 78 local peanut varieties in Guizhou., 2020, 39(11): 55–58 (in Chinese with English abstract).
[15] Xie K, Bai J, Yang Y Y, Duan N B, Ma Y M, Guo T, Yao F Y, Ding H F. The RNA-seq transcriptome analysis identified genes related to rice seed dormancy, 2019, 63: 308–313.
[16] Han Z, Wang B, Tian L, Wang S, Zhang J, Guo S, Zhang H, Xu L, Chen Y. Comprehensive dynamic transcriptome analysis at two seed germination stages in maize (L.), 2020, 168: 205–217.
[17] Li X, Qiao H, Wang Z, Han B, Xing Y, Yang Y. A Comparative transcriptome analysis reveals new insights into pre-harvest sprouting (PHS) in wheat., 2021, DOI: 10.21203/rs. 3.rs-910461/v1.
[18] Park M, Choi W, Shin S Y, Moon H, Lee D, Gho Y S, Jung K H, Jeon J S, Shin C. Identification of genes and microRNAs affecting pre–harvest sprouting in rice(L.) by transcriptome and small RNAome analyses, 2021, 12: 727302.
[19] Xu P, Tang G, Cui W, Chen G, Ma C L, Zhu J, Li P, Shan L, Liu Z, Wan S. Transcriptional differences in peanut (L.) seeds at the freshly harvested, after-ripening and newly germinated seed stages: insights into the regulatory networks of seed dormancy release and germination, 2020, 15: e0219413.
[20] Zhang J, Qian J Y, Bian Y H, Liu X, Wang C L. Transcriptome and metabolite conjoint analysis reveals the seed dormancy release process in Callery Pear, 2022, 23: 2186.
[21] Tai L, Wang H J, Xu X J, Sun W H, Ju L, Liu W T, Li W Q, Sun J, Chen K M. Pre-harvest sprouting in cereals: genetic and biochemical mechanisms, 2021, 72: 2857–2876.
[22] Bertioli D J, Jenkins J, Clevenger J, Dudchenko O, Gao D, Seijo G, Leal-Bertioli S C M, Ren L, Farmer A D, Pandey M K, Samoluk S S, Abernathy B, Agarwal G, Ballen-Taborda C, Cameron C, Campbell J, Chavarro C, Chitikineni A, Chu Y, Dash S, El Baidouri M, Guo B, Huang W, Kim K D, Korani W, Lanciano S, Lui C G, Mirouze M, Moretzsohn M C, Pham M, Shin J H, Shirasawa K, Sinharoy S, Sreedasyam A, Weeks N T, Zhang X, Zheng Z, Sun Z, Froenicke L, Aiden E L, Michelmore R, Varshney R K, Holbrook C C, Cannon E K S, Scheffler B E, Grimwood J, Ozias-Akins P, Cannon S B, Jackson S A, Schmutz J. The genome sequence of segmental allotetraploid peanut, 2019, 51: 877–884.
[23] Ernst J, Bar-Joseph Z. STEM: a tool for the analysis of short time series gene expression data, 2006, 7: 191.
[24] Liu D, Yu H L, Li F L, Guo H H. An analysis of dormancy and dormancy release in Taxus chinensis var. mairei seeds, 2011, 39: 29–43.
[25] Graeber K, Nakabayashi K, Miatton E, Leubner-Metzger G, Soppe W J. Molecular mechanisms of seed dormancy, 2012, 35: 1769–1786.
[26] Ali-Rachedi S, Bouinot D, Wagner M H, Bonnet M, Sotta B, Grappin P, Jullien M. Changes in endogenous abscisic acid levels during dormancy release and maintenance of mature seeds: studies with the Cape Verde Islands ecotype, the dormant model of, 2004, 219: 479–488.
[27] Cadman C S, Toorop P E, Hilhorst H W, Finch-Savage W E. Gene expression profiles ofCvi seeds during dormancy cycling indicate a common underlying dormancy control mechanism, 2006, 46: 805–822.
[28] Finch-Savage W E, Leubner-Metzger G. Seed dormancy and the control of germination, 2006, 171: 501–523.
[29] 崔维佩, 唐桂英, 徐平丽, 李鹏祥, 朱洁琼, 单雷. 花生种子萌发过程中内源激素含量的变化中国油料作物学报, 2020, 42: 869–877. Cui W P, Tang G Y, Xu P L, Li P X, Zhu J Q, Shan L. Changes of endogenous hormone content in peanut seeds during germination., 2020, 42: 869–877 (in Chinese with English abstract).
[30] Grappin P, Bouinot D, Sotta B, Miginiac E, Jullien M. Control of seed dormancy in: post-imbibition abscisic acid synthesis imposes dormancy maintenance, 2000, 210: 279–285.
[31] Shu K, Liu X D, Xie Q, He Z H. Two faces of one seed: hormonal regulation of dormancy and germination, 2016, 9: 34–45.
[32] Yang B, Cheng J, Wang J, Cheng Y, He Y, Zhang H, Wang Z. Physiological characteristics of cold stratification on seed dormancy release in rice, 2019, 89: 131–141.
[33] Merlot S, Gosti F, Guerrier D, Vavasseur A, Giraudat J. The ABI1 and ABI2 protein phosphatases 2C act in a negative feedback regulatory loop of the abscisic acid signalling pathway, 2001, 25: 295–303.
[34] Lee S, Cheng H, King K E, Wang W, He Y, Hussain A, Lo J, Harberd N P, Peng J. Gibberellin regulates Arabidopsis seed germinationRGL2, a GAI/RGA-like gene whose expression is up–regulated following imbibition, 2002, 16: 646–658.
[35] Ramaih S, Guedira M, Paulsen G M. Relationship of indoleacetic acid and tryptophan to dormancy and preharvest sprouting of wheat, 2003, 30: 939–945.
[36] Bai B, Novak O, Ljung K, Hanson J, Bentsink L. Combined transcriptome and translatome analyses reveal a role for tryptophan-dependent auxin biosynthesis in the control of DOG1-dependent seed dormancy, 2018, 217: 1077–1085.
[37] Pellizzaro A, Neveu M, Lalanne D, Vu B L, Kanno Y, Seo M, Leprince O, Buitink J. A role for auxin signaling in the acquisition of longevity during seed maturation, 2020, 225: 284–296.
[38] Preston J, Tatematsu K, Kanno Y, Hobo T, Kimura M, Jikumaru Y, Yano R, Kamiya Y, Nambara E. Temporal expression patterns of hormone metabolism genes during imbibition ofseeds: a comparative study on dormant and non-dormant accessions, 2009, 50: 1786–1800.
[39] Ayele B T, Ozga J A, Wickramarathna A D, Reinecke D M. Gibberellin metabolism and transport during germination and young seedling growth of pea (L.), 2011, 31: 235–252.
[40] Liu A, Gao F, Kanno Y, Jordan M C, Kamiya Y, Seo M, Ayele B T. Regulation of wheat seed dormancy by after-ripening is mediated by specific transcriptional switches that induce changes in seed hormone metabolism and signaling, 2013, 8: e56570.
[41] Fait A, Angelovici R, Less H, Ohad I, Urbanczyk-Wochniak E, Fernie A R, Galili G.seed development and germination is associated with temporally distinct metabolic switches., 2006, 142: 839–854.
[42] Rosental L, Nonogaki H, Fait A. Activation and regulation of primary metabolism during seed germination., 2014, 24: 1–15.
[43] Pandey M K, Pandey A K, Kumar R, Nwosu C V, Guo B, Wright G C, Bhat R S, Chen X, Bera S K, Yuan M, Jiang H, Faye I, Radhakrishnan T, Wang X, Liang X, Liao B, Zhang X, Varshney R K, Zhuang W. Translational genomics for achieving higher genetic gains in groundnut., 2020, 133: 1679–1702.
[44] Ullrich S E. Barley: Production, Improvement, and Uses. Chichester: John Wiley & Sons, 2010. p 137.
[45] Bryan A, Joseph L, Bennett J A, Jacobson H I, Andersen T T. Design and synthesis of biologically active peptides: a ‘tail’ of amino acids can modulate activity of synthetic cyclic peptides., 2011, 32: 2504–2510.
[46] Sato K, Yamane M, Yamaji N, Kanamori H, Tagiri A, Schwerdt J G, Fincher G B, Matsumoto T, Takeda K, Komatsuda T. Alanine aminotransferase controls seed dormancy in barley, 2016, 7: 11625.
[47] McClung C R. Plant circadian rhythms., 2006, 18: 792–803.
[48] Penfield S, Hall A. A role for multiple circadian clock genes in the response to signals that break seed dormancy in, 2009, 21: 1722–1732.
Comparative transcriptome profiling of dormancy regulatory network in peanut
WANG Fei-Fei1, ZHANG Sheng-Zhong1, HU Xiao-Hui1, CHU Ye2, CUI Feng-Gao1, ZHONG Wen3, ZHAO Li-Bo4, ZHANG Tian-Yu3, GUO Jin-Tao5, YU Hao-Liang6, MIAO Hua-Rong1,*, and CHEN Jing1,*
1Shandong Peanut Research Institute, Qingdao 266100, Shandong, China;2Department of Horticulture, University of Georgia Tifton Campus, Tifton 31793, GA, United States;3Shandong Seed Administration Station, Jinan 250100, Shandong, China;4Qingdao Agricultural Technology Extension Center, Qingdao 266071, Shandong, China;5Agricultural Service Center of Hezhuang, Xinzheng, Zhengzhou 451150, Henan, China;6Yantai Fenglin Foodstuff Co., Ltd, Yantai 264108, Shandong, China
Seed dormancy is an important and complex agronomic trait affecting yield and quality of peanut (L.). Seed dormancy and germination was reported to be regulated by the balance between abscisic acid (ABA) and gibberellic acid (GA). In this study, transcriptomic sequencing was performed with Huayu 52 (HY52), a peanut cultivar with strong dormancy, and two EMS mutant lines from HY52 with a weak level of dormancy. Seeds from these three lines were imbibed for 0, 12, and 24 h before tissue harvesting and RNA seq analysis. GA content of M23 and M67 was significantly higher than HY52 at 12 h after imbibition, however, the ABA content and ABA/GA ratio were lower than HY52. A total of 31,374 differentially expressed genes (DEGs) including biosynthesis and signal transduction related genes of plant hormones such as ABA and GA were discovered. We identified 50 genes related to ABA, 8 genes related to GA, 49 genes related to ethylene, and 13 genes related to auxin. Expression profiles of ABA and GA related genes was consistent with the higher GA and lower ABA content in the mutants compared with HY52 after 12 h and 24 h imbibition. In addition, many DEGs involved in carbohydrate and lipid metabolism, amino acid metabolism, and glutathione metabolism pathway were also identified. There were 5 carbohydrate metabolism related genes () and 4 lipid metabolism related genes. In addition, differentially regulated circadian rhythm pathways were found to involve in the process of peanut seed dormancy release. These results suggested that the regulation of dormancy maintenance and release was more complicated than phytohormone balance.
peanut; dormancy maintenance; dormancy release; transcriptome; plant hormone; amino acid metabolism
2022-08-10;
2023-02-21;
2023-03-03.
10.3724/SP.J.1006.2023.24186
通信作者(Corresponding authors):陈静, E-mail: mianbaohua2008@126.com: 苗华荣, E-mail: 1649813628@qq.com
E-mail: wangfeifeisj@163.com
本研究由国家自然科学基金青年基金项目(32001584, 32201876), 山东省自然科学基金面上项目(ZR2022MC045), 山东省农业良种工程(2020LZGC001), 山东省农业科学院创新工程(CXGC2022A03, CXGC2022A21), 青岛市民生科技计划项目(20-3-4-26-nsh)和新疆维吾尔自治区重大科技专项(2022A02008-3)资助。
This study was supported by the Youth Fund Project of the National Natural Science Foundation of China (32001584, 32201876), the General Project of Shandong Natural Science Foundation (ZR2022MC045), the Shandong Province Agriculture Improved Seed Project (2020LZGC001), the Innovation Project of Shandong Academy of Agriculture Sciences (CXGC2022A03, CXGC2022A21), the Qingdao People’s Livelihood Science and Technology Project (20-3-4-26-nsh), and the Major Science and Technology Program of Xinjiang Uygur Autonomous Region (2022A02008-3).
URL: https: //kns.cnki.net/kcms/detail/11.1809.S.20230302.1555.009.html
This is an open access article under the CC BY-NC-ND license (http: //creativecommons.org/licenses/by-nc-nd/4.0/).
