利用蛋白支架复合物提高大肠杆菌MG1655的L-苹果酸产量
2023-06-07周志东王晓瑜杨之帆
周志东,王晓瑜,张 同,李 琪,杨之帆,陈 俊
(1. 武汉科技大学化学与化工学院,湖北 武汉,430081;2. 湖北大学省部共建生物催化与酶工程国家重点实验室,湖北 武汉,430062)
苹果酸是一种四碳二羧酸,被广泛用于食品、化工、医药和农业领域[1],常用的生产方法有化学合成法、固定化酶催化法及发酵法等。化学合成法是将苯或丁烷在高温高压下氧化后再利用马来酸的水合作用产生D-苹果酸和L-苹果酸外消旋混合物[2],其中L-苹果酸存在于从细菌到人类的所有生物体中,而D-苹果酸在自然界中很少见且难以被人体吸收利用,故被禁止添加至婴幼儿及老年人食品中。酶催化法是利用固定化的富马酸酶或表达富马酸酶的全细胞,以富马酸为底物催化产生L-苹果酸[3],使用该法虽可获得单一的L-苹果酸,但富马酸酶的纯化成本较高,另外,产物L-苹果酸与反应残留的底物富马酸较难分离,所得L-苹果酸纯度不高。近年来,利用基因工程菌发酵已成为获取L-苹果酸的重要途径[4]。将来自解甘露醇罗尔斯顿菌的磷酸烯醇式丙酮酸(PEP)羧激酶导入pta突变型的大肠杆菌菌株WGS-10,经12 h好氧发酵后,所得L-苹果酸产量可达9.25 g/L[5]。除此之外,利用苹果酸酶催化丙酮酸也可以生成苹果酸。不过,丙酮酸是多种代谢途径的关键底物,将其转化为苹果酸并非易事。Dong等[6]通过敲除大肠杆菌W3110的ldhA、poxB、pflB、pta及ackA等基因来阻断丙酮酸的旁路消耗,再辅以苹果酸酶的过表达,所得苹果酸产量达到21.7 g/L。本课题组[7]敲除大肠杆菌MG1655的poxB、pta-ackA、ldhA、pflB、maeA、maeB以及pfkA等基因后,将所得工程菌株摇瓶发酵48 h,相应L-苹果酸产量达到9.893 g/L。利用基因敲除或过表达构建多基因的从头合成途径时,常面临中间产物的快速扩散和降解、对宿主细胞的毒害以及底物利用率低等问题,最终导致目标产物产量降低[8]。已有研究表明,通过模仿自然代谢控制机制,将途径酶共定位,在空间上形成多酶复合体,提高代谢物的局部浓度,在多酶复合体内形成底物通道,能减少底物在不同活性位点间的扩散距离,将代谢通量引向目标通路从而提高目标产物的得率[9-13]。Dueber等[14]直接截取后生动物信号传导蛋白相互作用域并融合表达后构建了一种(GBD)x-(SH3)y-(PDZ)z蛋白支架体系,通过优化蛋白支架中配体的比例、顺序以及酶的化学计量数,将最终产物滴度提高了77倍。Liu等[15]利用纤维素小体,将3种NAD+依赖性的脱氢酶于酵母表面组装来实现多酶级联反应,以甲醇作为底物所得NADH的最终产率达到了组装前相应值的5.1倍。Ji等[16]利用相互作用的蛋白PDZ和PDZ ligand将黄岑素和野黄岑素合成途径中的两个关键途径酶进行自组装,目标产物最终滴度分别提高了6.6和1.4倍。有鉴于此,本文选取遗传背景清楚且易于进行分子操作的大肠杆菌MG1655,借助RIAD-RIDD人工蛋白支架,将L-苹果酸生成途径关键酶Pykf和maeB进行共定位以形成酶复合体,再通过丙酮酸的积累来实现maeB的逆向催化,进而提高L-苹果酸的产量。
1 材料与方法
1.1 菌株、质粒、引物及仪器和试剂
大肠杆菌 DH5α、大肠杆菌 MG1655(DE3)和大肠杆菌 BL21(DE3)购于武汉力博瑞生物科技有限公司,载有荧光报告蛋白的支架片段以及N端或C端带有支架蛋白标签的目的基因片段由武汉金开瑞生物工程有限公司合成,质粒载体pET-23a和PRSFDuet-1为本实验室保存,构建载体所用引物均由生工生物工程(上海)股份有限公司合成。本研究所用菌株、质粒以及引物分别列于表1及表2,所用主要仪器有:Bio-Rad C1000型PCR仪,Sigma 1-14K型高速冷冻离心机,D-30M型高压细胞破碎仪,C006169(5 ml)和C006197(20 ml)重力型蛋白纯化空柱,Milipore超滤管,P680型高效液相色谱仪;主要试剂有:牛血清白蛋白,购自武汉天源生物技术有限公司,PIPES,购自北京鼎国昌盛生物技术有限责任公司,卡那霉素、壮观霉素,购自合肥博美生物科技有限责任公司,DNA Marker、质粒小提试剂盒、胶回收试剂盒、2×ESTaqMaster Mix,均购自南京诺唯赞生物科技股份有限公司,有机酸标准样品,购自Sigma公司,其它试剂均为国产分析纯试剂。
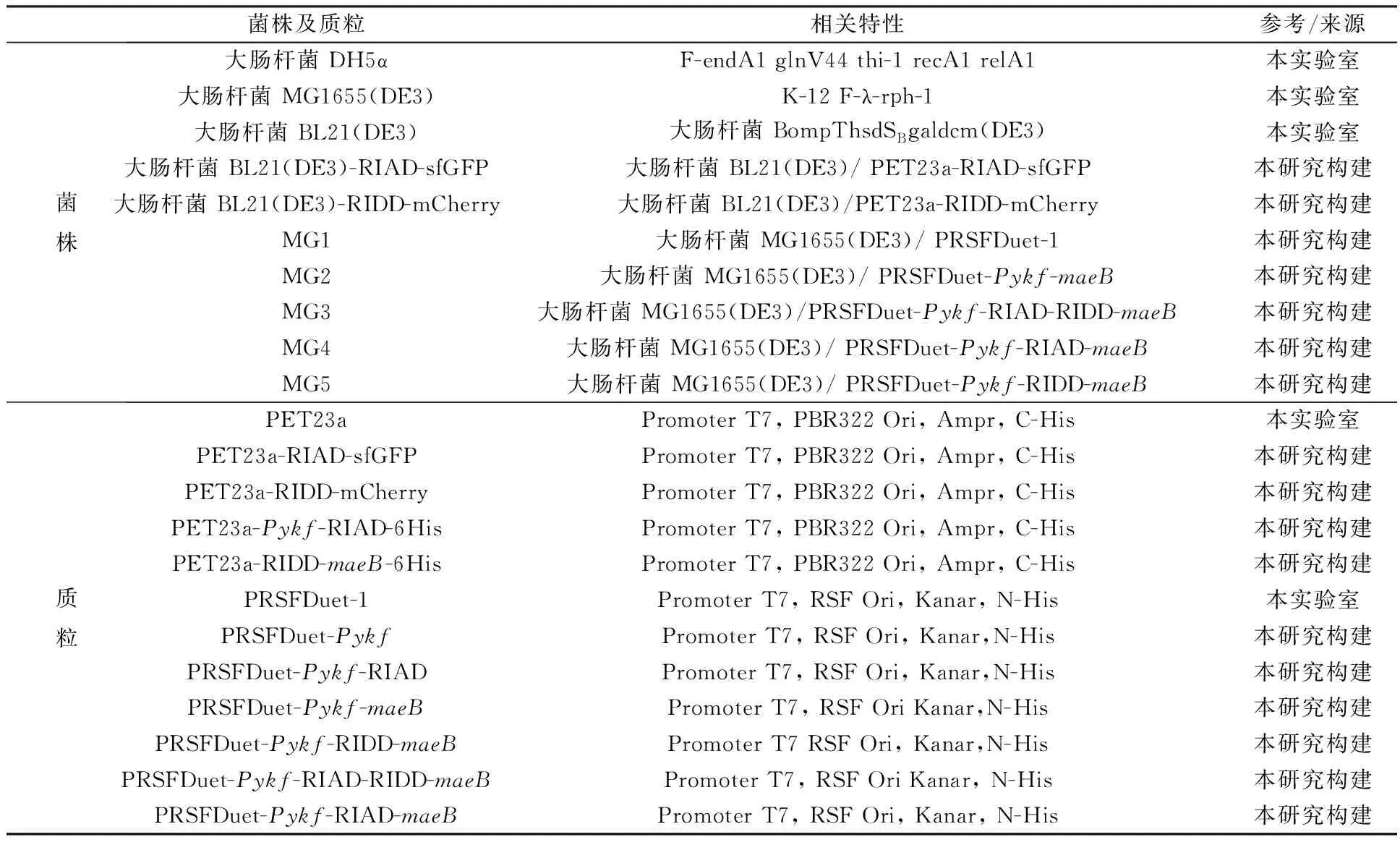
表1 菌株和质粒

表2 引物
1.2 酶支架系统的验证
1.2.1 荧光蛋白基因的装配
将红色荧光蛋白mCherry和绿色荧光蛋白sfGFP分别与RIDD和RIAD的C端融合并装配于载体pET23a上,构建重组报告质粒pET23a-RIAD-sfGFP和pET23a-RIDD-mCherry,sfGFP蛋白和mCherry蛋白靠近时会发出橙色荧光,由此判断RIAD-RIDD蛋白支架系统的结合情况[17]。
1.2.2 荧光蛋白的诱导表达
将构建成功的荧光报告质粒pET23a-RIDD-mCherry和pET23a-RIAD-sfGFP分别转化到大肠杆菌菌株BL21(DE3)中,相应得到菌株BL21(DE3)- RIDD-mCherry、BL21(DE3)- RIAD-sfGFP,于新鲜平板上分别挑取单菌落接种至5 mL氨苄青霉素终浓度为100 μg/mL的LB液体培养基中,在37 ℃下以200 r/min转速过夜培养,之后再以1%的接种量转接到100 mL相同氨苄青霉素浓度的LB液体培养基中,待OD600达到0.8~1.0时,加入终浓度为1.0 mmol/L的IPTG并在18 ℃、200 r/min的条件下培养16~18 h,待诱导结束后离心收集菌体并置于-20 ℃冰箱备用。
1.2.3 重组蛋白的纯化
将充分诱导蛋白表达后离心收集的大肠杆菌 BL21(DE3)-RIDD-mCherry、BL21(DE3)- RIAD-sfGFP菌体从冰箱取出并解冻,利用20 mL PBS缓冲液充分重悬后加入200 μL PMSF(0.01 mol/L)蛋白酶抑制剂,使用高压细胞破碎仪充分破碎细胞释放蛋白并收集上清液,将上清液移至重力柱中,再利用含有不同梯度浓度咪唑的PBS缓冲液洗脱后分别收集流出液,将纯度较高的流出液组分移至相应规格的超滤管中来进一步纯化浓缩目的蛋白。测定蛋白浓度后将其分装至1.5 mL EP管中,经液氮速冻后于-80 ℃保存。
1.3 工程菌中目的基因的组装
在丙酮酸激酶Pykf的C端和苹果酸酶maeB的N端分别添加RIAD和RIDD肽段标签合成RIAD-Pykf和RIDD-maeB后克隆至pET-23a载体上,所构建的载体依次为pET23a-Pykf-RIAD、pET23a-RIDD-maeB。借助特异性引物RSF-Pykf-RIAD-F/R,以质粒pET23a-Pykf-RIAD为模板,扩增获得Pykf-RIAD片段;利用MCS2-RIDD-maeB-F/R引物,以质粒pET23a-RIDD-maeB为模板,扩增获得RIDD-maeB片段。再以大肠杆菌 MG1655(DE3)基因组DNA为模板,分别使用引物RSF-Pykf-F/R、MCS2-maeB-F/R扩增相应的片段Pykf和maeB来构建无组装能力的对照组。图1所示为重组质粒pRSFDuet-Pykf-RIAD-RIDD-maeB的具体构建过程,其关键在于将目的片段Pykf-RIAD和RIDD-maeB依次装载到质粒pRSFDuet-1的第一和第二个MCS。按同样方法构建pRSFDuet-Pykf-maeB、pRSFDuet-Pykf-RIDD-maeB以及pRSFDuet-Pykf-RIAD-maeB等重组质粒。
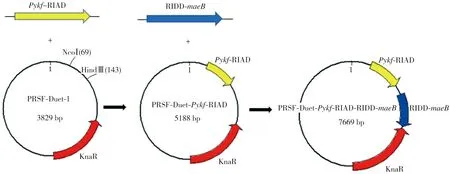
图1 重组质粒的构建
1.4 重组蛋白诱导表达条件
配制卡那霉素浓度为50 μg/mL的LB液体培养基。将构建成功的表达载体PRSFDuet-Pykf-RIAD-RIDD-maeB转化至大肠杆菌 MG1655(DE3)得到工程菌株MG3,挑取单菌落接种至5 mL 的LB液体培养基中于37 ℃下培养过夜,再以1%的比例将种子液接种至200 mL的LB液体培养基中于37 ℃下培养,待菌液OD600达到0.8~1.0时,取出一部分菌液分为若干组并加入终浓度为1.0 mmol/L的IPTG后分别于18、28、30、37 ℃下以200 r/min的转速诱导培养16~18 h,待诱导结束后破胞,使用SDS-PAGE检测蛋白表达情况,以此确定重组蛋白诱导表达的最佳温度。同时,将另一部分OD600达到0.8~1.0的菌液仍分为若干组,分别加入终浓度为0.2、0.4、0.6、0.8、1.0、1.2 mmol/L的IPTG,并转移至30 ℃的恒温摇床以200 r/min的转速诱导培养16~18 h,待诱导结束后破胞,使用SDS-PAGE检测蛋白表达情况,以此确定重组蛋白诱导表达的诱导剂IPTG最佳浓度。
1.5 重组蛋白的诱导表达及检测
确定最佳的培养温度和IPTG诱导剂浓度后,进行重组蛋白Pykf和maeB的诱导表达及检测,具体步骤参照荧光蛋白的诱导表达及重组蛋白的纯化。
1.6 工程菌株摇瓶发酵及检测
本研究采用好氧发酵方式。取出冻存的工程菌株,在添加了相应抗性的LB平板上划线,挑取单菌落接种于50 mL、卡那霉素浓度为50 μg/mL的LB液体培养基中,在37 ℃下以200 r/min的转速过夜培养,之后再以1%的接种量转接至50 mL、卡那霉素浓度为50 μg/mL、葡萄糖浓度为20 g/L的LBG液体培养基中,于37 ℃下培养至对数生长末期,待OD600达到0.8~1.0时,加入终浓度为0.2 mmol/L的IPTG并转移至30 ℃恒温摇床以200 r/min的转速发酵培养48 h,期间每隔6 h取出2 mL菌液,借助液相色谱仪检测其中的有机酸,分析条件如下:色谱柱为AcclaimTM120C18(4.6 mm×250 mm,5 μm);流动相为0.02 mol/L的KH2PO4,用H3PO4调节液体pH为2.8,进样量为20 μL,紫外检测波长为210 nm,流速为0.5 mL/min,柱温为25 ℃。
2 结果与分析
2.1 荧光蛋白的SDS-PAGE检测
荧光蛋白RIAD-sfGFP和RIDD-mCherry的SDS-PAGE检测结果如图2所示,其中,泳道1为流穿液,泳道2和泳道3为10 mmol/L的咪唑洗脱液,泳道4和泳道5为30 mmol/L的咪唑洗脱液,泳道6和泳道7为50 mmol/L的咪唑洗脱液,泳道8为100 mmol/L的咪唑洗脱液,泳道9为300 mmol/L的咪唑洗脱液,箭头指示目标蛋白条带。由图2(a)可知,荧光蛋白RIAD-sfGFP的诱导表达效果较好,当咪唑洗脱液浓度为30~100 mmol/L时(泳道4至泳道8),目标蛋白条带较单一,可以在此范围内收集蛋白;由图2(b)可知,荧光蛋白RIDD-mCherry的诱导表达效果也较好,在咪唑洗脱液浓度30~300 mmol/L范围内(泳道4至泳道9),均能获得较单一的目标蛋白条带,可在此范围内收集蛋白。
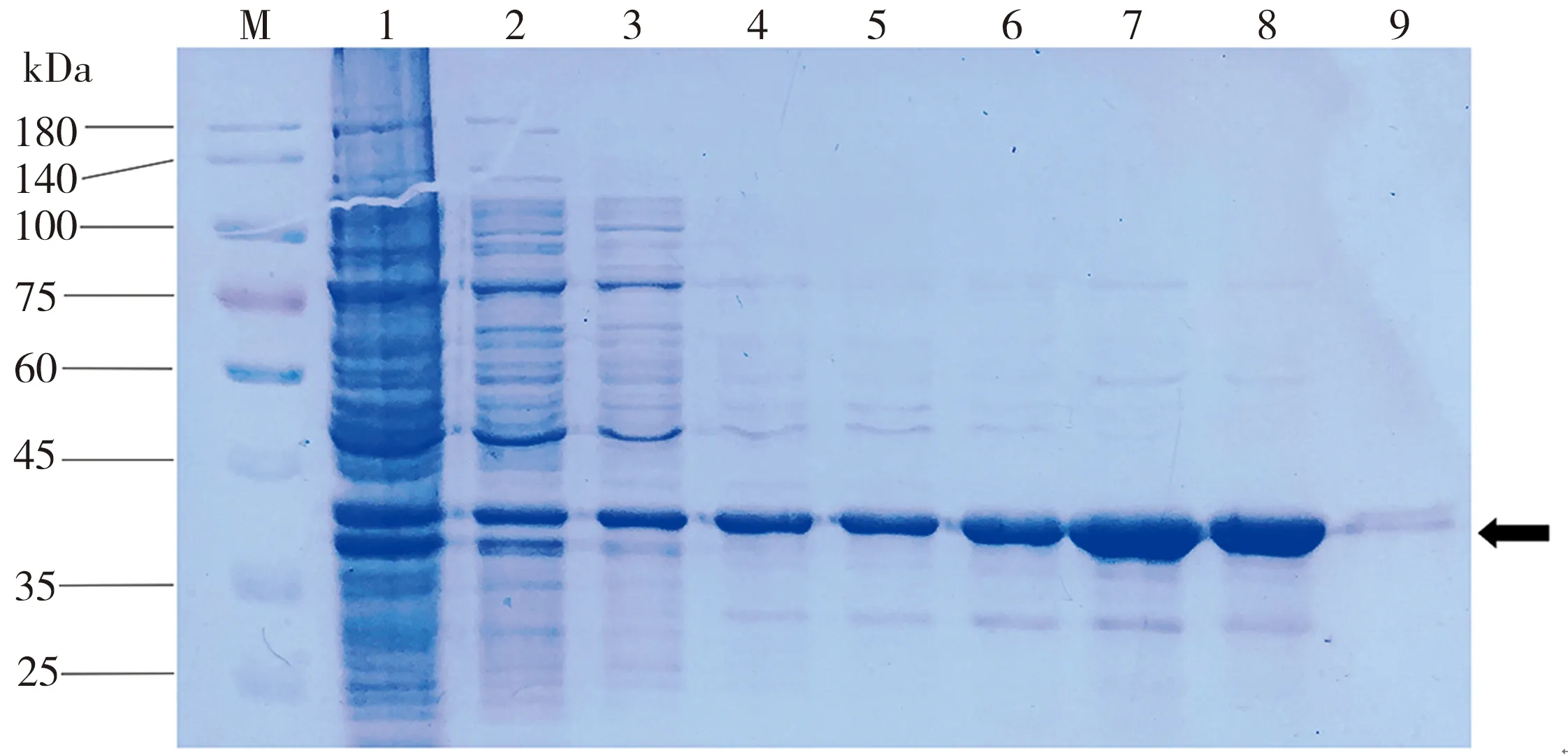
(a)RIAD-sfGFP
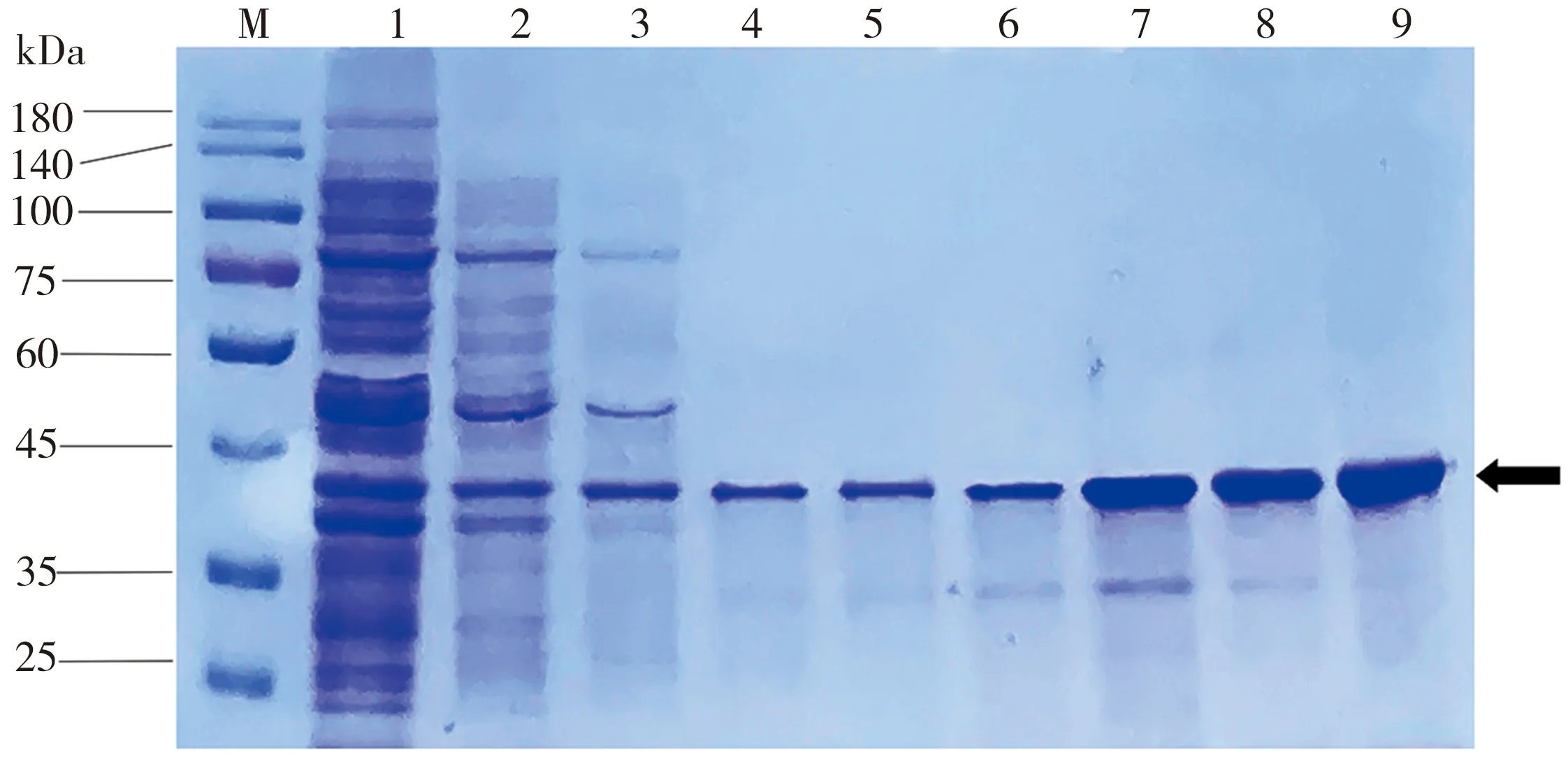
(b)RIDD-mCherry
2.2 蛋白支架功能完整性验证
将纯化的RIAD-sfGFP和RIDD-mCherry蛋白适当稀释,在25 ℃水浴锅中孵育30 min后分别取RIAD-sfGFP、 RIAD- mCherry以及二者的混合样品各600 μL于蓝光仪下观察,结果见图3。由图3可以看出,1号管中的RIDD-mCherry样品发出红色荧光,3号管中的RIAD-sfGFP样品发出绿色荧光,而2号管中RIDD-mCherry与RIAD-sfGFP的混合样品则发出橙色荧光,这表明,经体外孵育后,sfGFP和mCherry蛋白可通过蛋白支架RIAD和RIDD结合在一起,形成呈现橙色荧光的蛋白支架复合物RIAD-sfGFP-RIDD-mCherry。

图3 RIAD和RIDD的功能完整性验证
2.3 RIAD-sfGFP和RIDD-mCherry蛋白的互作验证
利用Native-PAGE检测RIAD-sfGFP蛋白和RIDD-mCherry的蛋白互作情况,结果如图4所示,其中泳道1为RIAD-sfGFP蛋白,泳道2为RIDD-mCherry蛋白,泳道3为二者复合体。从图4中可见,泳道2中RIDD-mCherry蛋白的尺寸增倍,这是因RIDD在近生理条件下极易形成二聚体所致。泳道3中RIAD-sfGFP蛋白与RIDD-mCherry蛋白的混合孵育物在Native-PAGE上迁移最慢,表明二者形成了蛋白复合体,这也再次证明RIAD-sfGFP蛋白和RIDD-mCherry蛋白确实存在相互作用。

图4 RIAD-sfGFP和RIDD-mCherry蛋白的Native-PAGE检测
2.4 重组载体的PCR鉴定
图5所示为重组载体的PCR检测结果。在图5(a)中,泳道1和泳道2分别以PRSFDuet-Pykf和PRSFDuet-Pykf-RIAD载体为模板,所用引物均为Duet-UP-1和Duet-Down-1;在图5(b)中,泳道1和泳道2均以载体PRSFDuet-Pykf-maeB为模板,所用引物分别为Duet-UP-1、T7 Terminator Primer和 DuetUP2 Primer、Terminator Primer,泳道3和泳道4均以载体PRSFDuet-Pykf-RIDD-maeB为模板,所用引物分别为Duet-UP-1、T7 Terminator Primer和 DuetUP2 Primer、Terminator Primer;在图5(c)中,泳道1和泳道2均以载体PRSFDuet-Pykf-RIAD-maeB为模板,所用引物分别为Duet-UP-1、T7 Terminator Primer和 DuetUP2 Primer、Terminator Primer,泳道3和泳道4均以载体PRSFDuet-Pykf-RIAD-RIDD-maeB为模板,所用引物分别为Duet-UP-1、T7 Terminator Primer和DuetUP2 Primer、Terminator Primer PCR。根据PCR检测结果可知,所有扩增的片段大小均符合预期,表明相应的目的基因已成功克隆至PRSFDuet-1载体,经测序验证全部正确。

(a)PRSFDuet-Pykf及PRSFDuet-Pykf-RIAD (b)PRSFDuet-Pykf-maeB及PRSFDuet-Pykf-RIDD-maeB (c)PRSFDuet-Pykf-RIAD-maeB及PRSFDuet-Pykf-RIAD-RIDD-maeB
2.5 重组蛋白诱导条件优化
在不同温度下诱导后,重组蛋白表达情况的检测结果如图6所示,其中,图6(a)中的泳道1和泳道2分别为重组蛋白于18 ℃诱导所得全菌体及破菌上清液,泳道3和泳道4分别为重组蛋白于28 ℃诱导所得全菌体及破菌上清液,图6(b)中的泳道1为未经诱导的全菌体,泳道2和泳道3分别为重组蛋白于30 ℃诱导所得全菌体及破菌上清液,泳道4和泳道5分别为重组蛋白于37 ℃诱导所得全菌体及破菌上清液,箭头指示目标蛋白条带。由检测结果可知,重组蛋白于18 ℃或30 ℃下诱导后的表达量较好,考虑到重组菌株MG3后期发酵培养对菌体生长速度及产酸的影响,最终确定重组蛋白诱导温度为30 ℃。

(a)18、28 ℃ (b)30、37 ℃
经不同浓度的诱导剂IPTG诱导后,重组蛋白表达情况的检测结果如图7所示,其中泳道1为未经诱导的全菌体,泳道2和泳道3分别为重组蛋白经0.2 mmol/L的IPTG诱导所得全菌体及破菌上清液,泳道4和泳道5分别为重组蛋白经0.4 mmol/L的IPTG诱导所得全菌体及破菌上清液,泳道6和泳道7分别为重组蛋白经0.6 mmol/L的IPTG诱导所得全菌体及破菌上清液,泳道8和泳道9分别为重组蛋白经0.8 mmol/L的IPTG诱导所得全菌体及破菌上清液,泳道10和泳道11分别为重组蛋白经1.0 mmol/L的IPTG诱导所得全菌体及破菌上清液,泳道12和泳道13分别为重组蛋白经1.2 mmol/L的IPTG诱导所得全菌体及破菌上清液,箭头指示目标蛋白条带。由检测结果可知,目的蛋白表达量随着IPTG浓度的提高而逐渐减少,考虑到高浓度的IPTG会毒害菌体,而采用较低浓度的IPTG能适当延长转录过程,有利于可溶性蛋白的表达[18],所以最终确定IPTG的浓度为0.2 mmol/L。

图8 重组蛋白Pykf-RIAD和RIDD-maeB的Native-PAGE检测
2.6 重组蛋白Pykf-RIAD和RIDD-maeB的互作验证
利用Native-PAGE检测重组蛋白Pykf-RIAD和RIDD-maeB的互作情况,结果如图8所示,其中,泳道1为经过纯化处理的Pykf-RIAD-RIDD-maeB蛋白样品,泳道2为流穿液经煮样处理后的蛋白样品,箭头指示目标蛋白条带。由图8可见,泳道2中的流穿液经高温处理后,蛋白支架间的结合作用解除,形成两条单一的条带分别对应RIDD-maeB和Pykf-RIAD蛋白,通过与泳道1中的Pykf-RIAD-RIDD-maeB蛋白复合物检测结果对比可知,重组蛋白maeB和Pykf在胞内表达后,能够通过蛋白支架RIDD和RIAD发生互作,形成Pykf-RIAD-RIDD-maeB蛋白复合物。
2.7 工程菌株发酵及产物检测
菌株MG1、MG2和MG3的发酵生长情况及产酸量检测结果分别如图9和图10所示,其中MG1为转入空载体PRSFDuet-1的大肠杆菌 MG1655(DE3)菌株,MG2为转入载体PRSFDuet-Pykf-maeB的大肠杆菌 MG1655(DE3)菌株,MG3为转入载体PRSFDuet-Pykf-RIAD-RIDD-maeB的大肠杆菌 MG1655菌株。从图9中可以看出,三组菌株的生长速度以MG3为最快,MG2次之,MG1最慢且与前两者差距较大。由图10可见,经过48 h摇瓶发酵,MG1菌株的L-苹果酸积累量极低,MG2菌株的L-苹果酸积累量也仅为0.4886 g/L,而MG3菌株的相应值则高达2.4012 g/L,同时,其丙酮酸积累量也达到了1.1342 g/L,明显高于菌株MG2(0.6409 g/L)与MG1(0.1290 g/L)的相应值,检测结果表明,借助蛋白支架实现Pykf和maeB共定位后,所形成的底物通道可有效促进PEP转化为丙酮酸,胞内丙酮酸不断积累有助于实现苹果酸酶的逆向催化,从而大幅提高了L-苹果酸的产量。
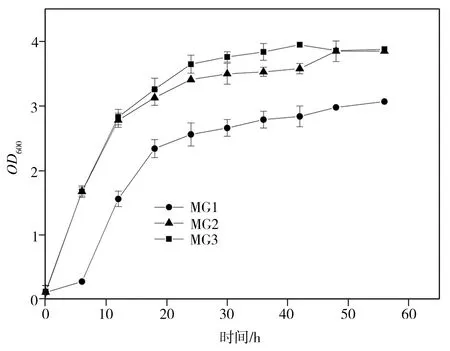
图9 菌株的生长曲线

图10 发酵液中的有机酸检测
2.8 单一支架标签对工程菌株发酵产酸的影响
构建重组菌株MG4(PRSFDuet-Pykf-RIAD-maeB)和MG5(PRSFDuet-Pykf-RIDD-maeB),以MG2(PRSFDuet-Pykf-maeB)为对照菌株,对三组菌株进行发酵培养,相应的生长情况及发酵48 h的产酸量检测结果如图11所示。由图11可见,三组菌株的生长情况及苹果酸产量差别不大,表明单一蛋白支架标签对菌株生长速度和产酸的影响较小,这也再次证实,MG3菌株快速生长且L-苹果酸产量大幅提高是Pykf和maeB共定位的结果,得益于蛋白支架RIAD、RIDD的相互作用。
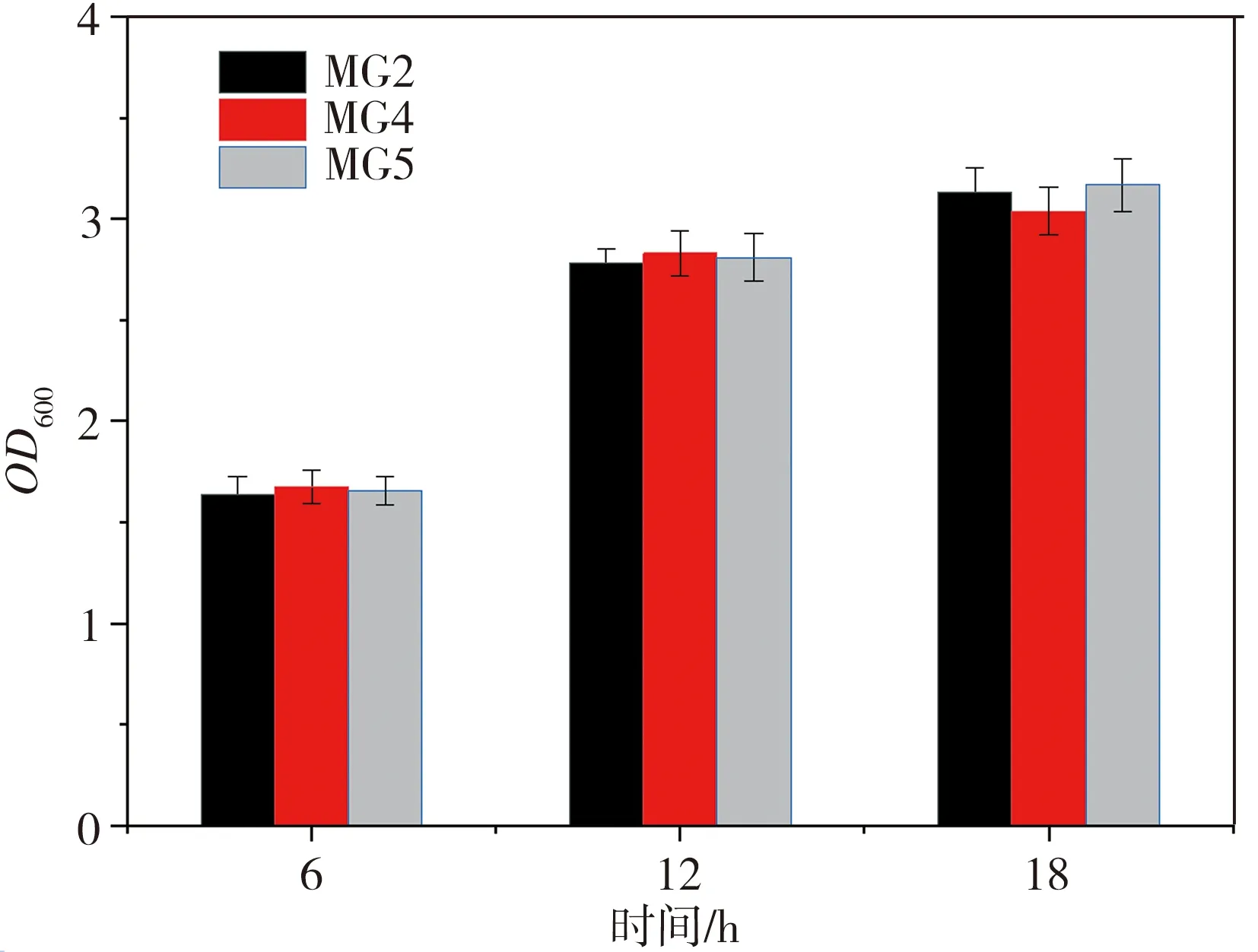
(a)菌株的生长
3 结语
为了在L-苹果酸与丙酮酸的可逆转化中逆向催化丙酮酸转化成L-苹果酸,进而积累目标产物L-苹果酸,本研究采用RIAD和RIDD蛋白支架将L-苹果酸合成途径中的关键酶Pykf和maeB锚定在一起,形成底物通道以增加丙酮酸的积累,促使苹果酸酶催化更多的丙酮酸转化成L-苹果酸。实际研究发现,利用蛋白支架RIAD和RIDD将Pykf与maeB共定位(Pykf-RIAD-RIDD-maeB)形成复合物并导入大肠杆菌MG1655后,所得重组菌株生长速度明显加快、L-苹果酸积累量大幅提高,这应归因于积累的丙酮酸直接被引往生成L-苹果酸的方向,减少了副产物如乙酸、乳酸等的产量,避免过多地消耗胞内还原力,从而使菌株生长速度加快,同时,苹果酸酶在催化丙酮酸转化为L-苹果酸时还可能伴随着NAD(P)H的生成,蛋白支架的引入使该反应过程加快,因此在胞内产生更多的NAD(P)H,进一步导致菌株生长加速,在菌株生长加速以及蛋白支架将酶共定位加速催化逆向反应的双重作用下,重组菌株的L-苹果酸产量大幅增加,这对于构建高产量的L-苹果酸工程菌株具有一定的参考价值。
