不同筛选方法的低密度SNP集合填充准确性比较
2023-05-15林雨浓王泽昭陈燕朱波高雪张路培高会江徐凌洋蔡文涛李英豪李俊雅高树新
林雨浓,王泽昭,陈燕,朱波,高雪,张路培,高会江, 徐凌洋,蔡文涛,李英豪,李俊雅,高树新
不同筛选方法的低密度SNP集合填充准确性比较

1内蒙古民族大学动物科技学院,内蒙古通辽 028042;2中国农业科学院北京畜牧兽医研究所,北京 100193;3通辽京缘种牛繁育有限责任公司,内蒙古通辽 028006
【目的】尝试通过在华西牛参考群高密度标记芯片位点中,使用两种标记筛选方法挑选具有代表性的且密度梯度不同的SNP位点集合,后利用基因组填充策略在相同填充参数下将低密度芯片数据填充至高密度继而进行后续基因组研究,从而达到降低华西牛基因型分型成本的目的。研究分别比较了不同标记集合填充准确性和填充一致性的差异,阐述了标记筛选方法、标记密度、最小等位基因频率和参考群体数量等4个因素对填充结果的影响,为华西牛低密度SNP填充芯片设计提供参考。【方法】将质控后剩余的1 233头华西牛群体随机分为参考群(986头)和验证群(247头)。使用等间距法(equidistance,EQ)和高MAF法(high MAF,HM)两种标记筛选方法分别从华西牛参考群体的Illumina Bovine HD 芯片位点集合中筛选出16种不同密度的SNP集合,共生成32种不同SNP梯度密度集合。随后在验证群体中利用Beagle(v5.1)软件将各低密度集合填充至770 k密度水平,计算填充准确性和填充一致性并对填充性能影响因素进行分析。【结果】32种低密度SNP集合的标记数量在100—16 000之间,窗口最大为24 176 kb,最小151 kb。随着标记密度升高,EQ和HM两种筛选方法填充一致性和准确性不断提升,但填充准确性和填充一致性增加的幅度越来越小。当标记集合密度超过12 k后均趋于平稳。SNP密度在16 k时两种方法的填充准确性达到最高(=0.8801,=0.8696)。当标记密度低于11 k时,不同标记密度梯度下HM方法填充一致性均高于EQ方法。然而当SNP集合密度超过11 k时,EQ筛选方法较表现出填充优势。与填充一致性结果趋势相似,在SNP集合密度低于10 k时,HM方法仍然具有较高的填充准确性,但当SNP集合密度高于10 k时,EQ方法的填充准确性则较高,且在SNP密度集合大于12 k后,EQ填充准确性趋于稳定。同时研究发现与低MAF标记位点相比,高MAF位点的填充准确性更高。填充过程中发现,填充一致性和填充准确性随着参考群体增大而提高。当参考群体数量在600—800时,位点填充准确性和一致性较高。【结论】在华西牛群体中,填充一致性和填充准确性随标记密度递增而上升,在标记密度为10 k—12 k区间,可获得较好的填充效果。当标记密度小于10 k时优先选择HM方法,更高密度时EQ方法较好。高MAF标记位点填充准确性更高。采用填充策略进行低密度标记填充时,参考群体数量在400头以上时填充效果较为理想。
填充准确性;低密度SNP芯片;华西牛;连锁不平衡;最小等位基因频率
0 引言
【研究意义】基因组选择(genomic selection,GS)作为畜禽育种的重要技术手段备受关注[1]。高密度分型带来的高成本是目前限制基因组选择技术在肉牛育种中推广应用的关键因素之一。通过基因型填充技术,使用较低分型成本获得的高密度基因型数据将会对肉牛育种及相关科研工作奠定基础。【前人研究进展】高密度SNP芯片对解析重要经济性状的群体遗传学分析、基因定位、基因组选择有着重要的意义。2007年12月,Illumina公司推出了Infinium BovineSNP50 BeadChip(BovineSNP50)商业芯片并在奶牛育种中得到了广泛应用[2]。但HAYES等指出在跨品种评估时只有当标记密度小于10 kb时,才可能在不同品种牛群中检测到相同LD分型[3]。BovineSNP50 芯片由于密度较低存在较大使用局限[4]。为了解决标记密度问题,Illumina公司在2010年1月推出了首款牛高密度芯片(BovineHD Genotyping BeadChip,BovineHD)[5]。CARVALHEIRO等的研究证实,相对于50k或更低密度的芯片,高密度SNP芯片可以通过连锁不平衡(linkage disequilibrium , LD)捕获更多的变异信息,从而获得准确性更高的基因组估计育种值[6]。VANRADEN等研究结果同样证实利用高密度SNP信息在进行GWAS关联分析时,可以得到更加准确的定位结果[7]。尽管随着当前生物技术快速发展,高密度SNP芯片的价格在不断降低,但大范围使用高密度SNP芯片带来的成本问题仍然限制了基因组育种技术的广泛应用。2003年,LI等提出基因组填充方法,获得了个体未被分型位点的基因型数据[8]。通过先期使用低密度SNP芯片对个体进行分型,随后使用填充技术将低密度数据填充到高密度水平为降低分型成本提供了一种可行的思路[9]。相关研究也在猪[10]、马[11]、鸡[12]等畜种中展开。【本研究切入点】增加标记密度可以提升育种值估计的准确性。在华西牛群体内针对填充准确性的研究鲜有报道。本研究利用填充策略将低密度芯片数据填充至高密度继而进行后续基因组研究,基于该设想,使用等间距法(equidistance, EQ)和高MAF法(high MAF , HM)两种筛选方法构建出不同梯度密度SNP集合。比较各集合填充性能的差异,探究筛选方法、标记密度、MAF和参考群体数量对填充准确性的影响。【拟解决的关键问题】针对华西牛群体,比较各集合填充性能的差异,探究筛选方法、标记密度、MAF和参考群体数量对填充准确性的影响。确定低密度SNP集合筛选最优参数组合,最终目的是实现填充后效果与Illumina Bovine HD芯片分型结果一致,为华西牛低密度芯片设计提供依据。
1 材料与方法
1.1 试验群体及SNP分型
本研究所使用的数据材料均来自中国农业科学院北京畜牧兽医研究所牛遗传育种创新团队组建的华西牛参考群体。该群体组建于2007年,群体分布于内蒙古锡林郭勒盟乌拉盖管理区。经过逐渐扩群,至2020年该群体共包含华西牛1 478头,每头均有87个表型测定记录、系谱记录及770 k高密度芯片分型数据。每年7—8月份,对基础群母牛及犊牛进行基础数据测定,10月份将育肥犊牛集中转运至北京或围场育肥场进行集中育肥,饲料配方保持一致。集中育肥期间每3个月全群测定一次体重和体尺数据。集中育肥6个月时,静脉采血20 mL,并取2 mL血液进行DNA提取工作,使用Illumina Bovine HD BeadChip芯片进行基因分型。
1.2 数据质控
使用PLINK v1.90[13]软件对常染色体基因型数据进行质量控制,质控顺序为先位点后个体。具体质控标准如下:一是删除基因分型检出率小于90%,最小等位基因频率小于0.01和哈代温伯格平衡检验值小于1×10﹣6的SNP位点;二是删除SNP缺失率大于10%的个体。质控后获得1 233头个体和671 164个SNP标记用于后续研究。
1.3 构建SNP集合
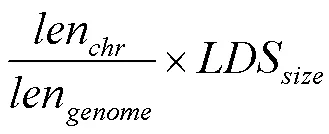
1.4 基因型填充及填充准确性评估
使用Beagle(v5.1)软件对32个SNP集合进行填充[16]。Beagle的填充程序分两阶段进行,首先对所有待填充SNP集合进行基因定相[17],随后进行基因型填充。所有SNP集合的定相和填充均采用Beagle的默认参数进行。
基因型填充时随机选择总群体20%的个体(247头)作为验证群体,剩余80%的个体(986头)作为填充参考群体。在计算参考群体大小对填充准确性的影响时,验证群体大小为总群的20%,在剩余个体中分别抽取100、200、400、600和800头组成参考群体,计算标记密度在7、10、13和16k时的填充结果。
采用填充一致性(concordance rate, CR)和填充准确性(imputation accuracy,2)[18]衡量基因型填充效果。其中填充一致性定义为验证群体中正确填充的位点数目与全部填充的位点数目的比值[19];填充准确性为填充位点的基因型(012编码)和真实基因型间的相关系数平方[20]。
2 结果
2.1 不同标记密度SNP集合筛选
使用EQ和HM两种位点筛选方法,在16组密度梯度中筛选出32个SNP集合。不同SNP集合包含的位点数量及窗口大小见表1。SNP集合中窗口最大为24 176 kb,最小为151 kb。
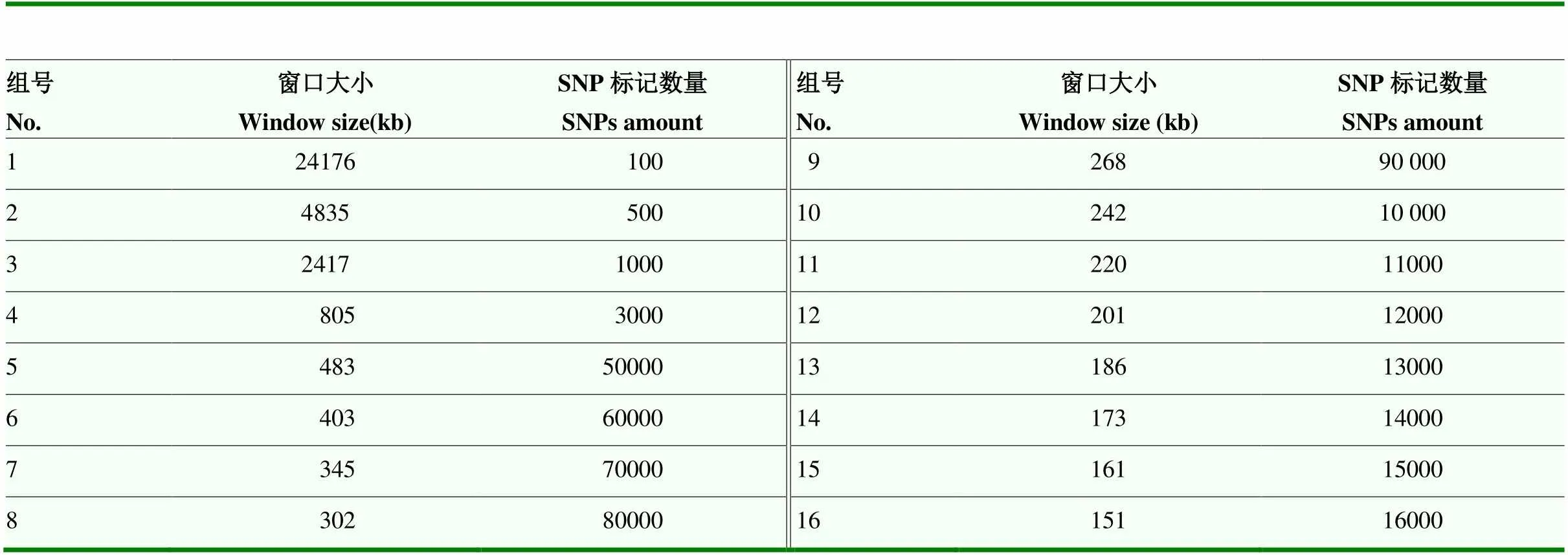
表1 不同密度集合窗口大小及SNP数量
2.2 标记密度对填充效果的影响
图1展示了EQ和HM方法筛选出的SNP集合的填充一致性和填充准确性。如图所示,EQ的填充一致性范围为0.5701—0.8863,HM的填充一致性范围为0.5743—0.8872。填充准确性的范围与填充一致性相似,EQ方法变化范围为0.0304—0.8801,HM方法变化范围为0.0393—0.8696。综合来看,随着SNP集合密度越来越大,填充一致性和填充准确性也随之增加,但SNP集合密度越高,填充准确性和填充一致性增加的幅度越来越小。SNP集合密度超过12 k后填充准确性和填充一致性变化趋于稳定,以EQ方法为例,从12 k到16 k增加了1.3%(填充一致性)和1.92%(填充准确性)。不同密度SNP集合的填充准确性和填充一致性结果见附表1。
2.3 标记筛选方法对填充效果的影响
如图1所示,EQ和HM两种方法的填充一致性和填充准确性在不同密度时存在差异。标记密度在11 k以下时HM方法具有较高的填充一致性和填充准确性,在标记密度大于11 k时EQ方法略好于HM方法。与填充一致性结果趋势相似,在SNP集合密度低于10 k时,HM方法仍然具有较高的填充准确性,但当SNP集合密度高于10 k时,EQ方法的填充准确性则较高,且在SNP密度集合大于12 k后,EQ填充准确性趋于稳定。
2.4 最小等位基因频率对填充效果的影响
填充准确性和填充一致性均与最小等位基因频率有关。图2中展示了标记密度为16 k时,EQ和HM筛选方法中填充一致性和填充准确性与标记MAF的分布。当MAF<0.1时,填充准确性较低但填充一致性较高。随着标记自身MAF的增加,在EQ筛选方法中,平均填充一致性平均值由0.9632下降到0.8276,填充准确性平均值由0.8533上升到0.8890;在HM筛选方法中,填充一致性平均值由0.9584下降到0.8508,填充准确性平均值由0.8220上升到0.8976(图3)。EQ和HM方法不同MAF区间填充一致性均值和填充准确性均值见附表2。
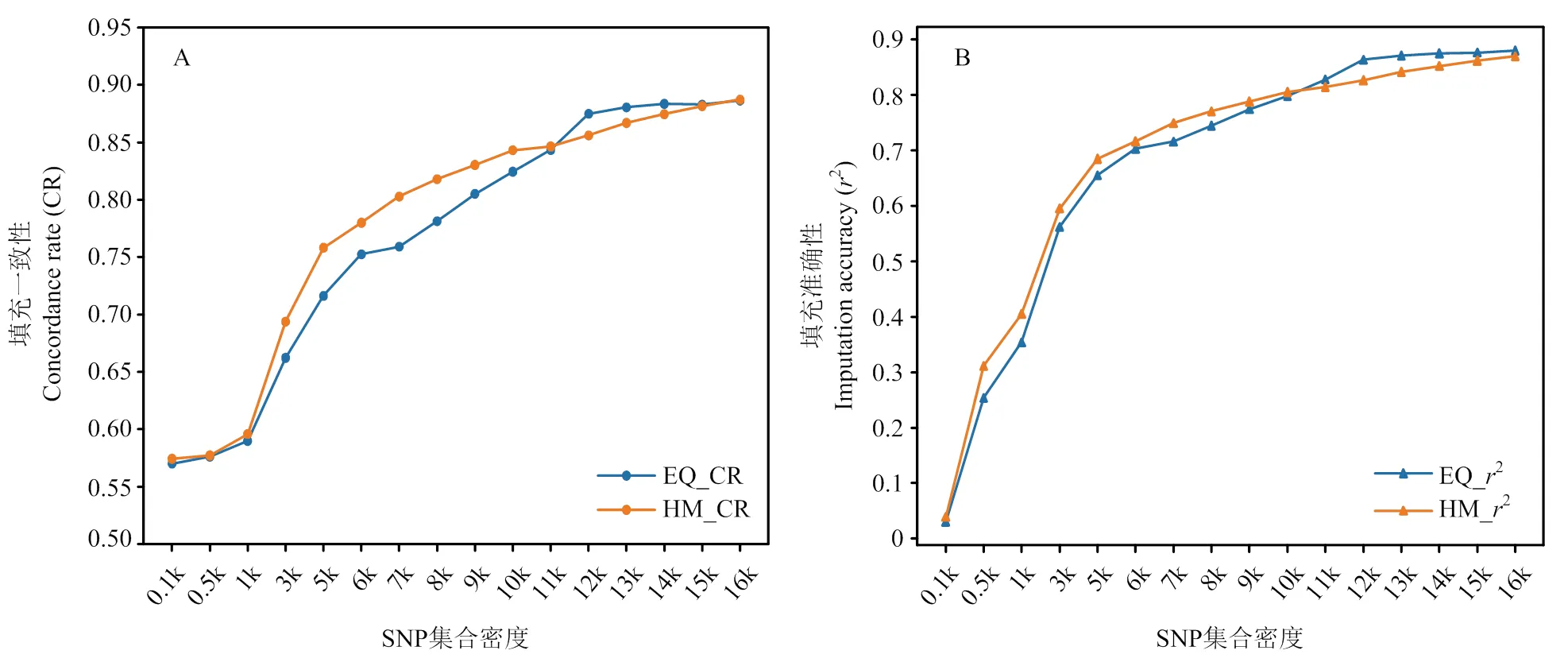
图1 不同筛选方法的填充一致性(A)和填充准确性(B)
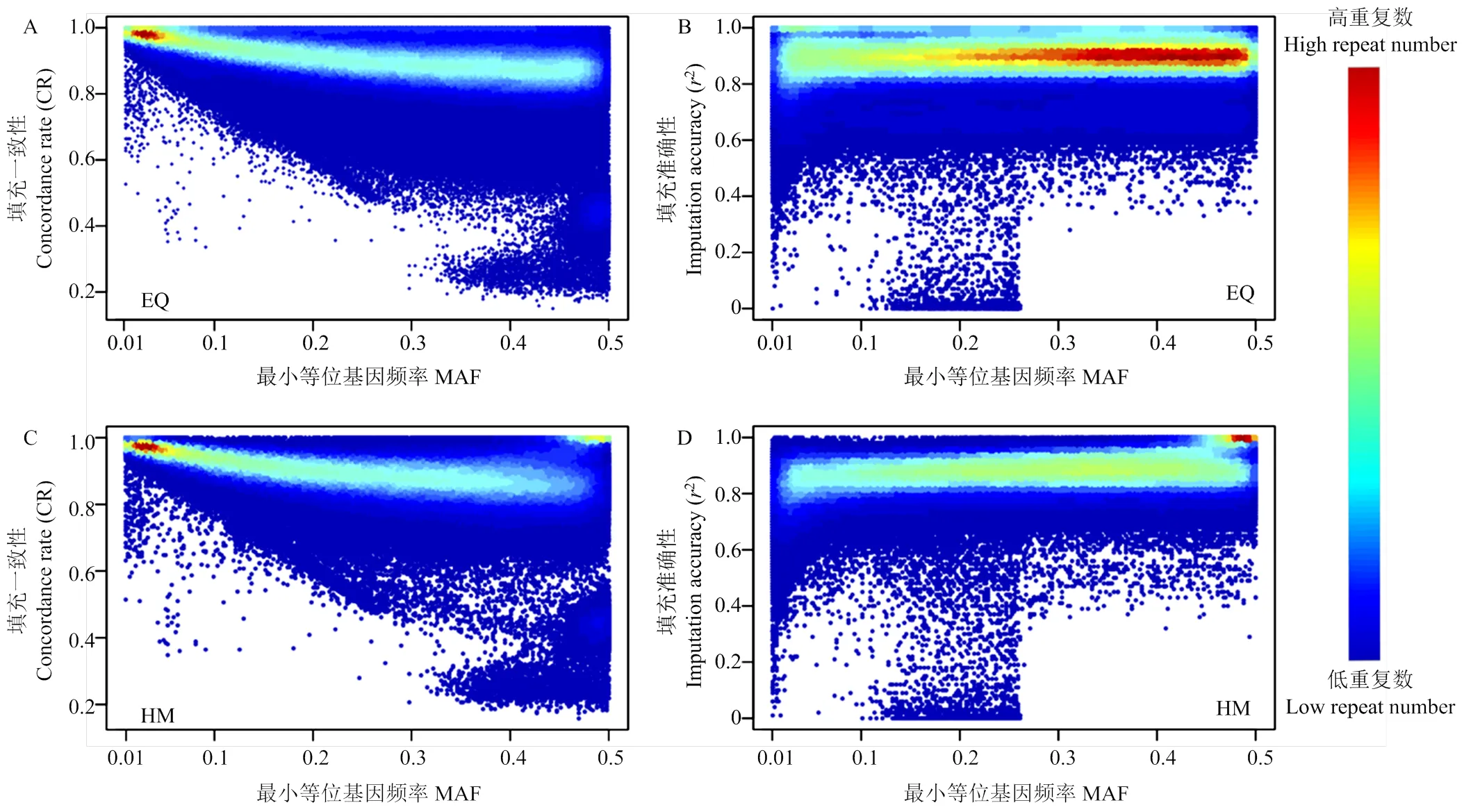
A:EQ筛选方法的填充一致性;B:EQ筛选方法的填充准确性;C:HM筛选方法的填充一致性;D:HM筛选方法的填充准确性。红色代表相同结果的位点数量多,蓝色代表相同结果的位点数量少
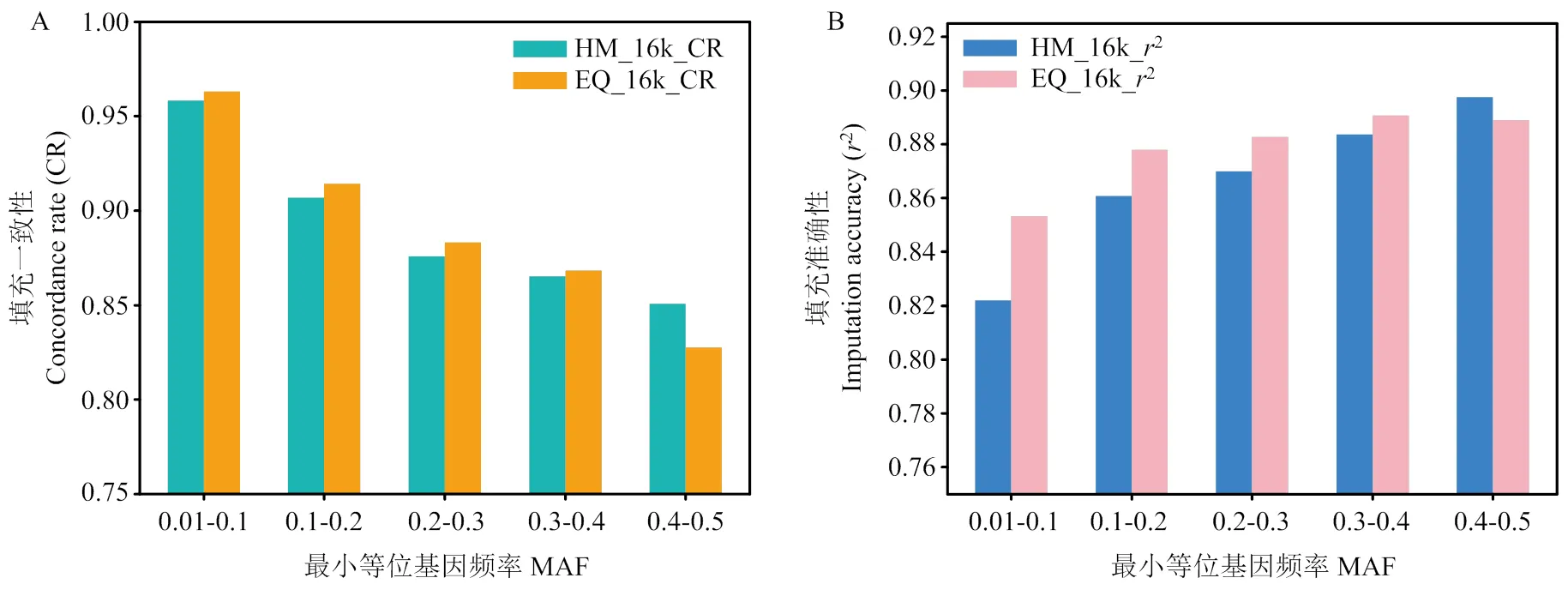
图3 密度为16 k时两种筛选方法的填充一致性平均值(A)和填充准确性平均值(B)与最小等位基因频率
2.5 参考群体对填充效果的影响
分别计算了标记密度在7、10、13和16 k时,参考群体大小分别为100、200、400、600和800时的填充一致性和准确性(图4)。结果发现,填充一致性和填充准确性随着参考群体增大而提高。以EQ筛选方法16 k密度为例,随着参考群体数量增加,相邻两个梯度之间的填充一致性增长率为:6.91%(100—200)、5.79%(200—400)、2.45%(400—600)和1.07%(600—800)。填充准确性增长率为:7.19%(100—200)、5.52%(200—400)、2.26%(400—600)和0.97%(600—800)。当参考群体数量在100—400头时,填充一致性和准确性的增速最快。当参考群体数量超过400时,填充一致性和填充准确性提升速度开始降低。当参考群体规模为800时,不同密度SNP集合填充一致性和填充准确性达到最高,填充一致性范围为0.7511(7 k)到0.8797(16 k)、填充准确性范围为0.7492(7 k)到0.8750(16 k),见附表3。
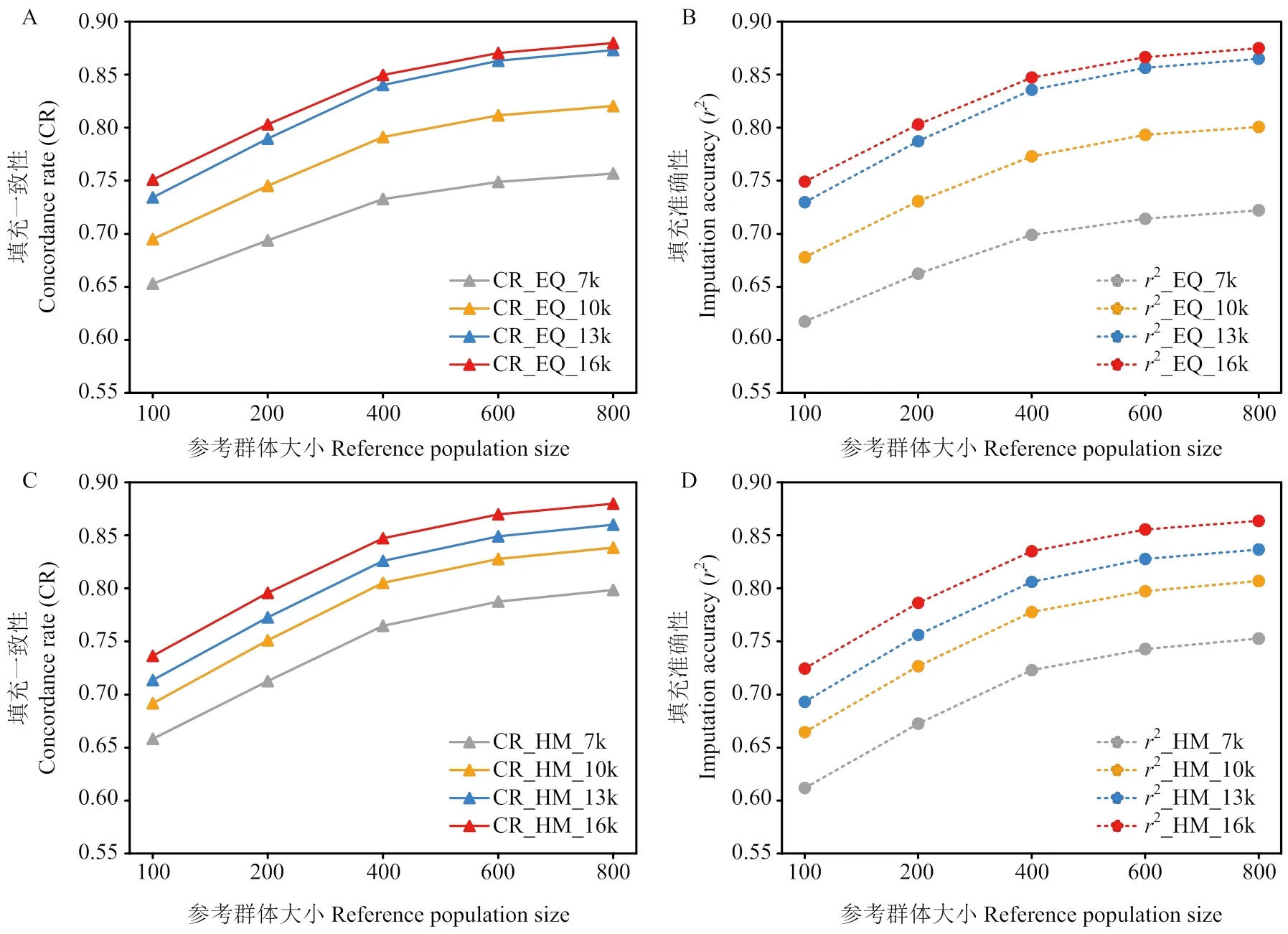
图4 参考群体大小对填充准确性和填充一致性的影响
3 讨论
3.1 标记密度对填充的影响
本研究目的是探究华西牛群体低密度SNP集合填充到770 k密度的填充效果。发现,低密度SNP集合的填充一致性和准确性均随标记密度增加而升高。在不同密度下SNP集合的填充一致性的提升趋势有显著差别,呈现出慢-快-慢的增长曲线。以EQ方法填充一致性为例,在密度较低时提升缓慢,从0.1 k到1 k仅提升3.51%。密度在1 k至12 k时提升较快,增长了48.31%。密度大于12 k后提升较慢,12 k到16 k仅提升1.30%。CALUS等报道,随着SNP芯片中标记不断增加,对应的参考单倍型将更加容易被识别出来,容易获得更高的填充准度[21]。因此可以推断,SNP集合密度对于填充效果的影响,本质上是基因组上单倍型的分布情况导致的。随着SNP标记密度增加,落在单倍型上的概率变大,连锁出附近其他位点的能力增强,因此填充准确性和一致性变高。
在本研究中,当集合密度大于12 k时,EQ和HM筛选方法的填充准确性和一致性均趋于稳定,因此推断在华西牛群体中最佳SNP填充密度标记在12 k左右。相关研究指出最佳SNP填充密度可能与基因组大小有关。WENG 等报道,在荷斯坦奶牛中,使用标记密度为7 k的芯片进行分型后,通过填充就可以获得较高填充准性的高密度SNP集合[22]。WANG等在关于鸡的研究中发现[23],同种群体中包含约400个均匀分布的标记,使用低密度芯片进行GEBV估计的准度相比于高密度芯片仅损失了6%。WELLMANN研究发现,在猪中使用含有3 000个位点的低密度芯片估算的GEBV,与密度为60 k的 SNP芯片估计得到的GEBV结果具有高度相关[24]。因此,在后续研究中可以继续探索在此密度上填充到高密度水平后的估计育种值准确性。
3.2 均匀分布对于填充的影响
当标记密度低于10 k时,HM方法具有较高的填充一致性和填充准确性。推断在标记密度低时,高MAF位点与周围QTL之间处于连锁不平衡状态的概率更高。此密度下使用EQ方法构建低密度SNP集合的填充准确性和一致性均较低。这是由于标记密度过低,相邻位点之间的连锁情况较差,从而使Beagle软件利用位点间的连锁进行填充的算法不能发挥出最优作用。当标记密度高于10 k时,EQ策略优于HM策略,其原因是等间距的筛选位点可以在位点数量有限的情况下,最大限度的利用分布在整个基因组上的遗传信息,因而相较于HM方法更具有优势。
3.3 最小等位基因频率对于填充的影响
本研究结果显示,随着标记自身MAF增加,填充一致性均值逐渐下降,填充准确性均值逐渐增加。同时观察到,在MAF小于0.1时,填充准确性较低。关于MAF对填充准确性的影响,在不同文章存在完全相反的观察结果,但均未深入展开讨论。Didier等针对Illuminate BovineLD芯片的研究结果表明,在多数肉牛品种中SNP位点的MAF高于0.3,在该情况下观察到高MAF位点有助于提高填充的准确性[14]。HERRY等同样利用等间距法挑选SNP位点用于填充,同样发现,低MAF的SNP位点填充错误率更高[25]。这可能是由于低MAF位点导致该位点的分型错误率提升,进而导致的填充错误率提高[26]。
但同样有研究报道,随着位点MAF的提升,填充准确性降低或对填充准确性没有影响。HOZÉ等在Montbéliarde品种的填充研究中,根据填充准确性将SNP分为两类。第一类是对于填充准确性大于0.9的SNP位点,没有观察到填充准确性与MAF存在相关关系。第二类对于填充准确性小于0.9的SNP位点,填充准确性随着MAF的增加而降低,他们猜测这可能与SNP位点的错误定位(miss mapping)有关,但未展开深入讨论[27]。罗汉鹏等使用Beagle 5.1和Minimac 3两款填充软件对荷斯坦奶牛基因组50 k芯片数据填充至150 k时,同样发现随着最小等位基因频率的增加,两款填充软件的填充一致性都表现较差,尤其平均最小等位基因频率在0.4—0.5时(位点约占待填充位点的29%),填充准确性相对较低[28]。上述两种观察结果与本研究相似。
3.4 参考群体对填充的影响
参考群体数量是影响填充效果的重要因素之一,在猪、牛、羊和鸡等不同物种类似研究中均有报道[21, 29-30]。本研究分别计算了在不同标记密度梯度下,参考群体数量分别为100、200、400、600和800时的填充一致性和填充准确性。
通过计算EQ方法16 k时参考群体数量对填充一致性的影响发现,当参考群体数量在100—400头时,填充一致性增长最快,分别提升了6.91%(100—200)和5.79%(200—400)。当参考群体的数量超过400时,填充一致性增速开始放缓,为2.45%(400—600)。当参考群体数量大于600时,填充一致性增长率仅为1.07%(600—800)。研究发现,参考群体对填充一致性和填充准确性的影响与密度因素的影响类似。当参考群体中华西牛数量不断增加时,填充一致性和填充准确性大幅提高,随着参考群体数量的增加,增速不断下降。上述结果表明,参考群体数量要维持在一个必要的大小,才能获得理想的低密度SNP芯片填充准确性。HOZÉ等报道,当参考群体数量小于400时,参考群体数量是影响填充效果最主要的因素[27]。随着参考群体数量的增加,可参考的单倍型库的规模也随之增加,在参考单倍型库中发现候选单倍型片段的概率随之增加[31-32]。这与UEMOTO等[33]、VENTURA等[18]和CALUS等[21]的结果一致。
4 结论
本研究使用EQ和HM两种标记筛选方法,评估了华西牛群体不同密度SNP集合填充到770 k的填充结果。研究发现填充一致性(CR)和填充准确性(2)受标记密度、最小等位基因频率、参考群体大小影响。填充一致性和填充准确性随密度递增而上升,在10—12 k区间内较适合用于填充,密度小于10 k时HM方法较好,密度大于10 k时EQ方法较好。低MAF位点对填充准确性的影响比较大。参考群体中个体数超过400时填充效果较好。
[1] 朱波, 王延晖, 牛红, 陈燕, 张路培, 高会江, 高雪, 李俊雅, 孙少华. 畜禽基因组选择中贝叶斯方法及其参数优化策略. 中国农业科学, 2014, 47(22): 4495-4505. doi:10.3864/j.issn.0578-1752.2014. 22.015.
ZHU B, WANG Y H, NIU H, CHEN Y, ZHANG L P, GAO H J, GAO X, LI J Y, SUN S H. The strategy of parameter optimization of Bayesian methods for genomic selection in livestock. Scientia Agricultura Sinica, 2014, 47(22): 4495-4505. doi:10.3864/j.issn.0578- 1752.2014.22.015. (in Chinese)
[2] VANRADEN P M, VAN TASSELL C P, WIGGANS G R, SONSTEGARD T S, SCHNABEL R D, TAYLOR J F, SCHENKEL F S. Invited Review: reliability of genomic predictions for North American Holstein bulls. Journal of Dairy Science, 2009, 92(1): 16-24.
[3] DE ROOS A P W, HAYES B J, SPELMAN R J, GODDARD M E. Linkage disequilibrium and persistence of phase in Holstein-Friesian, jersey and Angus cattle. Genetics, 2008, 179(3): 1503-1512.
[4] HAYES B J, BOWMAN P J, CHAMBERLAIN A C, VERBYLA K, GODDARD M E. Accuracy of genomic breeding values in multi- breed dairy cattle populations. Genetics, Selection, Evolution, 2009, 41: 51.
[5] MATUKUMALLI L K,SCHROEDER S,DENISE S, SONSTEGARD T, LAWLEY C T, GEORGES M. Analyzing LD blocks and CNV segments in cattle: Novel genomic features identified using the BovineHD BeadChip. 2011. www.scienceopen.com/document?vid= 0fb91f10-7679-4ec4- b5a9- ca39bd541f2e.
[6] CARVALHEIRO R, BOISON S A, NEVES H H R, SARGOLZAEI M, SCHENKEL F S, UTSUNOMIYA Y T, O'BRIEN A M P, SÖLKNER J, MCEWAN J C, VAN TASSELL C P, SONSTEGARD T S, GARCIA J F. Accuracy of genotype imputation in nelore cattle. Genetics, Selection, Evolution, 2014, 46: 69.
[7] VANRADEN P M, NULL D J, SARGOLZAEI M, WIGGANS G R, TOOKER M E, COLE J B, SONSTEGARD T S, CONNOR E E, WINTERS M, VAN KAAM J B C H M, VALENTINI A, VAN DOORMAAL B J, FAUST M A, DOAK G A. Genomic imputation and evaluation using high-density Holstein genotypes. Journal of Dairy Science, 2013, 96(1): 668-678.
[8] LI N, STEPHENS M. Modeling linkage disequilibrium and identifying recombination hotspots using single-nucleotide polymorphism data. Genetics, 2003, 165(4): 2213-2233.
[9] DRUET T, SCHROOTEN C, DE ROOS A P W. Imputation of genotypes from different single nucleotide polymorphism panels in dairy cattle. Journal of Dairy Science, 2010, 93(11): 5443-5454.
[10] GROSSI D A, BRITO L F, JAFARIKIA M, SCHENKEL F S, FENG Z. Genotype imputation from various low-density SNP panels and its impact on accuracy of genomic breeding values in pigs. Animal, 2018, 12(11): 2235-2245.
[11] CORBIN L J, KRANIS A, BLOTT S C, SWINBURNE J E, VAUDIN M, BISHOP S C, WOOLLIAMS J A. The utility of low-density genotyping for imputation in the Thoroughbred horse. Genetics, Selection, Evolution: GSE, 2014, 46(1): 9.
[12] YE S P, YUAN X L, LIN X R, GAO N, LUO Y Y, CHEN Z M, LI J Q, ZHANG X Q, ZHANG Z. Imputation from SNP chip to sequence: a case study in a Chinese indigenous chicken population. Journal of Animal Science and Biotechnology, 2018, 9: 30.
[13] CHANG C C, CHOW C C, TELLIER L C, VATTIKUTI S, PURCELL S M, LEE J J. Second-generation PLINK: rising to the challenge of larger and richer datasets. GigaScience, 2015, 4(1): s13742-15.
[14] BOICHARD D, CHUNG H, DASSONNEVILLE R, DAVID X, EGGEN A, FRITZ S, GIETZEN K J, HAYES B J, LAWLEY C T, SONSTEGARD T S, VAN TASSELL C P, VANRADEN P M, VIAUD-MARTINEZ K A, WIGGANS G R, CONSORTIUM B L. Design of a bovine low-density SNP array optimized for imputation. PLoS One, 2012, 7(3): e34130.
[15] BOLORMAA S, GORE K, VAN DER WERF J H J, HAYES B J, DAETWYLER H D. Design of a low-density SNP chip for the main Australian sheep breeds and its effect on imputation and genomic prediction accuracy. Animal Genetics, 2015, 46(5): 544-556.
[16] BROWNING B L, ZHOU Y, BROWNING S R. A one-penny imputed genome from next-generation reference panels. The American Journal of Human Genetics, 2018, 103(3): 338-348.
[17] MARCHINI J, HOWIE B. Genotype imputation for genome-wide association studies. Nature Reviews Genetics, 2010, 11(7): 499-511.
[18] VENTURA R V, MILLER S P, DODDS K G, AUVRAY B, LEE M, BIXLEY M, CLARKE S M, MCEWAN J C. Assessing accuracy of imputation using different SNP panel densities in a multi-breed sheep population. Genetics, Selection, Evolution: GSE, 2016, 48(1): 71.
[19] O’BRIEN A C, JUDGE M M, FAIR S, BERRY D P. High imputation accuracy from informative low-to-medium density single nucleotide polymorphism genotypes is achievable in sheep1. Journal of Animal Science, 2019, 97(4): 1550-1567.
[20] BROWNING S R, BROWNING B L. Rapid and accurate haplotype phasing and missing-data inference for whole-genome association studies by use of localized haplotype clustering. The American Journal of Human Genetics, 2007, 81(5): 1084-1097.
[21] CALUS M P L, BOUWMAN A C, HICKEY J M, VEERKAMP R F, MULDER H A. Evaluation of measures of correctness of genotype imputation in the context of genomic prediction: a review of livestock applications. Animal, 2014, 8(11): 1743-1753.
[22] WENG Z, ZHANG Z, ZHANG Q, FU W, HE S, DING X. Comparison of different imputation methods from low- to high-density panels using Chinese Holstein cattle. Animal, 2013, 7(5): 729-735.
[23] WANG C, HABIER D, PEIRIS B L, WOLC A, KRANIS A, WATSON K A, AVENDANO S, GARRICK D J, FERNANDO R L, LAMONT S J, DEKKERS J C M. Accuracy of genomic prediction using an evenly spaced, low-density single nucleotide polymorphism panel in broiler chickens. Poultry Science, 2013, 92(7): 1712-1723.
[24] WELLMANN R, PREUß S, THOLEN E, HEINKEL J, WIMMERS K, BENNEWITZ J. Genomic selection using low density marker panels with application to a sire line in pigs. Genetics, Selection, Evolution: GSE, 2013, 45(1): 28.
[25] HERRY F, HÉRAULT F, PICARD DRUET D, VARENNE A, BURLOT T, LE ROY P, ALLAIS S. Design of low density SNP chips for genotype imputation in layer chicken. BMC Genetics, 2018, 19(1): 108.
[26] YUAN M, FANG H Y, ZHANG H. Correcting for differential genotyping error in genetic association analysis. Journal of Human Genetics, 2013, 58(10): 657-666.
[27] HOZÉ C, FOUILLOUX M N, VENOT E, GUILLAUME F, DASSONNEVILLE R, FRITZ S, DUCROCQ V, PHOCAS F, BOICHARD D, CROISEAU P. High-density marker imputation accuracy in sixteen French cattle breeds. Genetics, Selection, Evolution, 2013, 45: 33.
[28] 罗汉鹏, 窦金焕, 安涛, 陈少侃, 王雅春. 基于荷斯坦牛群体基因组数据填充软件的准确性比较(Minimac 3与Beagle 5.1). 中国畜牧兽医, 2021, 48(5): 1664-1671.
LUO H P, DOU J H, AN T, CHEN S K, WANG Y C. Comparison of software (minimac 3 and beagle 5.1) for genomic imputation using Holstein cow population. China Animal Husbandry & Veterinary Medicine, 2021, 48(5): 1664-1671. (in Chinese)
[29] BOLORMAA S, CHAMBERLAIN A J, KHANSEFID M, STOTHARD P, SWAN A A, MASON B, PROWSE-WILKINS C P, DUIJVESTEIJN N, MOGHADDAR N, VAN DER WERF J H, DAETWYLER H D, MACLEOD I M. Accuracy of imputation to whole-genome sequence in sheep. Genetics, Selection, Evolution, 2019, 51(1): 1.
[30] HAYES B J, BOWMAN P J, DAETWYLER H D, KIJAS J W, VAN DER WERF J H J. Accuracy of genotype imputation in sheep breeds. Animal Genetics, 2012, 43(1): 72-80.
[31] VENTURA R V, LU D, SCHENKEL F S, WANG Z, LI C, MILLER S P. Impact of reference population on accuracy of imputation from 6K to 50K single nucleotide polymorphism chips in purebred and crossbreed beef cattle1. Journal of Animal Science, 2014, 92(4): 1433-1444.
[32] HEIDARITABAR M, CALUS M P L, VEREIJKEN A, GROENEN M A M, BASTIAANSEN J W M. Accuracy of imputation using the most common sires as reference population in layer chickens. BMC Genetics, 2015, 16: 101.
[33] UEMOTO Y, SASAKI S, SUGIMOTO Y, WATANABE T. Accuracy of high-density genotype imputation in Japanese Black cattle. Animal Genetics, 2015, 46(4): 388-394.
Comparison of Imputation Accuracy for Different Low-Density SNP Selection Strategies

1College of Animal Science and Technology, Inner Mongolia University for the Nationalities, Tongliao 028042, Inner Magnolia;2Institute of Animal Sciences, Chinese Academy of Agriculture Sciences, Beijing 100193;3Tongliao Jingyuan Breeding Cattle Breeding LLC, Tongliao 028006, Inner Magnolia
【Objective】To facilitate the low-cost genomic selection in Huaxi Cattle, the present study represented the first attempt to designed a new low-density Genotype chip to support imputation to higher density genotypes. The representative SNP markers with different density gradients were selected from high-density SNP chips in the Huaxi cattle reference population by using two SNP selection methods. And then, these marker sets were imputed to high-density sets with the same imputation parameters for subsequent genomic studies. Meanwhile, the current study compared the differences in imputation accuracy and concordance among SNP panels and illustrated the effects of four factors on imputation results, including marker screening method, marker density, minor allele frequency, and the number of reference population. This study could provide insights about the methods to select the low-density SNP markers for imputation in the current population and the representative SNPs, and aid in designing low-density SNP chip for Huaxi cattle.【Method】Totally 1,233 Huaxi cattle after genotypes filtered was randomly divided into reference (986) and validation (247) populations. , Based on Equidistance (EQ) and high MAF (HM), two SNP selection strategies were used to make 16 SNP sets with different densities from the Illumina Bovine HD chip in the reference population, respectively. Each of the 32 low-density set was then imputed to the 770K density level in the validation population by using Beagle (v5.1), while the imputation accuracy and concordance were calculated as the mean correlation between true and imputed genotypes. Finally, a comprehensive set of factors that influence the imputation performance were analyzed.【Result】The number of markers in the 32 low-density SNP sets ranged from 100 to 16 000, with a maximum window of 24 176 kb and a minimum window of 151 kb. The imputation accuracy and concordance of both EQ and HM methods went up with increasing marker densities. The imputation accuracy of both methods was the highest at 16k SNP density (=0.8801,=0.8696). When the marker density was below 11k, the imputation concordance of HM was higher than EQ for all marker density gradients. However, when the SNP density exceeded 11 k, EQ showed an imputation accuracy advantage over HM. Similar to the imputation concordance results, the HM method still had higher imputation accuracy when the SNP density was lower than 10 k, but the EQ method had higher imputation accuracy when the SNP pool density was higher than 10 k, and the EQ imputation accuracy tended to be stable after the SNP density was greater than 12 k. It was also found that the imputation accuracy of high MAF locus was higher. During the imputation process, it was found that the imputation accuracy and concordance increased with the increase of the reference panel. The imputation accuracy and concordance of loci were higher when the population of the reference panel was 600-800. 【Conclusion】In the Huaxi cattle population, the imputation accuracy and concordance increased with increasing marker density, and a better imputation effect could be obtained in the marker density of 10 k-12 k interval. The HM method was preferred when the marker density was less than 10 k, and the EQ method was better at high marker density. High MAF loci were more accurate for imputation. When the imputation strategy for low-density marker imputation was used, the number of reference panel should be at least 400 heads for better imputation effect.
imputation accuracy; low density SNP array; Chinese Simmental cattle; linkage disequilibrium; MAF
10.3864/j.issn.0578-1752.2023.08.013
2021-12-13;
2022-03-24
内蒙古自治区第五批“草原英才”工程产业创新创业人才团队专项、内蒙古自然科学基金面上项目(2019MS03077)、内蒙古自治区科技计划项目(KJXM2020002-05)、青年科学基金(32102505)
林雨浓,E-mail:Lin_Yunong@outlook.com。通信作者高树新,E-mail:shuxingao@126.com。通信作者李俊雅,E-mail:lijunya@caas.cn。
(责任编辑 林鉴非)
