玉米转录因子ZmEREB93负调控籽粒发育
2022-10-31庞浩婉傅乾坤杨青青张元元付凤玲于好强
庞浩婉,傅乾坤,杨青青,张元元,付凤玲,于好强
玉米转录因子ZmEREB93负调控籽粒发育
庞浩婉1,傅乾坤1,杨青青1,张元元2,付凤玲1,于好强1
1四川农业大学玉米研究所,成都 611130;2绵阳师范学院生命科学与技术学院,四川绵阳 621000
【目的】玉米作为重要的粮、经、饲多用作物,其产量的稳定对经济发展和粮食安全意义重大。AP2/EREBP(APETALA2/ ethylene response element binding protein,AP2/EREBP)转录因子在植物生长发育及逆境应答中发挥重要作用。前期研究发现,玉米ZmBES1/BZR1-5转录因子靶基因可能参与调控种子大小。克隆,并对其表达特性及功能进行分析,为深入解析其调控玉米籽粒发育的功能与机制奠定基础。【方法】从玉米自交系B73中克隆的全长序列,对其基因序列和编码氨基酸序列特征进行生物信息学分析。随后,通过实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)分析其组织表达模式,分别构建植物和酵母表达载体,进行亚细胞定位和转录激活活性分析。经农杆菌介导法将转入拟南芥,对转基因株系的种子表型进行分析。最后,通过体外染色质免疫共沉淀测序(chromatin immunoprecipitation sequencing,Chip-seq)和共表达分析筛选ZmEREB93可能调控的候选靶基因,并通过酵母单杂交(yeast one hybrid,Y1H)验证。【结果】成功克隆获得,序列分析结果表明,无内含子,开放阅读框长618 bp,编码205个氨基酸,有一个高度保守的AP2结构域,属于AP2家族的ERF亚类。qRT-PCR结果表明,在授粉后15和25 d的种子中表达量较高,其中,在25 d种子中表达量最高,约为15 d种子中表达量的11倍,在茎和根中有微量表达,在雄穗、花丝及苞叶中无表达。转录激活试验结果表明,ZmEREB93蛋白在酵母细胞中不具有转录激活活性。亚细胞定位结果显示,ZmEREB93蛋白定位于细胞核。与野生型株系相比,转基因拟南芥株系种子的长和宽显著变小且千粒重显著降低。体外Chip-seq与共表达分析结果表明,、、及为ZmEREB93转录因子的候选靶基因。Y1H试验表明,ZmEREB93蛋白可直接结合启动子。【结论】玉米ZmEREB93作为转录因子在种子中特异性表达,负调控种子大小。
玉米;转录因子;AP2/EREBP;籽粒
0 引言
【研究意义】玉米作为中国重要的粮、经、饲多用作物,其产量的稳定对中国经济发展和粮食安全意义重大。籽粒性状是决定玉米产量与品质的重要因素,除环境外,转录因子、激素等内部因子在籽粒发育中也发挥重要作用[1]。因此,挖掘玉米籽粒发育相关基因,可用于培育高产玉米新品种。【前人研究进展】转录因子指能够与基因启动子区域的顺式作用元件结合,进而激活或抑制靶基因转录的一类蛋白,在植物生长发育及逆境应答中起重要调控作用。植物中,转录因子被分为不同的类别,例如MYB、WRKY、bZIP以及AP2/EREBP等。其中,AP2/EREBP(APETALA2/ ethylene response element binding protein)作为植物中特有的一类转录因子,调控植物非生物胁迫响应、生长发育及免疫应答等途径[2]。1998年,JOFUKU等[3]率先从模式植物拟南芥中分离了第一个与花发育有关的AP2基因,编码蛋白含有2个AP2结构域。随后的研究中,根据AP2/ERF结构域数量及序列差异,AP2/EREBP家族成员被划分为AP2、RAV、ERF、Soloist等4个亚家族,但均有至少一个AP2/ERF结构域,由58—70个氨基酸残基组成,可特异性结合GCC-box元件(AGCCGCC)或DRE/CRT元件(序列A/GCCGAC)[4]。其中,AP2亚类有2个AP2保守结构域,RAV亚类有1个AP2和B3结构域,ERF和Solosit亚类均只有1个AP2保守结构域[5]。根据ERF亚类成员的AP2结构域第14位和第19位氨基酸的不同,又可以分为ERF(第14位丙氨酸,第19位天冬氨酸)和DREB/CBF(第14位缬氨酸,第19位谷氨酸)两类[6]。ERF亚类成员通过结合下游基因启动子区的GCC-box或DRE/CRT core顺式作用元件,调控靶基因表达,进而调控植物细胞内生理生化过程[7-9]。研究表明,AP2亚类主要参与调控开花、果实与种子发育等生长发育和逆境应答过程[10]。如,大麦HvAP2转录因子与MADS29互作,控制小花密度[11],过表达大豆可以促进开花[12],但过表达凤梨后则延迟开花[13]。而且,拟南芥和水稻个别AP2成员基因突变后,种子变大,重量增加[14-15]。过表达凤梨和莴苣会促使种子长度缩短,千粒重降低[13, 16]。但是,大豆GmAP2通过影响种子的长度,宽度和面积,进而正调控种子的大小[12]。目前,ERF亚类研究报道也较为深入,其功能涉及多方面,参与调控初级次生代谢、激素信号转导、生长发育及逆境应答等过程[17-20]。如菊花负调控胚的发育,干涉可促进胚胎发育和结实率[20]。番茄沉默株系促进叶片衰老,而过表达可延迟叶片衰老[17]。RAV亚类主要参与调控叶片衰老及生物与非生物胁迫,Soloist亚类的研究则鲜有报道[21]。玉米AP2家族共有214个成员,其中,AP2亚族成员IDS1和ANT调控维管和花发育,IDS1决定穗状花序分生组织的发育及成花数量,突变导致光合效率和生长率降低,以致产量降低[22-24]。此外,玉米AP2还调控节间和根系发育,其突变体节间变短和叶片起皱[25],过表达植株的支撑根数量增加,突变体的支撑根数量减少[26]。同时,研究发现玉米ERF亚类有166个成员,包括105个ERF成员和61个DREB成员,则主要参与逆境胁迫应答过程[22]。如表达受盐胁迫诱导,转化拟南芥的株系耐盐性显著提高[27],还可增强大斑病抗性,其突变体则易感大斑病[28],ZmEREB58通过促进表达,进而促进倍半萜的合成与积累[29]。【本研究切入点】目前,关于玉米ERF亚类参与调控种子发育的研究仍鲜见报道。前期研究中,发现ZmBES1/BZR1-5转录因子靶基因可能参与调控种子大小[30]。【拟解决的关键问题】本研究从B73克隆,并对其序列特征、表达模式、转录激活活性、亚细胞定位、调控籽粒发育的功能及调控靶基因进行分析,以期为后续深入研究的功能与调控机制提供参考依据。
1 材料与方法
1.1 材料准备
试验材料为玉米自交系B73,由四川农业大学玉米研究所保存。选取饱满的B73种子直接播种至大田,常规管理。待其进入生殖生长阶段,进行人工授粉自交,并取授粉前的雄穗、花丝、苞叶、茎、根,以及灌浆后10、15和25 d的籽粒,经液氮速冻并快速研磨至粉末状,装于1.5 mL离心管中,-80℃保存备用,每个组织设置3个生物学重复。
1.2 生物信息学分析
从MaizeGDB(https://maizegdb.org/)网站获取(Zm00001d010676)序列、编码序列及编码的氨基酸序列,利用在线工具GSDS(http://gsds. gao-lab.org/)进行结构可视化分析。同时,从NCBI(https://www.ncbi.nlm.nih.gov/)下载拟南芥、水稻和高粱等ERF氨基酸序列,利用DNAMAN软件进行氨基酸序列比对。
1.3 ZmEREB93的扩增
采用RNA提取试剂盒(大连宝生物工程有限公司)提取上述各样品总RNA,用cDNA反转录试剂盒(北京全式金生物技术有限公司)将其反转录成cDNA。同时,利用DNA提取试剂盒提取上述混合组织样品的基因组DNA(gDNA)。根据的开放阅读框(open reading frame,ORF)序列,采用Primer5设计扩增ORF序列引物(上游引物F:5′-ATGCAGCAGATGGAGTGGGACA-3′,下游引物R:5′-CGGAGACAGCGAGCCGAACAGC-3′)。分别以cDNA和gDNA为模板,用PrimeSTAR高保真DNA聚合酶进行PCR扩增,扩增体系为2×PrimeSTAR Buffer(Mg2+)12.5 μL、dNTPmix 2.0 μL、上游引物0.5 μL、下游引物0.5 μL、模板0.5 μL、PrimeSTAR 0. 25 μL和ddH2O 8.72 μL。扩增程序为94℃ 3 min,98℃ 10 s,66℃ 10 s,72℃ 50 s,35个循环;72℃ 5 min,12℃保存。产物经2%琼脂糖凝胶电泳检测,回收,并与载体pMD19-T连接,转化大肠杆菌,送至上海生工生物工程股份有限公司测序。
1.4 qRT-PCR分析
采用NCBI网站在线引物设计软件Primer-BLAST(https://blast.ncbi.nlm.nih.gov)设计的qRT-PCR引物(上游引物为93-F:5′-CCCTGGCACGT CGTCTATCT-3′,下游引物为93-R:5′-TCGTTGAGGT CGAACGGGAG-3′),扩增片段长185 bp。以玉米为内参基因(上游引物为EF-1a-F:5′-TGGGC CTACTGGTCTTACTACTGA-3′,下游引物为EF-1a- R:5′-ACATACCCACGCTTCAGATCCT-3′),扩增片段长135 bp。采用SYBR green法在CFX96TMReal- Time System进行qRT-PCR。qRT-PCR反应体系及程序按照SYBR Greensuper Mix试剂盒(北京全式金生物技术有限公司)说明,分别设置3个生物学和技术重复。采用2-ΔΔCT法计算在各组织中的相对表达量。
1.5 转录激活活性分析
采用Primer5设计的ORF扩增引物,根据同源重组酶及选取的限制性内切酶位点要求,在上下游引物分别引入pGBKT7多克隆位点侧翼序列(上游引物为93-BF:5′-aggccgaattcccggggatccATGCA GCAGATGGAGTGGGA-3′,下游引物为93-BR:5′- atgcggccgctgcaggtcgacCGGAGACAGCGAGCCGAA-3′。小写字母表示pGBKT7载体上的序列)。PCR扩增体系、程序及纯化回收参照1.3方法。采用HⅠ和Ⅰ限制性内切酶切割pGBKT7载体,使其线性化并纯化回收。使用同源重组试剂盒ClonExpress II One Step Cloning Kit(南京诺唯赞生物科技股份有限公司)构建pGBKT7-重组质粒,转化酵母AH109菌株。前期证实玉米ZmBES1/BZR1转录因子具有转录激活活性,以转化pGBKT7-ZmBES1/BZR1作为阳性对照,以转化pGBKT7空载体作为阴性对照。将转化后的菌液涂布在单缺SD/-Trp和三缺SD/-Trp/-His/ -Ade+X-α-gal的培养基上,28℃倒置培养3 d,观察菌落颜色及生长状况。
1.6 亚细胞定位
亚细胞定位使用植物表达载体pCAMBIA2300,同1.5引物设计方法,分别设计特异PCR引物(上游引物为93-GFPf:5′-ggacagggtacccggggatcc ATGCAGCAGATGGAGTGGGA-3′,下游引物去掉终止密码子TGA,引物为93-GFPr:5′-ggtactagtgtcga ctctagaCGGAGACAGCGAGCCGAA-3′)。参考1.3进行PCR扩增及扩增产物纯化回收。同1.5载体构建方法,用HⅠ和Ⅰ限制性内切酶切割pCAMBIA2300-载体,使其线性化并纯化回收,构建pCAMBIA2300-表达载体。参考Sun等[31]方法,将其转入GV3101农杆菌感受态,通过烟草注射的方法将其转化至本氏烟草中,正常培养36—48 h,于激光共聚焦显微镜下观察并拍照。
1.7 转基因拟南芥创制及表型分析
将上述pCAMBIA2300-表达载体转化GV3101农杆菌感受态,采用花絮浸染法转化拟南芥哥伦比亚生态型Col-0。收获T0代种子,播种至含50 mg·L-1卡那霉素的1/2 MS培养基,并于光照培养室培养2周,筛选阳性转化株系。每个株系独立收种,用同样方法筛选阳性株系至T3代纯合株系,并进行PCR检测,于激光共聚焦显微镜下观察荧光以确定目的基因是否表达。选取2个有目的基因插入且成功表达的纯合株系与野生型对照种植于相同条件下,直至成熟收种。每个株系随机取约1 000粒种子经1/10000天平进行称重,并用徕卡普通光学显微镜DM500于40倍物镜下拍照,测量每个株系种子的长和宽。每个株系种植30棵幼苗,并重复3次。
1.8 Chip-seq分析
体外Chip试验使用pCold-TF载体,同1.5引物设计方法,分别设计特异PCR引物(上游引物为93-Cf:5′-ctcggtaccctcgagggatccATGCAGCAGATGGA GTGGGA-3′;下游引物去掉终止密码子TGA,引物为93-Cr:5′-ctatctagactgcaggtcgacCGGAGACAGCGAG CCGAA-3′)。参考方法1.3进行PCR扩增及扩增产物纯化回收。同1.5载体构建方法,用HⅠ和Ⅰ限制性内切酶切割pCold-TF载体,使其线性化并纯化回收,构建pCold表达载体,使与His标签融合。随后,将其转化至大肠杆菌菌株Rosetta(DE3),在含有50 mg·mL-1氨苄青霉素的LB液体培养基培养OD600至0.6。参考Sun等[30]方法,用0.5 mol·L-1异丙基-β-D-硫代半乳糖苷(IPTG)诱导表达ZmEREB93蛋白,使用Ni-TED 1 mL SefinoseTM柱(上海生工生物工程股份有限公司)纯化ZmEREB93蛋白。随后,提取B73总DNA,超声波破碎为100—500 bp片段,并用乙醇沉淀纯化DNA。将纯化后DNA片段与带His标签的ZmEREB93蛋白加入孵化液(50 mmol·L-1Tris、1 mmol·L-1EDTA、100 mmol·L-1KCl、0.1% Triton X-100和1 mmol·L-1DTT,pH=7.0),4℃过夜。用孵育缓冲液洗涤3次,加入100 μL 5 mol·L-1NaCl,在65℃孵育2 h,解交联His-ZmEREB93和DNA片段。随后,提取DNA片段,并送至中国诺沃金公司进行Illumina测序,测序结果用于候选基因分析。
1.9 共表达分析
参考冯文奇等[32]方法,从MaizeGDB数据库(https://download.maizegdb.org/)下载B73转录组数据[33],利用R语言编写程序,将与玉米其他基因各组织中表达数据(FPKM值)进行相关性分析,选择相关系数大于0.7或者小于-0.7的基因作为候选目标。结合Chip-seq和共表达分析结果,筛选两方面共有的基因,并对这些基因起始密码子(ATG)上游2 000 bp序列进行分析,确认其启动子区域是否包含ERF结合基序GCC-box或DRE/CRT core元件,以鉴定ZmEREB93转录因子的候选靶基因。
1.10 酵母单杂
同1.5方法设计扩增候选靶基因启动子的特异PCR引物(分别为016pf:5′-tttgatattggatcggaattcTGTCA ACGTCGATCCGATGG-3′,016pr:5′-atacagagcacatgcc tcgagGGCTCCTCGTGCGCCTGG-3′;611pf:5′-tttgatatt ggatcggaattcCTATGACGACGGCGGCTCT-3′,611pr:5′-atacagagcacatgcctcgagCGGCGGCCGCGCCCGTCG-3′;488pf:5′-tttgatattggatcggaattcACAAACACAGGCA GAGAGGCA-3′,488pr:5′-atacagagcacatgcctcgagCGGT GAGTCGCGCGGCGC-3′;991pf:5′-tttgatattggatcggaa ttcACACACAGGTGGCTTTTCAAAGG-3′,991pr:5′-atacagagcacatgcctcgagGGAATAAGTTGGAGGAAAGGAAGC-3′;的ORF扩增引物为93f:5′-gattatgcctctcccgaattcATGCAGCAGATGGAGTGGGA-3′,93r:5′-agaagtccaaagcttctcgagCGGAGACAGCGA GCCGAA-3′)。参考Xiong等[34]方法,实施酵母单杂交试验。将的ORF构建至pJG4-5载体,靶基因启动子构建至pLacZi2u载体。利用酵母转化试剂盒(北京酷来搏科技有限公司),将已构建pLacZi2u启动子载体分别与pJG4-5空载体、pJG4-5与pLacZi2u启动子载体共同转化EYG48酵母感受态细胞(上海昂羽生物技术有限公司)。吸取2 μL转化后的菌液点至SD/-Trp/-Ura缺陷型培养基28℃培养2 d筛选阳性转化子,培养至菌斑2—3 mm。随后,挑取菌落于5 mL EP管过夜培养,取2 μL点于SD/-Trp/-Ura/X-gal(含20% Galactose+20% Raffiose+ 10 x Buffer salt)的缺陷型培养基28℃培养2 d并观察菌落生长情况与显色状态。
2 结果
2.1 ZmEREB93的序列特征
生物信息学分析结果表明,长618 bp,含一个外显子,无内含子和非编码区,编码205个氨基酸。ZmEREB93蛋白分子量21.62 kD,等电点10.24,亲水指数-0.33(<0),稳定系数43(>40),无跨膜区,预测定位于细胞核。其二级结构含19.02% α螺旋,8.29%β折叠,51.22%无规则卷曲。以上结果表明,ZmEREB93蛋白是一个不稳定的亲水蛋白。
以B73的cDNA和gDNA作模板,扩增的ORF,电泳结果显示,扩增产物大小一致且均位于500—700 bp(图1)。测序结果证实,Z的ORF长618 bp,无内含子。通过多序列比对发现,ZmEREB93与其他物种来源的ERF12具有较高的同源性,均含有一个高度保守的AP2结构域,属于ERF亚家族(图2)。
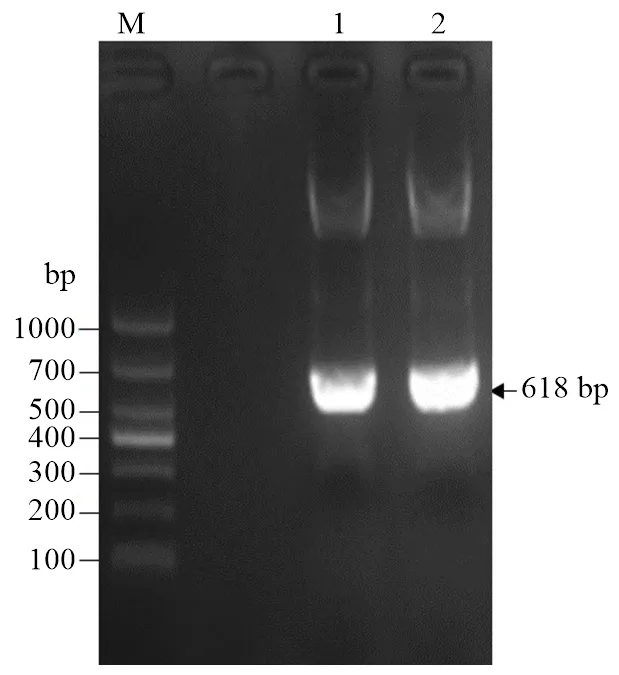
M:1000 bp DNA标准;1和2:分别以cDNA和gDNA为模板扩增
2.2 ZmEREB93在种子中的特异性表达
通过对玉米在不同组织中的qRT- PCR分析,结果表明,在授粉后15和25 d的种子中表达量较高,其中,在授粉后25 d的种子中表达量达到授粉后15 d种子中表达量的11倍左右。同时,成熟期的茎和根中也有表达,但是表达量较低,在雄穗、花丝和苞叶中无表达(图3)。
2.3 ZmEREB93无转录激活活性
为鉴定ZmEREB93转录激活活性,将其ORF构建至pGBKT7载体,以pGBKT7为阴性对照,pGBKT7为阳性对照,分别转化AH109酵母感受态细胞。结果(图4)表明,各载体转化菌株在SD/-Trp缺陷型培养基上均可正常生长,说明质粒转化成功。随后,将阳性转化子点至SD/-Trp/-His/ -Ade/X-α-gal三缺培养基上,转化pGBKT7的阳性对照菌株可正常生长且变蓝,而转化pGBKT7载体的阴性对照与pGBKT7菌株均不能生长。说明ZmEREB93在酵母细胞中以单体形式无转录激活活性。
2.4 ZmEREB93定位于细胞核
将和质粒转化农杆菌后,并注射烟草叶片进行瞬时表达。激光共聚焦显微镜成像结果显示,转化空载体的烟草叶片中,绿色荧光分布在整个细胞中。但是,在转化载体的烟草叶片中,荧光仅分布在细胞核(图5),该结果证实ZmEREB93转录因子位于细胞核发挥功能。
2.5 ZmEREB93负调控转基因拟南芥种子大小
对纯合转基因株系进行PCR和荧光检测(图6),在转基因株系L#1和L#2中可扩增到目的基因且检测到荧光,而野生型对照中不能检测到目的基因,说明插入拟南芥基因组且能够表达。对各株系的种子表型进行分析表明,与野生型对照相比,转基因株系L#1的粒长和粒宽均显著降低,转基因株系L#2种子的粒长无显著变化,但其粒宽显著降低。同时,L#1和L#2株系种子的千粒重均显著低于野生型株系(图7)。以上结果表明,负调控转基因拟南芥种子大小。
2.6 ZmEREB93靶基因
体外Chip-seq结果显示,通过Chip试验得到1 395个DNA片段,-value(-log10)值均大于2。进一步基因序列比对及GO注释发现,共注释1 226个基因。共表达分析结果显示,玉米中有100个基因的表达与表达相关性较高,相关系数大于0.7。结合两方面结果,筛选到共有基因4个,分别为、、和。随后,启动子元件分析发现,这4个基因启动子区均存在DRE/CRT core元件,无GCC-box(表1)。初步推测这些候选基因是ZmEREB93转录因子调控的靶基因。

AtERF12:拟南芥,AT1G28360;SbERF12:高粱,SORBI_3009G184300;OsERF12:水稻,OS05G0497200;SiERF12:谷子,XP_004970252.1;AlERF12:马绊草,QYY53011.1;ZjERF12:大枣,XP_015890508.1;DzERF12:榴莲,XP_022777342.1;CiERF12:山核桃,XP_042944848.1;CqERF12:藜麦,XP_021740383.1;ItERF12:三裂叶薯,XP_031112454.1
Y1H试验结果表明,在SD/-Trp/-Ura二缺培养基上,所有转化后的酵母均可正常生长形成菌落。但是,在SD/-Trp/-Ura/X-gal培养基上,只有共转化ZmEREB93与启动子载体的菌落呈现深蓝色,说明ZmEREB93可以直接结合的启动子(图8-a)。进一步构建启动子截短片段并实施Y1H,结果发现,ZmEREB93结合启动子的-1 621— -621 bp区段,该片段有3个DRE/CRT core元件(图8-b)。说明是ZmEREB93的靶基因。

图3 ZmEREB93的表达模式分析

图4 ZmEREB93转录激活活性分析
3 讨论
3.1 ZmEREB93的特征
在SUN等[30]前期研究中发现,玉米ZmBES1/ BZR1-5转录因子正向调控种子大小,作为其靶基因且受其负调控。因此,推测可能参与调控籽粒发育过程。本研究成功克隆到,发现其仅有一个外显子,无内含子(图1),在玉米种子中特异性表达,尤其在授粉后25 d的玉米种子中高表达(图3),表明其可能在玉米籽粒发育中起重要作用。ZmEREB93氨基酸序列仅有1个高度保守的AP2典型的特征结构域(图2),属于玉米ERF 家族成员[37]。进一步研究证实,ZmEREB93蛋白位于细胞核但无转录激活活性(图4—图5),可能的原因是ZmEREB93在酵母细胞中以单体形式存在不能发挥转录激活活性。已有研究证实,植物中一些转录因子单独存在时无法转录激活下游靶基因,只有形成二聚体或与其他蛋白互作后调控下游的表达。例如,玉米转录因子ZmBES1/BZR1-5单体无转录激活活性,但会自身形成二聚体[30]。水稻OsERF115转录因子与OsNF-YB1形成蛋白复合体进而发挥功能[38]。
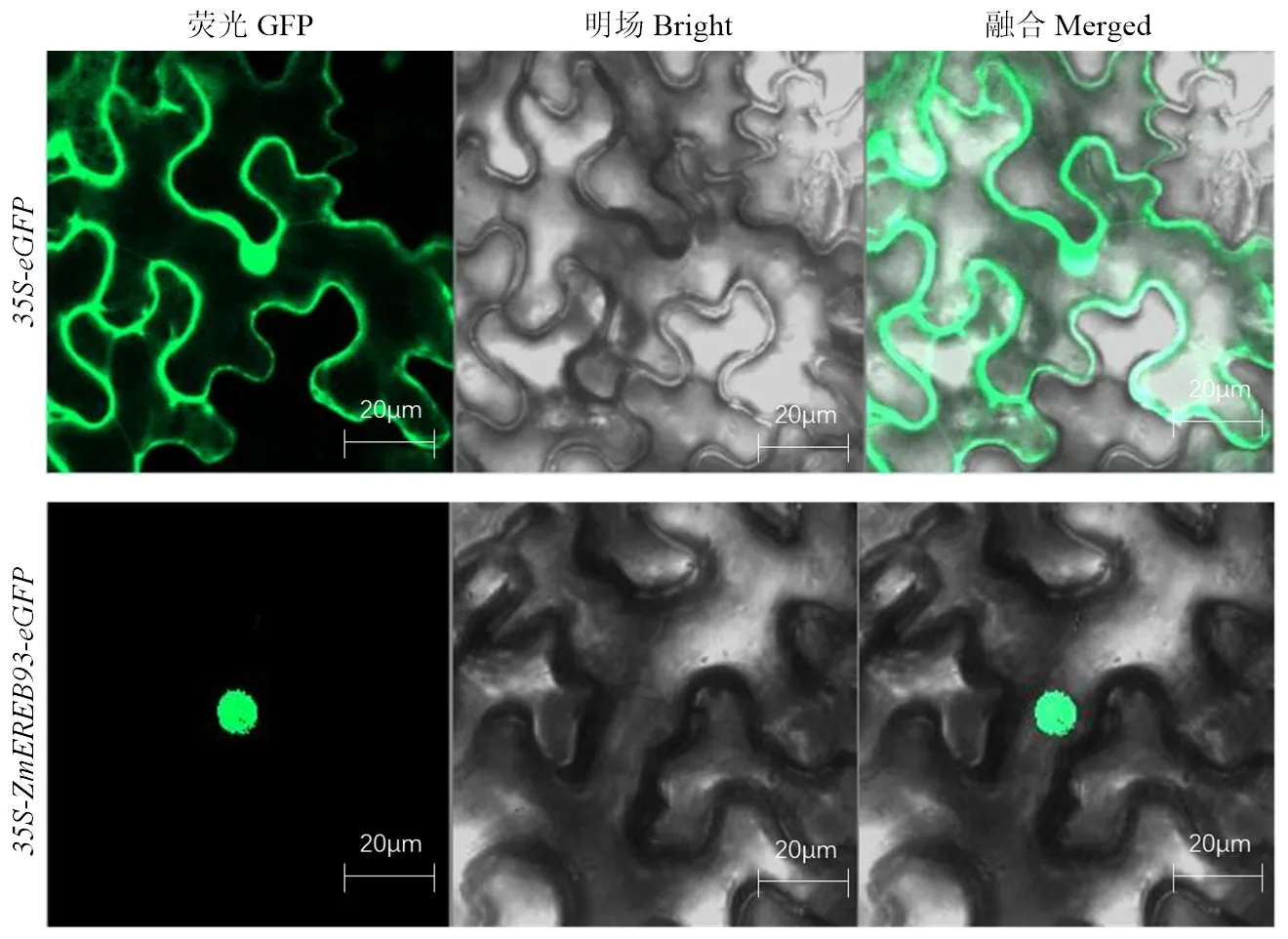
图5 ZmEREB93的亚细胞定位
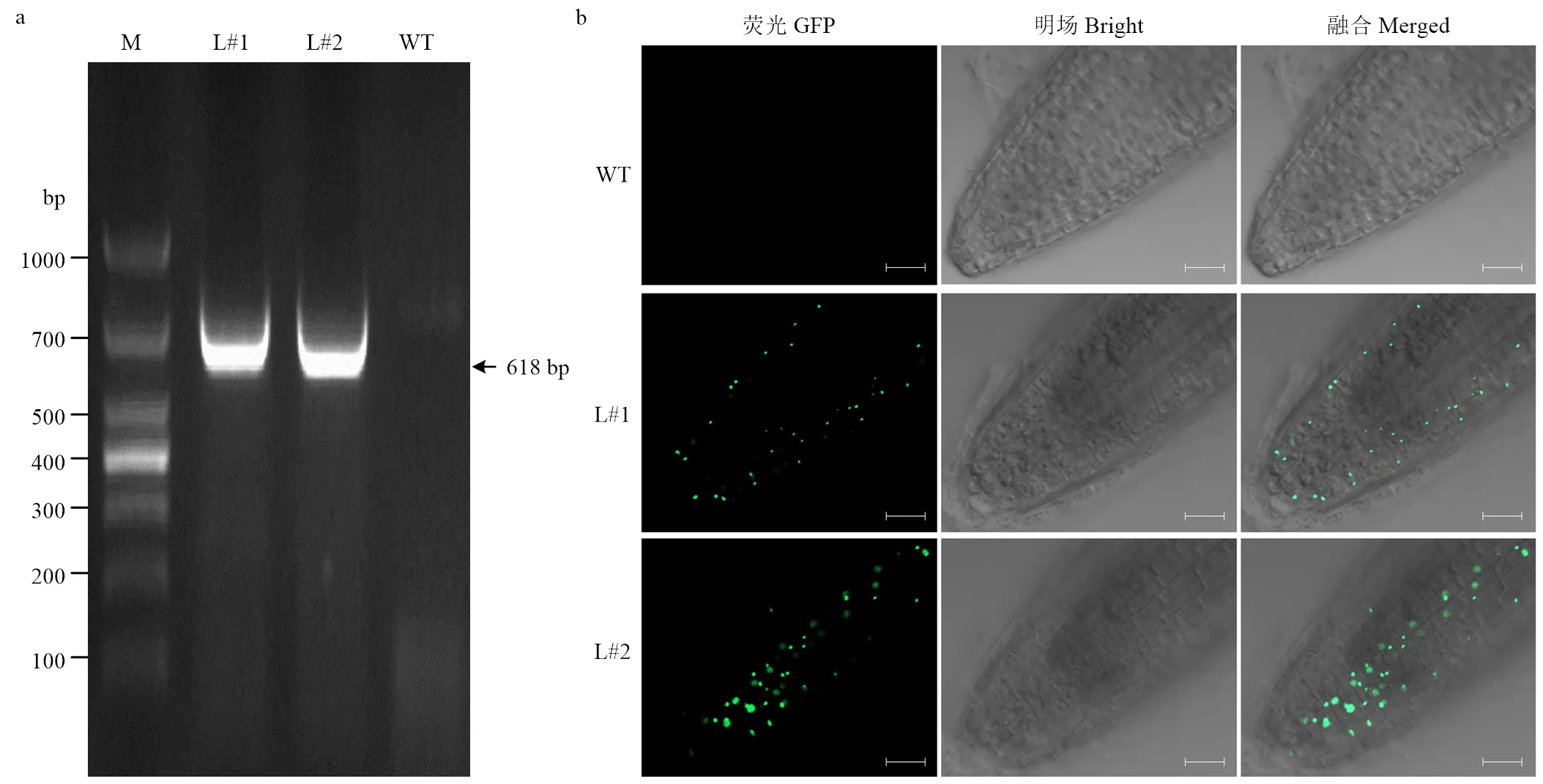
a:各株系PCR检测;b:各株系荧光检测;M:1000 bp DNA标准;L#1和L#2:分别为转化ZmEREB93的拟南芥株系;WT:野生型。下同
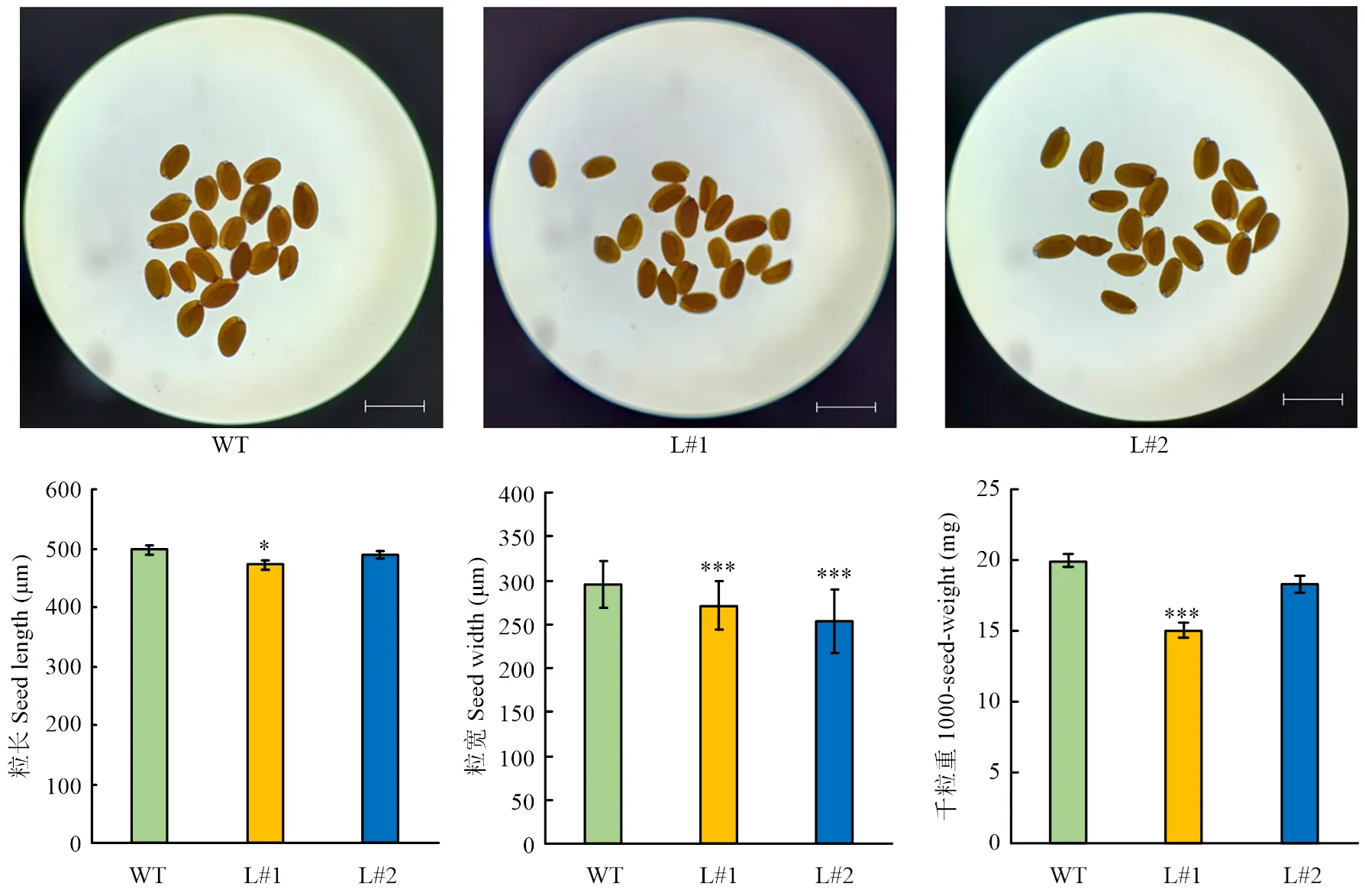
*:p<0.05;**:p<0.01;***:p<0.0001

a:Y1H验证ZmEREB93与Zm00001d013611、Zm00001d006016、Zm00001d027448、Zm00001d039991启动子结合。P016、P488、P991、P611表示Zm00001d006016、Zm00001d027448、Zm00001d039991、Zm00001d013611启动子;b:Y1H验证ZmEREB93与Zm00001d013611启动子不同片段结合。P1、P2、P3表示不同片段启动子;△表示DRE/CRT core元件
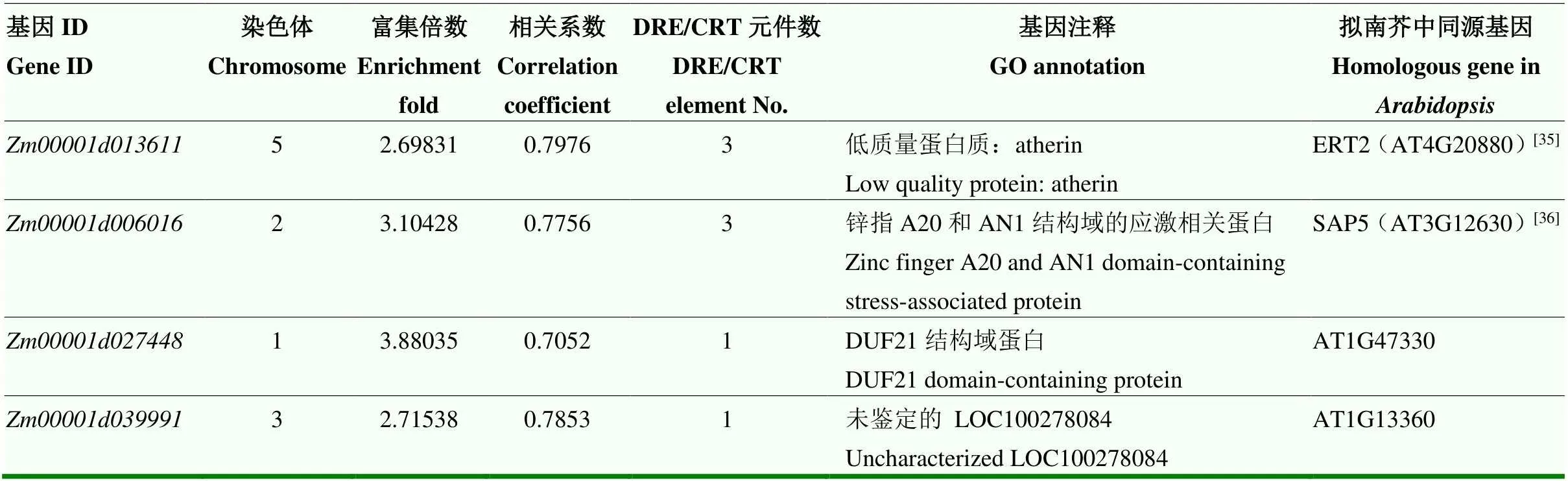
表1 ZmEREB93候选靶基因
3.2 ERF亚类转录因子调控植物生长发育
植物中,AP2转录因子家族的ERF亚类成员主要参与调控初级次生代谢、激素信号转导、生长发育及逆境应答等过程[17-20]。玉米中,AP2转录因子的AP2亚族成员主要调控生长发育过程[22-26],而ERF亚类主要参与调控逆境应答过程[22, 27-28]。但是,研究人员也发现,植物中ERF亚类成员作为转录抑制因子也参与调控籽粒发育过程。例如,菊花CmERF12与花和胚发育相关蛋白CmSUF4相互作用,进而抑制其靶基因(EGG CELL1),最终负调控胚的发育[39]。水稻OsERF115与OsNF-YB1形成蛋白复合体参与调控水稻籽粒灌浆和胚乳发育[38]。近期研究还发现,OsERF115通过抑制籽粒大小负调控因子OsGW2和OsGS6的启动子活性,进而调控籽粒大小和粒重[38, 40]。在本研究中,玉米在拟南芥中的同源基因参与调控花的发育[41],但是,将玉米转化拟南芥过量表达,转基因株系的种子较对照显著变小(图7),说明ZmEREB93转录因子抑制种子发育。
3.3 ZmEREB93调控的下游途径
众所周知,ERF亚类成员通过结合下游基因启动子区的GCC-box或DRE/CRT core顺式作用元件调控靶基因表达[7-9]。结合chip-seq与共表达分析筛选到4个候选基因可能作为ZmEREB93的靶基因,且它们的启动子区均有DRE/CRT core元件(表1)。其中,和在拟南芥中的同源基因分别为和,表达无组织特异性且受乙烯诱导,则参与逆境胁迫应答[34-35]。通过Y1H进一步证实,ZmEREB93可直接结合启动子的-1 621—-621 bp区段,该片段有3个DRE/CRT core元件(图8)。虽然,其余候选靶基因在Y1H试验中未得到验证,但不能排除其真实性,由于酵母表达系统存在假阳性和假阴性,仍需体内试验验证。同时,及均为未知基因,也可能调控籽粒发育。而且,在Chip-seq中鉴定到8个启动子上具有DRE/CRT顺式作用元件的PPR家族基因,而PPR成员通过影响线粒体发育,进而调控玉米的内核发育以及籽粒发育[41]。PPR突变通常造成线粒体的形态与功能缺陷,最终导致玉米籽粒发育的严重缺陷[42-43]。因此,在后续研究中,将通过转基因或敲除试验重点评价在玉米籽粒发育中的功能,并在体内进行其靶基因的验证及功能研究。
4 结论
从玉米B73中克隆到,长度为618 bp,不含内含子,编码205个氨基酸,有一个高度保守AP2结构域,属于AP2家族的ERF亚类。ZmEREB93在授粉后25 d的种子中高表达,定位于细胞核,且负调控转基因拟南芥的种子大小。为ZmEREB93调控的候选靶基因。
[1] DOLL N M, DEPEGE-FARGEIX N, ROGOWSKY P M, WIDIEZ T. Signaling in early maize kernel development. Molecular Plant, 2017, 10(3): 375-388.
[2] 高春艳, 吴芮, 袁玉, 刘同玥, 任莉萍. 植物AP2/ERF转录因子及其在非生物胁迫应答中的作用. 江汉大学学报(自然科学版), 2017, 45(3): 236-240.
GAO C Y, WU R, YUAN Y, LIU T Y, REN L P. Function of AP2/ERF transcription factors in plant tolerance to abiotic stress. Journal of Jianghan University (Natural Science Education), 2017, 45(3): 236-240. (in Chinese)
[3] JOFUKU K D, DEN BOER B G, VAN MONTAGU M, OKAMURO J K. Control offlower and seed development by the homeotic gene APETALA2. The Plant Cell, 1994, 6(9): 1211-1225.
[4] CHAKRAVARTHY S, TUORI R P, D’ASCENZO M D, FOBERT P R, DESPRES C, MARTIN G B. The tomato transcription factor Pti4 regulates defense-related gene expression via GCC box and non-GCC box cis elements. The Plant Cell, 2003, 15(12): 3033-3050.
[5] LICAUSI F, OHME-TAKAGI M, PERATA P. APETALA2/ethylene responsive factor (AP2/ERF) transcription factors: mediators of stress responses and developmental programs. New Phytologist, 2013, 199(3): 639-649.
[6] Wen K. The important role of AP2 functional genes in plant floral development. Biotechnology Bulletin, 2010, 20(2): 1-7.
[7] 丰锦, 陈信波. 抗逆相关AP2/EREBP转录因子研究进展. 生物技术通报, 2011(7): 1-7.
Feng J, Chen X B. Research progress of AP2/EREBP transcription factors in stress tolerance. Biotechnology Bulletin, 2011(7): 1-7. (in Chinese)
[8] RIECHMANN J L, MEYEROWITZ E M. The AP2/EREBP family of plant transcription factors. Biological Chemistry, 1998, 379(6): 633-646.
[9] RAO G, SUI J, ZENG Y, HE C, ZHANG J. Genome-wide analysis of the AP2/ERF gene family in. FEBS Open Bio, 2015, 24(5): 132-137.
[10] SHIGYO M, HASEBE M, ITO M. Molecular evolution of the AP2 subfamily. Gene, 2006, 366(2): 256-265.
[11] SHOESMITH J R, SOLOMON C U, YANG X, WILKINSON L G, SHELDRICK S, EIJDEN E V, COUWENBERG S, PUGH M L, ESKAN M, STEPHENS J, BARAKATE A, DREA S, HOUSTON K, TUCKER M R, MCKIM S M. APETALA2 functions as a temporal factor together with BLADE-ON-PETIOLE2 and MADS29 to control flower and grain development in barley. Development, 2021, 148(5): 1-5.
[12] JIANG W, ZHANG X, SONG X, YANG J, PANG Y. Genome-wide identification and characterization of APETALA2/ethylene-responsive element binding factor superfamily genes in soybean seed development. Frontiers in Plant Science, 2020, 11: 566647.
[13] LEI M, LI Z Y, WANG J B, FU Y L, XU L. Ectopic expression of theAPETALA2 gene AfAP2-2 reduces seed size and delays flowering in. Plant Physiology and Biochemistry, 2019, 139: 642-650.
[14] JOFUKU K D, OMIDYAR P K, GEE Z, OKAMURO J K. Control of seed mass and seed yield by the floral homeotic gene APETALA2. Proceedings of the National Academy of Sciences of the USA, 2005, 102(8): 3117-3122.
[15] JIANG L, MA X, ZHAO S, TANG Y, LIU F, GU P, FU Y, ZHU Z, CAI H, SUN C, TAN L. The APETALA2-Like transcription factor SUPERNUMERARY BRACT controls rice seed shattering and seed size. The Plant Cell, 2019, 31(1): 17-36.
[16] LUO C, WANG S, NING K, CHEN Z, WANG Y, YANG J, QI M, WANG Q. The APETALA2 transcription factor LsAP2 regulates seed shape in lettuce.Journal of Experimental Botany, 2021, 72(7): 2463-2476.
[17] CHEN Y, FENG P, TANG B, HU Z, XIE Q, ZHOU S, CHEN G. The AP2/ERF transcription factor SlERF.F5 functions in leaf senescence in tomato. Plant Cell Reports, 2022, 41: 1181-1195.
[18] RITONGA F N, NGATIA J N, WANG Y, KHOSO M A, FAROOQ U, CHEN S. AP2/ERF, an important cold stress-related transcription factor family in plants: A review. Physiology and Molecular Biology of Plants, 2021, 27(9): 1953-1968.
[19] YU Z X, LI J X, YANG C Q, HU W L, WANG L J, CHEN X Y. The jasmonate-responsive AP2/ERF transcription factors AaERF1 and AaERF2 positively regulate artemisinin biosynthesis inL.Molecular Plant, 2012, 5(2): 353-365.
[20] XU S, HOU H, WU Z, ZHAO J, ZHANG F, TENG R, CHEN F, TENG N.embryo development is negatively affected by a novel ERF transcription factor, CmERF12. Journal of Experimental Botany, 2022, 73(1): 197-212.
[21] FENG C Z, CHEN Y, WANG C, KONG Y H, WU W H, CHEN Y F.RAV1 transcription factor, phosphorylated by SnRK2 kinases, regulates the expression of ABI3, ABI4, and ABI5 during seed germination and early seedling development. The Plant Journal, 2014, 80(4): 654-668.
[22] ZHANG J, LIAO J, LING Q, XI Y, QIAN Y. Genome-wide identification and expression profiling analysis of maize AP2/ERF superfamily genes reveal essential roles in abiotic stress tolerance. BMC Genomics, 2022, 23(1): 125.
[23] CHUCK G, MEELEY R, HAKE S. Floral meristem initiation and meristem cell fate are regulated by the maize AP2 genes ids1 and sid1.Development, 2008, 135(18): 3013-3019.
[24] LIU W Y, LIN H H, YU C P, CHANG C K, CHEN H J, LIN J J, LU M J, TU S L, SHIU S H, WU S H, KU M S B, LI W H. Maize ANT1 modulates vascular development, chloroplast development, photosynthesis, and plant growth. Proceedings of the National Academy of Sciences, USA2020, 117(35): 21747-21756.
[25] LID S E, AL R H, KREKLING T, MEELEY R B, RANCH J, OPSAHL-FERSTAD H G, OLSEN O A. The maize disorganized aleurone layer 1 and 2 (dil1, dil2) mutants lack control of the mitotic division plane in the aleurone layer of developing endosperm. Planta, 2004, 218(3): 370-378.
[26] LI J, CHEN F, LI Y, LI P, WANG Y, MI G, YUAN L. ZmRAP2.7, an AP2 transcription factor, is involved in maize brace roots development.Frontiers in Plant Science, 2019, 10: 820.
[27] FU J, ZHU C, WANG C, LIU L, SHEN Q, XU D, WANG Q. Maize transcription factor ZmEREB20 enhanced salt tolerance in transgenic. Plant Physiology and Biochemistry, 2021, 159:257-267.
[28] ZANG Z, WANG Z, ZHAO F, YANG W, CI J, REN X, JIANG L, YANG W. Maize ethylene response factoris required for resistance to. Frontiers in Plant Science, 2021, 12: 630413.
[29] LI S, WANG H, LI F, CHEN Z, LI X, ZHU L, WANG G, YU J, HUANG D, LANG Z. The maize transcription factor EREB58 mediates the jasmonate-induced production of sesquiterpene volatiles.ThePlant Journal, 2015, 84(2): 296-308.
[30] SUN F, DING L, FENG W, CAO Y, LU F, YANG Q, LI W, LU Y, SHABEK N, FU F, YU H. Maize transcription factor ZmBES1/ BZR1-5 positively regulates kernel size. Journal of Experimental Botany, 2021, 72(5): 1714-1726.
[31] SUN F A, YU H Q, QU J T, CAO Y, DING L, FENG W Q, MUHAMMAD H B K, LI W C, FU F L. Maize ZmBES1/BZR1-5 decreases ABA sensitivity and confers tolerance to osmotic stress in transgenic. International Journal of Molecular Sciences, 2020, 21(3): 996.
[32] 冯文奇, 孙福艾, 丁磊, 郭新, 李晚忱, 付凤玲, 于好强. 玉米转录因子基因克隆及功能分析. 核农学报, 2020, 34(1): 17-25.
FENG W Q, SUN F A, DING L, GUO X, LI W C, FU F L, YU H Q. Cloning and function analysis ofgene in maize. Journal of Nuclear Agricultural Sciences, 2020, 34(1): 17-25. (in Chinese)
[33] WALLEY J W, SARTOR R C, SHEN Z, SCHMITZ R J, WU K J, URICH M A, NERY J R, SMITH L G, SCHNABLE J C, ECKER J R, BRIGGS S P. Integration of omic networks in a developmental atlas of maize. Science, 2016, 353(6301): 814-818.
[34] XIONG F, ZHANG B K, LIU H H, WEI G, WU J H, WU Y N, ZHANG Y, LI S. Transcriptional regulation of PLETHORA1 in the root meristem through an importin and its two. The Plant Cell, 2020, 32(12): 3812-3824.
[35] TRENTMANN S M. ERN1, a novel ethylene-regulated nuclear protein of. Plant Molecular Biology, 2000, 44(1): 11-25.
[36] KANG M, FOKAR M, ABDELMAGEED H, ALLEN R D.SAP5 functions as a positive regulator of stress responses and exhibits E3 ubiquitin ligase activity.Plant Molecular Biology, 2011, 75(4/5): 451-466.
[37] ZHANG J, LIAO J, LING Q, XI Y, QIAN Y. Genome-wide identification and expression profiling analysis of maize AP2/ERF superfamily genes reveal essential roles in abiotic stress tolerance. BMC Genomics, 2022, 23: 125.
[38] XU J J, ZHANG X F, XUE H W. Rice aleurone layer specific OsNF-YB1 regulates grain filling and endosperm development by interacting with an ERF transcription factor. Journal of Experimental Botany, 2016, 67(22): 6399-6411.
[39] XU S, HOU H, WU Z, ZHAO J, ZHANG F, TENG R, CHEN F, TENG N. Chrysanthemum embryo development is negatively affected by a novel ERF transcription factor, CmERF12. Journal of Experimental Botany, 2022, 73(1): 197-212.
[40] LIU C, MA T, YUAN D, ZHOU Y, LONG Y, LI Z, DONG Z, DUAN M, YU D, JING Y, BAI X, WANG Y, HOU Q, LIU S, ZHANG J S, CHEN S Y, LI D, LIU X, LI Z, WANG W, LI J, WEI X, MA B, WAN X. The OsEIL1-OsERF115-target gene regulatory module controls grain size and weight in rice. Plant Biotechnology Journal, 2022. Doi: 10.1111/pbi.13825.
[41] CHANDLER J W, WERR W. A phylogenetically conserved APETALA2/ETHYLENE RESPONSE FACTOR, ERF12, regulatesfloral development. Plant Molecular Biology, 2020, 102: 39-54.
[42] CHEN X, FENG F, QI W, XU L, YAO D, WANG Q, SONG R.encodes a PPR protein that affects cis-splicing of mitochondrial nad4 intron 1 and seed development in maize. Molecular Plant, 2017, 10(3): 427-441.
[43] CAI M, LI S, SUN F, SUN Q, ZHAO H, REN X, ZHAO Y, TAN B C, ZHANG Z, QIU F.encodes a mitochondrial PPR protein that affects the-splicing of nad2 intron 1 and seed development in maize. The Plant Journal, 2017, 91(1): 132-144.
Maize Transcription Factor ZmEREB93 negatively regulates kernel development
PANG HaoWan1, FU QianKun1, YANG QingQing1, ZHANG YuanYuan2, FU FengLing1, YU HaoQiang1
1Maize Research Institute, Sichuan Agricultural University, Chengdu 611130;2College of Life Science & Biotechnology, Mianyang Tearchers’ College, Mianyang 621000, Sichuan
【Objective】Maize, a kind of crucial crop, is widely used in food supply, livestock feed, and industry. AP2/EREBP (APETALA2/ethylene response element-binding protein) transcription factor (TF) plays an important role in plant growth, development, and stress response. Previous study showed thatmight regulate seed size as a target gene of ZmBES1/BZR1-5 TF.was cloned and used to analyze its expression pattern and function, which lays foundation to clarify the function and mechanism ofregulating seed size. 【Method】The full length ofwas cloned from maize inbred line B73 by PCR. The characters of nucleotide and amino acid sequences were analyzed by informatic methods. Subsequently, the tissue expression specificity ofwas analyzed via quantitative real time PCR (qRT-PCR). The expression vector in plant and yeast was constructed and used for subcellular localization and transcription activation assay, respectively.was transformed intomediated by agrobacterium transformation. The seed phenotype of transgenic lines was analyzed. Finally, the potential target genes of ZmEREB93 were screened by chromatin immunoprecipitation sequencing (Chip-seq) and co-expression analysis, and further confirmed by yeast one hybrid (Y1H). 【Result】Thegene was cloned by PCR. Sequence analysis showed thathad no intron and a 618 bp ORF, encoding 205 amino acids with a highly conserved AP2 domain and belongs to the ERF subclade of AP2 family. The results of qRT-PCR showed that thegene highly expressed in kernels of 15 and 25 days after pollination (DAP), and slightly expressed in stem and root, but did not express in tassel, silk and bract. The expression level ofwas the highest in 25 DAP kernels reached 11 times of that in 15 DAP kernels. The results of transcriptional activation and subcellular localization assay exhibited that ZmEREB93 protein had no transcriptional activation activity in yeast cells and was localized in the nucleus, respectively. Compared to wild type, the seeds of transgenic lines were significantly smaller and showed lower thousand-seed-weight. Chip-seq and co-expression analysis suggested that the,,andgenes were candidate target genes regulated by ZmEREB93 TF. The result of Y1H showed that ZmEREB93 directly bind topromoter. 【Conclusion】Maize ZmEREB93 TF specifically expressed in seeds and negatively regulated seed size.
maize; transcription factors; AP2/EREBP; grain

10.3864/j.issn.0578-1752.2022.19.001
2022-04-18;
2022-06-16
成都市科学技术局技术创新研发项目(2021-YF05-02024-SN)、四川省科学技术厅国际科技创新合作项目(2022YFH0067)
庞浩婉,E-mail:1421781500@qq.com。通信作者于好强,E-mail:yhq1801@sicau.edu.cn
(责任编辑 李莉)
