瘦肉型猪基因组中ROH 的应用及研究进展
2022-10-12李桂新王诗媛王小鹏郑恩琴
李桂新,王诗媛,杨 杰,王小鹏,郑恩琴
(华南农业大学动物科学学院,国家生猪种业工程技术研究中心,广东广州 510642)
在二倍体生物的基因组中,连续性纯合片段(Runs of Homozygosity,ROH)是指基因组中没有杂合子的纯合基因型的连续染色体片段。ROH 主要由群体历史变迁形成的,如种群瓶颈、遗传漂变和近亲繁殖等。另外,强烈的自然和人工选择也塑造着基因组区域不同的ROH 模式。较长的单倍型遗传自最近的共同祖先,较短的单倍型遗传自遥远的祖先。ROH 被认为是评估人类和动植物近亲繁殖的有效方法。优良性状的选择产生了畜禽品种显著的表型变化,并形成了基因组不同区域的ROH 模式。同时,选择也增加了基因座周围的纯合性。育种计划的选择强度和优良种畜的广泛应用导致近亲繁殖增加,引起群体的遗传多样性降低。使用中、高密度SNP 基因芯片扫描基因组中的ROH 是识别血缘同源(Identical By Descent,IBD)单倍型的有效方法。基因组ROH 可以提供一个种群过去和最近的群体变化信息,揭示世代间演变的历程。最近几十年,人类对瘦肉型猪的高强度选育可能导致近亲繁殖的累积,造成基因组中单倍型的多样性降低,等位基因的基因频率升高甚至接近固定存在于基因组中。本文主要围绕动物基因组ROH 研究的发展简史、ROH 的识别软件和相关参数,以及ROH 在瘦肉型猪的应用和研究进展进行综述,旨在为瘦肉型猪的育种提供参考依据。
1 ROH 发展简史
1999 年,Broman 等首次对人类基因组进行连续性纯合检测分析,推断纯合片段可能代表同合性,而且可能与人类健康具有很大的相关性。2006 年,Gibson 等利用高密度SNP 芯片首次在人类基因组中报道了ROH,揭示了整个基因组中ROH 的长度、频率和分布。2010 年,Sölkner 等、Ferenčaković 等首次将高密度SNP 芯片应用于畜禽基因组ROH 研究。随着重测序技术和SNP 芯片的发展、成熟和广泛引用及成本不断降低,出现了一系列关于畜禽基因组ROH 分析的相关研究。例如在猪基因组研究中,Bosse 等2012 年,首次研究瘦肉型猪基因组ROH 的群体历史(如群体瓶颈、近交繁殖等)、基因组特征(如GC 含量和重组率)和选择对基因组ROH 的影响。Silió 等利用系谱和基因组数据中评估了伊比利亚猪猪的近交水平和近交衰退。Herrero-Medrano 等利用高密度SNP 芯片通过ROH 分析,揭示了家猪和野猪的群体历史和近交繁殖。2014 年,Herrero-Medrano 等使 用60K SNP 芯片和重测序数据(~10X)进行ROH 检测,发现检测的ROH 和近交系数呈现很强的相关性。此后,瘦肉型猪基因组ROH 成为一个兴起的研究热点。
2 ROH 检测的软件和参数
目前,对于不同的数据集,关于不同软件间以及同一软件间不同参数的设置对ROH 检测影响的研究较少,使用不同软件之间的最佳参数难以确定。不同的研究采用了不同的软件和参数来识别ROH。目前,缺乏对ROH 的定义和识别标准,局限了ROH 相关研究的发展。不同的研究在检测ROH 时使用了各自的标准,有些研究采用了严格的标准,如Lencz 等采用至少100个连续SNPs,不允许存在杂合子,且需要在10 个或者更多个体间共享的ROH 为研究目标。而有些研究采用了较为宽松的标准,如Spain 等使用完全连锁不平衡和低连锁不平衡的数据集,研究了不同数量的SNPs和不同长度的ROH 对识别ROH 的影响,研究允许2%的杂合子出现,且取消了对SNPs 之间最大距离和最小SNPs 密度的限制。定义和识别ROH 标准的差异可能导致千差万别的结果,同时增加了假阳性出现的概率,并使不同研究结果之间的比较变得困难。
2.1 检测ROH 的软件 当前,根据不同的方式识别基因组ROH,可以将检测方法分为连续性(Consecutive Runs)识别和窗口滑动(Sliding Window)识别。连续性识别是一种沿着基因组对SNPs 位点连续扫描检测的方法,其设置参数类别包括一个ROH 中最小的SNPs数量、最大的杂合子与缺失SNPs 数量、相邻SNPs 位点的最大间隙以及最小运行长度等。应用连续性识别方法检测ROH 的软件有SVS(Golden Helix SNP &Variation Suite v.7.6.8)、SAS(SAS Institute)以 及最近比较热门的R 包—detectRUNS等。另一种检测方法是窗口滑动检测法,即设置窗口大小,以滑动窗口形式检测基因组ROH 的方法。常用软件及脚本有PLINK、GERMLINE、BEAGLE、cgaTOH、SVS(Golden Helix SNP &Variation Suite v.7.6.8)、BCFtools/RoH、RZooRoH和FORTRAN 脚本等。其中,SVS 软件和R 包—detectRUNS 能同时用这2 种检测方法来识别基因组ROH。目前PLINK 的窗口滑动和R 包—detectRUNS 的连续性检测方法被广泛应用于ROH 的研究。
2.2 检测ROH 的参数 无论是连续性运行还是窗口滑动识别ROH,都需要对多个参数和阈值进行设置。这些设置可能会对ROH 数量、大小和分布产生显著影响,而且默认设置值并不总是适合各种基因型数据,尤其是不同密度的商业SNP 芯片数据。Purfield 等使用牛50K 和HD SNP 芯片(777972 SNPs)分析ROH时,发现HD 芯片能更准确地识别ROH,50K 芯片的SNP 密度适用于大多数牛品种的ROH 检测。作者还比较了50K 和HD 芯片共有SNPs 数据集与HD 芯片识别ROH 的差异,发现二者都难以识别0.5~1 Mb 短长度的ROHs,且都能识别所有>5 Mb 长度的ROHs。同时,因为低密度SNP 芯片估计的ROH 水平与根据系谱的近交系数呈正相关。因此,常用商业基因芯片的标记密度在鉴定ROH 是合适的。进行ROH 分析之前,是否需要过滤最小等位基因型频率(Minor Allele Frequency,MAF)、偏离哈代-温伯格平衡(Hardy-Weinberg Equilibrium,HWE)和高连锁不平衡(Linkage Disequilibrium,LD)的SNPs 尚未得到共识。大多数研究都是采用过滤MAF<0.01 或者<0.05 的SNPs。有些研究也过滤LD,如Meyermans 等测试过滤不同LD 和MAF 值对检测ROH 的影响,发现随着过滤LD值的增加,基因组覆盖率迅速下降;同时发现MAF 过滤不仅影响固定区域的ROH 检测,而且在非固定区域检测到ROH 的发生率也存在较大差异。因此,在进行ROH 研究时,作者建议不要进行LD 和MAF 过滤。SNP 基因型分型错误是另一个可能影响ROH 检测的因素,它的存在会影响包含SNPs 数量多的长ROH 识别。当前研究的一个解决方案是允许一定数量的杂合SNP存在,但这是否会影响检测ROH 的可靠性尚未得到证实。此外,其他因素如ROH 中SNP 的最小密度、最大间隙、滑动窗口大小、窗口阈值、ROH 最小长度、ROH 包含的最少SNP 数量和最多缺失基因型数量在一定程度都会影响ROH 的检测。
目前,ROH 分析广泛应用于瘦肉型猪研究中,主流瘦肉型猪品种基因组ROH 的部分研究工作以及用于识别ROH 的相应参数和阈值均在表1 中展示。然而还有一些研究的参数设置没有明确地提到,不确定作者是否使用默认设置或调整参数,这样可能使得基因组的覆盖率被过高或过低估计。可见,ROH 的识别和定义标准在不同品种内和品种间都存在差异。
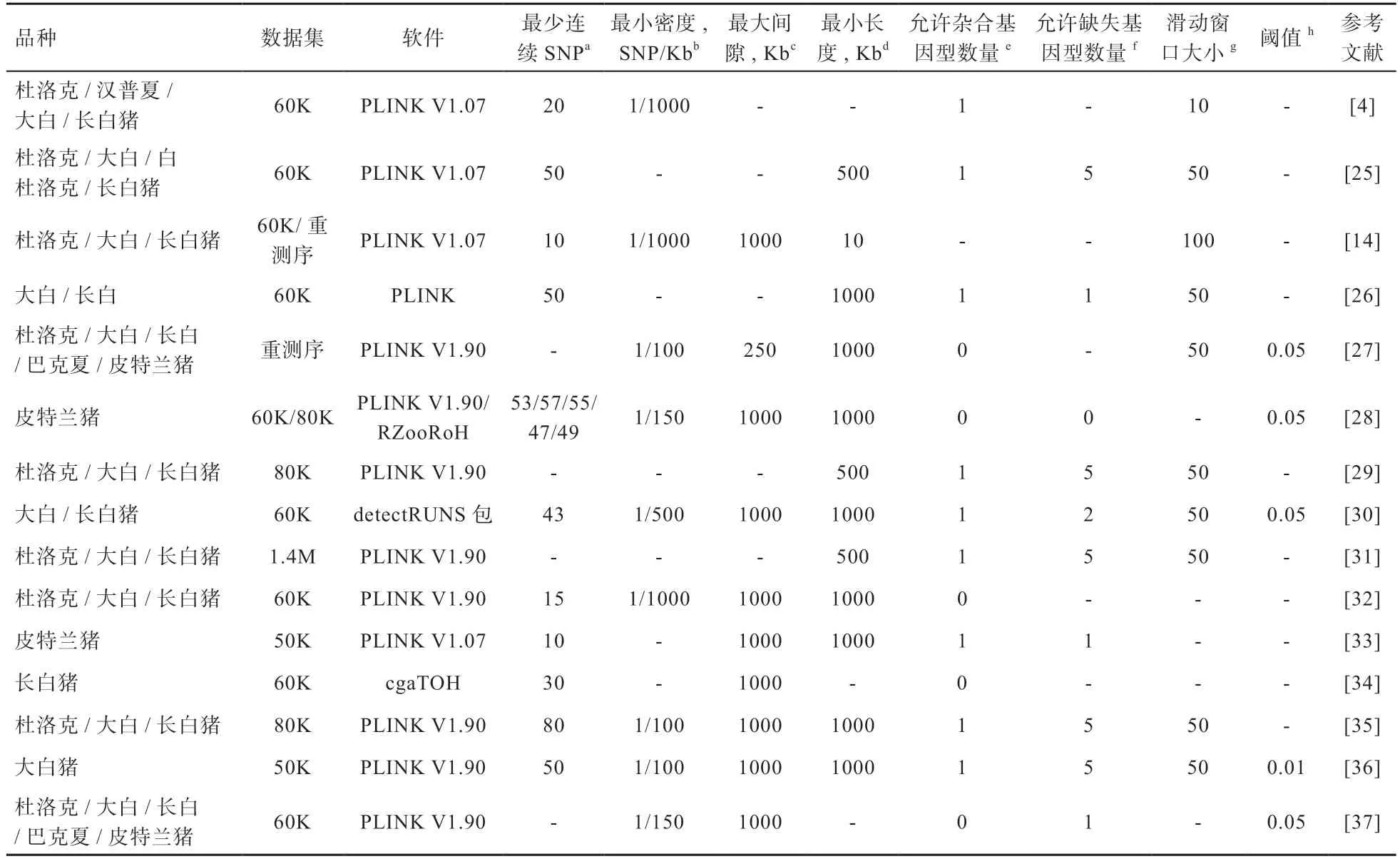
表1 不同瘦肉型猪品种ROH 识别和参数设置的比较
3 ROH 在瘦肉型猪基因组的应用
2010 年,Sölkner 等首次在牛基因组研究中报道ROH 后,越来越多学者对畜禽的ROH 进行了更深入的探索,尤其是瘦肉型猪ROH 的研究,包括遗传多样性分析、群体历史及近交系数的评估、选择信号的鉴别和不利ROH 检测等多个重点研究方向。
3.1 ROH 分析遗传多样性 基因组信息已被广泛用于评估畜禽的遗传多样性。经历过高强度人工选育的优良畜禽动物的表型变异减少,基因组单倍型的多样性降低,被选择位点周围的纯合性增加,出现ROH 模式,导致优良动物的遗传多样性降低、近交系数升高。
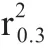
3.2 ROH 评估近亲繁殖水平 近交系数(Inbreeding Coefficient,F)是监测种群遗传变异和管理畜禽遗传资源的重要参数之一,准确地评估个体和群体的近交系数不仅是实际生产的切实需求,也是近交效应研究的重点。传统上,近交系数的估计是基于系谱信息(F),该方法度量的是个体任意位点上出现共享等位基因型的概率,估计值是固定的。F值是否能够真实反映群体的近交水平取决于个体系谱信息的完整性和准确性。然而,在实际生产中,由于各种因素导致系谱信息不完整或者记录错误的现象普遍存在,这将大大降低F估计值的可靠性。此外,F依赖于基础群信息,基于群体的基因组没有经历重组和个体不受选择的基础计算,也难以反映染色体中的纯合性,更不允许计算特定染色体区域的近交系数。
早期的低密度分子标记由于标记数量有限,可能存在抽样误差,导致估计值的准确性低。但随着重测序技术和高密度SNP 芯片的发展,DNA 分子信息显著地提高了评估近交系数的准确性。利用分子信息来评估群体的近交水平主要分为3 种方法:基于纯合子SNP 的近交系数(F);基于SNP 分子标记间的近交系数(F);基于ROH 的近交系数(F)。与F和F相比,F估计群体近交系数有以下几点优势:第一,可以区分是IBD 还是状态同源(Identical By State,IBS)。在使用单一分子标记计算估计近交水平时,难以区分IBD 和IBS,但使用ROH 却可以做到;第二,可以揭示近交发生的时期(近期还是遥远时期)。在减数分裂时重组事件可以打断较长的ROH 片段,因此ROH 的长度和距离共同祖先的世代数有很强的相关性。例如Shi 等检测到大白猪的近交水平可能是在近5 代积累导致,而且F和F之间的相关性比较低(0.18~0.37)。这个结果与其它瘦肉型猪的研究一致,并且F和F之间的相关性在不同长度的ROH没有明显变化。F不能区分IBD 和IBS,可能过高地估计了近交水平,除了与F之间的相关性较低外,与F和不同长度的F相关性都较高,这在皮特兰猪、杜洛克猪、长白猪以及不同品系大白猪的研究中都得到相同的结论。Zhan 等基于系谱信息和基因组信息评估了皮特兰猪的近交水平,F和F有显著的高相关性(r=0.949),F与F之间相关性较低,这也与Gorssen 等在其他皮特兰猪群体的报道一致,F和F都与F没有高相关性。这些结果表明F和F之间的相关性不高,可能是因为F和F应用的计算方法和原理不同,如F不能计算基因组中IBD 实际比例,也不能解释减数分裂期间重组的随机事件,这可能影响了计算的准确性,特别是没有或缺乏系谱信息时。同时基因组中某些单倍型可能是由于局部的低程度重组和高水平LD形成的。此外,相关研究表明短片段和中等长度片段的ROH 更有可能来自LD 或者遥远时期的近交事件,而通过排除LD 和随机效应的干扰,长片段ROH 估算的近交系数可以更准确地代表近期近交繁殖的水平。然而,不同群体的ROH 分类没有固定标准。因此,使用总长度的ROH 来估算近交水平是一个更为可行的方法。由评估瘦肉型猪和其他动物ROH 的相关研究表明,F可能是一种在理论上更有效和准确的度量畜禽近交水平的替代方法,被广泛用于评估个体和群体的近交水平。
3.3 ROH 识别人工选择的痕迹 欧洲家猪大约在9 000年前在近东被驯化,是最早被驯化的家畜之一。近一两百年来,随着人类对瘦肉型猪品种的需求和优良种畜的选育,不仅造成了瘦肉型猪外形和生长性状的变化,还在基因组中留下了选择的印迹。当基因组的特定区域受到长期、连续和高强度的正向选择,特别是人工选择,会表现出受选择区域的纯合频率迅速增加,甚至固定下来,出现ROH 片段。Bosse 等观察到,ROH在染色体中是非随机分布的,许多SNPs 分子标记在ROH 片段中具有异常频率,这些区域被称为“ROH 热点”或“ROH 岛”。因此,通过扫描基因组中的ROH 高频区域区段可以鉴定受到选择的基因组区域。
Gorssen 等对5 个不同品系皮特兰猪基因组进行ROH 分析,并与杜洛克猪、大白猪和长白猪进行比较,在8 号染色体上观察到一个90 Mb 的ROH 热点区域,约85%的个体存在共享ROH,著名的影响猪毛色的基因在这个区域也被鉴定到。此外,50~60 Mb区域处发现所有皮特兰品系、长白猪和大白猪存在的ROH 岛重叠。Zhan 等同样进行了皮特兰猪ROH分析,将16 个ROH 热点区域与猪数量性状基因座(Quantitative Trait Locus,QTL)进行比对,发现其与生长、肉质、胴体品质和繁殖等性状相关。同时,还鉴定到在生物学过程起关键作用的候选基因。Shi 等在大白猪基因组的4 个ROH 热点区域中检测到大量与繁殖相关的候选基因,这与大白猪繁殖力高、产仔数多的种质特性相一致。Gorssen 等利用全球共享的动物基因组数据进行了ROH 分析,观察到杜洛克猪在这些动物中ROH 热点区域最多,同时SNPs 发生频率也是最高的,很大部分超过80%,有些甚至达到100%。
经过长期的正向选择,控制重要经济性状的调控区域将高度纯合,这些特定区域也就更容易暴露在ROH高频区域。所以,观察到的ROH 高频区域有助于识别被选择的位点或区域,寻找与经济性状相关的候选基因,并应用于育种实践中。
3.4 检测不利ROH 单倍型 在瘦肉型猪育种工作中,重要经济性状的遗传增益是通过高强度选育实现的。然而,高强度选育会造成遗传多样性降低和近亲繁殖程度增加,而且可能会导致隐性有害等位基因的积累。这反过来又导致近交衰退,即平均表型性能的降低。近亲繁殖会产生ROH,大多数的ROH 对表型有中性甚至有利的影响,从而掩盖了少数不利的ROH 影响。由于近亲繁殖的模式在整个基因组中是不同的,因此,确定与表型相关的基因组ROH 区域可以使遗传增益和近亲繁殖水平之间得到更有效的平衡。有不少研究报道了ROHs 对经济性状会造成不利影响,但是没有考虑到不同ROH 片段对同一表型的影响和不同ROH 片段对多个表型的影响。Howard 等提出了一种能检测对表型不利的ROH,即能对个体内和个体间ROH 出现的联合效应进行描述的软件——Unfavorable Haplotype Finder。利用此软件在大白猪基因组中检测到13 个生长性状和繁殖性状相关的不利ROH(平均长度为1.54 Mb;长白猪中为4 个ROHs,平均长度为1.56 Mb),以及2 个品种中3 个与繁殖性状相关的共有区域。同时还指出LD、QTL 和ROH 在基因组中发生的频率对识别不利ROH 的能力产生很大影响。
4 小结与展望
ROH 分析是研究畜禽基因组特征的有效方法。本文针对瘦肉型猪基因组中ROH 在分析遗传多样性、评估近交繁殖水平和识别人工选择痕迹等应用进行了全面的综述。然而,在瘦肉型猪中有害突变的累积是否在ROH 出现的概率更高,以及ROH 是否与经济性状具有强相关性尚未有报道,仍需要进一步探究。
虽然目前对基因组中ROH 有很多研究,但仍存在许多科学问题值得进一步挖掘:①瘦肉型猪中ROH 的定义以及检测的参数和阈值缺乏一致的标准,很难直接比较不同研究的结果;②瘦肉型猪ROH 覆盖率高的ROH 区域是否存在有害突变,不同长度的ROH 中有害突变的频率是否有规律,对生长性状是否产生负面影响;③Unfavorable Haplotype Finder 软件既然能找出不利ROH 片段,那么是否可以能通过调整参数找到对瘦肉型猪最有利的ROH 片段或者杂合子片段(Runs of Heterozygosity,ROHet)以及不利的ROHet 片段,以用于现代育种计划。因此,关于瘦肉型猪基因组中ROH 和ROHet 仍需深入研究,进而为育种工作和解析瘦肉型猪遗传和表型差异的遗传机制提供理论依据。
