基于GEO数据库分析番茄干旱胁迫关键基因与信号通路
2022-07-26杨巍唐兵周麟笔马关鹏谭国飞瞿飞王天文曾庆鸿王洪亮邓英
杨巍,唐兵,周麟笔,马关鹏,谭国飞,瞿飞,王天文,曾庆鸿,王洪亮,邓英*
基于GEO数据库分析番茄干旱胁迫关键基因与信号通路
杨巍1,2,唐兵1,2,周麟笔1,2,马关鹏1,2,谭国飞1,2,瞿飞1,2,王天文1,2,曾庆鸿3,王洪亮3,邓英1,2*
1. 贵州省农业科学院园艺研究所, 贵州 贵阳 550000 2. 贵州省园艺工程技术研究中心, 贵州 贵阳 550006 3. 贵州省农业科技发展中心, 贵州 贵阳 550000
为研究干旱胁迫对番茄生长的影响,本研究利用生物信息学分析方法筛选番茄干旱胁迫的关键基因,通过检索GEO数据库中关于番茄干旱胁迫的基因芯片数据,获取GSE39894和GSE106317两个数据集矩阵数据,利用GEO2R分析工具进行差异表达基因筛选,应用DAVID在线数据库对差异表达基因进行GO功能分析和KEEG通路富集分析,运用String数据库和Cystoscopes软件构建差异表达基因的蛋白互作网络,并使用MCODE及Cytohubba插件筛选出参与干旱胁迫的最显著模块及关键基因。本实验筛选出1583个差异表达基因,其中748个上调基因,835个下调基因,GO功能分析和KEEG通路富集分析表明,这些差异基因在代谢通路、次生代谢产物的生物合成、植物激素信号转导、苯丙烷生物合成等方面显著富集,蛋白互作网络分析筛选出K4C9D8_SOLLC、K4B0Q1_SOLLC、CB13_SOLLC、PSBP_SOLLC、K4BCF4_SOLLC等10个关键基因,这些差异表达基因很可能是番茄干旱胁迫潜在的生物标志物。
GEO数据库; 番茄; 干旱胁迫; 生物信息学
番茄(L.或Mill.)属于茄科(Solanaceae),在世界范围内广泛种植[1],番茄果实热量低,富含番茄红素、维生素等营养物质,深受大众喜爱。然而番茄生长极易受到干旱等非生物胁迫的影响,导致减产与品质下降[2]。特别是贵州省地处云贵高原,厄尔尼诺现象频发,极易引起旱灾,对农业生产造成极为不利的影响[3]。从分子生物学水平研究干旱胁迫对番茄生长的影响,对番茄实际生产中提高其耐旱性具有实际积极意义。
GEO数据库全称为Gene Expression Omnibus (GEO, http://www.ncbi.nlm. nih.gov/geo/),是一个公共功能基因组数据存储库,收录了世界各国研究机构提交的高通量微阵列和高通量测序功能基因组数据集,并提供工具帮助用户查询和下载实验并策划基因表达谱,该数据库由美国国立生物技术信息中心(NCBI)创建并维护的基因表达数据库[4]。GEO数据库被广泛应用于生物医学领域,如肺鳞状细胞癌[5]、阿尔茨海默病[6]、乳头状甲状腺癌[7]、肺癌[8]等疾病的研究。而在植物逆境胁迫方面,Razieh R等分析了拟南芥的盐胁迫与干旱胁迫的基因芯片数据,HSF、AP2/ERF和C2H2三个转录因子家族可能在拟南芥的盐和干旱胁迫响应机制中起关键作用,增强拟南芥的耐盐性和耐旱性[9]。Tahmasebi A等整合棉花非生物胁迫GEO基因芯片数据,通过meta分析与系统生物学分析表明,差异表达基因主要在泛素依赖性过程、次生代谢产物的生物合成、植物激素和信号转导中显著富集[10]。由此可见利用生物信息学分析成为研究植物逆境胁迫的新思路。
本实验通过整合GEO数据库关于番茄干旱胁迫的基因芯片数据,使用DAVID数据库对筛选的差异表达基因(EDGs)进行GO功能分析和KEEG信号通路富集分析。利用string数据与Cytoscape软件构建蛋白互作网络(protein-protein interaction,PPI),利用MCODE及Cytohubba插件分析PPI网络,筛选出最显著模块与10关键基因,以期研究干旱胁迫对番茄生长发育的影响的分子机制。
1 方法与步骤
1.1 基因芯片数据获取及筛选
在基因表达数据库GEO(Gene Expression Omnibus)中,采用Search for Studies at GEO DataSets检索工具,以“drought stress”为关键字进行检索,然后以物种番茄与数据表达类型为“Expression profiling by array”进行筛选,并经人工筛选出数据集GSE39894(48个样本)与GSE106317(36个样本),二者均基于GPL4741平台,根据试验内容分别选取18个样本与6个样本进行分组分析。

表1 实验数据采集与分组
1.2 DEGs的筛选
采用GEO数据库中的在线分析工具Analyze with GEO2R进行在线分析,然后以<0.05与|logFC|≥2为标准进行基因数据筛选。
1.3 DEGs的GO分析与KEEG信号通路分析
利用在线数据库DAVID(https://david.ncifcrf.gov/)对DEGs进行GO功能分析与KEEG通路分析以及Gene ID转换,以<0.05为筛选标准。
1.4 蛋白互作网络构建
采用String数据库(https://www.string-db.org)对DEGs进行蛋白互作(PPI)网络分析,然后将分析结果导入Cytosacape3.8.2软件中进行调整与分析,构建PPI网络图。
1.5 最关键模块与关键基因的筛选
利用Cytosacape的MCODE插件筛选最关键模块,以Cytohubba插件筛选最关键模块的关键基因。
2 结果与分析
2.1 DEGs的筛选结果
采用GSE39894与GSE106317两个数据集的24个样本为研究对象,PCA分析见图1,利用Analyze with GEO2分析工具进行分析,该类型基因芯片(GPL4741平台)含有分子探针10209个,以<0.05与|logFC|≥2作为DEGs筛选标准,获得显著DEGs1583个,其中上调DEGs748个,下调DEGs835个,差异基因的分布火山图见图2。

图 1 样本PCA分析

图 2 DEGs火山图
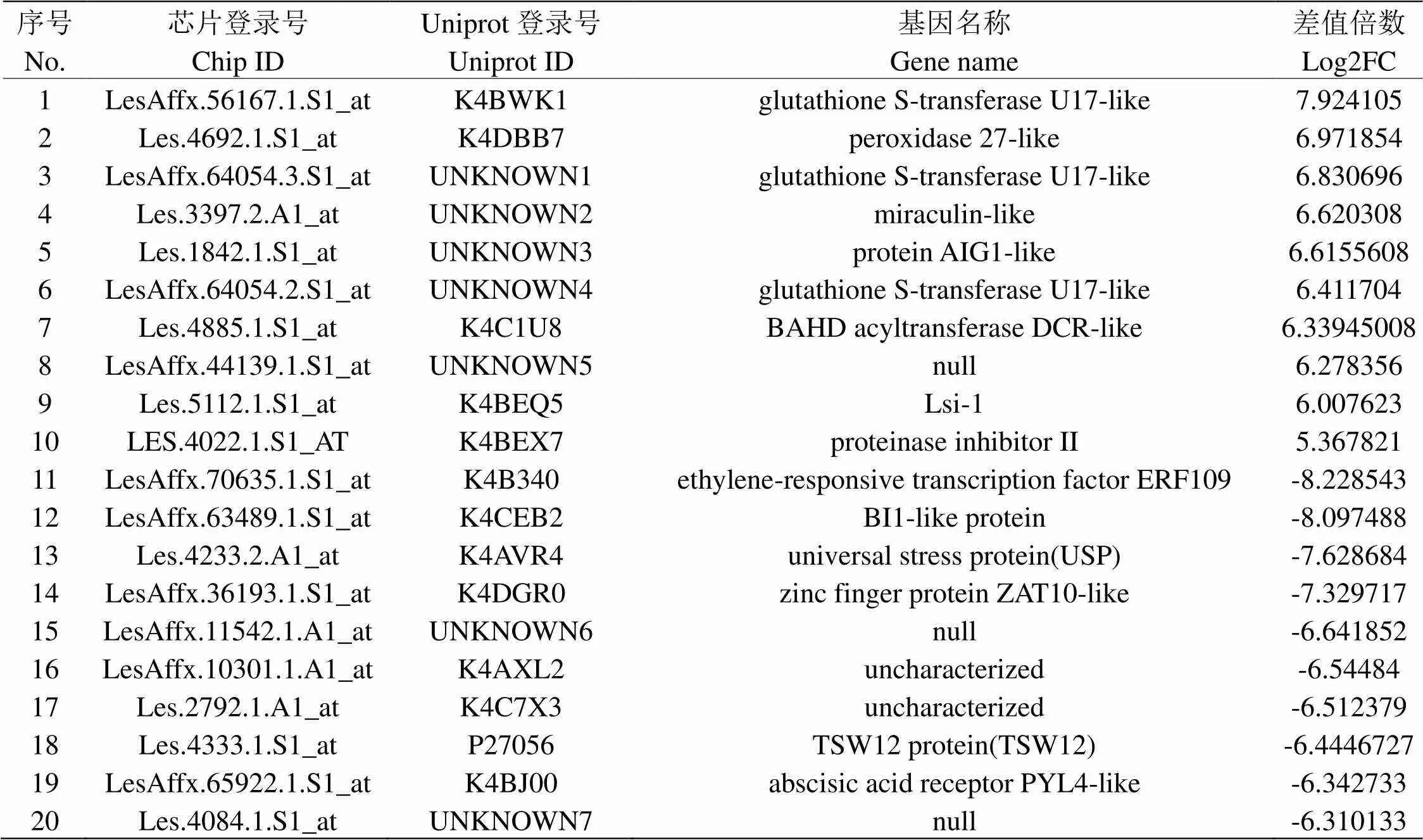
表2 前20显著DEGs

图 3 前20显著DEGs热图
2.2 DEGs的GO功能分析和KEEG信号通路分析
利用DAVID数据库对1583个DEGs进行GO功能分析与KEEG通路富集分析结果显示,这些DEGs主要参与了转录调控、氧化应激反应、过氧化氢分解代谢过程、细胞壁生物发生、防御响应等生物学过程;主要细胞成分位于细胞外区域、细胞壁、质外体、细胞膜等部位;主要分子功能涉及金属离子结合、转录活性因子、DNA结合、血红素结合、过氧化物酶活性等(图4)。KEEG信号通路分析显示,这些DEGs主要参与了代谢通路、次生代谢产物的生物合成、植物激素信号转导、苯丙烷类生物合成、淀粉与蔗糖的代谢等信号通路(图5)。
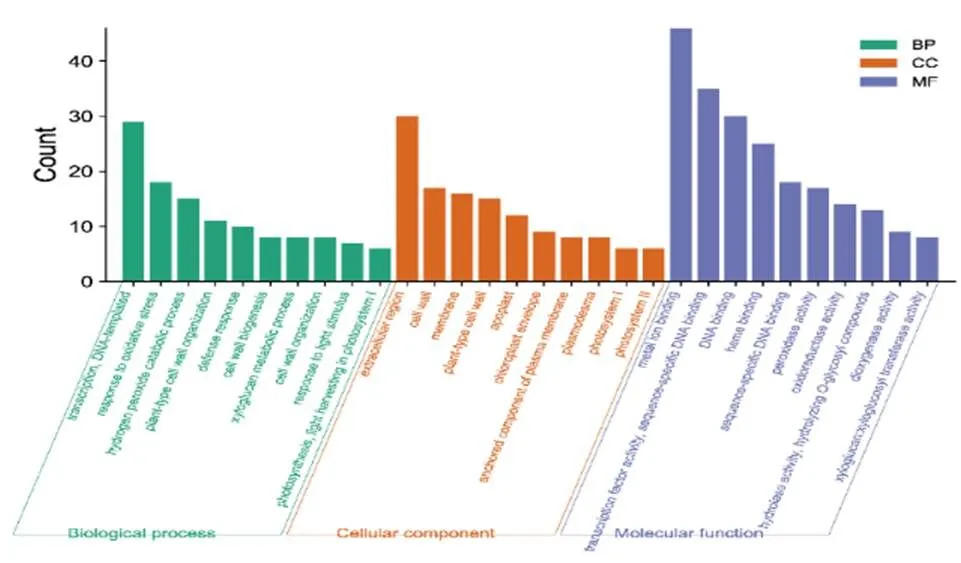
图 4 DEGs的GO功能富集图

图 5 DEGs的KEEG通路富集图

图 6 苯丙烷类生物合成途径(KEEG,map00940)
2.3 DEGs编码蛋白的PPI网络构建与关键基因的筛选

图 7 DEGs蛋白互作网络
注:a.利用string数据库与Cytoscape构建DEGs蛋白互作网络,b.利用MCODE插件从PPI网络中筛选出最显著模块,c.利用Cytohubba插件筛选最显著模块中10个关键基因。图a与图b上调DEGs标注为红色,下调DEGs标注为绿色。
Note: a. Use the STRING database and Cytoscape to construct protein interaction network of DEGs, b. Use the MCODE plug-in to screen out the most significant modules from the PPI network, c. Use the Cytohubba plug-in to screen the 10 key genes in the most significant modules. In Figures a and b, up-regulated DEGs are marked in red, and down-regulated DEGs are marked in green.
利用string数据库与Cytoscape软件对DEGs构建PPI网络,最终获得293个节点,581条相互作用的网络图(图7A)。通过Cytoscape中的MCODE插件从PPI网络筛选出最关键模块,设置参数为degree cutoff=2,node score cutoff=0.2,k-core=2和max. depth=100,最显著模块包含18个节点及117条相互作用(图7B),运用Cytohubba插件的MCC算法从最显著模块中筛选出排名前10位的关键基因,分别为K4C9D8_SOLLC、K4B0Q1_SOLLC、CB13_SOLLC、PSBP_SOLLC、K4BCF4_SOLLC、K4B7S8_SOLLC、K4CRD4_SOLLC、K4BLA0_SOLLC、K4CH43_SOLLC、K4BQ77_SOLLC(图7C,表3),这些关键基因的基因热图分析见图8。

表3 10个关键基因

图8 10个关键基因热图
3 讨 论
本实验基于GPL4741平台的2个数据集GSE39894与GSE106317的24个样本进行基因表达数据分析,最终筛选出DEGs1583个,其中上调DEGs748个,下调DEGs835个,通过分析前20个显著DEGs,发现表达量显著上调的K4BWK1、K4DBB7、UNKNOWN1、UNKNOWN4均与氧化还原代谢相关,特别是K4BWK1、UNKNOWN1、UNKNOWN4均参与了谷胱甘肽代谢过程,该代谢过程在植物体内具有抗氧化作用[11]。抗氧化作用基因表达上调可能是由于干旱胁迫引起细胞内ROS含量增加,这与Thirumalaikumar VP等的研究结论相一致[12]。UNKNOWN3(AIG1-like蛋白)类似于拟南芥中AIG1蛋白,是第一种被鉴定的IAN蛋白(immune-associated nucleotide-binding protein),可能在植物发育过程中发挥调节作用以及对生物和非生物胁迫的应答[13]。K4C1U8作为BSDH酰基转移酶家族成员,该酶类是植物中所特有的,参与多种植物次生代谢物的合成和修饰,如类黄酮、生物碱、萜类、多胺、花青素和挥发性酯类的酰基化修饰等,其介导的酰基化修饰在天然产物结构改造中普遍存在,对丰富植物次生代谢产物结构多样性,提高化合物的稳定性、脂溶性、改善生物利用度等方面均有重要意义[14,15]。K4BEQ5功能类似于水稻的硅内流转运蛋白,作为载体将硅从外部溶液转运到根细胞,硅有利于植物生长,提高植物抗生物和非生物胁迫能力[16,17]。而在显著下调DEGs中,K4B340作为乙烯响应因子,参与植物防御反应与乙烯激活信号通路,乙烯在植物的生物与非生物胁迫应答中具有重要作用。K4CEB2作为细胞膜的组成部分,表达量下调可能意味着细胞膜受损,K4AVR4被Yang ML等研究证明能显著提高海棠的耐旱性[17],K4DGR0作为锌指蛋白,对基因调控起着重要作用。K4BJ00是脱落酸(ABA)受体,参与激活ABA通路,ABA是重要的植物抗胁迫激素[18]。这些DEGs的显著表达说明干旱胁迫引起植物体内氧化胁迫,导致ROS上升破坏细胞膜结构,对抗环境胁迫的应激响应通路受阻,同时植物体内启动抗氧化与防御机制起到自我保护的作用。
通过对DEGs的GO功能富集分析表明,在转录调控、氧化应激反应、过氧化氢分解代谢过程、植物类细胞壁、防御反应等生物学过程中的相关基因参与了干旱胁迫的过程。KEEG信号通路富集分析结果显示,代谢通路、次生代谢产物的生物合成、植物激素信号转导、类苯基丙酸合成、淀粉与蔗糖的代谢等代谢途径均受到干旱胁迫的影响,以苯丙烷类生物合成途径(图6)为例,它是是植物3条主要次生代谢途径之一,丙氨酸经多步催化反应生成4-香豆酸辅酶A,进入特异性合成途径转化成不同的苯丙烷类代谢产物,如香豆素、类黄酮、萜类、木质素、花青素等,在植物的生长发育过程及应答逆境胁迫中发挥重要作用[19]。DEGs的PPI网络分析,筛选出10个关键DEGs均属于叶绿体蛋白,说明干旱胁迫直接影响了植物的光合作用。
本研究基于GEO数据库的基因芯片数据,使用生物信息学方法分析了番茄在干旱胁迫下的DEGs,通过GO功能富集与KEEG信号通路富集,在一定程度上阐明了干旱胁迫引起番茄代谢变化,由于生物信息学本身存在一定局限性,在后续的研究中,需要运用Q-PCR、Western Blot等实验技术对关键DEGs做进一步分析验证,同时结合蛋白质组学与代谢组学研究番茄干旱胁迫的基因表达谱,有助于从分子生物学水平更全面深入地分析干旱胁迫对番茄生长的影响。
[1] Klee HJ, Giovannoni JJ. Genetics and control of tomato fruit ripening and quality attributes [J]. Annual Review of Genetics, 2011,45:41-59
[2] Iovieno P, Punzo P, Guida G,. Transcriptomic changes drive physiological responses to progressive drought stress and rehydration in Tomato [J]. Front Plant Sci, 2016,7:371
[3] Lin Y, Liu F, Xu P. Effects of drought on infant mortality in China [J]. Health Economics, 2021,30(2):248-269
[4] Barrett T, Wilhite SE, Ledoux P,. NCBI GEO: archive for functional genomics datasets—update [J]. Nucleic Acids Research, 2013,41:991-995
[5] Li Y, Gu J, Xu FK,. Transcriptomic and functional network features of lung squamous cell carcinoma through integrative analysis of GEO and TCGA data [J]. Sci Rep, 2018,8:15834
[6] Ma GW, Liu MY, Du K,. Differential expression of mRNAs in the brain tissues of patients with Alzheimer's Disease based on GEO expression profile and its clinical significance [J]. BioMed Research Internationa, 2019(1):1-9
[7] Wan Y, Zhang XL, Leng HL,. Identifying hub genes of papillary thyroid carcinoma in the TCGA and GEO database using bioinformatics analysis [J]. Peer J, 2020,8(11):e9120
[8] Zhao J, Guo C, Ma Z,. Identification of a novel gene expression signature associated with overall survival in patients with lung adenocarcinoma: A comprehensive analysis based on TCGA and GEO databases [J]. Lung Cancer, 2020, 149:90-96
[9] Ghorbani R, Alemzadeh A, Razi H. Microarray analysis of transcriptional responses to salt and drought stress in[J]. Heliyon, 2019,5(11):e02614
[10] Tahmasebi A, Ashrafi-Dehkordi E, Shahriari AG,Integrative meta-analysis of transcriptomic responses to abiotic stress in cotton [J]. Progress in Biophysics and Molecular Biology, 2019,146:112-122
[11] Bachhawat AK, Yadav S. The glutathione cycle: Glutathione metabolism beyond the γ-glutamyl cycle [J]. Iubmb Life, 2018,70(7):585-592
[12] Thirumalaikumar VP, Devkar V, Mehterov N,. NAC transcription factor JUNGBRUNNE- N1 enhances drought tolerance in tomato [J]. Plant Biotechnology Journal, 2018,16:354-366
[13] Wang ZJ, Li X. IAN/GIMAPs are conserved and novel regulators in vertebrates and angiosperm plants [J]. Plant Signal Behav, 2009,4(3):165-167
[14] Panikashvili D, Shi JX, Schreiber L,. The Arabidopsis DCR encoding a soluble BAHD acyltransferase is required for cutin polyester formation and seed hydration properties [J]. Plant Physiol, 2009,151(4):1773-1789
[15] Manjasetty BA, Yu XH, Panjikar S,. Structural basis for modification of flavonol and naphthol glucoconjugates bymalonyltransferase (NtMaT1) [J]. Planta, 2012,236:781-793
[16] Ma JF, Tamai K, Yamaji N,A silicon transporter in rice [J]. Nature, 2006,440(7084):688-691
[17] Yang ML, Che SY, Zhang YX,. Universal stress protein inconfers enhanced drought tolerance [J]. J Plant Res, 2019,132(6):825-837
[18] Gaudet P, Livstone MS, Lewis SE,. Phylogenetic-based propagation of functional annotations within the Gene Ontology consortium [J]. Brief Bioinform, 2011,12(5):449-462
[19] Vogt T. Phenylpropanoid biosynthesis [J]. Molecular Plant, 2010,3(1):2-20
Analysis of Key Expressed Genes and Pathways in Tomato under Drought Stress Based on GEO Database
YANG Wei1,2, TANG Bing1,2, ZHOU Lin-bi1,2, MA Guang-peng1,2, TAN Guo-fei1,2, QU Fei1,2, WANG Tian-wen1,2, ZENG Qing-hong3, WANG Hong-liang3,DENG Ying1,2*
1.550000,2550006,3.550000,
In order to study the effect of drought stress on tomato growth, the key genes of drought stress in tomato were screened by bioinformatics analysis in this study. Retrieving the gene chip data of tomato under drought stress through GEO database, the matrix data of two datasets GSE39894 and GSE106317 were obtained. The differential expression genes were screened through GEO2R. The GO function and KEEG pathway enrichment analysis of differential expression genes were performed through DAVID online database. String database and Cytoscape software were used to construct protein interaction network of differentially expressed genes. The most significant modules and key genes involved in drought stress were screened by using MCODE and Cytohubba plug-in. In this study, 1583 differentially expressed genes were screened, including 748 up-regulated genes and 835 down-regulated genes. GO functional and KEEG pathway enrichment analysis showed that these differential genes were significantly enriched in metabolic pathways, biosynthesis of secondary metabolites, plant hormone signal transduction, and phenylpropanoid biosynthesis,10 key genes such as K4C9D8 _ SOLLC, K4B0Q1 _ SOLLC, CB13 _ SOLLC, PSBP _ SOLLC, K4BCF4 _ SOLLC were screened through protein interaction network analysis. These differentially expressed genes are likely to be potential biomarkers of drought stress in tomato.
GEO database; tomato; drought stress; bioinformatics
S641.2
A
1000-2324(2022)03-0355-07
10.3969/j.issn.1000-2324.2022.03.003
2021-06-17
2022-06-18
贵州省科技计划项目(黔科合支撑[2020]1Y090号);国家自然科学基金(31960595);贵州省基层农技推广体系改革与建设补助项目(GZNJTG-WN-2022-01);贵州省蔬菜现代农业产业技术体系项目(GZCYTX2022-01);贵州省农科院资源专项(黔农科院种质资源[2022]03号);贵阳市蔬菜种质资源研究中心建设项目(筑科合同[2021]5-1号)
杨巍(1985-),男,硕士,助理研究员,主要从事蔬菜育种与栽培. E-mail:yangwei139@sina.cn
Author for correspondence. E-mail:87928883@qq.com
