益生菌大肠埃希菌Nissle1917 鞭毛蛋白的结构分析及表位预测
2022-06-23杨颖邓永军张进区炳明朱国强文明
杨颖 ,邓永军 ,张进 ,区炳明 ,朱国强 ,文明
1.贵州大学动物科学学院,贵州 贵阳 550025;2.贵州省动物生物制品工程技术研究中心,贵州 贵阳 550025;3.肇庆学院生命科学学院,广东 肇庆 526061;4.扬州大学兽医学院,江苏 扬州 225009
大肠埃希菌Nissle 1917(EcN)是革兰阴性益生菌[1]。研究发现,EcN 对常见的胃肠道炎症疾病有较好的治疗效果[2-3],其作用机制在于EcN 能在人体肠道定殖,并阻止一些病原菌对肠道黏膜的侵袭,能保护和修护肠道黏膜屏障[4-5]。动物试验表明,EcN 可应用于治疗新生犊牛的腹泻疾病[6-7],还可用于构建益生菌活疫苗,预防F4+及F18+大肠埃希菌所致仔猪断奶后腹泻和水肿疾病等[8]。近年研究发现,EcN 还具有肿瘤靶向作用,是良好的药物载体,且与化疗药物联用可增强药物抗肿瘤的疗效[9-10]。
细菌鞭毛蛋白(FliC)由 D0、D1、D2、D3 4 个结构域组成,其中D0 和D1 是FliC 的保守区,位于蛋白序列的-NH3端和-COOH 端,由α 螺旋结构组成;D2 和D3 为其高变区,位于蛋白序列中部,由β 折叠构成。FliC 作为一种病原体相关分子模式(pathogen-associated molecular pattern,PAMP),通过结合细胞表面的Toll 样受体5(Toll-like receptor 5,TLR5)激活下游信号通路,从而激发固有和获得性免疫应答[11-13]。有研究报道,FliC 佐剂活性与触发TLR5 信号的能力有关,TLR5 靶序列位于FliC D1 保守区域[14];也有研究表明,D0 区域也参与了鞭毛蛋白与TLR5 的相互作用,但作用机制尚未明确[15];FliC 的 D2 和 D3 区域序列在不同种属细菌中高度可变,可诱导机体产生针对FliC 的抗体。有研究表明,FliC 高变区是其发挥黏膜佐剂活性的重要区域[16],也有研究表明,缺失FliC 高变区不影响其佐剂活性[17]。FliC 自身结构赋予其高效灵活的佐剂活性,通过调节结构与其佐剂活性的关系以适用于不同类型疫苗的研发,是目前国内外研究的热点[18-21]。本研究通过生物信息学方法分析EcN FliC的基本理化特性,对其结构及优势B 和T 细胞表位进行分析预测,为后续进一步研究其佐剂特性及应用奠定基础。
1 材料与方法
1.1 氨基酸序列 通过GenBank 数据库获得EcN FliC 氨基酸序列(AXY44884.1),序列如下:MAQVINTNSLSLITQNNINKNQSALSSSIERLSSGLRINSAKDDAAGQAIANRFTSNIKGLTQAARNANDGISVAQTTEGALSEINNNLQRIRELTVQASTGTNSDSDLDSIQDEIKSRLDEIDRVSGQTQFNGVNVLAKDGSMKIQVGANDGQTITIDLKKIDSDTLGLNGFNVNGSGTIANKAATISDLTAAKMDAATNTITTTNNALTASKALDQLKDGDTVTIKADAAQTATVYTYNASAGNFSFSNVSNNTSAKAGDVAASLLPPAGQTASGVYKAASGEVNFDVDAN -GKITIGGQEAYLTSDGNLTTNDAGGATAATLDGLFKKAGDGQSIGFNKTASVTMGGTTYNFKTGADAGAATANAGVSFTDTASKETVLNKVATAKQGTAVAANGD -TSATITYKSGVQTYQAVFAAGDGTASAKYADNTDVSNATATYTDADGEMTTIGSYTTKYSIDANNGKVTVDSGTGTGKYAPKVGAEVYVSANGTLTTDATSEGTVTKDPLKALDEAISSIDKFRSSLGAIQNRLDSAVTNLNNTTTNLSEAQSRIQDADYATEVSNMSKAQIIQQAGNSVLAKANQVPQQVLSLLQG。
1.2 EcN FliC 理化性质的分析 应用ProtParam 在线工具(https: / / web.expasy.org / protparam /)对 EcN FliC 理化特性进行分析,包括氨基酸数、相对分子质量,理论pI、正负电荷残基数,原子组成,原子总数和消光系数等。
1.3 EcN FliC 亲水性的分析 应用Protscale 在线工具(https://web.expasy.org /protscale /)对 EcN FliC亲疏水性进行分析。
1.4 EcN FliC 二级结构的分析 应用PSIPRED 软件(http: / / bioinf.cs.ucl.ac.uk / psipred /)及DNAstar 软件中的Protean 程序(包括Garnier-Robson 和Chou-Fasman 两种方法)预测分析 EcN FliC 的二级结构[22-23]。
1.5 EcN FliC 三级结构的分析 应用I-TASSER 在线软件(https: / / zhanglab.ccmb.med.umich.edu / ITASSER /)预测EcN FliC 的三级结构[24]。
1.6 EcN FliC B 及T 细胞表位的分析 应用在线DNAstar 中 Protean 软件(Kyte-Doolittle 法预测蛋白质的亲水性,Emini 法预测蛋白质的表面可及性,Karplus-Eisenberg 法预测蛋白质的柔韧性,Jameson-Wolf 法预测蛋白质的抗原指数)和ABCpred 软件综合预测EcN FliC 的B 细胞抗原优势表位;利用细胞毒性T 细胞(cytotoxic T cell,CTL)表位预测网站(http: / / www.imtech.res.in / raghava / propred /)预测EcN FliC 的T细胞表位[25]。
2 结 果
2.1 EcN FliC 的理化性质 EcN FliC 的相对分子质量为60 937.57,分子式为C2584H4201N745O940S6,理论pI 为4.68,脂肪氨基酸指数为76.74,不稳定指数为15.23,属于稳定蛋白。EcN FliC 由595 个氨基酸组成,包含17 种氨基酸,其中Ala 含量最高,为14.3%;其次是Thr,含量为12.3%;Met 和Pro 含量较低,分别为1%和0.8%;不包含Cys、His 和Trp。见图1。
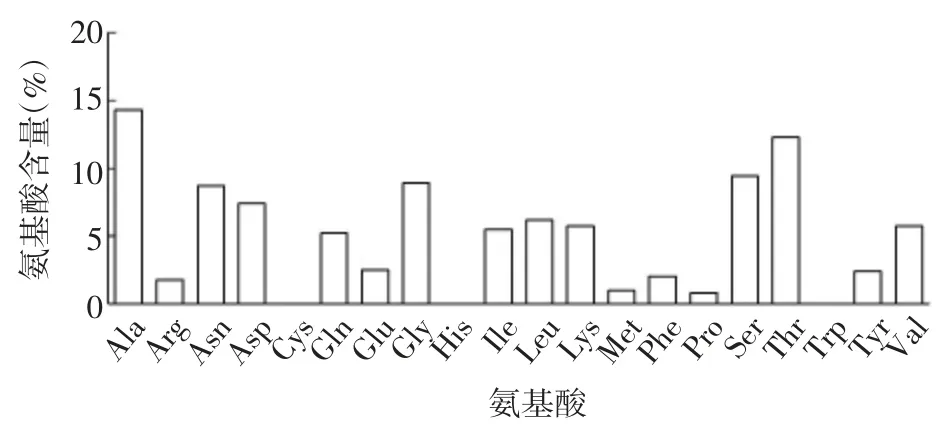
图1 EcN FliC 的氨基酸组成Fig.1 Animo acid composition of EcN FliC
2.2 EcNFliC 的亲水性 EcN FliC 带负电荷的氨基酸残基(Asp+Glu)共59 个,带正电荷的氨基酸残基(Arg+Lys)共45 个,亲水性平均值为-0.324,预测为亲水性蛋白。序列中第19 位Asn 亲水性最强,分值为-2.356;第267 位的Leu 疏水性最强,分值为1.467。见图2。

图2 EcN FliC 的亲水性分析Fig.2 Analysis of hydrophilicity of EcN FliC
2.3 EcN FliC 的二级结构 PSIPRED 分析表明,EcN FliC 的二级结构主要包括 α 螺旋、β 折叠、无规则卷曲3 种结构类型,其中α 螺旋占42.86%,β 折叠占12.77%,无规则卷曲占44.37%。二级结构中α螺旋和无规则卷曲结构占据整个蛋白的大部分区域,是EcN FliC 的主要结构。见图3。Garnier-Robson 及Chou-Fasman 法进一步预测EcN FliC 的二级结构表明,α 螺旋主要位于 9 ~ 32、45 ~ 98、104 ~ 127、505 ~592 等区段,β 折叠主要位于 2 ~ 6、134 ~ 139、143 ~151、155 ~ 161、225 ~ 229、234 ~ 240、296 ~ 300、410 ~ 417、438 ~ 444、445 ~ 450、466 ~ 470、485 ~489 等区段,无规则卷曲主要位于 101 ~ 105、150 ~ 154、251 ~ 286、315 ~ 321、332 ~ 335、396 ~ 403、443 ~ 446、497 ~ 501 等区段。见图4。

图3 PSIPRED 软件分析EcN FliC 的二级结构Fig.3 Analysis of secondary structure of EcN FliC by using PSIPRED software
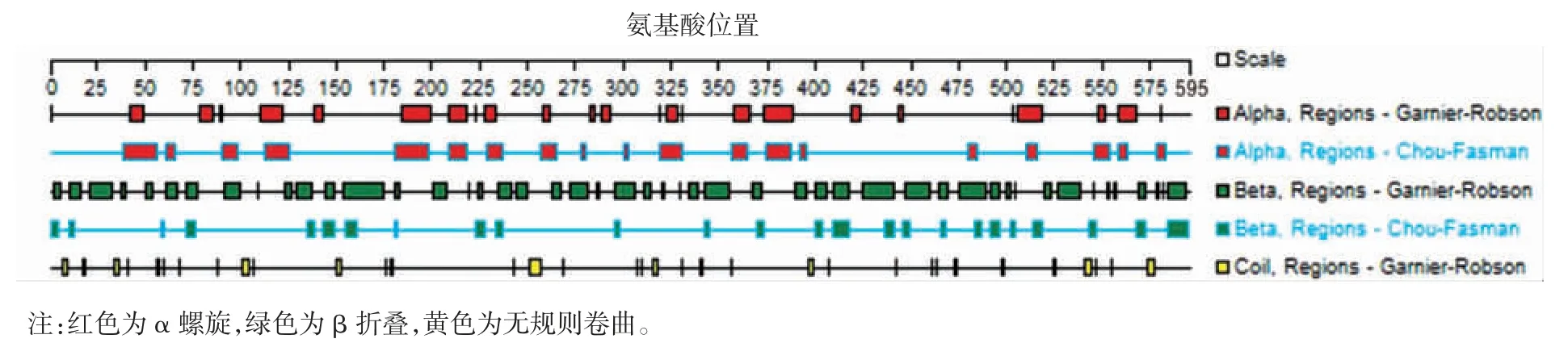
图4 DNAStar 软件分析EcN FliC 的二级结构Fig.4 Analysis of secondary structure of EcN FliC by DNAStar software
2.4 EcN FliC 的三级结构 EcN FliC 三级结构模型质量评估系数C-score 区间为-5 ~2,分值越高模型可信度越高;TM-score 代表结构相似度系数分值>0.5,表明模型具有正确的结构拓扑,可信度高。选择C-score 值最高且TM-score >0.5 的模型作为预测模型。见图5。

图5 EcN FliC 的三级结构预测Fig.5 Prediction of tertiary structure of EcN FliC
2.5 EcN FliC 的 B 细胞表位预测 DNAstar 软件预测结果表明,含B 细胞优势表位区域主要位于第9 ~19、35 ~ 50、89 ~ 104、157 ~ 178、251 ~ 265、282 ~ 301、302 ~ 322、327 ~ 339 位氨基酸,见图 6。在线软件ABCpred 预测结果表明,得分在0.68 以上的B 细胞优势表位区域见表1。结合两种方法综合筛选EcN FliC 的优势 B 细胞表位 13 个,分别为 9 ~ 18、39 ~ 48、80 ~ 89、93 ~ 102、122 ~ 131、157 ~ 166、217 ~ 226、254 ~ 263、283 ~ 292、288 ~ 297、294 ~ 303、312 ~ 321、332 ~ 341。
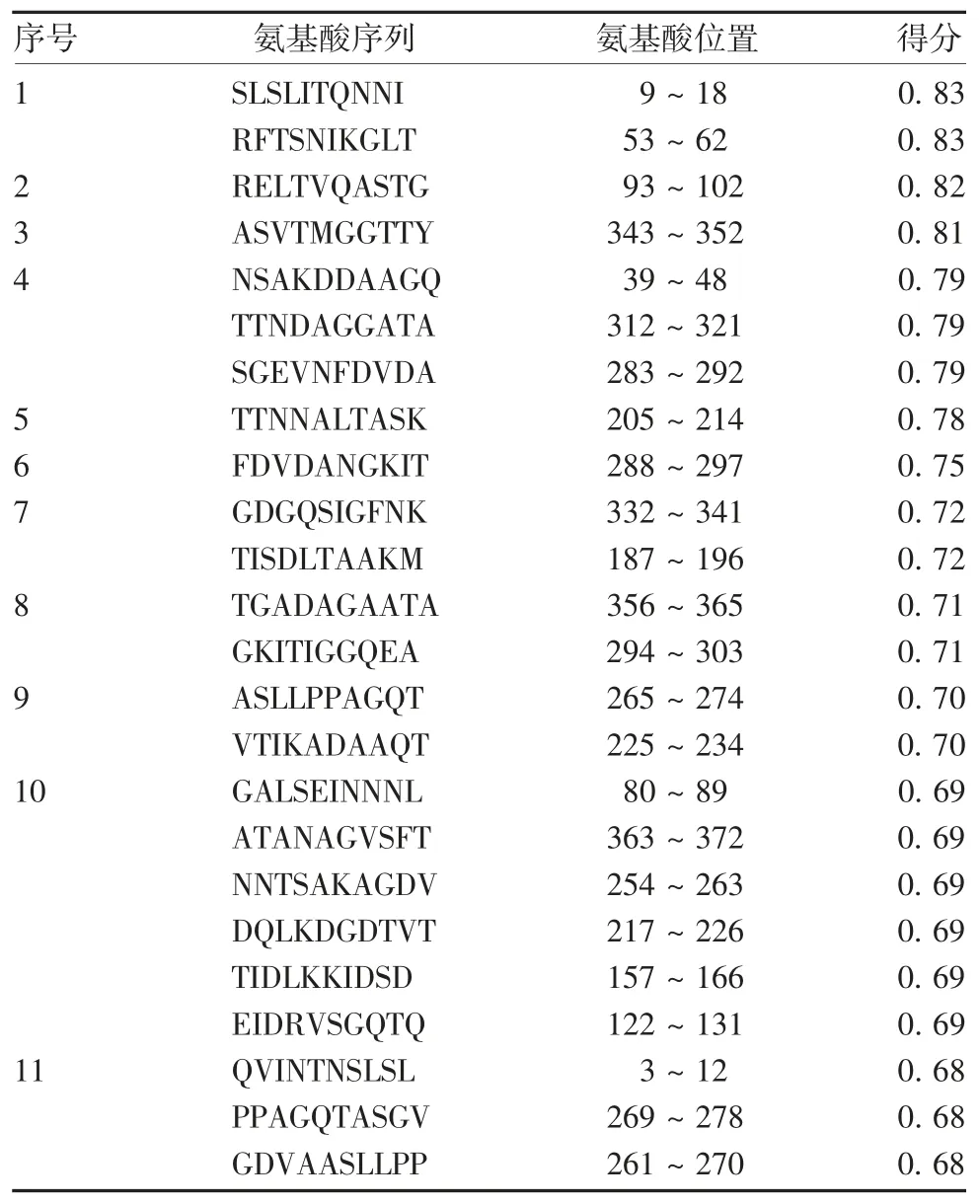
表1 ABCpred 软件预测EcN FliC B 细胞表位Tab.1 Prediction B cell epitopes of EcN FliC by ABCpred software
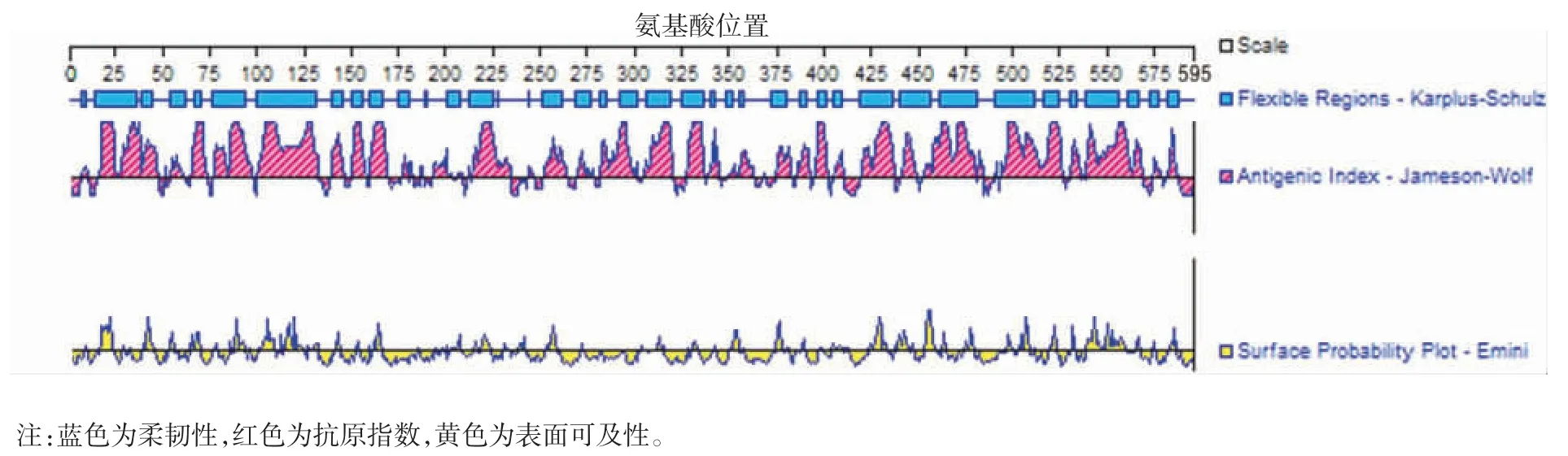
图6 EcN FliC 的柔韧性、抗原指数和表面可及性预测Fig.6 Prediction of flexibility,antigen coefficient and surfaceprobability of EcN FliC
2.6 EcN FliC 的T 细胞表位预测 CTL 表位预测网站预测结果表明,EcN FliC 有3 个CTL 优势表位,分别为NTSAKAGDV(255 ~264 位氨基酸)、TANAGVSFT(364 ~ 373 位氨基酸)和 VSANGTLTT(487 ~ 496 位氨基酸),得分均为1。
3 讨 论
鞭毛为细菌的运动器官,其丝状部由成百上千个FliC 组成,FliC 的佐剂活性是目前研究的热点,这有赖于该蛋白的结构特性,即蛋白两端高度保守区和中部高变区,不同区域对FliC 佐剂活性的作用尚未明确[14-17]。不同种属细菌的FliC 氨基酸组成差异较大[26-27],针对不同细菌FliC 的特性进行分析,以进一步研究其佐剂活性,开发高效佐剂产品。
本研究以益生菌EcN FliC 作为切入点,利用生物信息学方法和相关软件对EcN FliC 的氨基酸序列进行分析,分析结果表明,EcN FliC 为酸性、亲水性蛋白,总平均亲水性值为-0.324;二级结构中α 螺旋和无规则卷曲结构占比最多,β 折叠结构占比最少。由于EcN FliC 氨基酸序列与数据库中已有的模板序列同源度低,未能获取可信度高的预测结果,因此通过穿线法预测EcN FliC 三级结构模型,结果表明,EcN FliC 三级结构模型质量评估系数C-score 区间为-5 ~2,TM-score 代表结构相似度系数分值 >0.5,表明模型可信度较高,该结果与沙门菌FliC 的晶体结构相似[28]。
本研究预测B 细胞优势表位时,将EcN FliC 二级结构数据与亲水性区域、柔韧性区域、抗原指数及表面可及性等预测结果作为参数,结合ABCpred 软件预测结果,分析出具有更高可信度的B 细胞优势表位13 个,同时预测出3 个CTL 优势表位。FliC 的内在抗原性会导致特异性抗体的产生,进而在连续性治疗过程中阻碍其佐剂活性,这也成为FliC 临床开发和应用的障碍[29]。因此,今后将进一步研究FliC佐剂活性与抗原性相关决定簇之间的相关性。
