脲酶基因挖掘及其在枯草芽孢杆菌中的重组表达
2022-06-06景晓冉毕家华伍伦杰
周 霞,景晓冉,顾 洁,毕家华,伍伦杰,聂 尧,徐 岩
(江南大学 生物工程学院,江苏 无锡214122)
氨基甲酸乙酯(ethyl carbamate或urethane,简称EC),是一种存在于发酵食品和乙醇饮料(奶酪、酱油;黄酒、清酒、威士忌)中的氨类危害物[1-4]。2007年,国际癌症研究机构IARC(International Agency for Research on Cancer)正式将EC上升为2A类致癌物[5],与丙烯酰胺同等危险,引起人们对其安全的广泛关注。研究表明,尿素是黄酒中与乙醇反应生成EC的最主要前体物质。因而,去除黄酒中EC的主要策略是直接降低EC的含量和间接降低前体物质尿素的含量[6]。脲酶(Urease,EC 3.5.1.5)[7]因其不需更换酵母或生产工艺、直接高效的特性在解决黄酒中EC的食品安全问题中占据优势。
目前,已报道的脲酶通常来自发酵乳杆菌[8]、运动节杆菌[9]、肠杆菌属[10-11]、罗伊氏乳杆菌[12]、副地衣芽孢杆菌[13-15]、葡萄球菌[16]等,然而,这些微生物来源的脲酶基因簇的基因组成较为复杂,编码相应脲酶的活化过程需要所有辅助亚基UreE、UreF、UreG、UreD/UreH的参与,缺一不可,且相关辅助亚基的功能并不清晰。而来源于解淀粉芽孢杆菌[17]的基因簇通常包含UreA、UreB、UreC共3个结构亚基,基因异源表达及调控的可行性较高,然而,已报道来源于解淀粉芽孢杆菌B.amyloliquefaciensJP-21的脲酶基因簇仅在大肠杆菌系统中重组表达,难以用于传统发酵食品加工体系。
为达到食品安全应用的要求,脲酶基因的异源表达备受关注。枯草芽孢杆菌是当前研究得最好的食品安全级微生物(GRAS)[18-20],已被广泛用于生产各种工业酶。植酸酶[21]等在枯草系统中得以高效生产,但枯草系统用于表达重组脲酶的研究不多。当前仅有Liu等人[15]在枯草芽孢杆菌中实现了副地衣芽孢杆菌ATCC 9945a中含铁脲酶(Bp_Urease)的生产,摇瓶发酵酶活力只达到1 307 U/L。本研究在枯草芽孢杆菌中实现了基因挖掘所得的解淀粉芽孢杆菌IT-45来源的脲酶基因簇(ureABC)的表达,并通过密码子优化、核糖体结合位点优化重构脲酶基因簇以及调整基因ureC的位置来进一步提升脲酶活力,在酶法降解黄酒中的尿素应用上具有潜力。
1 材料与方法
1.1 实验材料
1.1.1 菌株及质粒E.coliJM109、B.subtilisWB600:分别为作者所在实验室保存的克隆和表达宿主菌株;pP43NMK:作者所在实验室保存的质粒;脲酶基因簇Ba-urease:从B.amyloliquefaciensIT-45全基因组(GenBank:CP004065.1)中获得。
1.1.2 主要试剂 蛋白胨、酵母粉:购自美国Oxoid公司;NaCl、K2HPO4·3H2O、KH2PO4、Na2HPO4·12H2O、NaH2PO4·2H2O、NaOH、NaClO、苯酚、亚硝基铁氰化钠、甘油、尿素、三氯乙酸、柠檬酸、柠檬酸钠:购自国药集团化学试剂公司;甲醇 (HPLC)、乙腈(HPLC):购自上海阿达玛斯试剂有限公司;PrimeSTAR®MAX DNA Polymerase、QuickCut Enzyme、Marker:购自宝生物工程(大连)有限公司;ClonExpress®II One Step Cloning Kit:购自南京诺唯赞生物科技有限公司。
显色剂I:苯酚15 g,亚硝基铁氰化钠0.625 g,用超纯水定容至250 mL。
显色剂II:NaOH 13.125 g,NaClO 7.5 mL,用超纯水定容至250 mL。
1.1.3 主要仪器 超声波细胞破碎仪:南京新辰生物科技有限公司;DK-S34型水浴锅:上海森信实验仪器有限公司;Five Easy Plus型pH计:上海METTLER TOLEDO公司;C1000 Touch PCR仪:美国Bio-rad公司;核酸电泳装置:美国Bio-Rad公司;AlphaImager Mini凝胶成像仪:美国Protein Simple公司;CYTATION3酶标仪:美国BioTek公司;Agilent 1260 InfinityⅡ型高效液相色谱仪:美国安捷伦科技公司。
1.2 实验方法
1.2.1 基因挖掘 采用NCBI(https://www.ncbi.nlm.nih.gov/)的BLAST分析工具以及Clustal Omega软件(https://www.ebi.ac.uk/Tools/msa/clustalo/)对脲酶编码基因进行核苷酸序列比对、氨基酸序列比对。采用iTOL v5(https://itol.embl.de/itol.cgi)对序列比对文件进行系统发育树分析。比较不同来源的脲酶基因结构,选定研究出发的基因序列。
1.2.2 重组菌的构建与表达 构建质粒所用的引物见表1。以基因合成所得的包含B.amyloliquefaciensIT-45中脲酶基因簇的质粒为模板,用引物U-F/U-R扩增脲酶基因簇Ba-urease(ureABC)。以质粒pP43NMK为模板,用引物P-F/P-R进行质粒线性化。利用ClonExpress®II One Step Cloning Kit将经胶回收后的基因片段Ba-urease和线性化质粒pP43NMK进行连接,并转化至E.coliJM109,挑取转化子并验证正确,获得质粒pP43NMK-Ure。

表1 质粒构建所用引物Table 1 Primers used in plasmid construction
为强化脲酶的表达,通过密码子优化(http://www.kazusa.or.jp/codon/)以及核糖体结合位点(RBS)优 化(https://salislab.net/software/)重 新 组 装所有基因,获得基因Ba-urease-1。为了对结构亚基UreC进行强化表达,将ureC移置至ureA和ureB的上游,从而获得基因Ba-urease-2。用引物U-F1/U-R1、U-F2/U-R2分别扩增基因Ba-urease-1、Baurease-2,用引物P-F1/P-R2、P-F2/P-R2分别进行质粒pP43NMK线性化,利用ClonExpress®II One Step Cloning Kit将经胶回收后的基因片段和线性化质粒进行连接,并转化至E.coliJM109,挑取转化子验证正确,分别获得质粒 pP43NMK-Ure-1、pP43NMK-Ure-2。
最后,将构建成功的重组质粒转化至B.subtilisWB600中,获得重组表达菌株B.subtilisWB600/pP43NMK-Ure、B.subtilisWB600/pP43NMK-Ure-1、B.subtilisWB600/pP43NMK-Ure-2。
采用所构建的重组B.subtilis单菌落,接种于含有50μg/mL卡那霉素的5 mL LB液体培养基中,在37℃、200 r/min的摇床中振荡培养10 h。按照3%的接种体积分数转接于含有50μg/mL卡那霉素的100 mL TB液体培养基中,在37℃、200 r/min的摇床中振荡培养50 h,每隔一定时间测定菌体浓度及脲酶活力。
1.2.3 酶活测定 酶活单位(U)定义:在常压、37℃、pH 7.0的条件下,每分钟分解1μmol尿素所需要的酶量。脲酶活力采用Berthelot反应比色法[22]测定:脲酶水解尿素生成氨,氨与苯酚-次氯酸钠反应生成靛酚蓝,从而可以通过检测该显色反应在625 nm下的OD值变化来表征酶活。
取粗酶液200μL,添加至200μL含有30 g/L尿素的10 mmol/L磷酸缓冲液(pH 7.0)中,混匀。在37℃水浴下反应20 min,立即加入200μL 10%三氯乙酸,混匀终止反应。随后,依次加入200μL显色剂Ⅰ和200μL显色剂Ⅱ,混匀后在37℃水浴下反应20 min。反应结束后,8 000 r/min离心5 min,适当稀释,以灭活的酶液作为空白对照,测定其OD625nm的 变 化。使 用0、0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9、1.0 mmol/L NH4Cl溶液进行酶活反应,做出氨离子标准曲线,用于计算反应体系生成氨的总量并计算酶活。
最适反应温度:将粗酶液和尿素混合,在不同温度(20、25、28、30、35、37、40℃)下反应后测定酶活,以最高酶活为100%,分别计算不同温度下酶活的相对值。
最适反应pH:用10 mmol/L的柠檬酸、柠檬酸三钠溶液以及10 mmol/L的磷酸氢二钾、磷酸二氢钾溶液调节反应体系的pH(4.0、4.5、5.0、5.5、6.0、6.5、7.0)并测定酶活,以最高酶活为100%,分别计算不同pH下酶活的相对值。
1.2.4 摇瓶发酵条件优化 挑取所构建的重组B.subtilis单菌落,接种于含有50μg/mL卡那霉素的5 mL LB液体培养基中,在37℃、200 r/min的摇床中振荡培养10 h。
按3%的接种体积分数分别转接于含有50μg/mL卡那霉素的100 mL LB、TB、M9液体培养基中,在37℃、200 r/min的摇床中振荡培养50 h,每隔一定时间测定脲酶活力。
分别按3%、5%、7%、9%的接种体积分数转接于含有50μg/mL卡那霉素的100 mL TB液体培养基中,在37℃、200 r/min的摇床中振荡培养50 h,每隔一定的时间测定脲酶活力。
按3%的接种体积分数转接于含有50μg/mL卡那霉素的100 mL TB液体培养基中,在37℃、200 r/min的摇床中培养8 h后,变温(17、25、28、30、37℃)诱导,每隔一定的时间测定脲酶活力。
按3%的接种体积分数转接于含有50μg/mL卡那霉素、不同Ni2+浓度(0、1.0、2.0、3.0、4.0、5.0、6.0、7.0 mmol/L)的100 mL TB液体培养基中,在37℃、200 r/min的摇床中振荡培养50 h,每隔一定时间测定脲酶活力。
1.2.5 尿素降解的测定 反应体系:将500μL粗酶液加至500μL尿素溶液(300 mg/L)中混匀,37℃反应1 h。
反应体系中的尿素含量通过高效液相色谱法(HPLC)测定[23],样品的处理方法和液相色谱条件参照文献[24]。
2 结果与分析
2.1 基因挖掘与结构分析
通过NCBI核苷酸数据库中脲酶各种亚基的编码基因的对比分析发现,不同种属之间的脲酶结构基因同源性很低,但其结构亚基对应的蛋白质氨基酸序列之间同源性较高,其中ureC编码的α结构亚基保守性最高[25]。在传统酒精饮料发酵过程中会产生乳酸杆菌属、芽孢杆菌属等同属于厚壁菌门的菌株[26],且对酒精饮料没有安全隐患。所以在厚壁菌门中挖掘UreC的蛋白质编码序列,并采用Clustal Omega软件进行氨基酸序列的比对以及系统发育树的分析,见图1。
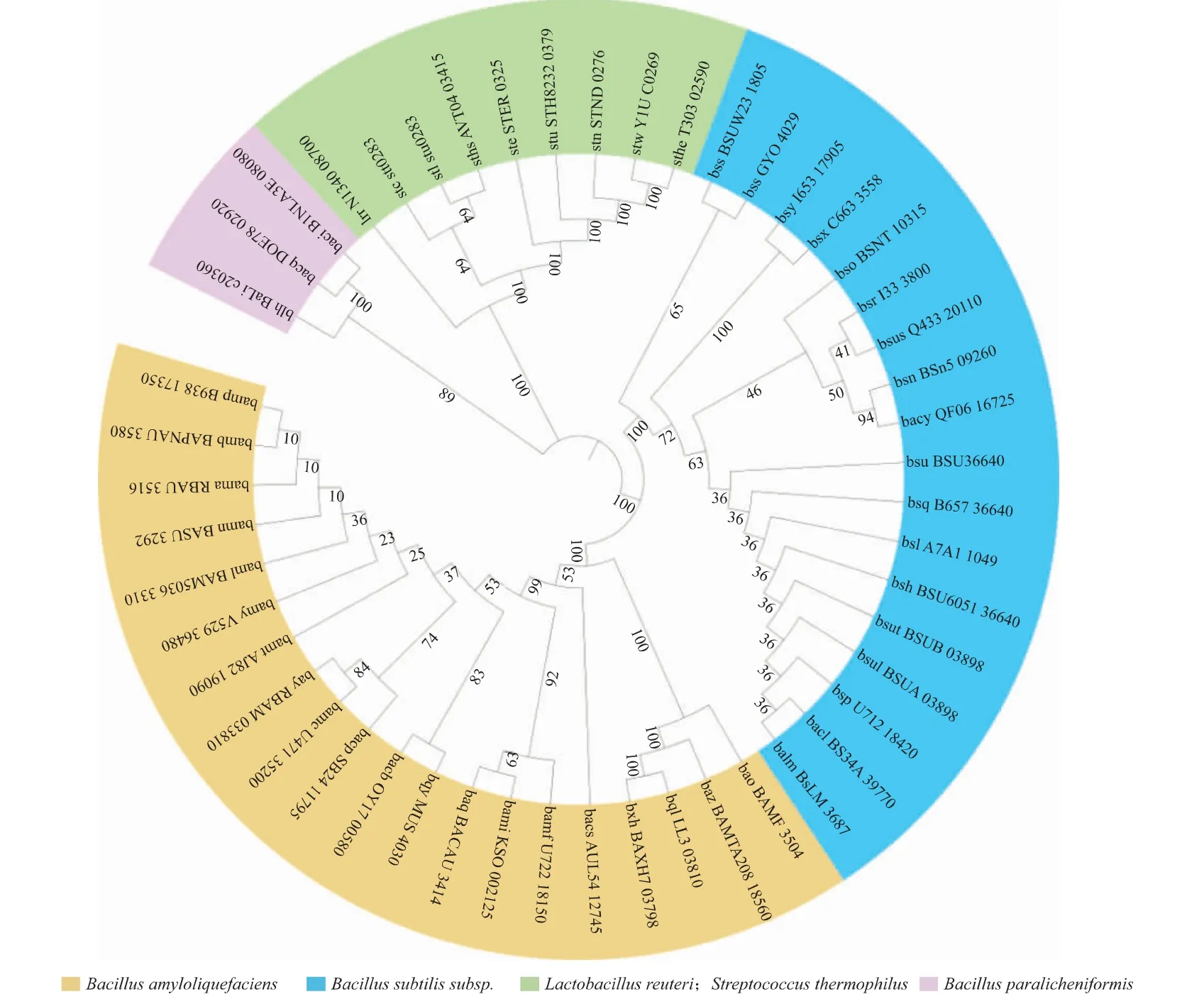
图1 系统发育树Fig.1 Evolutionary tree
对挖掘所得的不同来源的脲酶基因进行结构分析,见图2。发现B.amyloliquefaciensIT-45基因组(GenBank:CP004065.1)中 只 有ureA、ureB、ureC这3个结构基因,无其他辅助基因,且与枯草芽孢杆菌属具有相似的特性。与其处于系统发育树同一分支的B.amyloliquefaciensJP-21来源脲酶基因簇已在大肠杆菌中成功表达,并通过点饱和突变提高活性[17],但在枯草芽孢杆菌中的重组表达有待研究。因此,选择来源于B.amyloliquefaciensIT-45的脲酶基因簇Ba-urease(ureABC)作为研究出发的脲酶结构基因。
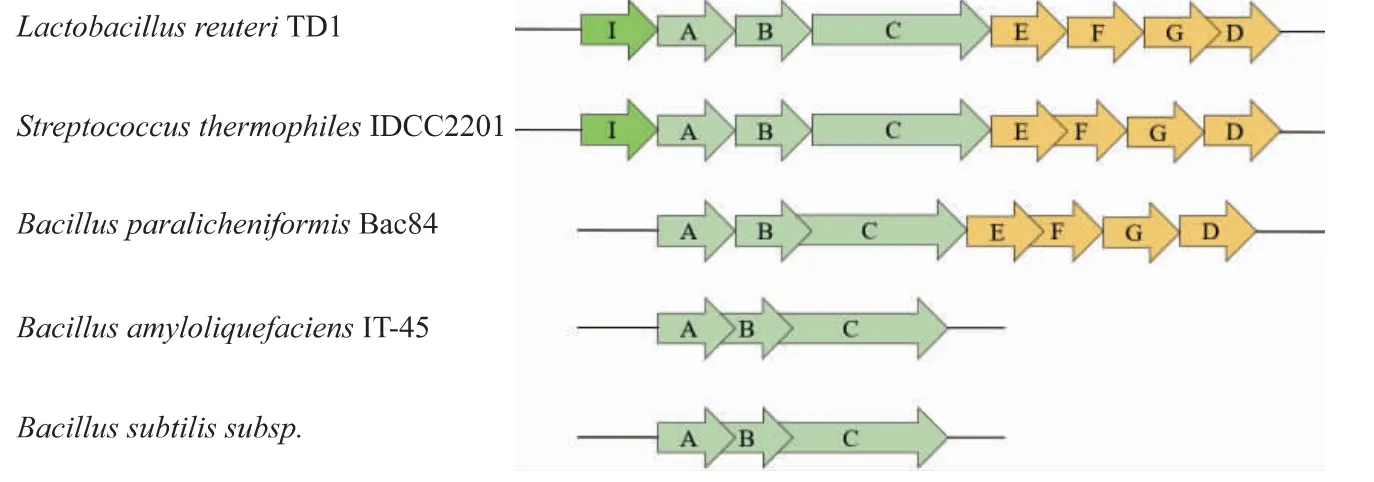
图2 不同来源脲酶的基因簇Fig.2 Structures of urease gene clusters in different microorganisms
2.2 脲酶基因的克隆与表达
脲酶基因簇Ba-urease由3个结构基因ureA、ureB、ureC组成,细菌来源的脲酶是由结构亚基UreA、UreB、UreC形成的异源多聚体(UreABC)3[27]。基于脲酶的活化机制,成熟的脲酶蛋白质结构中的结构亚基(UreA、UreB、UreC)的数量为1∶1∶1[28],因而可以通过提高结构亚基的表达量来提高脲酶的产量。而作为大亚基的UreC是催化亚基,其对脲酶的活力至关重要。此外,文献[29]表明,在同一个操纵子中表达多个基因时,离启动子越远的基因表达量越低。对脲酶基因簇Ba-urease加以分析,发现各亚基基因之间有重叠,因此,在本研究中对脲酶基因簇进行了完整基因归位、枯草密码子优化、RBS优化以及将大亚基UreC调换至靠近启动子处来进行重构,见图3。核酸电泳验证见图4(a)。由图4(b)的SDS-PAGE电泳分析可知,发酵样品经浓缩后,有符合UreC的61 150大小的条带,超声破碎后上清液有微弱条带,大部分条带均在沉淀中。
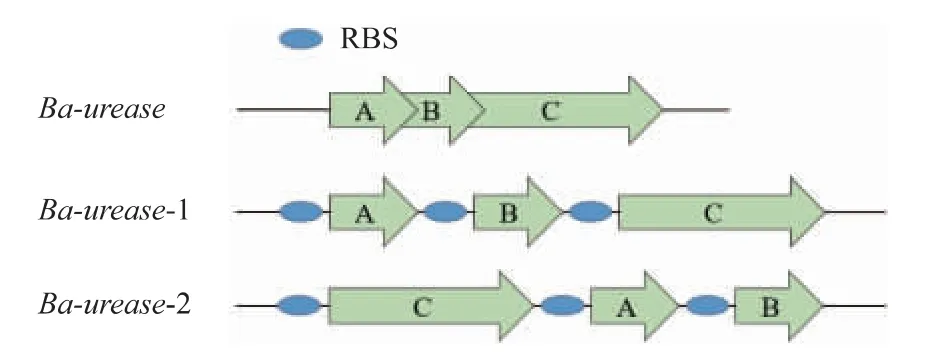
图3 Ba-urease基因簇变换流程Fig.3 Manipulation of the Ba-urease gene cluster
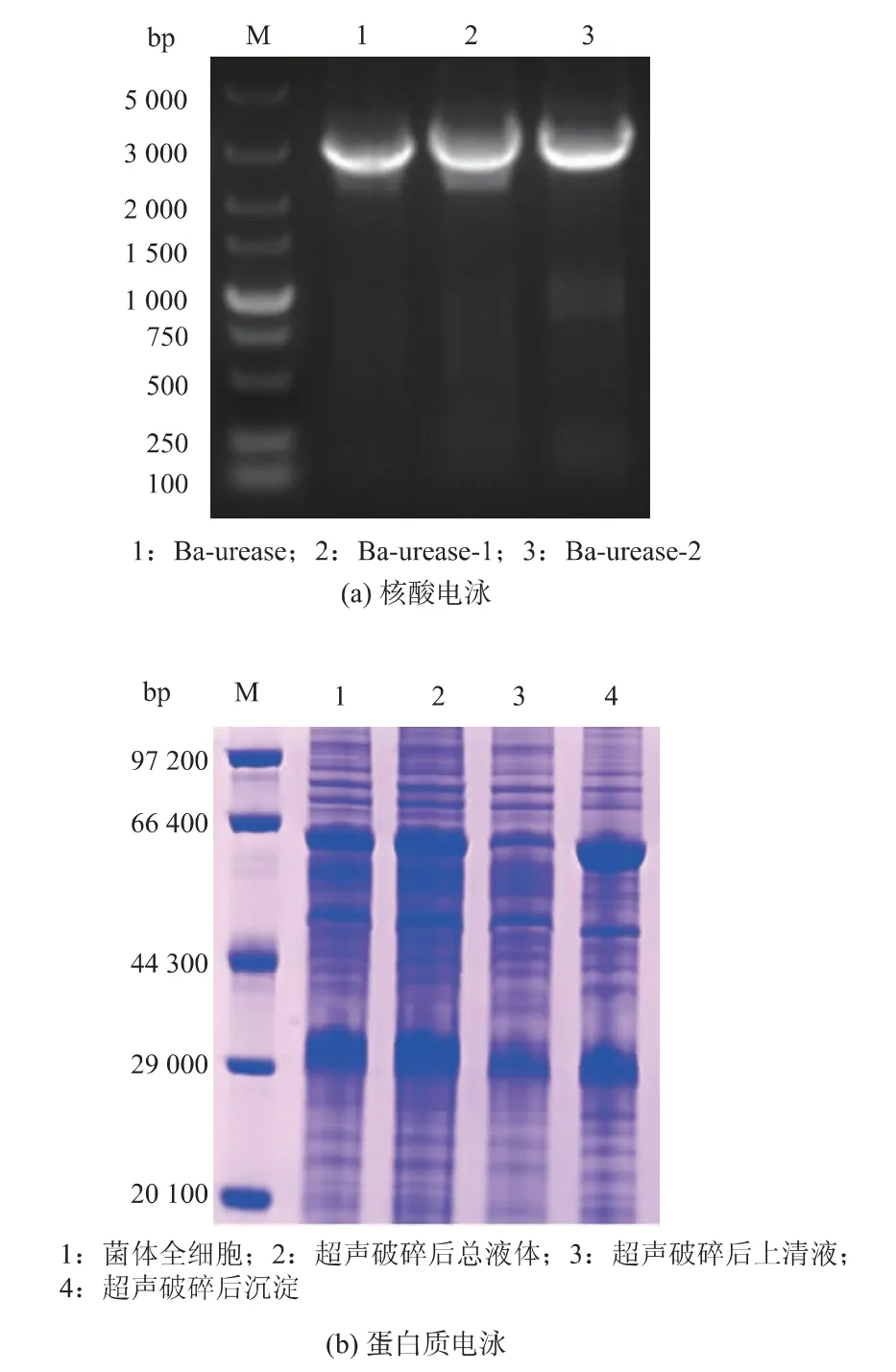
图4 重组菌株的验证Fig.4 Verification of recombinant strains
重组菌株B.subtilisWB600/pP43NMK-Ure表达原始脲酶基因簇(Ba-urease)时,发酵后仅能在菌体全细胞时检测到脲酶活力(6.85 U/mL),见图5。
图5 酶活检测的显色反应Fig.5 Color reaction of enzyme activity detection
对重组菌株B.subtilisWB600/pP43NMK-Ure、B.subtilisWB600/pP43NMK-Ure-1、B.subtilisWB600/pP43NMK-Ure-2发酵50 h,前期每隔5 h取样,后期10 h取样,平行测OD以及酶活,见图6。重组菌B.subtilisWB600/pP43NMK-Ure-1摇瓶发酵后,脲酶活力最高可达9.01 U/mL,相对于原始脲酶基因簇提高了31%。重组菌B.subtilisWB600/pP43NMKUre-2摇瓶发酵后,脲酶活力最高可达10.15 U/mL,相对于原始脲酶基因簇提高了48%,相对于密码子、RBS优化重构的基因簇提高了13%。
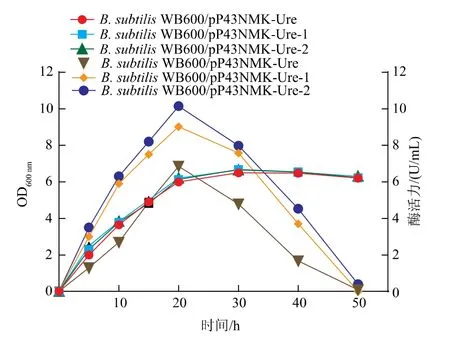
图6 重组菌株摇瓶发酵酶活力随时间变化曲线Fig.6 Time courses of enzyme activity in recombinant strains in shake-flasks
2.3 酶学性质表征
酶分子与底物之间的相互作用和结合概率可以通过提高反应温度来增强,从而提高酶活,但是反应温度过高会破坏酶分子的空间结构,导致酶活丧失,因此需要选择合适的反应温度来充分发挥酶的催化功能。在20、25、28、30、35、37、40℃下进行测定,结果见图7(a)。重组脲酶的最适反应温度为37℃,且在30~37℃范围内可以保持30%以上的酶活力。
探究重组脲酶的最适反应pH,可以用来评价此重组脲酶能否用于降解黄酒中的尿素。在其他条件不变的情况下,测定重组脲酶在pH 4.0~7.0范围内的酶活力,结果见图7(b)。重组脲酶在pH 6.5时活力最高,与pH 7.0时相差0.03倍,可得最适反应pH范围为6.5~7.0。
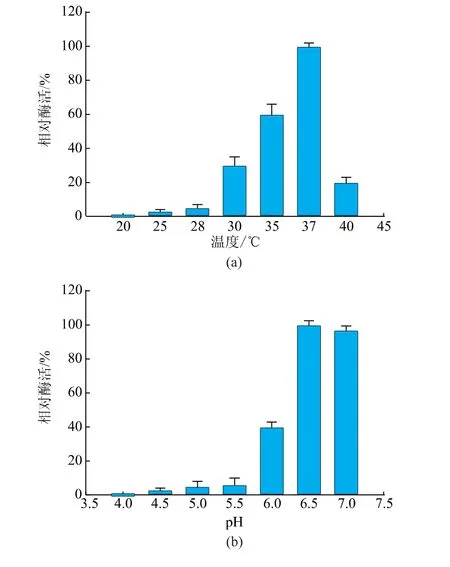
图7 最适反应条件Fig.7 Optimal reaction conditions
发酵乙醇饮料一般为酸性体系,其中黄酒的pH约为3.5~4.8。目前,对黄酒中尿素降解有一定效果的是酸性脲酶,只有日本和美国的少数企业实现了工业化生产,其使用因安全问题在国内有一定的限制[30]。国内对酸性脲酶的研究仍处于实验阶段,尚未达到商业化供应的水平,这大大限制了酸性脲酶处理黄酒的工业化应用。我国目前市售的脲酶几乎均是植物来源的中性脲酶,其来源广泛且价格低廉。该酶在中性条件时活性高,但在酸性条件下酶活极低,一般认为这种中性脲酶不适用于黄酒中尿素的降解[13],需要进行耐酸性改造或固定化研究才有工业化应用的可能性。
2.4 摇瓶发酵优化表征
培养基营养成分和接种体积分数都影响着细胞的生长和蛋白质的表达。选取LB、TB、M9培养基,分别按照3%、5%、7%、9%的接种体积分数进行转接,探究其对产酶能力的影响。如图8(a)、8(b)所示,营养元素丰富的TB培养基发酵产生的脲酶酶活最高,为10.15 U/mL;接种体积分数为5%时,酶活为10.43 U/mL。因而,确定TB培养基中5%接种体积分数为脲酶重组菌B.subtilisWB600/pP43NMK-Ure-2的最佳条件。
在一定温度范围内,较高的诱导温度可以加速菌体生长和蛋白质合成,但是过快的蛋白质合成速率会导致新生肽链来不及折叠从而形成无活性的包涵体,导致酶活低下甚至没有活性;诱导温度过低会减缓菌体生长,导致蛋白质合成的速率过低,同样会造成酶活低下。为提高脲酶的表达量及活性,需要确定合适的诱导温度。如图8(c)所示,当诱导温度为28℃时,脲酶酶活达到11.3 U/mL,相对于37℃发酵时提高了11%。因而,确定28℃为重组菌B.subtilisWB600/pP43NMK-Ure-2表达脲酶的最佳诱导温度。
脲酶是依赖于金属离子的水解酶,在其催化亚基UreC中存在2个Ni2+结合位点[31],适量添加Ni2+可以有效提高脲酶酶活。Ni2+添加量过少,无法完全激活脲酶;Ni2+添加过量,会对菌体产生毒害,影响菌体生长和脲酶酶活。如图8(d)所示,当Ni2+添加量为4 mmol/L时脲酶活力最高,脲酶酶活力达到12.3 U/mL,相对于28℃发酵且不添加Ni2+提高了约9%。因而,确定4 mmol/L为脲酶重组菌B.subtilisWB600/pP43NMK-Ure-2的最佳Ni2+添加量。
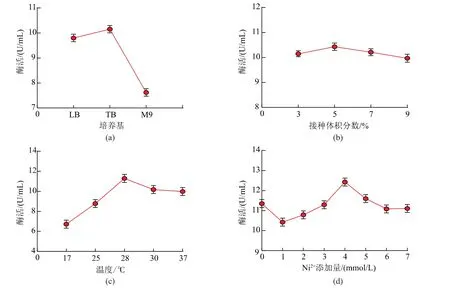
图8 发酵条件的优化Fig.8 Optimization of fermentation conditions
2.5 尿素降解的测定
对重组菌株B.subtilisWB600/pP43NMK-Ure-2进行最佳条件的发酵,在常压、37℃、pH 7.0的反应条件下,检测到脲酶酶活为12.5 U/mL。取该条件发酵下得到的粗酶液500μL,加至500μL尿素溶液(300 mg/L)中混匀,37℃反应1 h。通过高效液相色谱法检测尿素质量浓度,结果见图9。反应体系中的尿素质量浓度显著降低,表明该重组脲酶可以有效降解尿素。
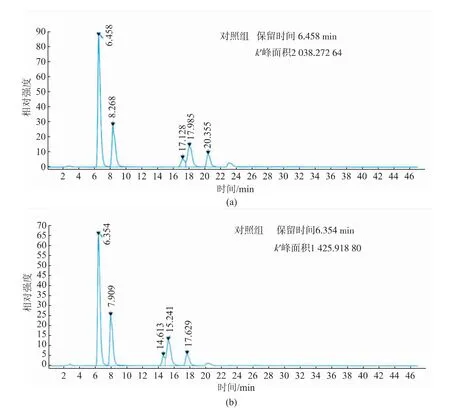
图9 高效液相色谱荧光检测法测定尿素色谱图Fig.9 Chromatogram of urea by HPLC-FLD
3 结语
在本研究中,作者利用食品级系统B.subtilisWB600/pP43NMK实现了来源于Bacillus amyloliquefaciensIT-45的脲酶Ba-urease的摇瓶发酵表达,并验证了其可以有效降解尿素。
通过前期的脲酶基因挖掘和结构分析,获得了来源于B.amyloliquefaciensIT-45的只包含3个结构亚基的脲酶基因簇Ba-urease。所有亚基的表达对活力产生都必不可少,因此采用对所有基因进行枯草芽孢杆菌密码子优化、将各亚基重叠部分完全分开并在中间插入优化的核糖体结合位点重新组装脲酶基因簇Ba-urease的策略,使Ba-urease的酶活力从6.85 U/mL提升至9.01 U/mL;通过调整基因ureC至ureA、ureB前,强化了UreC的表达,脲酶活力进一步提升至10.15 U/mL,相对于原始脲酶活力提高了48%。在此基础上,探究了重组脲酶的酶学性质,可得:最适反应温度为37℃,且在30~37℃范围内可以保持30%以上的酶活力;最适反应pH范围为6.5~7.0,为中性脲酶。对重组脲酶Ba-urease在100 mL摇瓶发酵的基础上进行条件优化,最终确定培养基为TB、接种体积分数5%、诱导温度28℃、Ni2+添加量4 mmol/L的最佳发酵条件,酶活力达到了12.5 U/mL,相对于原始脲酶活力提高了82%,这是目前为止解淀粉芽孢杆菌来源脲酶食品级摇瓶发酵的最高值。
基于工业化应用的需求,将重组脲酶Ba-urease加入尿素溶液中反应,通过高效液相色谱检测到尿素质量浓度显著降低。该重组脲酶安全有效、操作方便,反应pH及其稳定性仍需进一步提高。本研究为食品工业中酶法降解黄酒中的尿素提供了一种方法,具有广阔的应用前景。
