牛源致脑炎大肠杆菌FimD、PilN蛋白的生物信息学分析和优势B细胞表位预测
2022-05-31海永慧李蓓蓓任静静蒋建军王鹏雁
海永慧 , 钦 倩 , 李蓓蓓 , 任静静 , 马 勋 , 蒋建军 , 王鹏雁
(石河子大学动物科技学院 , 新疆 石河子 832003)
肠外致病性大肠杆菌(Extra-intestinal pathogenicEscherichiacoli,ExPEC)可通过其特有的毒力因子定殖肠道外的其他组织中,最终导致宿主发病[1]。ExPEC包括新生儿脑膜炎大肠杆菌(Neonatal meningitis causingEscherichiacoli,NMEC)、尿道致病性大肠杆菌(UropathogenicEscherichiacoli,UPEC)、败血症大肠杆菌(SepticemicEscherichiacoli,SEPEC)[2]和禽致病性大肠杆菌(Avian pathogenicEscherichiacoli,APEC)[3]。近年来,新疆多个规模化奶牛养殖场的犊牛出现了以神经症状为主的病例,剖检发现患牛脑膜严重出血,从脑组织中分离出来的病原菌经鉴定为肠外致病性大肠杆菌,这给奶牛养殖场带来了经济损失,同时也给牛源ExPEC的防控带来了挑战[4]。
目前用于ExPEC的疫苗主要以传统灭活苗为主,但此类疫苗免疫保护效果相对比较局限,且副作用大,此外,可用于牛源致脑炎大肠杆菌的疫苗鲜有报道。多表位候选疫苗可克服传统疫苗的缺点,能更高效地刺激机体产生特异性免疫应答,并消除针对不良表位的免疫应答[5]。多表位疫苗可以刺激机体保守和有利的表位,产生免疫反应,具有更高安全性,还可以通过抗原表位的工程设计和预测来提高疫苗的广度和效力[6]。
牛源致脑炎大肠杆菌S9922分离自新疆某奶牛场患病犊牛脑组织中,本课题组前期已对其进行全基因组测序并将E.coliS9922全基因组序列分别与GenBank上公布的56个具有全基因组序列的大肠杆菌进行比较分析,得到了69个E.coliS9922特有毒力基因,因此本试验从中选取了与黏附有关的FimD和PilN两个菌毛蛋白,利用生物信息学工具分析其蛋白理化性质、结构和优势B细胞表位,为牛源致脑炎大肠杆菌多表位疫苗的研发提供基础数据。
1 材料与方法
1.1 材料 牛源致脑炎大肠杆菌S9922分离自新疆某奶牛场患病犊牛脑组织中,并已被鉴定为肠外致病性大肠杆菌,由石河子大学动物科技学院微生物与免疫学实验室保存。将S9922进行全基因组测序并进行比较基因组学研究,筛选到与细菌致病有关的特异性黏附蛋白FimD、PilN等。
1.2 方法
1.2.1 蛋白的理化性质分析 运用在线服务器ProtParam(https://web.expasy.org/protparam/),分析FimD、PilN蛋白的氨基酸组成、分子质量、理论等电点、原子组成、不稳定指数和平均亲水系数等理化性质。
1.2.2 蛋白跨膜区、信号肽、糖基化位点和磷酸化位点分析 运用在线服务器TMHMM server v.2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)、SignalP-5.0 server (http://www.cbs.dtu.dk/services/SignalP/)、NetNGlyc1.0(http://www.cbs.dtu.dk/services/NetNGlyc/)和NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/),分别分析FimD、PilN蛋白氨基酸序列的跨膜区、信号肽区域、糖基化位点和磷酸化位点。
1.2.3 蛋白二级结构分析 利用SOMPA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)分析蛋白的β转角、β折叠、α螺旋、无规则卷曲等结构。同时用DNASTAR软件中的Protean板块分析蛋白抗原性、亲水性、柔韧性、表面可及性较好的肽段。
1.2.4 蛋白三级结构分析 将FimD、PilN蛋白的氨基酸序列提交至在线服务器SWISS-MODEL(https://swissmodel.expasy.org/interactive),FimD蛋白选取3rfz.1.B作为模板,PilN蛋白选取3ezj.1.A作为模板,构建FimD、PilN蛋白的三级结构模型。
1.2.5 蛋白优势B细胞表位预测 利用BepiPred(http://www.cbs.dtu.dk/services/BepiPred)、Immunomedicine Group(http://imed.med.ucm.es/Tools/antigenic.pl)对FimD、PilN蛋白的B细胞表位进行预测。综合DNASTAR软件中的Protean板块的抗原性、柔韧性、亲水性和表面可及性以及信号肽、跨膜区、二级结构,预测FimD、PilN蛋白的B细胞优势抗原表位。
2 结果
2.1 FimD、PilN蛋白的理化性质分析 FimD蛋白由382个氨基酸组成,其中含量较多的氨基酸:Gly占10.7%,共有41个;Ser占10.2%,共有39个;Asn占9.4%,共有36个;Thr占8.1%,共有31个;Leu占7.1%,共有27个;带负电荷的残基(Asp+Glu)总数为28,带正电荷的残基(Arg+Lys)总数为25,理论等电点为6.16。FimD蛋白的原子组成为C1828H2798N510O588S9,相对分子质量为41 615.93 Da,不稳定系数为28.31,小于阈值40,归类为稳定蛋白。平均亲水系数(GRAVY)为-0.435,GRAVY的范围为-2~2,负值表明蛋白为亲水蛋白,FimD蛋白归类为亲水蛋白。
PilN蛋白由560个氨基酸组成,其中含量较多的氨基酸:Thr占12.7%,共有71个;Ser占11.1%,共有62个;Leu占9.1%,共有51个;Ala占7.7%,共有43个;Gly占7.5%,共有42个;带负电荷的残基(Asp+Glu)总数为43,带正电荷的残基(Arg+Lys)总数为44,理论等电点为7.61。PilN蛋白的原子组成为C2586H4184N724O858S22。相对分子质量为59 851.32 Da,不稳定系数为36.5,小于阈值40,归类为稳定蛋白。GRAVY为-0.274,GRAVY的范围为-2~2,负值表明蛋白为亲水蛋白,FimD蛋白归类为亲水蛋白。
2.2 FimD、PilN蛋白的跨膜区、信号肽、糖基化位点和磷酸化位点分析 利用在线服务器TMHMM分析FimD蛋白和PilN蛋白的跨膜区,结果如图1所示,FimD蛋白不存在跨膜区,PilN蛋白在1~50位氨基酸上可能出现跨膜区。利用在线服务器SignalP-5.0 分析FimD蛋白和PilN蛋白的信号肽,结果如图2所示,FimD蛋白不存在信号肽;PilN蛋白在26~27位氨基酸上出现信号肽。

图2 蛋白信号肽分析
利用在线服务器NetNGlyc1.0进行蛋白糖基化位点分析,FimD蛋白共有7个潜在的糖基化位点,PilN蛋白共有2个潜在的糖基化位点。利用在线服务器NetPhos 3.1进行蛋白磷酸化位点分析,分析结果如彩版封二图3所示,FimD蛋白共有潜在的48个磷酸化位点,其中有23个Ser位点,15个Thr位点,10个 Tyr位点;PilN蛋白共有85个潜在的磷酸化位点,其中有47个Ser位点,33个Thr位点,5个Tyr位点。

图3 磷酸化位点分析
2.3 FimD、PilN蛋白的二级结构分析 利用在线服务器SOPMA对蛋白的二级结构进行分析,FimD蛋白结果如彩版封二图4所示,α螺旋占6.54%,延伸链占32.94%,β转角占5.50%,无规则卷曲占56.02%;PilN蛋白结果如彩版封三图5所示,α螺旋占23.75%,延伸链占20.54%,β转角占4.29%,无规则卷曲占51.43%。

图4 FimD蛋白二级结构预测

图5 PilN蛋白二级结构预测
利用DNASTAR软件中的Protean板块分析蛋白抗原性、亲水性、柔韧性、表面可及性的结果如彩版封三图6所示,综合分析得出亲水性、柔韧性、抗原性和表面可及性都较好的肽段(表1)。
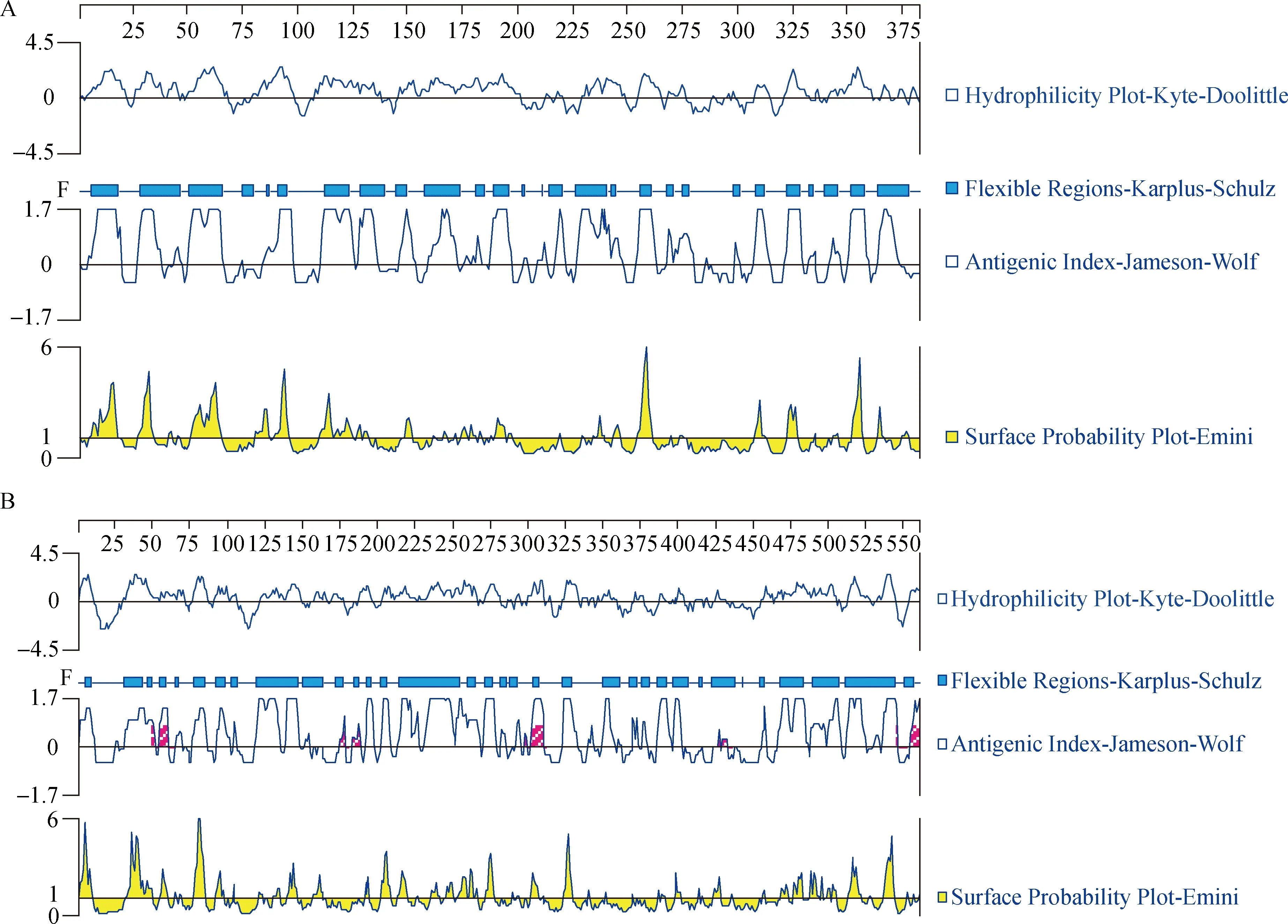
图6 DNASTAR软件预测蛋白亲水性、柔韧性、抗原性和表面可及性

表1 FimD、PilN蛋白亲水性、柔韧性、抗原性和表面可及性都较好的肽段预测
2.4 FimD、PilN蛋白的三级结构分析 利用SWISS-MODEL进行同源建模,同源建模也可称比较建模,是利用已知的同源蛋白质的结构建立目的蛋白质的结构模型。FimD蛋白、PilN蛋白三级结构预测结果如图7所示,FimD蛋白选取3rfz.1.B作为模板,全局模型质量评估(GMQE)结果为0.78,QMEAN为-2.01,相似性为56.81%;PilN蛋白选取3ezj.1.A作为模板,GMQE结果为0.10,QMEAN为-4.44,相似性为12.78%,所有图中都存在α-螺旋、β-折叠、β-转角和无规则卷曲,与二级结构分析的结果相符合。

图7 FimD蛋白、PilN蛋白三级结构预测
2.5 FimD、PilN蛋白的优势B细胞表位预测 采用BepiPred预测的蛋白B细胞表位结果如表2所示,Immunomedicine Group预测的蛋白B细胞表位结果如表3所示。参照高瞻等[7]的方法进行最终的蛋白优势B细胞表位预测,由于表位大多出现在表面可及性、抗原性、柔韧性、亲水性都较好的肽段,并且目前预测B细胞表位的在线工具都未达到百分之百的准确率,因此需要选取用BepiPred预测的B细胞表位结果和用Immunomedicine Group预测的B细胞表位结果的共同部分,并综合DNASTAR软件、SOPMA分析的结果,选出表面可及性、抗原性、柔韧性、亲水性都较好并且没有α螺旋的肽段,预测出最终的蛋白优势B细胞表位。最终预测出FimD蛋白潜在的优势B细胞表位为:7~18、144~152、307~315、365~373;PilN蛋白潜在的优势B细胞表位为:77~86、138~149、516~524。

表2 BepiPred方法预测蛋白B细胞表位的肽段位置

表3 Immunomedicine Group方法预测蛋白B细胞表位的肽段位置
3 讨论
大肠肝菌(Escherichiacoli,E.coli)引起的脑膜炎是一个复杂的多步骤过程,包括从肠黏膜的定植到进入血液循环并形成高水平的菌血症,然后进一步穿过血脑屏障,进入脑脊液最终引起脑膜炎[8]。黏附是NMEC致病机理中的第一步,也是穿越血脑屏障的重要一步。E.coli对构成血脑屏障的脑微血管内皮细胞的黏附被认为是其侵入中枢神经系统的重要因素[9]。菌毛与E.coli的黏附、定植和致病都有着非常密切的关系[10],FimD和PilN都属于菌毛的亚单位蛋白,在菌毛的组装中起着重要的作用[11-12]。
表位是蛋白抗原性的基础,而且与机体的固有免疫、获得性免疫等密切相关,表位的研究对于疾病的诊断和分子疫苗的设计有至关重要的意义[13]。随着生物信息学研究领域的不断发展和创新,生物信息学被广泛地应用[14],目前,对蛋白质结构与功能进行生物信息学技术分析已经成为大多表位和疫苗研究的首选方法[15],与传统的试验方法相比,生物信息学技术使蛋白结构分析和表位的预测更加准确和简便[16]。表位大多出现在蛋白表面可及性强、柔韧性好、亲水性和抗原指数高的肽段[17],以及结构较松散,易扭曲、盘旋并展示在蛋白表面的β转 角及无规则卷曲和该蛋白的膜外区域,从而有利于抗体结合[18]。
本试验利用生物信息学方法综合分析FimD和PilN表面可及性、柔韧性、蛋白亲水性、抗原指数、二级结构、三级结构等参数来预测B细胞表位。结果显示,FimD和PilN蛋白的膜外区域占较高比例,因此可能被抗原递呈细胞接触并引发免疫应答。蛋白二级结构和三级结构分析结果显示,FimD蛋白的β转角和无规则卷曲占61.52%,PilN蛋白的β转角和无规则卷曲占55.72%,2个蛋白的β转角和无规则卷曲所占比例之和大于50%。BepiPred-2.0基于随机森林算法提供了最先进的基于B细胞表位序列的预测[19],与其他可用工具相比,该表位被认为具有更高的质量,并且确实显著提高了预测能力[20]。Immunomedicine Group使用Kolaskar和Tongaonkar的方法预测表位,只有当最小氨基酸残基大小为8个时才被报告,该方法的准确性约为75%[21]。IEDB为科学界提供了表位数据的开放访问,以及表位预测和分析工具,收集了最广泛的试验验证的B细胞和T细胞表位数据[22]。分别使用在线预测工具BepiPred和Immunomedicine Group预测出FimD蛋白具有11个和14个B细胞表位肽段,PilN蛋白具有12个和23个B细胞表位肽段。现已有的预测B细胞表位的在线工具均未达到百分之百的准确性。本试验选取除去信号肽和跨膜区后的肽段,综合分析FimD、PilN蛋白在不同在线预测工具得出的B细胞表位预测结果以及DNASTAR软件分析得出的2个蛋白表面可及性、柔韧性、抗原性、亲水性较好的肽段,选取共同部分最终确定潜在的优势B细胞表位。综上所述,FimD、PilN蛋白具有很多潜在的抗原表位,推测这2个蛋白具有良好的免疫原性,但还需要在后期的试验中进一步验证。本试验预测筛选得到的FimD、PilN蛋白潜在表位可为牛源致脑炎大肠杆菌多表位疫苗的研发提供基础数据。
