细胞分裂周期相关蛋白(CDCAs)与乳腺癌发生、发展和转移的生物信息学分析*
2022-05-05刘婧婷隆建萍金凤玲林碧玉王文第裴建赢王晶晶
刘婧婷, 隆建萍, 金凤玲, 林碧玉, 王文第, 裴建赢, 王晶晶
兰州大学1 第一临床学院 3第一医院感染管理科,兰州 730000 甘肃省妇幼保健院2 乳腺一科 4妇幼保健科研中心,兰州 730050
乳腺癌(breast cancer,BC)是全球女性最常见的恶性肿瘤之一,2018年新诊断的乳腺癌约210万例,全球死亡人数626679人[1]。虽然对乳腺癌发生的潜在机制进行了广泛的研究,但乳腺癌患者,尤其是女性患者的生存率仍然较低。因此,创新有效的治疗方法和新药的研发是非常重要的。细胞分裂是生命的关键过程。许多研究已经证明,细胞分裂过程中的功能障碍会导致肿瘤的发生[2-6]。细胞分裂周期相关蛋白(cell division cycle-associated protein,CDCAs)家族由8个成员组成(CDCA1~8)。CDCA1对核分裂和微管的稳定性至关重要[7]。CDCA2编码细胞周期相关蛋白的靶向亚基——蛋白磷酸酶1γ(protein phosphatase 1γ,PP1γ),在细胞周期中参与核膜的重组并调控DNA损伤[8]。CDCA3的作用是调节细胞周期,在细胞周期的G1期通过蛋白降解和转录调控表达水平[9]。CDCA4是细胞周期的调节因子,其编码的蛋白属于E2F转录因子家族,主要分布在核有丝分裂器上,与G1/S期的转变有关[10]。CDCA5是细胞分裂过程中姐妹染色单体内聚合和分离的重要调控因子,在DNA修复中起着重要的作用[11]。CDCA7是c-Myc的直接靶基因,该基因的过表达可促进淋巴母细胞样细胞的转化[12]。CDCA8是有丝分裂的重要调控因子,其编码的复合物可调控染色质诱导的微管稳定和纺锤体形成[13]。然而,CDCAs的差异表达及其在乳腺癌中的预后价值仍有待阐明。因此,本研究利用大样本、高通量多数据库分析乳腺癌中CDCAs的表达情况及临床意义,以期为探索乳腺癌的发生、发展、预后判断等提供依据。
1 材料与方法
1.1 数据库
基因表达数据取自公共基因芯片数据库(Gene Expression Omnibus, GEO,https://www.ncbi.nlm.nih.gov/geo/)。检索词为“breast cancer”和“Homo sapiens[porgn:__txid9606]”,总共检索到81400个关于人乳腺癌数据集。纳入研究的数据需同时包括乳腺癌组织样本和正常乳腺组织样本,且每组样本在10个以上。筛选后选择3个基因表达谱(GSE45827、GSE65194、GSE61304),这3个数据库基于平台GPL570([HG -U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array)。从TCGA(The Cancer Genome Atlas)数据库下载原始数据,通过RSEM标准化。纳入的数据基本信息见表1。

表1 本文纳入的乳腺癌基因芯片基本信息Table 1 Basic information of four GEO datasets of breast cancer
1.2 差异表达基因(differentially expressed genes,DEGs)的筛选
从GEO和TCGA数据库下载的乳腺癌相关微阵列数据使用R软件处理(版本3.6.1,https://cran.r-project.org/)。使用limma包在R软件中分析乳腺癌组织与正常乳腺组织之间的DEGs,计算FC(fold-change)值,并根据P<0.01和|log FC|≥2的标准进一步选择。在GEPIA(Gene Expression Profiling Interactive Analysis)数据库(http://gepia.cancer-pku.cn/)验证所选取的DEGs在不同肿瘤中的表达情况[14],在“Single Gene Analysis”模块输入所选取的DEGs,设置“P<0.05、|log FC|≥1”作为筛选条件,Jitter Size设置为0.4,对照样本选择“Match TCGA normal and GTEx data”。
1.3 Oncomine数据库分析
Oncomine(https://www.Oncomine.org)是一个用于全基因组表达分析公开的癌症基因表达谱数据库[15]。该数据库包含715个数据集,86733例正常组织和肿瘤样本。本文利用Oncomine分析了乳腺癌及相应正常组织中CDCAs的转录水平。
1.4 Kaplan-Meier Plotter数据挖掘和处理分析
Kaplan-Meier Plotter(www.kmplot.com)是一个在线工具,能够评估21种癌症类型中54000多个基因对生存率的影响,数据集包括乳腺癌(6234例)、卵巢癌(2190例)、肺癌(3452例)和胃癌(1440例)[16],该数据库纳入了GEO、TCGA数据库中基因表达信息及临床生存信息。本文使用Kaplan-Meier Plotter评价CDCAs表达水平的预后价值,采用Kaplan-Meier绘图仪计算CDCAs表达中位数,将样本分为高表达组和低表达组,采用log-rank检验分析Kaplan-Meier生存曲线,计算相对危险度(hazard ratio,HR)、95%可信区间和P值,以P<0.05为差异有统计学意义,主要评估乳腺癌患者的整体生存率(overall survival,OS)、无复发生存率(recurrent-free survival,RFS)和无远处转移生存率(distant metastasis-free survival,DMFS)这3个指标。
1.5 DEGs的功能富集分析
使用基因本体(Gene Ontology,GO)数据库来分析3个独立类别的丰富基因功能:生物过程(biological process,BP)、分子功能(molecular function,MF)和细胞成分(cellular component,CC)[17]。使用基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genome,KEGG)来分析基因的相关通路[18]。使用DAVID(Database for Annotation Visualization and Integrated discovery)在线工具(https://david-d.ncifcrf.gov/)进行DEGs的GO和KEGG信号通路分析[19],以P<0.05为差异具有统计学意义。
1.6 蛋白-蛋白相互作用(protein-protein interaction,PPI)网络分析
STRING(The Search Tool for the Retrieval of Interacting Genes,https://string-db.org/)在线工具用于检测DEGs之间的蛋白质相互作用,用组合得分>0.9提取PPI,通过Cytoscape软件进行可视化PPI网络[20]。利用Cytoscape软件中的MCODE(Molecular Complex Detection Technology)插件构建PPI网络中的功能模块,CytoHubba插件用于计算每个蛋白质节点的MCC(maximal clique centrality),前30个基因被确定为关键基因。
2 结果
2.1 乳腺癌患者中的DEGs
根据P<0.01和|logFC|≥2的标准,在数据集GSE45827、GSE65194、GSE61304中,共有2879个DEGs,其中显著上调的有2015个,包括细胞分裂周期相关蛋白家族成员CDCA1、CDCA2、CDCA3、CDCA5、CDCA7、CDCA8,显著下调的有864个。随后进行维恩分析以得到DEGs维恩图,见图1。

图1 乳腺癌基因芯片数据集中DEGs分析Fig.1 Identification of DEGs in breast cancer microarray datasets
2.2 CDCAs在乳腺癌组织和正常组织之间的表达差异
首先,利用Oncomine数据库分析了不同癌症类型和相应正常组织中CDCAs的转录水平,见图2。与正常组织相比,CDCA1/2/3/5/7/8在肿瘤组织中mRNA水平升高,特别是在乳腺癌、肺癌、结直肠癌以及中枢神经系统肿瘤中。在肿瘤组织中,CDCA3在48个数据集中显著升高,4个数据集中下降;CDCA5在52个数据集中表达升高,4个数据集中表达降低;CDCA8在41个数据集中高表达,在3个数据集中低表达。
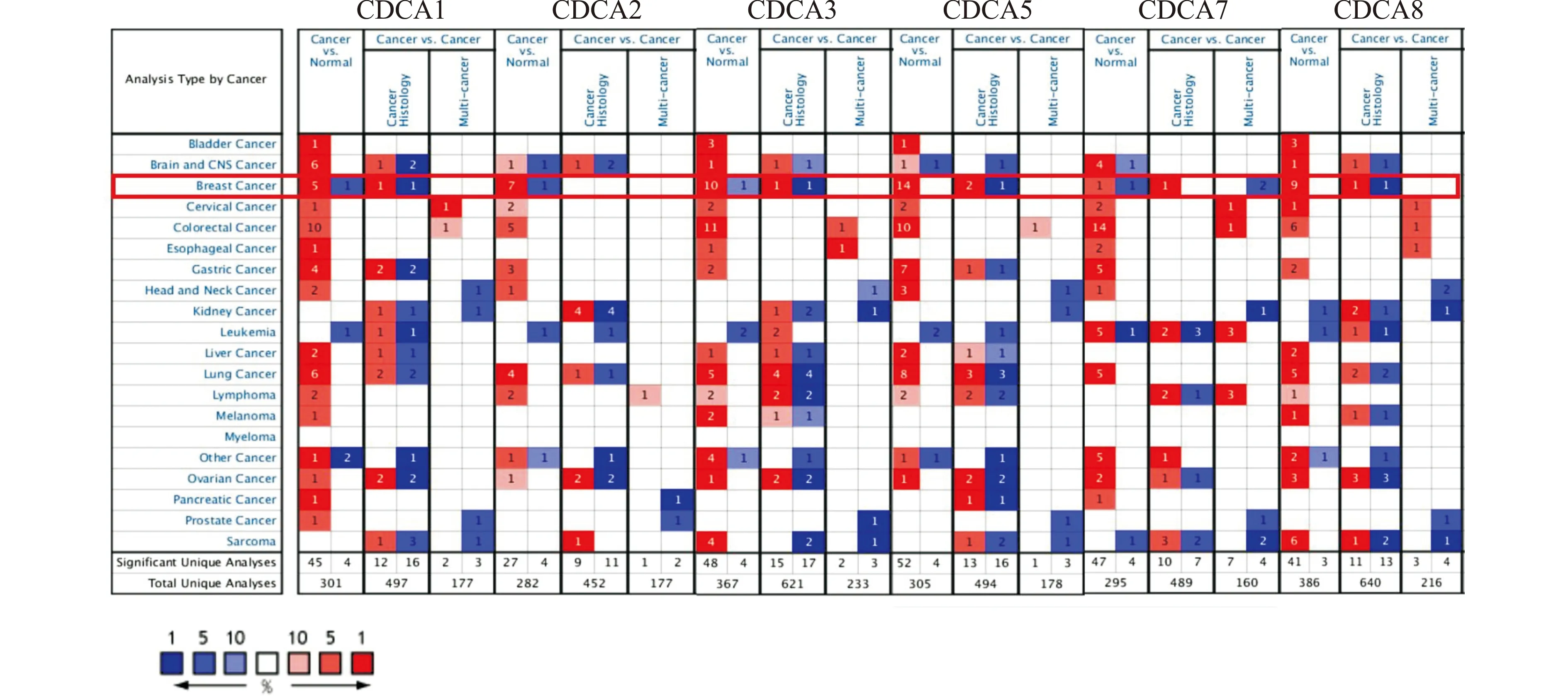
数据类型为mRNA;P值≤0.05;|logFc|≥2;基因排名≤10%;基因表达水平由格子内颜色的深度来表示;与正常组织相比,红色代表肿瘤组织中靶基因的过表达,而蓝色则代表基因表达下调图2 Oncomine数据库中不同肿瘤CDCAs mRNA表达水平的差异Fig.2 Difference in mRNA expression levels of CDCAs in various cancers based on Oncomine analysis
TCGA数据库中CDCAs在不同肿瘤组织的表达有差异,见图3。在乳腺癌、宫颈癌、卵巢癌、子宫内膜癌癌组织中,CDCA1、CDCA2、CDCA3、CDCA5、CDCA8表达量明显高于正常组织(均P<0.05;其中,乳腺癌样本n=1085与正常样本n=291)。为了进一步分析CDCAs表达水平与乳腺癌分期的关系,在“Single Gene Analysis”模块中的“Stage Plot”页面进行分析,输入所选取的CDCAs,CDCA1、CDCA2、CDCA3、CDCA5、CDCA8表达水平在乳腺癌不同分期均有显著差异(均P<0.01),F值和P值见图4中右上角标注信息。

*P≤0.05图3 CDCA基因在TCGA数据库各种癌组织和正常组织中的表达差异Fig.3 Differential expression data for CDCA genes in a variety of normal and cancerous human tissues in TCGA database
2.3 CDCAs对乳腺癌预后的影响
使用Kaplan-Meier Plotter在线分析工具评估CDCAs的表达水平与乳腺癌患者预后的关系,结果显示:CDCAs的表达水平与乳腺癌患者的总生存时间之间存在关联,总体来说,CDCAs的表达水平增高提示患者预后不良。与CDCA2/3/5/8高表达组相比,CDCA2/3/5/8低表达组乳腺癌患者的OS更长,预后更佳(均P<0.05),相对危险度(HR)及其95%可信区间及P值分别在图中右上角标注,见图5A。乳腺癌患者RFS与CDCA1/2/3/5/7/8高度相关,CDCA1/2/3/5/7/8表达较高者,RFS较短(均P<0.05),见图5B。CDCA2/5/7/8表达较低的乳腺癌患者DMFS更长(均P<0.05),见图5C。
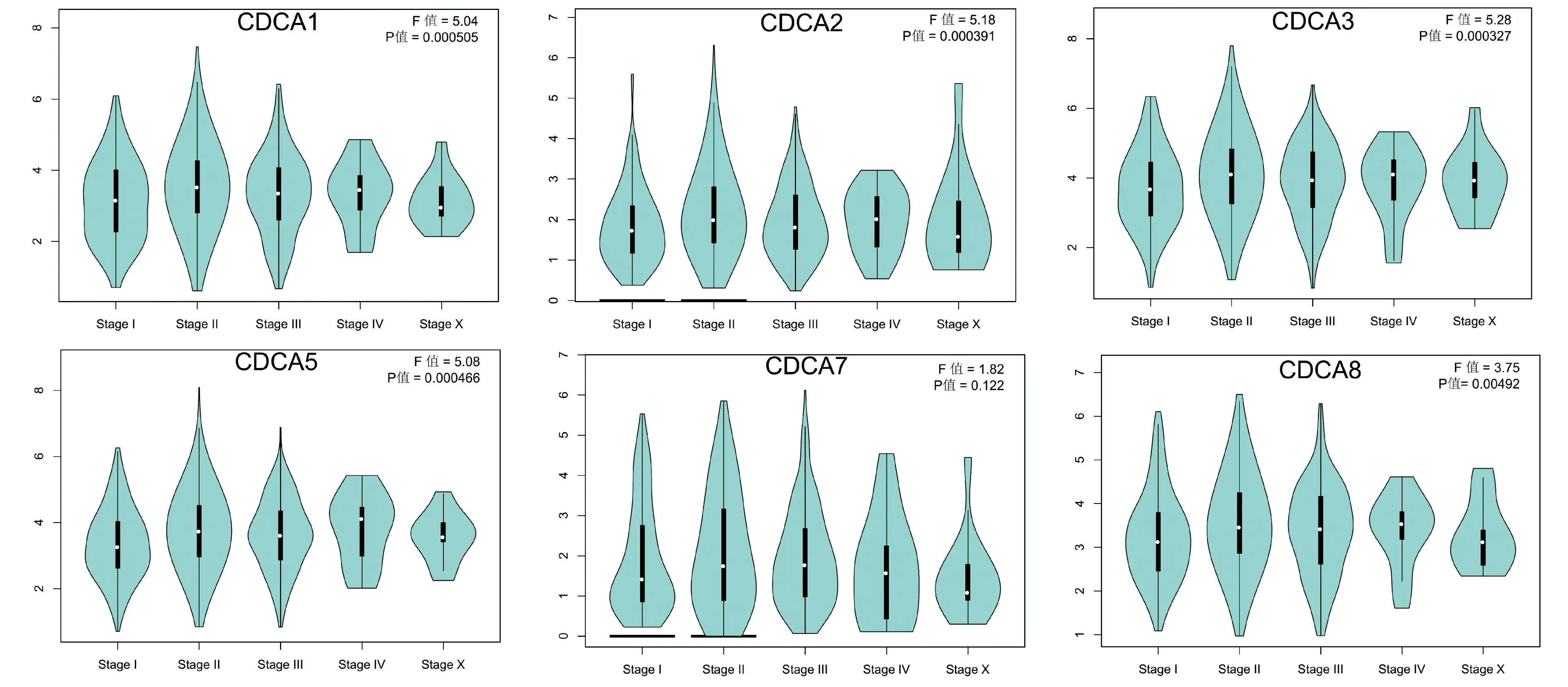
图4 CDCA基因在TCGA数据库中乳腺癌组织不同分期的表达差异Fig.4 Differential expression of CDCA genes in breast cancer tissues of all stages in TCGA database

A:CDCAs在乳腺癌患者中的预后价值-整体生存率;B:CDCAs在乳腺癌患者中的预后价值-无复发生存率;C:CDCAs在乳腺癌患者中的预后价值-无远处转移生存率图5 CDCA1/2/3/5/7/8在乳腺癌患者中的预后价值Fig.5 Prognostic value of CDCA1/2/3/5/7/8 in breast cancer patients
2.4 DEGs的GO、KEGG富集分析和通路分析
使用DAVID在线网站进行DEGs的GO富集分析,共有2630个基因的探针号能够被识别。筛选条件设为P≤0.01,筛选CDCA相关的DEGs得到:
①生物过程(BP)中主要作用于细胞周期、有丝分裂细胞周期、细胞有丝分裂M期、细胞分裂、细胞器分裂、核分裂、染色姐妹单体分离等,详见图6A;②细胞成分(CC)作用点主要作用于染色体、着丝粒、纺锤体、核仁、微管细胞骨架、非膜结合的细胞器,详见图6B;③分子功能(MF)主要作用于染色质结合和蛋白丝氨酸/苏氨酸磷酸酶活性,详见图6C。使用DAVID在线网站进行KEGG信号通路富集分析,筛选条件设为P≤0.01,得到27条信号传导通路,详见图6D。

A:生物过程;B:细胞成分;C:分子功能;D:KEGG信号通路富集分析图6 乳腺癌中DEGs的GO、KEGG功能分析Fig.6 GO and KEGG functional analysis of DEGs in breast cancer
2.5 CDCAs蛋白相互作用网络预测及功能分析
PPI网络共涉及1278个节点和6040个边缘,见图7A。通过PPI网络中的连通度评估了MCC前30个基因,见图7B。

A:蛋白-蛋白相互作用(PPI)网络的可视化;B:PPI网络MCC排名前30的基因图7 乳腺癌DEGs蛋白-蛋白相互作用网络的可视化Fig.7 Visualization of the PPI network of DEGs in breast cancer
结果显示KIF2C是最突出的基因(Rank=1),其次是CDC20、CCNB1、CDK1、NDC80、CDCA8、BUB1、CCNB2、BUB1B、CENPF、BIRC5(Rank=2);AURKB(Rank=13)、PLK1(Rank=14)、MAD2L1(Rank=15);CENPE、SMC3、WAPAL、ZWINT、SMC1A、RAD21、CKAP5、RANGAP1、NUF2、ESPL1、PPP2R5C、PPP2R1B、KNTC1、ZWILCH、CDCA5(Rank=16)。
3 讨论
乳腺癌是全球女性最常见的恶性肿瘤之一,以其较高的病死率严重威胁着女性的健康。为了预防乳腺癌的发生,尽可能做到“早诊断、早治疗”,寻找针对乳腺癌早期分子标志物的药物靶点,对乳腺癌患者的预后极为重要。CDCAs是细胞增殖过程中的调控因子,在细胞增殖过程中发挥重要作用。在本研究中,我们发现在乳腺癌组织和正常乳腺组织中,CDCA基因家族有不同的mRNA表达水平,CDCA2、CDCA3、CDCA5和CDCA8的过度表达与乳腺癌患者整体生存期相关。许多研究表明,细胞分裂过程中的失调都可能导致恶性肿瘤的发生,有丝分裂主要涉及染色体事件(如:染色体浓缩、姐妹染色单体分离)和细胞骨架事件(如:核被膜破裂、染色体运动、胞质分裂等),这些过程是由蛋白质激酶和磷酸酶的竞争作用调控的,如发生异常则会导致肿瘤的发生[21-23]。目前,以CDCA基因家族作为预后因子的研究引起了人们的关注[24-26]。然而,关于CDCA基因家族在乳腺癌发生和进展过程中的作用机制还缺乏系统的分析。
本研究基于生物信息学的方法,发现在乳腺癌组织中CDCA1/2/3/5/7/8的表达水平显著升高,说明CDCAs可能在乳腺癌的发生和进展过程中发挥作用。CDCA2的功能是结合蛋白磷酸酶1γ(PP1γ)和细胞周期控制DNA损伤反应[27]。Shi等[28]的研究表明,CDCA2可下调Cyclin E1(CCNE1)表达,进而使得肺腺癌细胞G1期延长,而CDCA2的过表达则可上调CCNE1,促进肺腺癌细胞的增殖分化。本研究中,通过GO富集分析结果显示,CDCAs可介导细胞有丝分裂过程,与Shi等的研究结果部分一致。CDCA3可触发有丝分裂并控制细胞周期过程,研究表明,CDCA3的表达异常与肝癌和口腔鳞状细胞癌等的发生有关[29-30]。Adams等[31]的研究证实,CDCA3在非小细胞肺癌中高表达,高水平的CDCA3与患者预后不良相关。同样,在本文的研究中,CDCA3在乳腺癌组织中表达水平高于正常乳腺组织,且与较短的OS、较差的预后高度相关。CDCA5也被认为是癌基因,有文献报道在多种类型的癌症中存在表达水平异常[32]。CDCA5在DNA修复中起着至关重要的作用,并参与姐妹染色单体的聚合和分离过程[33]。Nguyen等[34]的研究表明,CDCA5过表达与非小细胞肺癌预后不良有关,同样本文的研究也证实CDCA5的表达与乳腺癌患者的整体生存率、无复发生存率和无远处转移生存率存在明显的相关性。CDCA8被认为是一种潜在的癌基因,研究表明,其在肺癌、胃癌组织中表达上调,但在正常组织中表达非常低或缺失[35-36]。Yu等[37]的研究表明,CDCA8在他莫昔芬耐药的乳腺癌细胞系(MCF7/ TamR和T47D/TamR)中显著升高,CDCA8表达的下调显著抑制了乳腺癌细胞的增殖和他莫昔芬耐药。相反,CDCA8过表达显著促进了他莫昔芬敏感的乳腺癌细胞系(MCF7和T47D)的增殖并诱导其对他莫昔芬的耐药性。CDCA8是乳腺癌中三苯氧胺耐药的关键调控因子,提示CDCA8可能是乳腺癌治疗的潜在靶点。
本研究中,CDCA1/2/3/5/7/8在乳腺癌中表达显著升高,提示可能与乳腺癌的发展和进展存在相关性。GO和KEGG富集分析结果显示,CDCAs可能参与介导细胞有丝分裂、染色姐妹单体分离等过程,同时还可能参与调节染色体、着丝粒、纺锤体、核仁等细胞成分的合成。此外,生存分析结果显示,CDCA2/3/5/8低表达组的乳腺癌患者其总体生存期更长,预后更佳;而CDCA1/2/3/5/7/8表达水平增高时,患者复发率也会有一定程度的增加。因此,这些CDCAs有望成为乳腺癌潜在的治疗靶点。本研究系统分析了乳腺癌中CDCAs的表达水平和预后价值,认为CDCA2/3/5/8可能是治疗乳腺癌的潜在靶点,为深入了解乳腺癌分子生物学的复杂机制提供了依据。然而,本文的研究数据仅来源于GEO、TCGA等数据库,虽然多个数据库间的研究结果可以相互印证,但未通过临床标本及分子实验进行进一步验证,并且关于CDCAs调控miRNAs及其参与机制仍不清楚,因此,仍需要进行大量的研究来发现这些机制,从而找到合适的癌症临床治疗方法。
