miR-139555及其靶基因PtNBC在三疣梭子蟹适应盐度胁迫中的表达调控
2022-04-13赵笑颜吕建建
赵笑颜 吕建建, 张 文 吴 捷 金 玲 刘 萍,
(1. 上海海洋大学水产科学国家级实验教学示范中心, 上海 201306; 2. 中国水产科学研究院黄海水产研究所,农业农村部海洋渔业可持续发展重点实验室, 青岛 266071; 3. 青岛海洋科学与技术国家实验室,海洋渔业科学与食物产出过程功能实验室, 青岛 266235)
三疣梭子蟹(Portunus trituberculatus), 属节肢动物门(Arthropoda), 甲壳纲(Crustacea), 十足目(Decapoda), 梭子蟹科(Portunidae), 梭子蟹属(Portunus), 分布于我国黄渤海、东海, 日本、朝鲜、马来西亚群岛等诸多海域, 是重要的海水经济蟹类之一[1,2]。三疣梭子蟹作为海水“弱”高渗调节型种类[3],养殖水体盐度骤变容易影响其体内离子及渗透平衡, 进而影响其蜕皮、摄食及生存[4—6]; 盐度变化也会引起其机体免疫能力下降, 增加疾病发生的几率[7]。探讨三疣梭子蟹盐度适应机制, 对其健康养殖及耐盐良种培育有十分重要的意义。
通过鳃上皮细胞的离子跨膜转运维持机体内的渗透压和离子平衡是甲壳动物进行渗透压调节的主要方式[8]。microRNA(miRNA)是一类具有重要功能的非编码小分子RNA, 能够在转录后调控靶基因的表达, 在生物发育、分化及抗逆过程中发挥巨大作用[9,10]。已有研究表明, miRNA在水生动物的渗透调节中发挥重要作用[11], 如在斑马鱼(Danio rerio)中miR-200a和miR-200b直接调控Na+/H+exchanger基因的表达, 影响离子细胞中Na+的跨膜运输[12]; 抑制罗非鱼(Oreochromis niloticus)体内miR-206的表达, 可显著提高IGF-1mRNA水平, 从而间接调控离子转运基因Na+/K+-ATPase和NKCC的表达[13,14]; 在刺参(Apostichopus japonicas)中, miR-10通过调控TBC1D5(胞内转运和应激诱导的自噬之间的一个关键的分子开关)基因的表达来适应盐度胁迫[15,16]。目前, 甲壳类中有关miRNA的研究较少, 且研究内容主要为性别和免疫等领域[17—20], 少有研究关注 miRNA 在甲壳动物渗透压调节中的功能。
在前期工作中, 通过比较转录组和生物信息学方法筛选到了在低盐胁迫下三疣梭子蟹鳃组织中差异表达的miR-139555, 预测其靶基因为离子转运相关的PtNBC基因(碳酸氢钠协同转运基因, 英文名称: Sodium bicarbonate cotransporter, 缩写NBC)。本研究克隆了PtNBC基因并进行了序列特征分析,开展了该基因和miR-139555在三疣梭子蟹主要组织中的表达分布规律及在低盐胁迫后鳃中的表达模式分析。研究结果将明确miR-139555与其潜在靶基因PtNBC的调控关系, 有助于系统解析三疣梭子蟹盐度适应机制。
1 材料与方法
1.1 miRNA的筛选及靶基因预测
本实验室前期对低盐胁迫处理的三疣梭子蟹鳃组织进行高通量测序, 获得了转录组及miRNA数据库。首先, 从miRNA数据库中筛选到了差异表达的miR-139555。使用RNAhybrid软件进行miRNA-139555的靶基因预测。根据miRNA与靶基因的序列匹配、miRNA-mRNA双链之间的热稳定性, 筛选出最小自由能低于‒30 kCal/mol 的候选目标, 最终获得miR-139555的靶基因为PtNBC。miRNA-139555与PtNBC的结合位点在该基因mRNA 5′UTR的427—433 bp, 两者结合的最小自由能(mfe)为–36.1 kCal/mol。
1.2 材料
实验动物均取自山东省昌邑市海丰水产养殖有限公司黄海水产研究所实验基地。随机选取80日龄活力良好的健康三疣梭子蟹, 平均体重(25±3) g。实验动物在室内养殖车间的水泥池暂养7d, 暂养期间, 盐度34, 温度(26±2)℃, 持续充氧, 每天更换 1/3 体积的海水且定时投喂新鲜饵料。随机选取9只暂养后的三疣梭子蟹, 设置3个平行, 每个平行3只, 分别取肌肉、心脏、胃、脑、眼柄、血细胞、肝胰腺和鳃共8个组织, 液氮储存备用。
随机选取暂养的三疣梭子蟹进行低盐胁迫实验。实验设置2组: 对照组(海水盐度34)和低盐11胁迫组(72h-LC50为11[21]), 每组设置3个平行, 每个平行30只。在3个水泥池中注入正常海水, 另选3个水泥池将海水盐度调节至11(用淡水勾兑), 盐度稳定后将各组三疣梭子蟹分别放置于池中。分别在胁迫0、12h、24h、48h和72h各时间点取鳃组织, 每个平行取3只, 于液氮中保存备用。
1.3 PtNBC基因cDNA全长克隆
用TRIzol法提取未经低盐胁迫处理的三疣梭子蟹鳃、肝胰腺和肌肉等组织的RNA, 核酸定量仪(NanoDrop 2000 Thermo Scientific)检测RNA的浓度及质量、1.0%的琼脂糖凝胶电泳检测其完整性。将各组织中的总RNA均匀混合, 使用TaKaRa公司的SMARTer®RACE cDNA Amplification Kit试剂盒合成3′和5′ RACE cDNA模板。从转录组数据库中筛选获得了PtNBC序列, 用Primer Premier 5.0软件设计特异的3′和5′RACE引物, 引物由上海生工生物公司合成(表 1)。使用南京诺唯赞生物公司的Phanta Max Super-Fidelity DNA Polymerase进行3′和 5′的巢式扩增, PCR产物使用胶回收试剂盒(购自TaKaRa公司)进行目的片段回收, 连接(全式金pEASY-T1 Cloning), 转化(全式金Trans1-T1 Phage Resistant Chemically Competent Cell), 挑取阳性单克隆, 进行菌落 PCR 鉴定(M13引物), 将目的单克隆菌液测序(上海生工生物工程有限公司)。

表1 本研究中所用引物序列Tab. 1 Primers used in this study
1.4 序列生物信息学分析
使用Contig Express软件将测序获得的3′序列、5′序列与验证的基因序列进行拼接, 得到PtNBC基因cDNA全长。利用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)预测该基因的开放阅读框。使用 Signal4.1(http://www.cbs.dtu.dk/services/SignalP/)和SMART(http://smart.embl-heidelberg.de/)在线软件预测该基因编码蛋白的信号肽、跨膜结构和功能结构域。目的基因与其他物种的一致性及同源性分析采用Blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)。氨基酸序列多重序列比对使用DNAMAN 5.2.9软件。进化树构建使用MEGA 6.0软件(邻接法Neighbor-joining)。
1.5 PtNBC基因和miRNA-139555的组织表达及各处理组的表达特征分析
运用TRIzol法提取三疣梭子蟹全组织及各实验组的总RNA, 并检测质量(核酸定量仪NanoDrop 2000 Thermo Scientific)和完整性(1.0%琼脂糖凝胶电泳)。
利用PrimeScript RT reagent Kit with gDNA Eraser试剂盒(TaKaRa)进行反转录, 合成cDNA模板, 用于PtNBC基因定量分析。利用Mir-X miRNA First-Strand Synthesis Kit试剂盒(TaKaRa)进行miRNA cDNA第一链合成, 用于miRNA-139555定量分析。
依据克隆获得的三疣梭子蟹PtNBC序列及miRNA-139555成熟序列, 使用Primer Premer 5.0及miRNA Design V1.01软件设计特异性RT-PCR引物,分别以β-actin和U6作为内参(表 1), 使用ABI 7500 Real Time PCR仪和SYBR GreenProTaqHS qPCR Kit(艾科瑞公司)试剂对样品进行定量分析。PCR反应体系为: 2× SYBR®Green ProTaqHS Premix II 5 μL, Primer F 0.4 μL, Primer R 0.4 μL, Template cDNA 1 μL, ddH2O 3 μL。用2–∆∆Ct法分析PtNBC基因及miRNA-139555的相对表达量, 利用SPSS 19.0软件对数据进行分析, 使用OriginPro和EXCEL对数据进行处理,P<0.05 表明具有显著性差异。
1.6 体外双荧光素酶实验
PtNBC基因的5′-UTR野生型(wt)和突变型(mut)目的片段由上海生工生物公司合成。miRNA-139555与PtNBC-5′-UTR结合位点如图 1所示。本实验所用双荧光素酶报告基因表达载体为psiCHECK-2。目的片段插入位点为XhoⅠ和NotⅠ,将载体酶切后胶回收备用。用T4连接酶(Fermentas公司)进行目的基因片段与载体的连接, 利用DH5a感受态细胞(上海生工)进行转化(抗性:Amp),37℃, 过夜培养。转化后平板挑菌, 37℃ 250转/分钟摇菌14h, 用菌液进行PCR鉴定, 并将阳性克隆菌液送测序进一步验证, 最终成功构建了PtNBC-5′-UTR-wt/mut载体。

图1 miRNA-139555与PtNBC-5′UTR靶位点结合示意图Fig. 1 Schematic diagram of miRNA-139555 binding to PtNBC-5′UTR target site
将293T细胞接种到96孔板中, 待细胞密度达到50%—70%时进行转染。0.16 μg的5′UTR-wt、5′UTR-mut目的质粒分别与5 pmol的miR-139555、NC mimics(miRNA对照)两两混合, 再各加入10 μL DMEM充分混匀后室温放置(溶液A)。将10 μL DMEM与0.3 μL的转染试剂(浓度为0.8 mg/mL)充分混匀(溶液B), 室温放置5min。将溶液A与B充分混匀, 室温放置20min。转染前为细胞换取新鲜培养基, 之后将转染混合物加入混匀。37℃, 5%CO2培养。转染6h后换取新鲜培养基, 转染48h后收集细胞检测, 使用Promega Dual-Luciferase system试剂盒进行荧光素酶活性检测并记录Renilla luciferase值。
2 结果
2.1 三疣梭子蟹PtNBC基因cDNA序列特征分析
本实验通过克隆获得了三疣梭子蟹碳酸氢钠协同转运基因cDNA全长, 命名为PtNBC, Gen-Bank登录号为MW323548。cDNA全长5308 bp, 包含570 bp的5′-UTR、3570 bp的ORF和1168 bp的3′-UTR, 编码1189个氨基酸, 预测到该基因的分子量为133.66 kD, 理论等电点(pI)为6.07。利用Signal4.1及SMART在线软件预测分析氨基酸的结构特征。Signal4.1未预测到信号肽, SMART预测结果显示,PtNBC包含10个跨膜结构域(TM)和2个功能结构域(Pfam: Band_3_cyto和HCO3_cotransp)。
2.2 PtNBC基因氨基酸序列同源性分析及进化树构建
三疣梭子蟹PtNBC氨基酸序列与其他物种的NBC(碳酸氢钠协同转运)基因编码的氨基酸进行同源性比对, 结果显示, 该氨基酸序列与麦龙螯虾(Cherax cainii)C. cainii_NBC1的同源性最高(85.37%); 其次是天空蓝魔虾(Cherax destructor)C.destructor_NBC1(84.63%)和凡纳滨对虾(Litopenaeus vannamei)Lv_NBC(81.30%)。系统进化树分析显示, 三疣梭子蟹与凡纳滨对虾首先聚为一支,然后与麦龙螯虾和天空蓝魔虾相聚, 与鱼类、昆虫类较远(图 2)。
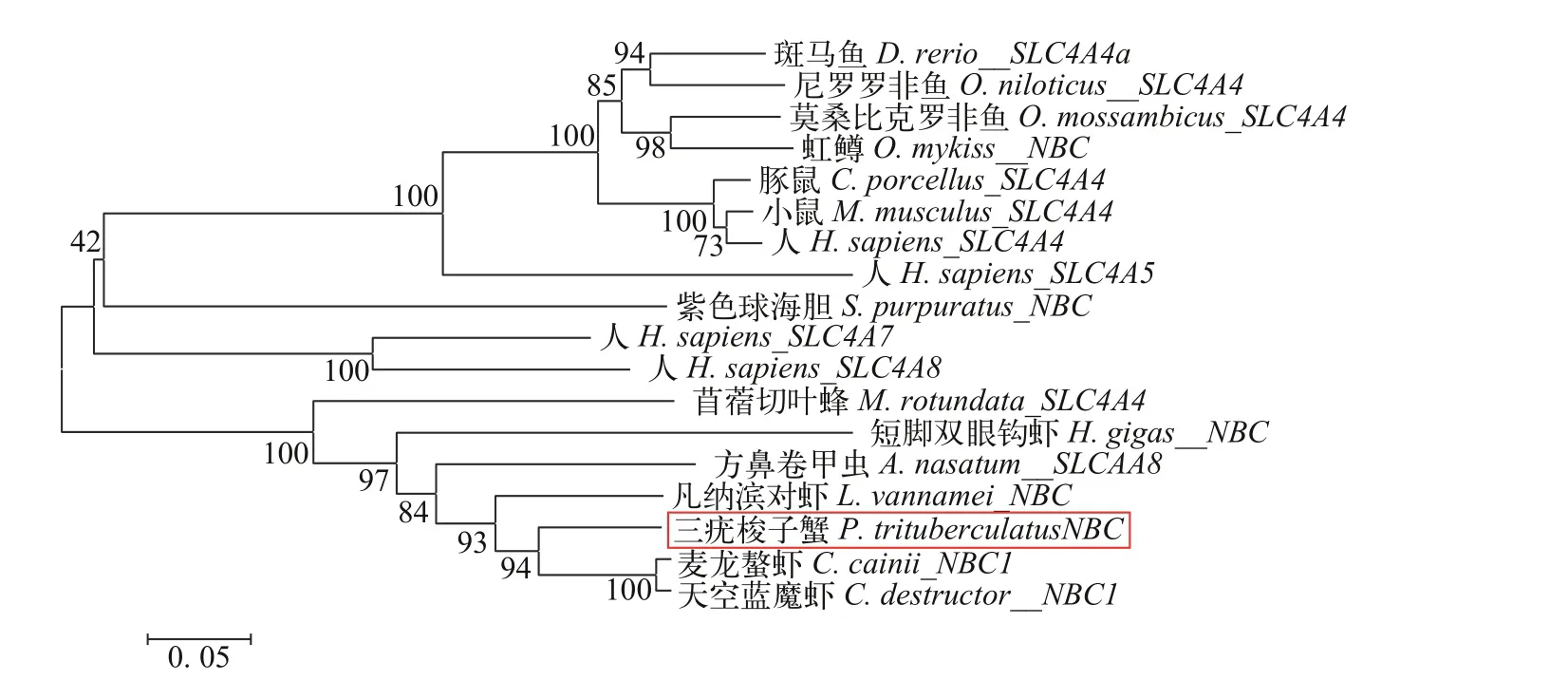
图2 NBC氨基酸序列NJ进化树Fig. 2 NJ evolutionary tree of NBC amino acid sequence各物种名称及GenBank登录号(Name and GenBank accession number of each species): 三疣梭子蟹P. trituberculatus(MW323548); 凡纳滨对虾L. vannamei(AQX43455.1); 麦龙螯虾C. caini(AJO70192.1); 天空蓝魔虾C .destructor(AJO70010.1); 小鼠M. musculus SLC4A4(AAI48293.2); 斑马鱼D. rerio SLC4A4a(NP_001030156.1); 短脚双眼钩虾H. gigas NBC(LAC22142.1); 尼罗罗非鱼O. niloticus SLC4A4(XP_005451600.1); 莫桑比克罗非鱼O. mossambicus SLC4A4(BAJ49842.1); 虹鳟O. mykiss NBC(NP_001117797.1); 紫色球海胆S. purpuratus NBC(NP_001073019.1); 豚鼠C. porcellus SLC4A4(NP_001166501.1); 方鼻卷甲虫A. nasatum SLC4A8(KAB7498207.1);H. sapiens SLC4A7(XP_016863016.1); 苜蓿切叶蜂M. rotundata SLC4A4(XP_012151659.1); H. sapiens SLC4A4(AAF21718.1); H.sapiens SLC4A5(AAI09222.1); H. sapiens SLC4A8(AAH25994.1)
2.3 miR-139555及PtNBC基因组织表达分布分析
利用RT-PCR分析miR-139555及PtNBC基因在三疣梭子蟹不同组织中的相对表达情况。如图 3所示, miRNA-139555及PtNBC基因在肌肉、心脏、胃、脑、血细胞、眼柄、肝胰腺和鳃8个组织中均有表达, miRNA-139555在鳃组织中表达量最高, 之后是在肌肉、胃和肝胰腺中表达较高, 与其他组织相比表达量有显著性差异(P<0.05)。与miRNA-139555相同,PtNBC基因也在鳃组织中有最高表达,在肌肉、胃和脑中的表达次于脑, 与其他组织相比表达量有显著性差异(P<0.05)。
2.4 低盐胁迫下鳃组织中miR-139555及PtNBC基因的表达
如图 4所示, 在低盐胁迫72h内, 与对照组相比, miR-139555整体呈现上调表达的趋势, 在胁迫48h达到最高峰, 为对照组的4.9倍(P<0.05); miR-139555在胁迫24h及48h有下调表达的趋势, 但与对照组相比, 均为显著上调表达(P<0.05)。而PtNBC基因在胁迫72h内显著下调表达, 且各时间点表达量均低于对照组, 在12h表达量最低, 为对照组的0.06倍(P<0.05); 在胁迫12—72h,PtNBC基因呈现上调表达趋势, 但与对照组相比均显著下调表达。综上, 在低盐胁迫下miR-139555与PtNBC基因在三疣梭子蟹鳃组织中的表达趋势呈现明显的负相关。
2.5 miR-139555作用于PtNBC基因的mRNA的体外验证结果
利用双荧光素酶实验进一步验证miRNA-139555与PtNBC基因是否具有直接的作用关系。如图 5所示, 与其他对照组相比, 仅在miRNA-139555与PtNBC-5′-UTR-wt共同转染入细胞的情况下, 荧光素酶的表达量显著下降(P<0.001), 表明PtNBC基因的表达被抑制。目的基因的突变型载体作为对照组, 排除了miRNA-139555与载体其他位点结合而影响实验结果的可能。
3 讨论
3.1 低盐胁迫下miR-139555的表达分析
microRNA(miRNA)是一类具有重要功能的非编码小分子RNA, 能够在转录后调控靶基因的表达, 在生物抗逆过程中发挥重要作用[22—24]。为了探索miRNA在三疣梭子蟹盐度适应中的作用, 基于前期对三疣梭子蟹低盐胁迫前后鳃组织样品的高通量miRNA测序分析, 我们筛选到一个在低盐胁迫下显著差异表达的、新预测的miRNA, 命名为miRNA-139555。本研究分析了miRNA-139555的组织表达分布及低盐胁迫下在鳃组织中的表达规律。我们发现miRNA-139555在鳃组织中的表达明显高于其他组织, 且低盐胁迫后鳃组织中的miR-139555呈现显著上调表达的趋势, 在48h表达量最高, 相较于对照组上调4.9倍(图 4)。鳃被认为是甲壳类离子交换和渗透调节的主要器官[25]。miR-139555在鳃中高表达且受低盐胁迫诱导, 表明其可能在三疣梭子蟹渗透压调节中发挥重要作用。
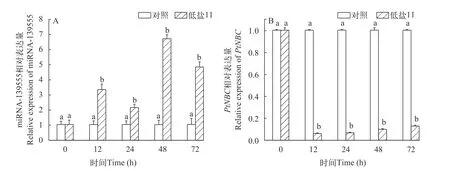
图4 低盐胁迫下miR-139555及PtNBC在三疣梭子蟹鳃组织中的表达Fig. 4 Expression of miR-139555 and PtNBC in the gills of P. trituberculatus under low salt stress
3.2 miR-139555可调控靶基因PtNBC的表达
已有研究表明, 盐度变化可诱导水生动物体内某些miRNA差异表达, 而这些miRNA的靶基因为渗透调节相关基因[26,27]。本研究预测到miR-139555的潜在靶基因为离子转运相关的PtNBC基因。该基因cDNA全长5308 bp, 编码1189个氨基酸。Signal4.1未预测到信号肽, 表明PtNBC不是分泌蛋白。SMART预测结果显示,PtNBC基因含有10个TM结构域, 和以往报道的SLC4家族成员普遍含有10-14个TM结构域一致[28]。并且与多数NBC1基因相同, 包含2个功能结构域(Pfam: Band_3_cyto和HCO3_cotransp)。进化树分析显示,C. destructor_NBC1与PtNBC进化关系最接近,该基因含有12个TM区域, 其中10个TM区域与PtNBC完全相同[29]。
王萍等[30]对青海湖裸鲤(Gymnocypris przewalskii)的研究表明, slc4a4(NBC)基因在主要组织中泛表达。在本研究中,PtNBC基因也显示泛组织表达的模式(图 3)。然而, 值得注意的是, 与miR-139555相同,PtNBC基因也在鳃组织中具有最高的表达。并且在PtNBC基因相对高表达的组织中(如肌肉、胃等), miR-139555也有较高表达, 显示了该基因和miR-139555具有组织共定位的趋势, 该现象与我们预测的miR-139555的靶基因为PtNBC基因相符。靶基因预测结果显示, miR-139555作用于PtNBC基因mRNA的5′UTR区。以往的多数研究关注miRNA与靶基因3′UTR结合发挥调控功能, 但随着对miRNA调控机制研究的不断深入, 也有不少关于miRNA靶定基因5′UTR[31]和ORF[32]区域参与调控基因表达的报道。例如: Wigar等[33—35]研究发现,let-7能够靶定lin-41 mRNA 5′UTR区, 影响斑马鱼的胚胎发育[36]。另外, 多个研究证实miRNA可通过靶定相关基因的5′UTR或ORF, 抑制或增强肿瘤细胞的生长。基于以上相关报道, 为进一步证明miR-139555与其潜在靶基因PtNBC间的调控关系, 我们在细胞水平开展了体外双荧光素酶实验。研究结果表明, 在体外细胞水平, miR-139555能够直接抑制PtNBC基因的表达, 与低盐胁迫下两者呈现负相关的趋势一致(图 5)。

图3 miR-139555及PtNBC在三疣梭子蟹不同组织中的表达Fig. 3 Expression of miR-139555 and PtNBC in different tissues of P. trituberculatusM. 肌肉muscle; H. 心脏heart; S. 胃stomach; Br. 脑brain; E. 眼柄optic stalk; B. 血细胞blood cell; He. 肝胰腺hepatopancreas; G. 鳃gill; 不同小写字母代表表达量的差异显著性Different lowercase letters indicated significant differences (P<0.05); 下同The same spplues below
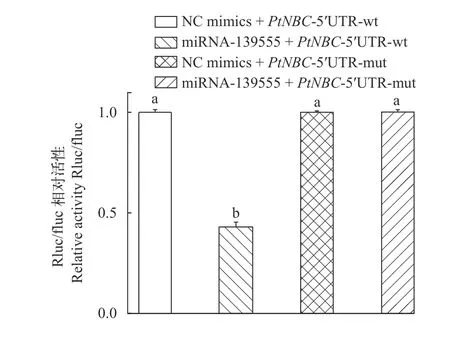
图5 双荧光素酶报告检测miR-139555与PtNBC-5′UTR的相互作用Fig. 5 Dual luciferase report to detect the interaction between miR-139555 and PtNBC-5′UTR
3.3 miR-139555及其靶基因PtNBC在渗透压调节中的作用
多数研究表明, miRNA通过与靶基因的完全或不完全互补性结合降解或沉默靶基因, 从而调节基因的表达, 两者为负调控关系[37]。比如尼罗罗非鱼(Oreochromis niloticus)通过下调鳃组织中miR-429的表达使渗透胁迫转录因子(OSTF1)相应的上调表达, 来适应高盐胁迫[38]; 在低盐胁迫下, 刺参(Apostichopus japonicas)Aja-miR-22上调表达, 其靶基因(棘皮动物微管相关蛋白EMAP)下调表达。本实验中我们也发现了相似的结果, 在低盐胁迫12h,三疣梭子蟹鳃组织中miR-139555上调表达, 之后与对照组相比, 处于持续上调表达的状态; 而PtNBC在低盐胁迫12h下调表达, 之后持续处于下调表达的状态, 两者表达趋势呈现典型的负相关(图 4)。该结果同样也支持miR-139555的靶基因为PtNBC基因。
Deigweiher等[39]的研究表明, 在海水硬骨鱼类中,NBC1基因定位在鳃上皮细胞基底质膜上, 主要作用是协调向体内转入Na+和HCO3–, 与水生生物的渗透压调节或盐度适应密切相关。Ali等[29]对C.cainii和C. destructor两种淡水龙虾的研究表明,NBC基因在两个物种中均有5个亚型且均具有参与调节盐度的功能。目前, 除PtNBC基因外, 在三疣梭子蟹转录组中我们并没有发现其他与盐度适应相关NBC基因亚型。梁从飞等[40]在尼罗罗非鱼的研究中发现, NBC基因在高盐胁迫下呈现上调表达的趋势, 而我们的结果显示PtNBC基因在低盐胁迫后显著下调表达, 两个研究的结果相对应。我们推测, 低盐水体中Na+的浓度较低, 为了维持体内渗透压平衡, 三疣梭子蟹通过microRNA途径主动降低PtNBC基因的表达而以减少Na+的吸收, 从而维持体内外的渗透压平衡。
总而言之, 我们初步证实了PtNBC基因是miR-139555的靶基因, miR-139555可能通过调控鳃上皮细胞PtNBC基因的表达而发挥重要的渗透压调节功能。本研究尝试从转录后水平探究三疣梭子蟹的渗透调节机制, 有助于系统解析该物种盐度适应机制, 同时为三疣梭子蟹耐盐良种的鉴定和培育提供新的思路。
