冠状病毒突刺糖蛋白的免疫表位预测分析*
2022-03-29姜海明李姗珊张立新
焦 娟 ,姜海明 ,李姗珊 ,张立新 ,刘 杰△
1.中国人民解放军总医院第七医学中心检验科,北京 100700;2.滨州医学院烟台附属医院重症医学科,山东烟台 264100
冠状病毒是一类RNA 病毒,通过人的呼吸道或接触传播,感染该病毒后常见临床表现有发热,以及咳嗽、气促和呼吸困难等呼吸道症状,严重者可导致肺炎、严重急性呼吸综合征、肾衰竭,甚至死亡。
近期,新型冠状病毒(SARS-CoV-2)疫苗相继问世(灭活疫苗、腺病毒载体疫苗和mRNA 疫苗等),其作用机制即通过激活人自身的免疫系统来对抗病毒,其主要的作用靶点为分布于病毒表面的突刺糖蛋白(Spike蛋白)。部分学者在SARS-CoV-2致病机制方面的基础研究也做出了巨大的努力,从最初的SARSCoV-2基因组的测定完成[1],到SARS-CoV-2全病毒三维结构的解析[2],都显示Spike蛋白是SARS-CoV-2的主要致病因素:Spike蛋白与人体细胞表面血管紧张素转换酶2(ACE2)的结合介导了SARS-CoV-2对人体细胞的入侵[3-4]。因此SARS-CoV-2的Spike蛋白已经成为人们诊断、治疗、预防SARS-CoV-2感染的主要靶点。本研究拟通过生物信息学软件,通过横向比较和分析SARS-CoV-2的Spike蛋白的二级结构、三级结构和B 细胞表位,探讨Spike蛋白潜在的高效免疫表位,为SARS-CoV-2检测试剂盒、疫苗制备、治疗药物的研发提供可靠的生物学信息和实验基础。
1 材料与方法
1.1 蛋白结构和序列的选择 根据已公布的蛋白结构数据库(https://www.rcsb.org/),比对Spike蛋白相关的结构信息,下载对应的结构信息文件(pdb文件),其登录号为6XR8/6XRA(SARS-CoV-2),5X58(SARS-CoV),5X59(MERS);同时,下载相对应的氨基酸序列。结合下载的序列,比对GenBank数据库,分别下载各蛋白的完整蛋白序列,其登录号分别为YP_009724390,P59594.1,AGN70929.1。
1.2 二级结构预测 抗原免疫表位主要位于蛋白质表面,具有较强的亲水性,含有或邻近柔性区域,易于形成典型的二级结构等。因此分析目的蛋白质的二级结构,对了解其免疫表位分布具有重要的参考作用。本研究应用PHD(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=/NPSA/npsa_phd.html)和Jpred4(http://www.compbio.dundee.ac.uk/jpred/)等在线软件分别预测目的蛋白质的α螺旋、β折叠、转角以及卷曲等二级结构的组成。
1.3 B细胞表位预测 根据目的蛋白的蛋白序列应用在线软件Bepipred Linear Epitope Prediction 2.0(http://tools.iedb.org/bcell/)分别预测目的蛋白的B细胞表位,该软件运用随机森林算法对目的蛋白的表位和非表位氨基酸进行预测,同时利用自身表位数据库对预测序列进行多重优化[5]。本研究同时比对目的蛋白的二级结构和三维结构的数据,进一步筛选优势抗原表位区段,得出Spike蛋白的优势B细胞免疫表位。
2 结果
2.1 氨基酸序列分析 Spike蛋白的结构信息非常丰富:目前在蛋白质结构数据库中,Spike蛋白的高分辨结构有上百个,均为冷冻电子显微解析的结构。这些结构信息显示,Spike蛋白有多种构象形式:侵染细胞前状态、侵染细胞后状态和与中和抗体结合状态等,这些状态为分析Spike蛋白的免疫表位提供了丰富的分析线索。本文以Spike蛋白三维结构为基础模型,以多种预测软件为辅助,分析和研究严重急性呼吸综合征冠状病毒(SARS-CoV)、SARS-CoV-2 和中东呼吸综合征冠状病毒(MERS-CoV)共3种冠状病毒Spike蛋白的B细胞表位信息。首先在GeneBank数据库中下载相关蛋白的一级序列信息:SARS-CoV的Spike蛋白序列(YP_009724390)与SARS-CoV-2的Spike 蛋白(P59594.1)具有76% 同一性,与MERS-CoV的Spike 蛋白具有35%的同一性(AGN70929.1)。
2.2 Spike蛋白二级结构分析 运用PHD 和Jpred4两种在线软件,对SARS-CoV、SARS-CoV-2、MERSCoV的Spike蛋白二级结构进行预测。SARS-CoV的Spike 蛋白α 螺旋主要分布于5~11、51~56、409~419、729~769、797~805、849~861、871~1 018、1 129~1 140、1 151~1 184、1 195~1 213等肽段。β折叠分布于146~166、173~178、181~190、198~230、241~258、277~279、309~377、421~428、442~450、465~484、552~582、584~601、610~646、693~724、1 032~1 058、1 075~1 087、1 100~1 115、1 213~1 219、1 247~1 255等肽段。自由卷曲位于α螺旋和β折叠之间。见表1。

表1 3种病毒Spike蛋白二级结构组成(%)
SARS-CoV-2的Spike蛋白α螺旋主要分布于361~371、747~788、815~823、867~879、887~1 035、1 147~1 159、1 175~1 123、1 134~1 151等肽段。β折叠分布于2~8、36~50、61~77、90~108、116~145、167~177、190~206、262~288、298~329、376~381、395~403、418~425、432~437、450~455、507~517、538~577、583~588、595~600、608~613、589~695、711~729、1 048~1 075、1 093~1 105、1 120~1 132、1 151~1 156、1 190~1 195 等肽段。自由卷曲位于α螺旋和β折叠之间。
MERS-CoV的Spike 蛋白α 螺旋主要分布于107~112、225~231、277~281、304~318、405~418、430~436、806~837、846~856、887~895、908~912、941~954、963~1 110、1 230~1 241、1 262~1 285、1 297~1 316等肽段。β折叠分布于2~10、30~35、64~70、84~89、98~104、116~123、129~131、162~172、179~188、202~206、236~242、262~271、287~296、321~337、360~377、399~405、440~449、464~471、478~483、567~576、603~664、670~676、713~718、754~765、779~804、1 121~1 127、1 134~1 151、1 202~1 213、1 317~1 320等肽段。自由卷曲位于α螺旋和β折叠之间。
2.3 B细胞表位预测 运用Bepipred Linear Epitope Prediction 2.0在线软件,对上述病毒Spike蛋白B细胞表位完成预测,阈值设定为0.5。SARS-CoV的Spike蛋白B 细胞表位区段分布于16~38、60~84、134~148、173~181、203~212、236~249、317~351、355~412、427~473、505~519、602~630、642~651、663~689、768~796、811~827、1 089~1 100、1 115~1 156、1 234~1 248 等肽段。SARS-CoV-2的Spike蛋白B细胞表位区段分布于13~37、59~81、138~156、177~189、206~221、250~260、306~322、370~393、409~425、440~450、458~501、516~536、617~632、634~644、656~666、672~690、695~710、786~800、806~816、828~842、1 108~1 118、1 133~1 172、1 198~1 206、1 252~1 263、1 275~1 306等肽段。MERS-CoV的Spike蛋白B 细胞表位区段分布于16~38、60~84、134~148、173~181、203~212、236~249、317~351、355~412、427~473、505~519、602~630、642~651、663~689、768~796、811~827、1 089~1 100、1 115~1 156、1 234~1 248等肽段。
2.4 结合蛋白三维结构的B细胞表位区段的筛选以上的表位信息结果均源于软件的预测,这些结果很可能与实际情况差异较大,因此本研究将以上B细胞表位区段预测信息,在高分辨的蛋白三维结构和二级结构基础上,同时结合B细胞抗原表位结构和生化特点,进一步人工筛选SARS-CoV(5X59)[6]、SARSCoV-2(6XR8)[3]、MERS-CoV(5X58)[6]Spike蛋白的B细胞表位区段,见表2~4。

表2 SARS-CoV(5X59)Spike蛋白的B细胞表位区段

表3 SARS-CoV-2(6XR8)Spike蛋白的B细胞表位区段
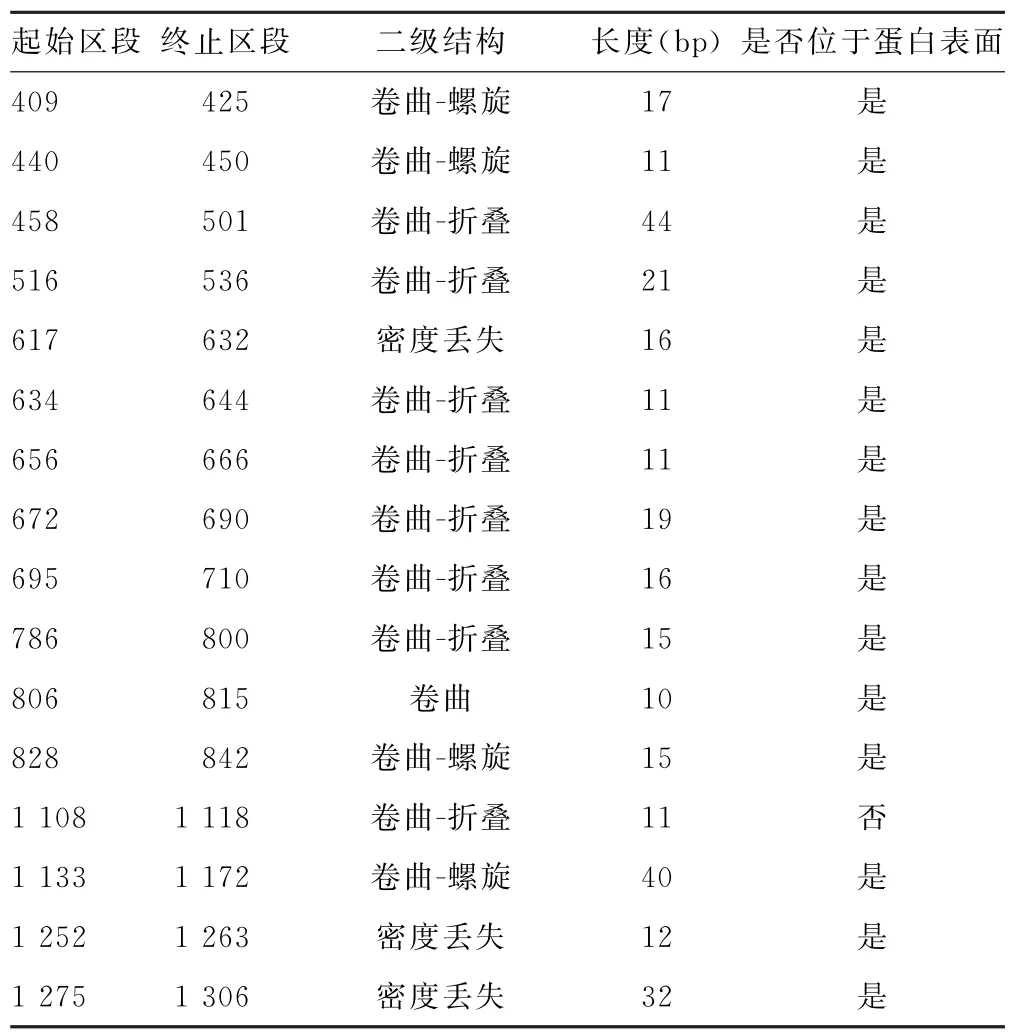
续表3 SARS-CoV-2(6XR8)Spike蛋白的B细胞表位区段

表4 MERS-CoV(5X58)Spike蛋白的B细胞表位区段
由于蛋白三维结构信息来自于蛋白晶体衍射数据,因此存在结构密度信息的部分缺失,另一方面,结构密度信息的缺失一般意味着蛋白这部分区域处于无序排列的状态,即在周边的环境中可以自由摆动或存在结构变化,说明该区域亲水性较强并位于蛋白三维结构的表面。
将B 细胞表位信息,SARS-CoV、SARS-CoV-2、MERS-CoV 3种病毒Spike蛋白的二级结构信息和三级结构信息整合在一起筛选,可提高数据分析的可信性。蛋白抗原表位区段基本分布于蛋白三维结构的表面,与二级结构有所差别。SARS-CoV Spike蛋白抗原表位区段中卷曲-螺旋占17.0%,卷曲-延伸占50.0%,卷曲占33.0%。SARS-CoV-2 Spike蛋白抗原表位区段中卷曲-螺旋占22.0%,卷曲-延伸占59.0%,卷曲占19.0%。MERS-CoV Spike蛋白抗原表位区段中卷曲-螺旋占25%,卷曲-延伸占37.5%,卷曲占37.5%。
综合分析结果发现,具有优势B细胞表位区段中存在一定二级结构和一定柔韧性结构组合的占比较高,比如β折叠。这一特点完全符合抗原蛋白B细胞表位的基本特征。
3 讨论
Spike蛋白是SARS-CoV、SARS-CoV-2、MERSCoV 等冠状病毒关键性致病因子。Spike蛋白的结构已经较为清晰,其致病的分子机制已较为明了[3-4]:首先,Spike蛋白的受体结合域(RBD)向外伸展与人体细胞表面的ACE2受体结合;其次,RBD 与ACE2的结合启动了Spike蛋白自身在S位点的剪切作用,使Spike蛋白发生“脱冠”变化;最后,“脱冠”的Spike蛋白HR 结构域得以暴露出来,在HR 结构域辅助下,病毒完成和人体细胞的融合和遗传物质的注入过程。因此,Spike蛋白RBD 和ACE2识别和结合,是冠状病毒侵染细胞的最关键环节,从目前已有的 Spike蛋白-中和抗体的三维结构中,也可以证明这一结论。
在蛋白结构数据库中,SARS-CoV、SARS-CoV-2、MERS-CoV 等病毒的 Spike蛋白和其中和抗体的结构较多,如6NB6/7(SARS-CoV)、6WPT(SARSCoV-2)、7K4N (SARS-CoV-2)、6NB3/4(MERSCoV)等[7-9]。对这些复合体的结构分析后发现,中和抗体与 Spike蛋白表面结合位点主要集中于RBD 结构域和CTD 结构域。同时本研究发现ACE2 与Spike蛋白结合位点也位于RBD 结构,且与中和抗体的结合位点重叠,这进一步证明了中和抗体在抗病毒时的作用机制。以SARS-CoV-2 Spike蛋白为例,在7KNB结构中,ACE2 结合于449~502aa[10];7K8W结构中,中和抗体结合于444~501aa[11];本研究结果与上述研究结果相似,表明本研究分析结果具有一定的可靠性和前瞻性。
本研究同时发现 Spike蛋白RBD 和CTD 结构中,含有多个优势B 细胞表面抗原表位的多肽区段,这为检测试剂、抗体药物的开发等提供了较好的作用靶点。然而,冠状病毒的RBD 结构域、三维结构非常复杂,主要由内部的β折叠和外围的无规则卷曲所构成,说明RBD 结构域外围表面结构十分灵活,RBD 在与ACE2或中和抗体结合时,可能存在局部的构象重建;同时在病毒侵染过程中RBD 本身还会存在巨大的结构变化,使基础和临床研究难度大大增加,因此更多的结构和B 细胞表面抗原表位信息将提供更多的研究思路和实验基础。
