大豆PP2C家族基因鉴定与响应盐胁迫的转录组分析
2022-03-03杨昕霞唐满生
杨昕霞,唐满生,张 斌,*
(湖南科技学院,a.后勤处;b.化学与生物工程学院,湖南 永州 425199)
植物在生长过程中经常会受到一些不利的环境因素影响,如干旱、盐、极端温度等。随着全球气候变暖,环境胁迫出现的更加频繁且持续时间更长,这将对植物的生长、发育和农作物产量产生重大不利影响。为了适应不利环境,植物进化出多种分子和生理机制,如ABA信号途径、SOS信号途径、转录因子调控、蛋白质修饰等。其中,植物信号转导过程中蛋白激酶(protein kinases,PKs)和蛋白磷酸酶(protein phosphatases,PPs)可通过蛋白去磷酸化调节蛋白功能,因此其可参与许多生物过程,如植物发育、信号转导、非生物胁迫响应等。而蛋白磷酸酶主要包括3类:STP(Ser/Thr phosphatases)、PTP(protein Tyr phosphatases)和DSPTP(dual-specificity phosphatases)。蛋白磷酸酶2C(Type 2C protein phosphatase,PP2C)属于PTP类,已有报道表明,植物PP2C家族蛋白能够通过影响植物体内的代谢过程、激素水平、生长因子等在环境胁迫(干旱、盐、碱、病原菌等)响应中发挥作用。
多个植物物种中已鉴定出PP2C家族成员,如拟南芥()、水稻()、短柄草()、辣椒()、香蕉()、马铃薯()、棉花()、玉米()、小麦()、野生大豆()等。在拟南芥中,PP2C家族成员的功能研究较多。最早在拟南芥中鉴定出80个PP2C家族基因,其中,73个PP2C家族基因被分为13个亚族(A~E、F1、F2、G~L),另外有7个未被分组。亚族A的PP2C蛋白在ABA信号网络中发挥重要作用,参与调节生理过程和胁迫响应,例如干旱、极端温度和盐胁迫等。正常条件下,拟南芥亚族A中的PP2C蛋白能够与SnRK2s蛋白激酶结合抑制其活性;而在植物响应非生物胁迫时,PYR/PYL蛋白能够耦合脱落酸(abscisic acid,ABA),进而结合亚族A中的PP2C蛋白,使其释放SnRK2s蛋白激酶,进而激活下游转录因子以适应逆境胁迫。Yu等研究表明,小麦PP2C亚家族A的成员能够与TaSnRK2.1和TaSnRK2.2蛋白激酶互作,并且来源于A亚族的2135基因在拟南芥中过表达可降低其ABA敏感性。Chen等研究表明,拟南芥PP2C家族基因25、26、27和29对碱胁迫表现出不同的响应。拟南芥AtPP2C1蛋白可以与CIPK9相互作用,以调节低K条件下植物根的生长和种子发育,并且还能够与MPK4或MPK6激酶协同作用,在拟南芥受到损伤时缓解病原胁迫。棉花PP2C家族基因的表达能够响应低温、高温、干旱和盐胁迫,说明其在非生物胁迫中起重要作用。
以上研究初步探究了PP2C家族基因在植物发育和环境胁迫中的作用,但是其在大豆中的作用却鲜见报道。Bai等研究表明,大豆GmPP2C1(GLYMA.13G106800,即本研究中的GmPP2C79)以ABA依赖的方式与ABA受体GmPYL1相互作用,参与植物激素信号转导过程。Lu等证明了大豆2-1(Glyma.17G221100,即本研究中的2104)参与种子发育过程,该基因过表达能够提高种子产量。Chen等对野生大豆PP2C家族D亚族中的13个成员进行了系统的生物信息学分析。而基于盐胁迫下栽培大豆()PP2C家族基因的全基因组学鉴定与分析的研究尚未见报道。大豆是我国重要的经济作物,也是食用油的主要来源,但是土壤盐渍化导致的大豆减产问题尤为严重。因此,大豆PP2C基因家族成员的鉴定和耐盐性功能探究是非常必要的。本研究对栽培大豆全基因组数据中的PP2C家族成员进行了鉴定和分析,同时对盐处理前后的大豆地上部(茎和叶)进行了转录组学分析,重点对差异表达基因中的PP2C家族基因进行分析,包括GO富集分析、响应盐胁迫的表达模式、蛋白互作网络预测和酵母双杂交验证,这将为阐明大豆PP2C家族基因在响应盐胁迫时的生理功能和分子机制奠定基础。
1 材料与方法
1.1 序列检索与理化性质分析
利用已报道的80个拟南芥AtPP2C家族成员(AtPP2C1-80)的保守蛋白序列在Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Gmax)进行比对,初步筛选AtPP2C在栽培大豆中的同源基因。然后利用NCBI数据库中的在线工具CDD(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进一步鉴定这些蛋白序列是否含有PP2C蛋白特有的保守结构域(PF00481),剔除不含该保守结构域的蛋白序列,得到大豆PP2C家族成员。通过在线工具ExPASy(https://web.expasy.org/protparam/)预测大豆PP2C家族成员的蛋白分子量(ku)、不稳定系数、总平均亲水性(grand average of hydropathicity,GRAVY)与理论等电点(pI)。
1.2 染色体定位与进化树构建
从Phytozome数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Gmax)检索得到栽培大豆PP2C家族成员在不同染色体上的定位信息,从NCBI数据库(https://www.ncbi.nlm.nih.gov/genome/?term=Glycine+max)下载大豆20条染色体全长信息,根据PP2Cs基因的染色体定位与染色体全长,利用Map Chart 2.2软件绘制染色体定位示意图。利用Clustal X软件将拟南芥80个PP2C成员与大豆126个PP2C成员的氨基酸序列进行多重比对,利用MAGA 7软件构建系统进化树,参数设置:NJ(Neighbor-joining)法,校验参数Bootstrap为1 000,泊松模型。
1.3 基因与蛋白结构分析
栽培大豆PP2C的基因、CDS和蛋白序列分别从Phytozome数据库获得。利用大豆PP2C的基因和CDS序列在GSDS网站(http://gsds.cbi.pku.edu.cn/index.php)绘制基因结构图;利用MEME网站(http://meme-suite.org/tools/meme)预测大豆PP2C蛋白的保守基序(Motif),参数设置:保守基序最小长度为6,最大长度为50,最大数量设置为10。
1.4 大豆幼苗培养与盐处理
实验所用栽培大豆材料为天隆1号,将大豆种子浸泡在蒸馏水中6 h后平铺在润湿的滤纸上萌发2 d,待根长1.0~1.5 cm时移至石英砂中,用Hoagland营养液进行水培。约10 d后大豆幼苗的第一、三出复叶展开,以Hoagland营养液培养的材料为对照,同时以含有150 mmol·LNaCl的Hoagland营养液培养的大豆材料为处理组,6 h后取地上部(茎和叶)于50 mL离心管中,液氮速冻后放于超低温冰箱保存。
1.5 转录组测序与蛋白互作网络预测
大豆样品(对照组和处理组分别3组生物重复)送上海欧易生物医学科技有限公司利用Illumina HiSeqTM 2500测序平台进行转录组测序并进行初步分析。将|log2 fold change|≥1且Q<0.05(Q值是值校正值)的基因定义为差异表达基因(DEGs)。使用转录组数据中PP2C家族基因的FPKM(fragments per kilobase million)值,利用Heml 1.0软件绘制基因表达热图。使用在线数据库PlantRegMap(http://plantregmap.cbi.pku.edu.cn/)对大豆PP2C家族基因进行GO富集分析。利用在线工具STRING(https://string-db.org/cgi/input.pl)预测大豆PP2C家族蛋白间的互作关系。
1.6 实时荧光定量PCR(qRT-PCR)分析
用含有150 mmol·LNaCl的Hoagland营养液处理生长10 d的大豆幼苗,分别在0、3、6、12 h时取地上部(茎和叶)用于总RNA提取。利用反转录试剂盒(ThermoFisher scientific)合成cDNA,使用Bio-Rad CFX96定量PCR仪(USA,Bio-Rad)进行PCR扩增。反应体系为20 μL:10 μL 2×SYBR Green Mix(康为世纪),上下游引物各0.5 μL,9 μL稀释后的cDNA。程序设置:95 ℃预变性3 min,60 ℃退火延伸30 s,共40个循环。利用2法计算基因的相对表达量。qRT-PCR分析所用引物通过NCBI网站的Primer Blast工具设计,如表1所示。

表1 qRT-PCR引物
1.7 酵母双杂交(Y2H)
酵母双杂交分析参照Zhao等的方法进行。根据蛋白互作预测结果,克隆289和2113基因的CDS序列,分别连接到pGBKT7载体上,构建重组bait载体BD-289和BD-2113。克隆247、253、258、2122基因的CDS序列,连接到pGADT7载体上,获得重组prey载体AD-247、AD-253、AD-258和AD-2122。将bait载体和prey载体共转化酵母菌株Y2H,在SD-Leu-Trp和SD-Leu-Trp/-His培养基上培养。以pGADT7空载体与BD-289或BD-2113共转化的Y2H菌株作为阴性对照。
2 结果与分析
2.1 大豆PP2C家族基因鉴定及其染色体定位
利用80个拟南芥PP2C成员(AtPP2C1~80)在Phytozome数据库检索,并通过CDD工具检测其蛋白保守结构域,最终鉴定出126个大豆PP2C基因,根据这些基因所在的染色体顺序及其基因编号,将其命名为21~2126(表2)。不同PP2C蛋白含有的氨基酸残基数量有差异,为208~1 073 aa,其中,氨基酸残基数量最多的是GmPP2C9(1 073 aa),其分子量也最大;而最少的是GmPP2C64(208 aa),相应地其具有最小的分子量。大多数大豆PP2C蛋白(93个)的不稳定系数大于40,为不稳定蛋白;其余33个则为稳定蛋白。此外,GmPP2C蛋白的等电点和总平均亲水性分别在4.62~9.57和-0.695~-0.014。
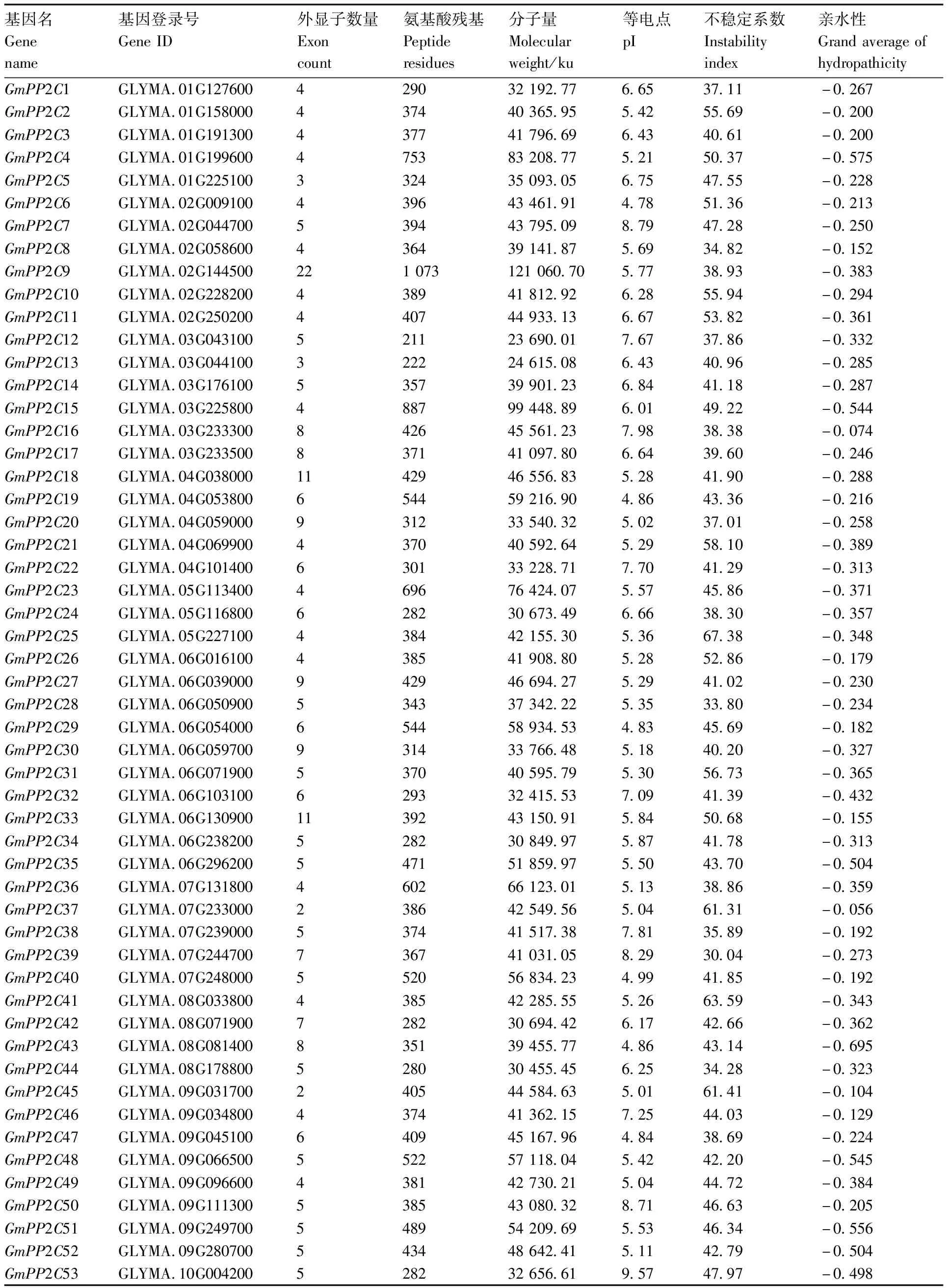
表2 大豆PP2C家族成员的理化性质
利用Mapchart 2.2 软件绘制大豆PP2C家族基因的染色体定位图,发现126个2基因不均匀地分布在大豆20条染色体上(图1)。其中,Chr10染色体上2基因数量最多,有12个;仅有1个基因(295)定位在16号染色体上。最短的染色体Chr11上共有8个2基因,而最长的Chr18染色体上只有6个。

图1 大豆PP2C家族基因的染色体定位
2.2 基因结构与氨基酸保守基序分析
在基因结构方面,2基因的内含子数量介于3~20,亚族I/J中仅有的一个成员29基因序列最长,其外显子数量(22个)也最多;L亚族中的233次之,有11个外显子;除了亚族L和I/J之外,其他亚族成员的外显子数量均少于10,并且每个亚家族的基因成员都具有相似的基因结构,如亚族B、亚族C和亚族D,几乎所有的基因都含有4个外显子,亚族F基因的外显子数量介于5~8(图3-a)。利用MEME数据库对GmPP2C家族成员的保守结构域进行预测,发现保守基序Motif 1、Motif 2、Motif 3和Motif 5在GmPP2C家族中非常保守,所有成员都具有这4种保守基序。有些Motif仅在某一亚族或是某几个亚族中存在,如Motif 6仅出现在亚族E中,而Motif 8仅在亚族D中存在,Motif 9仅在亚族C中出现,除C、D、E、K的所有亚家族中均含有Motif 10(图3-b)。

进化树中包含126个大豆PP2C家族成员,80个拟南芥PP2C家族成员,共分为11个亚家族。

图3 大豆PP2C家族成员的基因结构(a)与蛋白质结构(b)
2.3 系统进化分析
为了研究栽培大豆PP2C家族与其他物种的关系,利用已有报道获得的拟南芥PP2C序列和本文鉴定的栽培大豆PP2C蛋白序列进行多重比对,在MEGA7软件中对两个物种的PP2C蛋白进行聚类。参考拟南芥PP2C家族的分组方式,大豆PP2C家族被分为11个亚族(A-H、I/J、K-L)(图2)。其中,亚族E中GmPP2C数量最多,有22个;亚族F次之,有21个;而亚族I/J、K和L中GmPP2C数量最少,分别都只有1个。
2.4 PP2C家族基因对盐胁迫的响应
对正常条件和盐胁迫处理的大豆幼苗进行转录组测序分析,结果显示,在盐处理后共有1 235个差异基因(logFC>1,Q value<0.05),其中466个上调,769个下调。这些差异基因中包含9个PP2C家族基因,其中有7个上调,2个下调(图4-b)。根据所有2基因在盐胁迫下的FPKM值绘制基因表达热图(图4-c),241、248、272、284、289、2107和2113在盐处理后表达水平显著上调,227和2114在盐处理后表达水平显著下调。利用qRT-PCR检测这9个基因在盐处理不同时间的表达水平,结果与转录组测序结果一致,证明转录组测序结果的可靠。上述7个上调基因中,241和2107在盐处理后表达量持续上升,而248、272、289和2113在盐处理6 h表达量最高。227和2114在盐处理后表达量下调,227随着盐处理时间的延长表达量逐步趋于稳定,2114则在盐处理6 h时表达量最低。

a,对照和盐处理样品的相关分析;b,差异基因数量统计;c,大豆PP2C基因在盐处理前后的表达模式;d,9个差异基因的qRT-PCR结果。
2.5 PP2C家族差异基因的GO富集与蛋白互作预测
为了更好地探究大豆PP2C家族成员潜在的功能,对大豆PP2C家族中9个差异基因进行GO富集分析。发现这些基因均显著富集在分子功能一类中,其中,显著富集的GO term主要包括离子结合(ion binding)、阳离子结合(cation binding)、金属离子结合(metal ion binding)、磷酸蛋白磷酸酶活性(phosphoprotein phosphatase activity)、磷酸酶活性(phosphatase activity)、磷酸酯水解酶活性(phosphoric ester hydrolase activity)和水解酶活性(hydrolase activity)等(图5)。对PP2C家族成员进行蛋白互作预测分析,发现有一部分GmPP2C蛋白之间存在蛋白互作现象(图6-a),其中差异表达基因编码蛋白GmPP2C89可能与GmPP2C53、GmPP2C58和GmPP2C122存在互作关系;而GmPP2C113蛋白可能与GmPP2C47、GmPP2C58和GmPP2C122互作。

MF,分子功能;CC,细胞组分;右边的数值为Q值。
2.6 酵母双杂交(Y2H)分析
根据大豆PP2C家族成员的蛋白互作预测结果(图6-a),构建了289和2113基因重组bait载体BD-289和BD-2113,以及对应的重组prey载体AD-247、AD-253、AD-258和AD-2122。bait载体和prey载体共转化Y2H菌株,在不同的培养基上培养。结果显示,BD-289分别与pGADT7载体、AD-253、AD-258和AD-2122共转化的Y2H菌株在SD-Leu-Trp培养基上均能长出菌斑,而在SD-Leu-Trp/-His培养基上均无法生长(图6-b);BD-2113分别与pGADT7载体、AD-247、AD-258和AD-2122共转化的Y2H菌株在SD-Leu-Trp培养基上均能正常生长,而在SD-Leu-Trp/-His培养基上仅BD-2113和AD-247共转化的菌株能够长出菌斑(图6-c),表明GmPP2C113蛋白能够和GmPP2C47互作,可能在植物生长或响应胁迫过程中协同发挥功能。

a,蛋白互作预测网络图;b、c,酵母双杂交验证蛋白互作,SD/-L/-T为缺失亮氨酸和色氨酸的酵母培养基,SD/-L/-T/-H为缺失亮氨酸、色氨酸和组氨酸的酵母培养基。
3 讨论
不同物种的PP2C家族成员数量存在一定的差异,拟南芥中有80个,水稻中有78个,马铃薯中有78个,二倍体亚洲棉()和中棉()中则分别包含87个、99个PP2C成员。126个大豆PP2C成员在基因外显子数量、氨基酸残基数量、分子量、等电点等方面存在较大差异。染色体定位分析表明,大豆126个2基因不均匀地分布在栽培大豆的20条染色体上(图1)。参考拟南芥中2基因的分组方式可将大豆中的126个成员分为11个亚族(A~H、I/J、K~L),总体上与之前有关植物PP2C家族的报道分组一致,仅在亚家族数量上存在差异。如在拟南芥和小麦中可以分为13个亚族,棉花和马铃薯中均可以分为12个亚族,而在水稻中仅可分为11个亚家族。大豆PP2C基因相同亚族的成员总体上具有类似或相同的基因结构和保守基序,说明相同亚族内的基因可能发挥类似的功能。
盐处理后栽培大豆共有1 235个差异基因,其中466个上调,769个下调。而在这些差异基因中共包含9个PP2C家族基因(7个上调,2个下调),即241、248、272、284、289、2107和2113在盐处理后表达水平上调,227和2114在盐处理后表达水平下调,qRT-PCR结果也与此一致。进一步分析发现,这9个差异基因中,有7个基因(241、248、272、284、289、2107和2113)属于A亚族。研究表明,拟南芥、小麦、马铃薯、大豆等物种中PP2C家族A亚族中的蛋白能够通过调控ABA信号转导,进而调节植物多种生理过程和胁迫响应,例如干旱、极端温度和盐胁迫等,说明栽培大豆A亚族的这7个基因可能在盐胁迫响应中发挥作用。对这9个差异基因进行GO富集分析,发现主要富集的GO term为离子结合、磷酸蛋白磷酸酶活性、磷酸酶活性、磷酸酯水解酶活性和水解酶活性等,说明大豆PP2C可能主要通过其磷酸酶活性使目标蛋白去磷酸化进而参与到胁迫响应过程中。通过STRING预测大豆PP2C家族126个成员蛋白互作,发现有部分成员间可能存在互作关系,包括两个差异基因编码蛋白GmPP2C89和GmPP2C113。STRING数据库是基于已公开的基因组数据进行蛋白互作预测的工具,但是预测结果需要进行实验验证,因为同源蛋白在不同物种中可能表现出不同的功能。通过酵母双杂交实验发现,预测的蛋白互作网络中仅GmPP2C113和GmPP2C47存在互作关系。
本研究对栽培大豆PP2C家族成员进行了全基因组学鉴定与生物信息学分析,同时通过转录组测序与qRT-PCR验证,最终筛选到9个差异表达的2基因,其中,属于亚家族A的7个成员可能在大豆盐胁迫响应中发挥重要作用,后续研究中将会针对这7个基因进行深入研究。
